Helicobacter pylori 26695基因组尺度代谢研究进展
游丽斌,刘冬娟,李 娟 ,方慧生*
(1.中国药科大学生命科学与技术学院,江苏南京210009;2.南京市鼓楼医院血液科,江苏南京210008)
近些年,基因组学的迅速发展为科学家们探究生命规律提供全新的方法。全基因组测序拓宽了我们对细菌基因组的了解,序列信息为研究细菌的遗传学改变和适应性突变体的特征带来新的启发。另一方面,利用序列信息可以识别功能基因、重构功能单位网络甚至整个通路网络。在基因组测序和海量注释的基础上,基因组尺度代谢网络的重构发展成熟起来,它通过结合序列和基因功能注释信息,把基因编码的蛋白质所催化的生化反应构建成代谢网络,反映出参与代谢过程的基因-蛋白质-反应之间的相互关系,并有效地转化为数学模型在计算机上进行模拟、分析[1]。基因组尺度代谢网络模型可应用于:与高通量技术结合并有效地分析、处理高通量数据;指导代谢工程;基于假设指导有目的性的发现研究;探究物种间关系;分析网络特性[2]。
幽门螺杆菌(Helicobacter pylori,H.pylori),是从胃黏膜中分离出来的一种弯曲样杆菌。它是导致慢性胃炎和消化性溃疡的主要原因,1994年世界卫生组织/国际癌症研究机构(WHO/IARC)将幽门螺杆菌定为Ⅰ类致癌原。1997年,H.pylori 26695菌株的全基因组测序完成,2003 年,Boneca[6]进行了第一次H.pylori 26695基因组注释工作,该版本注释包含编码序列1 590条,其中功能基因1 080个。Schilling和Thiele等先后在2002年和2005年重构了H.pylori 26695的代谢网络模型,分别是iCS291[4]和 iIT341[5]。iIT341 模型对目的基因预测的准确率不高,模型的质量仍有待进一步提高。2013 年,Resende等[6]对 H.pylori 26695 基因组进行了重新注释,该版本包含1 573个CDS,功能基因1 212个,与代谢相关基因712个,并找到191个新功能蛋白,该版本注释可为构建新的高质量代谢网络模型提供基础。一般重构过程分为三个步骤:初步构建、验证完善、数学模拟。第一步一般可编程或用软件自动完成,最重要的验证完善步骤贯穿重构始终,数学模拟的目的也包括评估网络的质量。目前完善一个高质量的代谢网络仍需要大量人工校对工作,如验证基因-蛋白质-反应关系需要查阅大量相关文献。结合更多幽门螺杆菌特异性的研究成果,将基因的预测信息和实验结果结合有助于指导针对H.pylori的治疗。
本文以几个重要代谢通路为主线,结合近几年H.pylori 26695与代谢有关的酶与基因组关系的研究成果,阐述H.pylori 26695的主要代谢通路及其特点。除另有说明外,本文讨论的 H.pylori即 H.pylori 26695。以“HP”开头的基因号和以“Hp”开头的蛋白质名称表示H.pylori特有的基因编号和蛋白质名称。
1 H.pylori的营养需要
H.pylori是一种嗜二氧化碳并且微量需氧的微生物,在大气中需提供5% ~10%CO2,它即可适应微氧条件(<5%O2),也可适应有氧条件(一般为21%O2)。H.pylori某些特性会随着培养环境中氧含量而变化,包括溶血素的产生、甲硝哒唑耐药性、铁氧还蛋白氧化还原酶活性、诱导上皮细胞的改变的能力[7]。目前认为H.pylori生长必需氨基酸包括:精氨酸、组氨酸、亮氨酸、甲硫氨酸、苯丙氨酸、颉氨酸、丙氨酸、半胱氨酸、脯氨酸。Testerman等[8]进一步研究发现,在以上氨基酸为基础的化学限定培养基上同时加入非必需的色氨酸和异亮氨酸有助于细菌生长,而单独加入时没有效果,而且在有色氨酸和异亮氨酸的培养基上再加入谷氨酸或谷氨酰胺都能大大加速细菌生长。离子浓度是影响细菌生长的限制性因素,而氨基酸浓度对细菌生长没有影响。该研究证明H.pylori生长还需要钠、氯化钾、硫胺素、铁、锌、镁、次黄嘌呤和丙酮酸,而铜不是其生长所需。
关于H.pylori生长必需氨基酸,基因组分析从另一个角度为以上结果做出佐证。比如,重构H.pylori甲硫氨酸的从头合成途径时发现,在H.pylori基因组中只找到一个编码硫醚γ-合成酶的基因——HP0106,而缺少编码从头合成过程其他酶的基因。另一方面,虽然甲硫氨酸补充途径普遍存在于所有类型生物体中,但在H.pylori基因组中除了HP0089,还未发现其他编码甲硫氨酸补充途径所需酶的基因,故认为H.pylori无法合成甲硫氨酸。再如,苏氨酸脱氨和乙酰醇酸合成是亮氨酸合成的前提,但H.pylori基因组中缺少编码以上两种酶的基因,所以亮氨酸是维持菌株生长所必需。H.pylori无法从分支酸合成苯丙氨酸,也是因为缺少编码磷酸甘油酸脱水酶的基因。H.pylori基因组缺少合成硫胺素前体的几个关键酶的基因,但是 Barison等[9]发现HP1287所编码的4-氨基-2-甲基-5-(氨甲基)嘧啶氨基水解酶可能参与了合成硫胺素前体的补充途径,他们克隆纯化了该蛋白,其晶体结构显示酶活性位点改变,而且酶活力实验显示该酶对4-氨基-2-甲基-5-(氨甲基)嘧啶底物的亲和力不强,这些变化所具有的重要意义需要进一步证实。
2 H.pylori重要代谢通路
2.1 中心代谢
2.1.1 糖代谢
H.pylori所属的弯曲杆菌属的其他细菌大都无法分解代谢碳水化合物,但H.pylori可以通过氧化和发酵方式代谢D-葡萄糖或D-半乳糖。HP1174编码一种钠离子依赖性转运蛋白,H.pylori利用该转运蛋白将D-葡萄糖或D-半乳糖主动转运至细胞内进而参与代谢[10]。基因分析结果也表明H.pylori糖代谢途径与大肠杆菌相同,包括糖酵解/糖异生途径、磷酸戊糖途径、杜德洛夫途径(Entner-Doudoroff pathway)。参与上述途径的相应基因在H.pylori基因组中几乎都能找到,除了糖酵解途径中编码磷酸果糖激酶的基因(pfk)和编码丙酮酸激酶的基因(pyk),还有糖异生途径中编码葡萄糖-6-磷酸酶的基因(g6p)和磷酸戊糖途径中编码6-磷酸葡萄糖酸脱氢酶的基因(gnd)[11]。丙酮酸是糖酵解和杜德洛夫途径的共同产物,它可能代谢为乳酸、乙酸进而与三羧酸循环、呼吸链联系起来。
2.1.2 三羧酸循环
三羧酸循环在细胞代谢过程中主要有两种作用:为其他合成代谢提供小分子前体,如α-酮戊二酸、乙酰乙酰辅酶A、草酰乙酸;氧化乙酰单位产生CO2,通过生成还原型核苷酸,将能量存于ATP中。因为H.pylori缺少编码α-酮戊二酸脱氢酶复合物的基因,但存在编码铁氧还蛋白氧化还原酶的相似基因HP0588-HP0591,所以 H.pylori三羧酸循环与标准型的三羧酸循环有所不同,H.pylori三羧酸循环如图1所示。H.pylori三羧酸循环中富马酸酶将富马酸转为苹果酸,实验表明,富马酸酶可与铋离子结合,而铋治疗是现今治疗胃炎的一种方法,该结果提示三羧酸循环通路可成为H.pylori铋治疗的靶标[12]。Kather等提出,苹果酸生成草酰乙酸的反应由HP0086编码的醌氧化还原酶介导,该酶不同于苹果酸脱氢酶以NAD+为电子受体而以醌为电子受体[13]。由于幽门螺杆菌临床分离株具有多样性和变异性,临床上还没有诊断和疫苗的“金标准”抗原,以NAD(P)为辅因子的异柠檬酸脱氢酶被认为是潜在的血清学诊断监测的分子靶标。Huang等利用克隆、表达、重组等技术研究H.pylori SS1菌株异柠檬酸脱氢酶的生化特性,为临床上诊断、监测H.pylori感染情况提供了有用信息[14]。
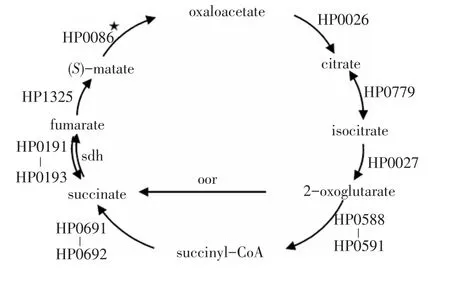
图1 H.pylori三羧酸循环概略图Fig.1 Krebs cycle of H.pylori
2.2 氨基酸代谢
氨基酸代谢是H.pylori代谢的重要环节,因为它是碳、氮、能量的主要来源。H.pylori的大部分氨基酸代谢过程遵循与大肠杆菌一样的过程,体现一定的物种间保守性,但更重要的是有一部分基因体现出H.pylori的物种特异性,利用物种特异性可以发现分子靶标,开发抗菌药物。比如,HP0290编码合成赖氨酸所需的二氨基庚酸脱羧酶,它被认为是抗菌药物的靶点,分析晶体结构发现酶的诱导契合机理——下游活性位点loop区环住催化中间体进而释放产物,该结构分析有助于找到酶的特异性抑制剂[15]。
Shibayama等发现,H.pylori无法吸收胞外的天冬酰胺,而是通过HP0723编码的天冬酰胺酶把胞外的天冬酰胺水解为天冬氨酸而运至细胞内[16]。与天冬酰胺的情况类似,H.pylori也无法吸收谷氨酰胺和谷胱甘肽,而是将其在胞外水解为谷氨酸,水解谷氨酰胺的酶是γ-谷氨酰转移酶,它由两个亚基组成,源于一个HP1118编码的前体蛋白[17]。胞外水解产物谷氨酸由HP1506编码的钠离子依赖性谷氨酸转运蛋白转运至细胞内[18]。以上两种水解酶与幽门螺杆菌感染的致病机制密切相关,天冬酰胺酶抑制T细胞增殖[16],γ -谷氨酰转移酶诱发细胞凋亡、抑制胃细胞和T细胞的增殖[19]。Rimbara等研究临床上幽门螺杆菌感染引起的胃癌、胃溃疡病人的分离株,并分析天冬酰胺酶与γ-谷氨酰转移酶活性[20]。他们提出,γ-谷氨酰转移酶的高活性而引起谷氨酰胺的消耗是导致强烈炎症反应的原因,并非谷胱甘肽和天冬酰胺的消耗。并且,在动物模型中添加额外的谷氨酰胺显示出积极效果[21],但额外的谷氨酰胺是否能降低幽门螺杆菌感染后胃癌发生风险有待进一步分析。
HP0549编码谷氨酸盐消旋酶,该酶介导D-型和L-型谷氨酸盐的可逆转换,它是细胞壁合成的必需酶。Mixcoha等用分子动力学模拟揭示谷氨酸盐消旋酶的催化机理,催化反应用三至四步完成四次连续的质子转移[22]。吡唑并嘧啶二酮类化合物是谷氨酸盐消旋酶的选择性抑制剂[23],在化合物中心支架加上咪唑环能够增加药物口服生物利用度[24]。
分支酸是合成芳香族氨基酸的前体,分支酸合成途径又叫做“莽草酸途径”——以磷酸烯醇丙酮酸和磷酸赤藓糖为起点,经过7种酶的介导合成分支酸(见图2)。莽草酸途径是所有微生物和植物的基本代谢功能,而动物体内不存在,所以该通路的所有酶成为抗菌药物设计的靶标。例如,第二步与第四步酶分别对NAD+和NADP+具有依赖性,可通过找到抑制物阻碍酶与NAD+、NADP+结合从而阻断莽草酸途径。Liu等[25]测定H.pylori脱氢奎尼酸合成酶-NAD+复合物晶体结构,找到了抑制物结合口袋,最后利用GOLD对接软件筛选出可能的抑制物。Han等[26]则是通过表达、分离纯化莽草酸脱氢酶,高通量筛选出五个可能是酶抑制剂的化合物。通过研究酶结构与活性的关系有助于抑制剂的开发,如 Cheng等[27]找到H.pylori莽草酸激酶结构中与莽草酸直接作用的重要保守残基(D33、F48、R57、R116、R132),并详细分析酶催化活性位点的配体结合与构象柔性变化。
其他必需、非必需氨基酸的生物合成与代谢通路虽然通过基因组分析可以找到编码大部分酶的相似基因,但还需通过进一步实验证明。

图2 H.pylori分支酸合成概略图Fig.2 Chorismate synthesis pathway of H.pylori
2.3 维生素与辅因子的生物合成
甲基萘醌,即维生素K2,是作为病原菌呼吸链中电子转运链的电子载体。大部分细菌都采用以大肠杆菌为模板的甲基萘醌生物合成通路,而Hiratsuka等[28]在链霉菌属发现了一种新的甲基萘醌生物合成通路——Futalosine Pathway。Dairi[29]进一步证明了H.pylori采用Futalosine通路的变型通路,首先由mqnA编码的酶介导生成AFL(6-amino-6-deoxyfutalosine),继而在甲基硫代腺苷核苷酶(Methylthioadenosine Nucleosidase,MTAN)作用下直接形成DHFL(dehypoxanthine futalosine)。基因组中找不到mqnA的相似基因,而HP0089编码MTAN。MTAN是一种多功能酶,它位于7个基本细胞代谢通路的节点,包括甲基萘醌合成、甲硫氨酸补充途径、S-腺苷甲硫氨酸利用、聚胺合成、嘌呤补充合成、S-腺苷甲硫氨酸自由基通路、II类自诱导分子合成,特异性的抑制 MTAN 活性可能抑制 H.pylori生长[30]。HpMTAN晶体结构中5’-烷基硫醇结合位点与底物(S-腺苷同型半胱氨酸)的相互作用特点可为发现MTAN抑制剂提供思路[30]。DHFL先后转为cyclic DHFL、1,4-Dihydroxy-2-naphthoate,最后生成甲基萘醌[30]。H.pylori变型的 Futalosine通路的具体过程有待进一步研究。
生物素,也叫做维生素H,是所有生物体生长必不可少的辅因子。Lin等[31]提出大肠杆菌的生物素合成途径如图3所示,他们认为生物素合成途径的开端模仿脂肪酸合成途径。最后四步生成生物素的过程在所有生物体中保守,依次经过 BioF、BioA、BioD、BioB 的作用合成生物素[32]。在 H.pylori基因组中几乎都找到了上述酶的相似基因(见图3的括号内),但针对H.pylori生物素合成通路的研究还很少。Przemyslaw等[33]纯化了 HpBioD的结构,并与其他物种的BioD比较分析,总结出BioD的家族特点,提出该酶是抗菌药物设计的新靶点。

图3 大肠杆菌生物素合成通路Fig.3 Biotin synthesis of E.coli
维生素B6被公认为许多细胞代谢反应的辅因子,Grubman等[34]首次提出维生素B6与细菌致病机制密切相关。他们证明维生素B6对人类病原体的鞭毛结构、糖基化、运动能力具有重要作用,并找到H.pylori的两个致病因子——HpPdxA和 HpP-dxJ,分别由HP1583和HP1582编码。
2.4 核苷酸代谢
嘌呤和嘧啶是合成核苷三磷酸(NTPs)的基础,是核酸的前体。H.pylori无法从头合成嘌呤,而是通过补充途径合成嘌呤。一方面,H.pylori的生长需要嘌呤(Abstract no.2273,Miller EF.& Maier RJ,Annual Meeting of the American Society for Microbiology,May 2011),而生物信息学分析表明,嘌呤从头合成第一步需要的IMP无法获得,因为合成IMP需要10种酶,但基因组中只能找到两种酶的相似基因(HP1218、HP1112)。另一方面,Liechti等[35]证实,H.pylori从环境中吸收原料,如:肌苷、腺苷、鸟嘌呤、鸟苷、黄嘌呤、黄苷,然后通过补充途径合成IMP,IMP继而分别生成ATP、GTP(见图4)。补充途径所需原料由HP1180编码的转运蛋白配合质子泵转运至胞内[36]。HP0930编码固定相生存蛋白,它将 GMP、XMP、IMP、AMP分别水解为相应的核苷[35]。之前的研究认为H.pylori存在腺嘌呤脱氨酶活性,但最新研究反驳了这一观点,该酶的脱氨作用针对的是腺苷而不是腺嘌呤[36],但基因组中没找到对应的基因。

图4 H.pylori嘌呤补充合成途径概略图Fig.4 Purine salvage pathway of H.pylori
UTP、CTP是RNA合成的前体,经9种酶从头合成。暂时认为H.pylori嘧啶的从头合成通路以大肠杆菌为模板,编码这9种酶的相似基因依次是:HP0919/HP1237、HP1084、HP0581/HP0266、HP1011、HP1257、HP0005、HP0777、HP0198、HP0349。HP0777 编码UMP激酶(UMPK),而且与其他物种以Mg2+作为辅因子不同,它更倾向于把 Mn2+作为辅因子[37]。Chu等[38]进一步揭示GTP对HpUMPK的别构调节机制,GTP与HpUMPK结合成一个稳定的六聚体,该齐聚反应状态不易释放产物UDP,但GTP结合的同时引发UDP结合区域(α2)变构而使紧密状态松散,从而帮助释放UDP。除了HP0777,其他酶-基因关系和脱氧核苷酸的生物合成途径还没有新的研究进展,故不赘述。
3 H.pylori其他特征
幽门螺旋杆菌的质子门控尿素通道即HpUREI是其能够生存在胃酸性环境中的重要原因。Strugatsky等[39]揭示了幽门螺旋杆菌J99尿素的质子门控通道,这种六聚体结构是尿素及其他小酰胺溶质的新渗透模式的基础。通道在中性pH条件下关闭,在酸性pH条件下打开,并允许尿素迅速进入细胞质与脲酶接触。脲酶产生的NH3和CO2能够中和进入胞内的质子,所以即使胃液pH低于2.0,细胞周质pH仍维持在6.1左右。HP0071是编码该通道蛋白的相似基因。Cornally等[40]提出HP1193编码的醛酮还原酶可能也与细菌适应酸环境生长有关。
H.pylori表达一种致病因子——CagA(cytotoxin-associated gene A),CagA上调胃粘膜上皮细胞的精胺氧化酶水平[41]。精胺氧化酶分解精胺产生H2O2,从而导致细胞凋亡和DNA损伤。Lee等在H.pylori没有检测到精胺,而有亚精胺[42]。亚精胺由HP0832编码的亚精胺合酶(spermidine synthase,SPMS)催化腐胺和脱羧的S-腺苷甲硫氨酸而生成,亚精胺合成通路是H.pylori聚胺的主要来源。HpSPMS与其他物种的SPMS序列相似性低于20%,而且缺少一段信号序列。晶体结构显示HpSPMS有一个N端-beta折叠区和一个C端-罗斯曼样结构域[43],两个结构域间有一个与众不同的结合口袋,且酸性静电表面电位较少,还有一个大的包藏空间,这些特点可能使HpSPMS成为潜在的抗菌药物靶标,因为SPMS是菌体细胞保持活力必不可少的。虽然亚精胺合成通路的前几个酶(speB、speC、speD)在基因组中没找到,但是不能排除也像HpSPMS一样序列相似性太低的情况。
4 总结与展望
全基因组尺度代谢网络的构建,从系统的角度,由生物体内各种分子的相互作用的研究到代谢通路、网络、模块,最终对整个生命活动的路线图有了完整的理解,进而对生物代谢功能进行重新设计和合理的工程改造。代谢网络模型重构过程非常复杂,尤其是基因-蛋白质-反应关系的鉴别、网络模型的评估验证等步骤工作量大,耗费时间长。利用代谢网络重构工具[44]可以缩短一定时间,提高工作效率,但构建高精度代谢网络仍离不开大量人工验证工作。一般先利用生物数据库或软件对代谢网络进行初步重构,再根据生物文献和生物实验数据对初步重构的生物代谢网络模型进行验证和修正。目前H.pylori代谢网络模型对必需基因的预测准确性不高,重构更加连续、完整的高精度模型是最终目标。
本文对H.pylori特异性的重要代谢通路进行了较为全面的综述,一方面,从基因水平阐述代谢通路与基因的关系,为重构更高质量代谢网络提供参考;另一方面,本文详细分析了关键酶对H.pylori生理的重要作用,为开发抗幽门螺杆菌药物、诊断幽门螺杆菌感染情况带来启示。H.pylori代谢网络大部分是保守的,多采用与大肠杆菌或其他近缘物种非常相似的代谢形式,然而,在进化过程中一部分H.pylori特异性的代谢过程对实际应用具有更大价值。生物体的代谢过程与转录调控、信号转导机制密切相关,为了更加精确地做出预测,更加真实地模拟生物体生命活动的路线图,需要在代谢网络基础上,整合转录调控网络和信号转导网络。整合的H.pylori调控网络、信号网络和代谢网络模型将是未来的一个研究方向。
References)
[1] THIELE I,PALSSON B Ø.A protocol for generating a high-quality genome-scale metabolic reconstruction[J].Nat Protoc,2010,5(1):93-121.
[2] OBERHARDT M A,PALSSON B Ø,PAPIN J A.Applications of genome-scale metabolic reconstructions[J].Mol Syst Biol,2009,5:320.
[3] BONECA I G,DE R H,EPINAT J C,et al.A revised annotation and comparative analysis of Helicobacter pylori genomes[J].Nucleic Acids Research,2003,31(6):1704-1714.
[4] SCHILLING C H,COVERT M W,FAMILI I,et al.Genome-scale metabolic model of Helicobacter pylori 26695[J].J Bacteriol,2002,184(16):4582-4593.
[5] THIELE I,VO T D,PRICE N D,et al.Expanded metabolic reconstruction of Helicobacter pylori(iIT341 GSM/GPR):an in silico genome-scale characterization of single-and double-deletion mutants[J].J Bacteriol,2005,187(16):5818-5830.
[6] RESENDE T,CORREIA D M,ROCHA M,et al.Reannotation of the genome sequence of Helicobacter pylori 26695[J].J Integr Bioinform,2013,10(3):233.
[7] COVERT L.Perspectives on methodology for in vitro culture of Helicobacter pylori[J].Methods Mol Biol.2012,921:11-15.
[8] TESTERMANT L,CONN P B,MOBLEY H L,et al.Nutritional requirements and antibiotic resistance patterns of Helicobacter species in chemically defined media[J].J Clin Microbiol.2006,44(5):1650-1658.
[9] BARISON N,CENDRON L,TRENTO A,et al.Structural and mutational analysis of TenA protein(HP1287)from the Helicobacter pylori thiamin salvage pathway-evidence of a different substrate specificity[J].FEBS J,2009,276(21):6227-6235.
[10] PSAKISG,SAIDIJAM M,SHIBAYAMA K,et al.The sodium-dependent D-glucose transport protein of Helicobacter pylori[J].Mol Microbiol,2009,71(2):391-403.
[11] MARAIS A,MENDZ G L,HAZELL S L,et al.Metabolism and genetics of Helicobacter pylori:the genome era[J].Microbiol Mol Biol Rev,1999,63(3):642-674.
[12] ZHUO C,QING L Z,RUI G G.Inhibition of fumarase by bismuth(III):Implications for the tricarboxylic acid cycle as a potential target of bismuth drugs in Helicobacter pylori[J].Biometals,2012,25(1):95-102.
[13] KATHER B,STINGL K,VANDER R M,et al.Another unusual type of citric acid cycle enzyme in Helicobacte rpylori:themalate:quinoneoxidoreductase[J].J Bacteriol,2000,182(11):3204-3209.
[14] HUANG D,LIU J,SHEN G.Cloning,expression,and enzymatic characterization of isocitrate dehydrogenase from Helicobacter pylori[J].Protein J,2009,28(9-10):443-447.
[15] HU T,WU D,CHEN J,et al.The catalytic intermediate stabilized by a“down”active site loop for diaminopimelate decarboxylase from Helicobacter pylori.Enzymatic characterization with crystal structure analysis[J].J Biol Chem,2008,283(30):21284-21293.
[16] SHIBAYAMA K,TAKEUCHI H,WACHINO J,et al.Biochemical and pathophysiological characterization of Helicobacter pylori asparaginase[J].Microbiol Immunol,2011,55(6):408-417.
[17] SHIBAYAMA K,WACHINO J,ARAKAWA Y,et al.Metabolism of glutamine and glutathione via γ-glutamyltranspeptidase and glutamate transport in Helicobacter pylori:possible significance in the pathophysiology of the organism[J].Mol Microbiol,2007,64(2):369-406.
[18] LEDUC D,GALLAUD J,STINGL K,et al.Coupled amino acid deamidase-transport systems essential for helicobacter pylori colonization[J].Infect Immun,2010,78(6):2782-2792.
[19] SCHMEES C,PRINZ C,TREPTAU T,et al.Inhibition of T-cell proliferation by Helicobacter pylori gamma-glutamyl transpeptidase[J].Gastroenterology,2007,132(5):1820-1833.
[20] RIMBARA E,MORI S,KIM H,et al.Role of γ -glutamyltranspeptidase in the pathogenesis of Helicobacter pylori infection[J].Microbiol Immunol,2013,57(10):665-673.
[21] HAGEN S J,OHTANI M,ZHOU J R,et al.Inflammation and foveolar hyperplasia are reduced by supplemental dietary glutamine during Helicobacter pylori infection in mice[J].J Nutr,2009,139(5):912-918.
[22] MIXCOHA E,GARCIA-VILOCA M,LLUCH J M,et al.Theoreticalanalysis ofthe catalytic mechanism of Helicobacter pylori glutamate racemase[J].J Phys Chem B,2012,116(41):12406-12414.
[23] DE JONGE B L,KUTSCHKE A,URIA-NICKELSEN M,et al.Pyrazolopyrimidinediones are selective agents for Helicobacter pylori that suppress growth through inhibition of glutamate racemase(MurI)[J].Antimicrob Agents Chemother,2009,53(8):3331-3336.
[24] BASARAB G S,HILL P,EYERMANN C J,et al.Design of inhibitors of Helicobacter pylori glutamate racemase as selective antibacterial agents:incorporation of imidazoles onto a core pyrazolopyrimidinedione scaffold to improve bioavailabilty[J].Bioorg Med Chem Lett,2012,22(17):5600-5607.
[25] LIU J S,CHENG W C,WANG H J,et al.Structure-based inhibitor discovery of Helicobacter pylori dehydroquinate synthase[J].Biochem Biophys Res Commun,2008,373(1):1-7.
[26] HAN C,WANG L,YU K,et al.Biochemical characterization and inhibitor discovery of shikimate dehydrogenase from Helicobacter pylori[J].FEBS J,2006,273(20):4682-4692.
[27] CHENG W C,CHEN Y F,WANG H J,et al.Structures of Helicobacter pylori shikimate kinase reveal a selective inhibitor-induced-fit mechanism[J].PLoS One,2012,7(3):e33481.
[28] HIRATSUKA T,FURIHATA K,ISHIKAWA J,et al.An alternative menaquinone biosynthetic pathway operating in microorganisms[J].Science,2008,321:1670-1673.
[29] DAIRI T.Menaquinone biosyntheses in microorganisms[J].Methods Enzymol,2012,515:107-22.
[30] MISHRA V,RONNING D R.Crystal structures of the helicobacter pylori MTAN enzyme reveal specific interactions between S-Adenosylhomocysteine and the 5’-Alkylthio binding subsite[J].Biochemistry,2012,51(48):9763-9772.
[31] LIN S,HANSON R E,CRONAN J E.Biotin synthesis begins by hijacking the fatty acid synthetic pathway[J].Nat Chem Biol,2010,6:682-688.
[32] COREY J F,JOSEPH T J.Biotin synthase:Insights into radical-mediated carbon-sulfur bond formation[J].Biochemistry,2012,49(31):6746-6760.
[33] POREBSKI P J,KLIMECKA M,CHRUSZCZ M,et al.Structural characterization of Helicobacter pylori dethiobiotin synthetase reveals differences between family members[J].FEBS J,2012,279(6):1093-1105.
[34] GRUBMAN A,PHILLIPS A,THIBONNIER M,et al.Vitamin B6 is required for full motility and virulence in Helicobacter pylori[J].Mbio,2010,1(3):e00112-10.
[35] LIECHTI G,GOLDBERG J B.Helicobacter pylori relies primarily on the purine salvage pathway for purine nucleotide biosynthesis[J].J Bacteriol,2012,194(4):839-854.
[36] MILLER E F,VAISH S,MAIER R J.Efficiency of purine utilization by helicobacter pylori:Roles for adenosine deaminase and a NupC homolog[J].PLoS ONE,2012,7(6):e38727.
[37] LEE M J,CHIEN-LIANG L,TSAI J Y,et al.Identification and biochemical characterization of a unique Mn2+-dependent UMP kinase from Helicobacter pylori[J].Arch Microbiol,2010,92(9):739-746.
[38] CHU C H,LIU M H,CHEN P C,et al.Structures of Helicobacter pylori uridylate kinase:insight into release of the product UDP[J].Acta Crystallogr D Biol Crystallogr,2012,68(7):773-783.
[39]STRUGATSKY D,MCNULTY R,MUNSON K,et al.Structure of the proton-gated urea channel from the gastric pathogen Helicobacter pylori[J].Nature,2013,493:255-258.
[40] CORNALLY D,MEEB,MACDONAILL C,et al.Aldo-keto reductase from Helicobacter pylori-role in adaptation to growth at acid pH[J].FEBS J,2008,275:3041-3050.
[41] CHATURVEDI R,DE SABLET T,PEEK R M,et al.Spermine oxidase,a polyamine catabolic enzyme that links Helicobacter pylori CagA and gastric cancer risk[J].Gut Microbes,2012,3(1):48-56.
[42] LEE M J,HUANG C Y,SUN Y J,et al.Cloning and characterization of spermidine synthase and its implication in polyamine biosynthesis in Helicobacter pylori strain 26695[J].Protein Expr Purif,2005,43:140-148.
[43] LU P K,TSAI J Y,CHIEN H Y,et al.Crystal structure of Helicobacter pylori spermidine synthase:a Rossmann-like fold with a distinct active site[J].Proteins,2007,67(3):743-54.
[44] PITKNEN E,ROUSU J,UKKONEN E.Computational methods for metabolic reconstruction[J].Current Opinion in Biotechnol,2010,21(1):70.

