苎麻纤维素合成酶基因BnCesA4 cDNA序列的克隆与表达分析
刘昱翔,陈建荣,彭 彦,黄 妤,赵 燕,黄丽华,郭清泉,张学文*
(1湖南农业大学生物科学技术学院,长沙410128;2长沙学院生物工程与环境科学系,湖南长沙410003)
苎麻纤维是最优良的植物韧皮纤维之一,具有极佳的弹性和柔软性,是植物界最长的优质纤维。纤维的组成单位纤维素大分子是由多个D-吡喃葡萄糖残基以β-1,4-糖苷键联结而成的不分枝多糖,在植物体内纤维素构成纤维素糖苷网,是承受机械压力的支撑结构,并调控植物形态学上的某些特性与特征[1]。
Pear等从棉纤维cDNA文库中鉴定出2个纤维素合成酶基因GhCesA1和GhCesA2[2]。植物纤维素合成酶包含保守的D,D,D区和QXXRW结构域,该结构中的第一个D,天冬氨酸残基在Mg2+存在的情况下是唯一能够与尿苷二磷酸-葡萄糖(UDP-glc)结合的位点,而第三个D,TED则可能起到催化作用[3,4]。植物纤维素合成酶在氨基酸序列的N-端具有锌指结构域,这个锌指结构表明植物纤维素合成酶蛋白至少和一个其他蛋白有互作[5,6]。植物纤维素合成酶共有8个跨膜结构域,2个位于N-端,6个位于C-端,跨膜结构域在细胞质膜上形成供新合成糖苷链进入细胞壁的通道[7~9]。植物纤维素合成酶基因表达状况的研究发现:不同的纤维素合成酶基因在植物不同部位表达,一种纤维素合成酶基因可以在多个部位表达,也可能在某一特定部位专一性地表达[10~12]。同一细胞中有多个纤维素合成酶共同表达,而不同的纤维素的表达受到不同的调控因素的控制[13,14]。
2008年田志坚等利用同源克隆的方法克隆获得了第一个苎麻纤维素合成酶基因BnCesA1缺少5'端部分碱基的cDNA序列,随后蒋杰等以此为基础获得了BnCesA基因的全长cDNA序列。半定量实验表明,BnCesA1在根、茎、叶、芽4种组织中均有表达,且表现出茎>叶>芽>根的表达规律[15~17]。本研究在克隆获得新的苎麻纤维素合成酶基因基础上,对其序列进行生物信息学分析和荧光定量RTPCR,以为进一步研究苎麻韧皮纤维的合成过程提供一定的理论依据。
1 材料与方法
1.1 材料
苎麻(Boehmeria nivea(Linn.)Gaud.)栽培种湘苎1号、湘苎3号、湘潭大叶白、城步青麻均由湖南农业大学苎麻研究所提供。
大肠杆菌DH5菌株,由湖南农业大学细胞生物学实验室保存。
1.2 方法
1.2.1 苎麻纤维素合成酶基因BnCesA4 cDNA序列克隆
通过GenBank下载多种已公布的植物纤维素合成酶基因序列构建数据库,利用本地BLAST工具与前期研究中获得的苎麻转录组数据进行序列比对。从苎麻转录组数据中发掘出与多种纤维素合成酶具有较高同源性的片段CL6473。针对该序列设计一对特异性引物:
CL6473 Primer F:5'-AATGCCGATGAGGTTTC CAGAG-3'
CL6473 Primer R:5'-AACTGCTCGTTACTCCA CCAATC-3'
以生长旺盛的湘苎3号苎麻中上部茎秆组织为材料,提取苎麻总RNA,反转录合成cDNA第一链,以其为模板,扩增片段 CL6473。扩增产物连接pMD18-T载体,转化大肠杆菌,筛选阳性克隆送交铂尚生物公司测序,并将测序结果与转录组数据进行比较。
利用TaKaRa公司3'及5'RACE试剂盒分别扩增片段CL6473的3'及5'末端cDNA序列。参照试剂盒说明设计3'RACE引物 CL6473 RACE3:5'-TACTGTACCTTGCCTGCTATCTG-3'和 5'RACE巢式PCR引物CL6473 5'GSP1:5'-GATTTCCCTCTC TTCTCTCGTAC-3';CL6473 5'GSP2:5'-GCTCTC CGTCCACAGTCACTTC-3'。3'及5'RACE 所得片段送交铂尚生物公司测序。
将CL6473核心片段与3'及5'RACE克隆获得的两端序列进行拼接,最终得到BnCesA4基因全长cDNA序列。
1.2.2 苎麻纤维素合成酶基因BnCesA4的生物信息学分析
将BnCesA4序列与GenBank核酸数据库进行联机比对,分析BnCesA4序列与不同植物纤维素合成酶基因的同源关系。使用软件DNAMAN查找开放阅读框,并翻译蛋白质,预测BnCesA4蛋白的分子量、等电点等基本理化性质。通过NCBI网站Conserved Domain Search Service工具分析蛋白质的保守结构域。使用 http://www.cbs.dtu.dk/services/TMHMM/网站预测BnCesA4蛋白的跨膜结构域。
1.2.3 苎麻纤维素合成酶的进化分析
下载多种植物纤维素合成酶氨基酸序列共66条,与3种苎麻纤维素合成酶BnCesA1、BnCesA3和BnCesA4通过MEGA软件以Neighbor-Joining的方式共同构建系统发育树。
1.2.4 苎麻纤维素合成酶基因BnCesA4表达量的荧光定量PCR分析
分离湘苎1号、湘苎3号、湘潭大叶白及城步青麻等4个品种的成熟阶段茎秆,分韧皮部和木质部组织并提取总RNA。这4个品种的苎麻在原麻产量和纤维支数上都有明显差异,湘苎1号高产中支,湘苎3号高产高支,湘潭大叶白中产低支,城步青麻低产高支[18]。依据荧光定量引物设计原则设计BnCesA4基因荧光定量引物:
RT F:5'- ATGGAGGAATGGAGGAAGAAG -3'
RT R:5'- AGAATCACAATCCGAAGAAGG -3'
扩增片段长度200 bp。选择肌动蛋白Actin1作为内照基因。Actin1基因上下游引物分别为Actin F:5'-GCTCCGTTGAACCCTAAG-3';Actin R:5'-CGATTGTGATGATTT-3'。使用TaKaRa公司SYBR Premix Ex Taq(TaKaRa)荧光定量试剂盒完成荧光定量实验,分析BnCesA4基因在4个代表性苎麻品种的木质部与韧皮部中的表达差异。
2 结果分析
2.1 苎麻纤维素合成酶基因BnCesA4 cDNA序列克隆与生物信息学分析
将核心片段CL6473与通过3'及5'RACE获得的两端序列进行拼接,得到长度为4 008 bp的cDNA序列。BnCesA4基因编码区全长3 270 bp,5'端非编码区236 bp,3'端非编码区502 bp,推测其编码一个1 090 aa的蛋白质,预测该蛋白质的分子质量123.0 kD,理论等电点为7.63。苎麻纤维素合成酶基因BnCesA4 cDNA核苷酸序列及相对应的蛋白质序列如图1所示。


图1 BnCesA4基因cDNA序列及推测的蛋白质序列Fig.1 The cDNA sequence and its putative protein sequences of BnCesA4 gene
将BnCesA4序列与GenBank进行联机BLAST分析,结果显示其核酸序列与亮叶桦(Betula luminifera CesA8 KC204972)、欧洲/美洲山杨(Populus tremula×Populus tremuloides CesA2 AY573572)、栽培种毛白杨(Populus tomentosa cultivar CV-BJHR01 CesA5 HQ585873)、陆地棉(Gossypium hirsutum CesA9 JQ345699)、蓖麻(Ricinus communis CesA6 XM_002524253)等序列同源达到80%以上;其氨基酸序列与可可(Theobroma cacao CesA6 XP_007019545)、蓖麻(Ricinus communis CesA6 XP_002524299)、亮叶桦(Betula luminifera CesA8 AGV22110)、陆地棉(Gossypium hirsutum CesA9 AFB18639)、毛白杨(Populus tomentosa CesA AFZ78561)序列同源均超过85%。将BnCesA4氨基酸序列与AtCesA1和两种已在GenBank中登录的苎麻纤维素合成酶BnCesA1、BnCesA3进行多重序列比对,结果如图2所示。比对结果显示,BnCesA4在氨基酸序列上与3种纤维素合成酶有很高的相似度,这表明克隆获得的BnCesA4序列为BnCesA基因家族成员。BnCesA4蛋白TMHMM预测显示具有8个跨膜结构域(287~304、311 ~330、867 ~889、901 ~923、938 ~960、985~1007、1022~1044、1 049~1 071),2个位于 N 端,6个位于C端。氨基酸序列N端39~82位置上具有锌指结构域,该结构域被认为在纤维素合成酶各亚基之间的相互作用上起到重要作用,在氨基酸829~833的位置上有与底物结合和催化相关的QVLRW保守结构域。研究显示,纤维素合成酶是N-糖基化的,在拟南芥的研究中也证实CesA蛋白是被磷酸化的,而这些修饰对蛋白质的活性是极为重要的。motif预测显示,BnCesA4有5个糖基化位点(123~126、142~145、577 ~580、822 ~825、964 ~967)。BnCesA4具有1个CAMP磷酸化位点(691~694)和9个酪氨酸激酶(CK2)磷酸化位点(30~33、148 ~151、343 ~346、369 ~372、492 ~495、727 ~730、743 ~746、787 ~790、917 ~920)。

图2 AtCesA1、BnCesA1、BnCesA3与BnCesA4蛋白质序列比对结果Fig.2 Multiple Alignment of protein sequence of AtCesA1,BnCesA1,BnCesA3 and BnCesA4
2.2 苎麻纤维素合成酶的进化分析
从GenBank下载多种植物纤维素合成酶氨基酸序列与苎麻纤维素合成酶BnCesA1、BnCesA3和BnCesA4共同构建系统发育树(图3),以了解纤维素合成酶之间的系统发育关系,实心圆指示出3种苎麻纤维素合成酶。
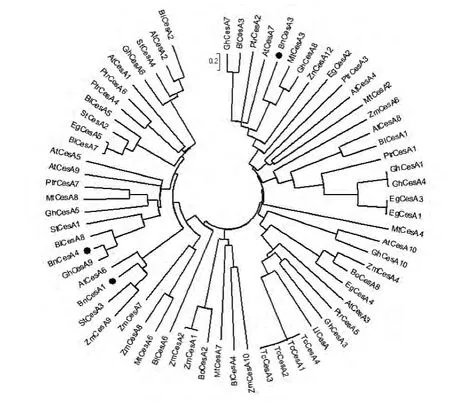
图3 纤维素合成酶基因系统发育树Fig.3 Phylogenetic tree of cellulose synthase genes from different species
系统发育树显示木本植物与草本植物CesA在系统发育树中呈分散分布,没有出现木本植物或草本植物CesA聚集的分枝,推测CesA的分化应早于草本植物与木本植物的分化。而不同植物的纤维素合成酶在系统发育树中的分布也分散,CesA的分化早于这些物种出现。某些同一物种的多个CesA分布在同一分枝上,如可可纤维素合成酶TcCesA1、2、3、4,可能是相同基因产生的多个拷贝,纤维素合成酶家族成员之间可能在功能上存在冗余。BnCesA4在进化关系上与GhCesA9最为接近。
2.3 苎麻纤维素合成酶基因BnCesA4表达量的荧光定量PCR分析
荧光定量实验选择在BnCesA4基因高可变区设计引物,有效避免了具有较高同源性的纤维素合成酶家族其他基因的影响,溶解曲线峰值单一,引物特异性良好。采用双Δct法对荧光定量结果进行统计,获得BnCesA4基因在不同品种的木质部与韧皮部的表达量关系如图4所示。
BnCesA4基因在4个品种的苎麻木质部和韧皮部都有表达,其中在城步青麻韧皮部表达量最高,在湘苎1号木质部中表达量最低,湘苎1号中韧皮部表达量是木质部表达量的2倍,而其他品种中韧皮部与木质部的表达量差距都不大。除湘苎3号中木质部表达量略高于韧皮部外,在其他3个品种中都表现出韧皮部的表达量高于木质部。BnCesA4基因在4个品种韧皮部表达水平差异较小,说明BnCesA4在4个品种的苎麻韧皮纤维的合成过程中表达量稳定。4个品种苎麻韧皮部表达水平与苎麻的原麻产量和纤维支数之间并没有发现直接联系,如湘潭大叶白属于中产低支的苎麻品种,但其韧皮部BnCesA4基因表达量高于属于高产高支品种的湘苎3号。

图4 苎麻品种间木质部与韧皮部BnCesA4基因表达差异Fig.4 Differential expression of BnCesA4 in phloem and xylem among different ramie varieties
3 结论与讨论
田志坚和蒋杰克隆获得了第一个苎麻纤维素合成酶基因BnCesA1,并通过半定量RT-PCR实验发现BnCesA1基因在根、茎、叶和芽组织中均有表达,但表达量有差异。苎麻具有发达的韧皮部纤维,是重要的纺织工业原料,而利用苎麻木质纤维素生产生物能源成为苎麻研究的热点,木质纤维的质量直接关系到生产成本与转化过程中的二次污染。本研究克隆获得了BnCesA4基因全长cDNA序列,利用多种已知植物纤维素合成酶氨基酸序列构建系统发育树,更重点关注了该基因在苎麻茎秆的韧皮部及木质部中的表达。BnCesA4基因在苎麻茎秆韧皮部与木质部都有表达,其参与了苎麻韧皮纤维的合成过程,但不具有韧皮部表达的特异性。同时笔者也发现在纤维素集中合成的韧皮部中,BnCesA4基因的表达量并没有极显著的高于木质部,而BnCesA4的表达量与苎麻纤维产量与品质也没有直接的联系。其可能原因:首先,纤维素的生物合成过程是多个纤维素合成酶共同参与的结果,某一种纤维素合成酶并不能决定纤维素的产量与品质。其次,纤维素合成酶需要与其他多种蛋白构成复合体来行使功能,纤维素合成酶直接合成纤维素,而调节纤维素的产量与质量则由其他类型的蛋白完成,如纤维素内切酶对合成的葡聚糖链的修饰与加工过程,最终使其转化为纤维素微纤丝[19~21]。第三,原麻产量与纤维支数取决于多种因素的共同作用,如原麻胶质含量、苎麻纤维分子结晶度等[18]。所以要实现利用基因工程手段改良苎麻纤维产量与品质,需要克隆获得更多苎麻纤维素合成酶基因家族成员,并对其表达规律有充分的认识,在此基础上完善对纤维素合成酶复合体的研究,从分子层面上进一步阐明苎麻特殊的纤维发育机制。
[1]Brown RMJr,Saxena IM.Cellulose biosynthsis:a model for understanding the assembly of biopolymers[J].Plant Physiol Biochem,2000,38(1/2):57 -67.
[2]Pear J,Kawagoe Y,Schreckengost W,et al.Higher plants contain homologs of the bacterial CelA genes encoding the catalytic subunit of the cellulose synthase[J].Proc Natl Acad Sci USA,1996,93:12637 -12642.
[3]Charnock SJ,Davies GJ.Structure of the nucleotidediphospho - sugar transferase,SpsA,from Bacillus subtilis,in native and nucleotide - complexed forms[J].Biochemistry,1999,38:6380 -6385.
[4]Morgan JLW,Joanna Strumillo,Jochen Zimmer.Crystallographic snapshot of cellulose synthesis and membrane translocation[J].Nature,2013,493:181 -186.
[5]Kawagoe Y,Delmer DP.Pathway and genes in involved in cellulose biosynthesis[J].Genet Eng,1997,19:63 -87.
[6]Kawagoe Y,Delmer DP.Cotton CelA1 has a LIM - like Zn binding domain in the N-terminal cytoplasmic region[J].Plant Physio,1997b,114:85.
[7]Darby M Harris,Kendall Corbin,Tuo Wang,et al.Cellulose microfibril crystallinity is reduced by mutating C-terminal transmembrane region residues CESA1A903V and CESA3T942I of cellulose synthase [J].Proc Natl Acad Sci USA,2012,109:4098 -4103.
[8]Zhang B,Deng L,Qian Q,et al.A missense mutation in the transmembrane domain of CESA4 affects protein abundance in the plasma membrane and results in abnormal cell wall biosynthesis in rice [J].Plant Mol Biol,2009,71:509 -524.
[9]Kurek I,Kawagoe Y,Jacob - Wilk D.Dimerization of cotton fiber cellulose synthase catalytic subunits occurs via oxidation of the zinc - binding domains[J].Proc Natl Acad Sci USA,2002,99:11109 -11114.
[10]Turner SR,Somerville CR.Collapsed xylem phenotype of Arabidopsis identifies mutants deficient in cellulose deposition innthe secondary cell wall[J].Plant Cell,1997,9:689-701.
[11]Taylor NG,Scheible WR,Cutler S,et al.The irregular xylem 3 locus of Arabidopsis encodes a cellulose synthase required for secondary cell wall synthesis[J].Plant Cell,1999,11:769 -779.
[12]Olawole OO,Evert J,Vincken JP,et al.Differential expression of cellulose synthase(CesA)gene transcriptsin potato as revealed by QRT - PCR[J].Plant Physiology and Biochemistry,2009,47:1116 -1118.
[13]Richmond TA,Somerville CR.The cellulose synthase superfamily[J].Plant Physiol,2000,124:495 -498.
[14]Monika S Doblin,Isaac Kurek,Deborah Jacob - Wilk,et al.Cellulose biosynthesis in plants:from genes to rosettes[J].Plant and Cell Physiology,2002,43(12):1407 -1420.
[15]田志坚,易 蓉,陈建荣,等.苎麻纤维素合成酶基因cDNA 的克隆及表达分析[J].作物学报,2008,34(1):78-83.
[16]易 蓉,田志坚,黄丽华,等.苎麻纤维素合成酶基因cDNA的功能分析[J].湖南农业大学学报(自然科学版),2009,35(1):13 -16.
[17]蒋 杰,揭雨成,周清明,等.苎麻纤维素合酶基因BnCesA1全长cDNA的克隆与表达分析[J].植物遗传资源学报,2012,13(5):851 -857.
[18]郭清泉,胡日生,孙焕良,等.苎麻胶质的基因型差异与成因及育种中利用研究Ⅰ.苎麻胶质及其组分含量的基因型差异[J].湖南农业大学学报(自然科学版),2000,26(5):340 -342.

