嗜血杆菌属CODEHOP PCR检测方法的建立及在树鼩检测中的应用
邢 进,冯育芳,岳秉飞,贺争鸣,孙晓梅,代解杰
嗜血杆菌属CODEHOP PCR检测方法的建立及在树鼩检测中的应用
邢 进1,冯育芳1,岳秉飞1,贺争鸣1,孙晓梅2,代解杰2
目的 建立树鼩中嗜血杆菌的CODEHOP PCR快速检测方法,为树鼩微生物质量控制提供参考。方法 应用CODEHOP在线简并引物设计工具,比对NCBI中6株嗜血杆菌的外膜蛋白序列设计简并引物。通过4株嗜血杆菌标准菌株和24株它属菌株进行特异性和敏感性评价,将建立CODEHOP PCR检测方法,应用于树鼩嗜血杆菌的检测。结果 简并引物HF1/HR8和HF3/HR3扩增标准菌株副鸡嗜血杆菌(ATCC 29545)、溶血嗜血杆菌(ATCC 33390)、流感嗜血杆菌(ATCC 33391)、副流感嗜血杆菌(ATCC 33392)的目的片段分别为506 bp~535 bp和344 bp~390 bp,经测序和NCBI Blast比对,结果准确。两对引物组合,能够有效的区分嗜血杆菌属菌株与它属菌株,检测限最低可达2.1 pg/μL。在受试的野生树鼩呼吸道样本中嗜血杆菌阳性率为33.3%(20/60)。结论 所建立的方法具有较好的特异性和敏感性,操作简便,能够用于动物样品的嗜血杆菌检测。
嗜血杆菌;CODEHOP PCR;树鼩;简并引物;检测
Supported by the Standardization of Experimental Tree Shrews and the Integrated Demonstration of Creation and Application of Major Human Disease Models of Tree Shrews (No. 2014BAI01B01) Corresponding author: He Zheng-ming, Email: zhengminghe57@163.com
嗜血杆菌属(Haemophilus.spp)是一群革兰氏阴性,不抗酸、不运动、无芽孢的短杆菌或球杆菌,绝大部分存在于人和动物的呼吸道粘膜[1]。引起人感染的主要有流感嗜血杆菌、副流感嗜血杆菌、溶血嗜血杆菌、副溶血嗜血杆菌、杜克雷嗜血杆菌等[2],其中流感嗜血杆菌的致病性最强,能够引起人,特别是婴幼儿的脑膜炎、呼吸道和生殖道感染[3]。副流感嗜血杆菌能够引起肺炎和细菌性心内膜炎,杜克雷嗜血杆菌能够引起软性下疳等[4]。引起动物疾病的嗜血杆菌中,副猪嗜血杆菌能够引起猪格拉泽氏病[5],副鸡嗜血杆菌能够引起传染性鼻炎,引发急性呼吸道疾病、关节炎及败血症[6]。如果能够同时检测嗜血杆菌的多种菌株,对于有效的防止疾病和感染的发生具有重要意义。但是某些动物源性的嗜血杆菌会转变为巴斯德菌或放线杆菌[4],某些嗜血杆菌的培养条件苛刻,需要X、V因子和二氧化碳。因此在检测中,将嗜血杆菌与巴斯德菌科的其它菌属相区别十分困难。
共有序列简并杂合寡核苷酸引物(Consensus Degenerate Hybrid Oligonucleotide Primers,CODEHOP)PCR方法能够扩增相同功能的蛋白基因。该方法通过网络数据库(http://blocks.fhcrc.org/codehop.html),将已知的不同微生物的同源蛋白进行比对,设计出高扩增效率的简并引物,实现对同源目的蛋白基因的扩增[7-8],解决了某些菌株没有已知蛋白编码基因序列的限制。
树鼩作为一种新型的实验动物,已经广泛的应用于微生物感染模型、呼吸系统、内分泌、神经系统疾病和肿瘤等方面的研究[9]。本研究旨在利用CODEOP PCR检测树鼩中嗜血杆菌的携带情况,为树鼩的进一步实验动物化和微生物质量控制奠定基础。
1 材料与方法
1.1 菌株 副鸡嗜血杆菌(ATCC 29545),溶血嗜血杆菌(ATCC 33390),流感嗜血杆菌(ATCC 33391)副流感嗜血杆菌(ATCC 33392)等4株嗜血杆菌购自美国标准生物品收藏中心(ATCC)。另有24株对照菌株用于检测方法的特异性测定,见表2。
1.2 设备及试剂 上海安亭WGP-600恒温培养箱,德国Hettich22R离心机、美国Thermo Nanodrop超微量分光光度计、美国ABI veriti96 PCR仪,美国Bio-Rad Powerpac HC电泳仪,美国Kodak GL212pro紫外凝胶成像系统。DNA提取试剂盒购自德国Qiagen公司,PCR试剂购自宝生物工程(大连)有限公司。
1.3 引物设计 将流感嗜血杆菌(GenBank登录号AAF97580.1)、副猪嗜血杆菌(GenBank登录号ADJ38784.1)、副溶血嗜血杆菌(GenBank登录号WP_005706527.1)、副流感嗜血杆菌(GenBank登录号WP_014064876.1)、溶血嗜血杆菌(GenBank登录号EIJ73911.1)和杜克雷嗜血杆菌(GenBank登录号EIJ73911.1)的外膜蛋白(OMP)氨基酸序列在CODEHOP在线引物设计工具中进行比对,并设计简并引物[10]。在所得引物中选择分数较高,简并度低的引物,由生工生物工程(上海)股份有限公司合成,见表1。共选择了5条上游引物(HF1~HF5)和8条下游引物(HR1~HR8)进行配对组合,根据特异性、敏感性测定,确定最佳引物对。
1.4 样本来源及DNA提取 受试样品来自昆明郊区采集的60只滇西亚种野生树鼩咽拭子及气管。用Qiagen DNA提取试剂盒(Cat.No.69504)提取上述样品的DNA,提取步骤根据试剂盒说明进行。本研究中所用菌株的DNA同样按此方法提取。
1.5 PCR反应体系和条件 PCR反应体系定为20 μL,其中10×PCR Buffer 2 μL,10 mmol/L dNTP Mix 1.6 μL,5 U/μL Taq HS DNA聚合酶0.2 μL,10 μmol/L上下游引物各0.5 μL,模板DNA 0.5 μL,无核酸酶水14.7 μL。扩增条件经过退火温度和反应时间优化后确定为如下反应条件:94 ℃预变性3 min;94℃变性30 s,54℃退火30 s,73 ℃延伸45 s,35个循环;最后72 ℃延伸7 min。PCR产物用1%琼脂糖在0.5%TBE中,以120V/60 mA电泳40 min。扩增产物经琼脂糖电泳后送北京擎科新业生物技术有限公司测序。
1.6 特异性测定 将所选上下游简并引物两两配对组合,对4株嗜血杆菌及24株它属菌株基因组DNA进行扩增,检测各引物对的特异性优劣。
1.7 敏感性测定 将4株嗜血杆菌的DNA分别做10倍系列稀释至10-6。用上述PCR方法进行扩增,检测该方法的最低检测限。
2 结 果
2.1 检测方法的建立和特异性 经引物的配对组合,两组引物HF1/HR8和HF3/HR3均能对4株嗜血杆菌扩增出单一的目的条带,引物对HF1/HR8扩增4株嗜血杆菌标准菌株目的片段大小分别为:流感嗜血杆菌535 bp,副鸡嗜血杆菌506 bp,副流感嗜血杆菌529 bp,溶血嗜血杆菌520 bp。引物对HF3/HR3扩增4株嗜血杆菌标准菌株目的片段大小分别为:流感嗜血杆菌345 bp,副鸡嗜血杆菌368 bp,副流感嗜血杆菌344 bp,溶血嗜血杆菌390 bp。目的片段经测序,流感嗜血杆菌ATCC33391的产物片段经Blast比对(http://blast.ncbi.nlm.nih.gov/Blast.cgi),结果准确无误,与GenBank中序列(登录号为LN831035.1)相似度为99%。其他3株标准菌株的产物片段经比对,未得出高相似度序列结果。
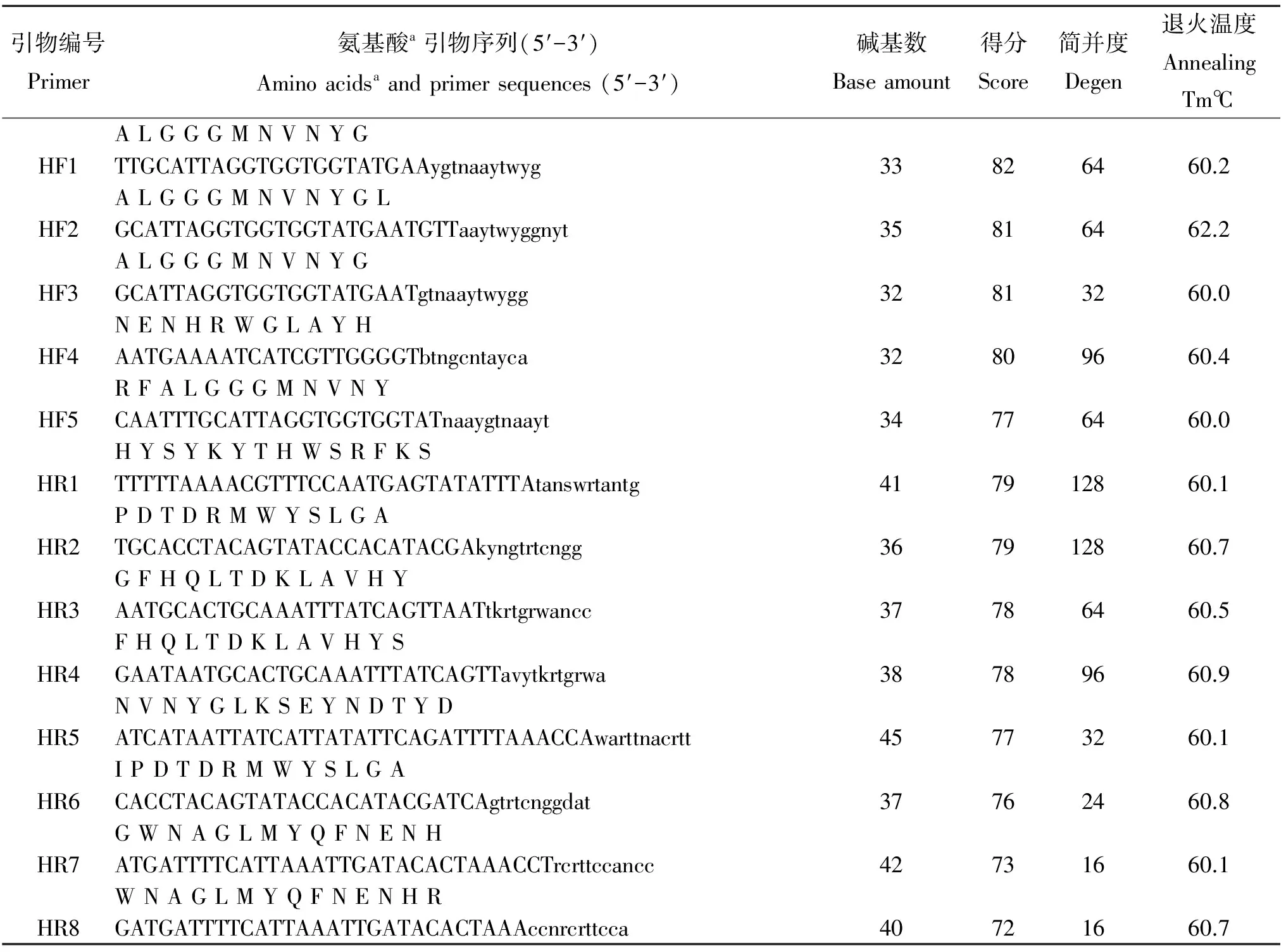
表1 本研究中的CODEHOP引物序列及其编码氨基酸序列Tab.1 Degenerate primers designed by CODEHOP used in this study
注:a, 下游引物HR1~HR8的碱基序列与氨基酸序列相反。
Note: a, the sequence of downstream primers HR1-HR8 with the animo acids are reverse.
2.2 所有配对引物扩增其它参考菌株,没有引物对能够实现对嗜血杆菌完全的特异性扩增。其中引物对HF1/HR8和HF3/HR3扩增的交叉反应最少,HF1/HR8与4株它属菌株有交叉反应,包括多杀巴斯德杆菌、达科马巴斯德杆菌、肉芽肿巴斯德杆菌和小鼠放线杆菌。HF3/HR3与产气巴斯德杆菌和鸟巴斯德杆菌存在交叉反应。因此利用两对引物扩增结果相组合的形式能够实现对嗜血杆菌的区分和检测,结果见表2。

1. 100 bp DNA Marker; 2-6. HF1/HR8扩增副鸡嗜血杆菌ATCC29545、溶血嗜血杆菌ATCC33390、流感嗜血杆菌ATCC33391和副流感嗜血杆菌ATCC33392的目的片段; 7-11. HF3/HR3扩增副鸡嗜血杆菌ATCC29545、溶血嗜血杆菌ATCC33390、流感嗜血杆菌ATCC33391和副流感嗜血杆菌ATCC33392的目的片段.
1: 100 bp DNA Marker; 2-6: Target fragments of HF1/HR8 amplifiedH.paragallinarumATCC29545,H.haemolyticusATCC33390,H.influenzaeATCC33391andH.parainfluenzaeATCC33392; 7-11: Target fragments of HF3/HR3 amplifiedH.paragallinarumATCC2954,H.haemolyticusATCC33390,H.influenzaeATCC33391 andH.parainfluenzaeATCC33392.
图1 HF1/HR8和HF3/HR3扩增嗜血杆菌阳性目的片段
Fig.1 Positive fragments of HF1/HR8 and HF3/HR3 primers amplified 4 Haemophilus strains respectively

1. 100 bp DNA Marker; 2-25. 引物对HF1/HR8扩增它属参考菌株结果,依次为嗜肺巴斯德杆菌ATCC12555、ATCC35149、多杀巴斯德杆菌ATCC43137、支气管鲍特杆菌ATCC19395、产气巴斯德杆菌ATCC27883、鸟巴斯德杆菌ATCC29546、溶血曼海姆杆菌ATCC33369、达科马巴斯德杆菌ATCC43325、肉芽肿巴斯德杆菌ATCC49244、粪产碱杆菌ATCC8750、鸡白痢沙门氏菌CMCC50047、奇异变形杆菌DJ150030、蜡样芽胞杆菌CMCC63301、绿脓杆菌ATCC47085、假结核耶尔森氏菌CMCC53518、念珠状链杆菌ATCC14647、鼠棒状杆菌CMCC65013、肺炎链球菌CMCC36001、乙型溶血性链球菌CMCC32210、大肠杆菌CMCC44110、泰泽氏菌RJ株、肺支原体PG34株、 痢疾志贺菌CMCC51252、小鼠放线杆菌ATCC49577.
1: 100 bp DNA Marker; 2-25: Results of primer pair HF1/HR8 amplified other species strains. From lanes 2 to 25, in order, isP.pneumotropicabiotypeHeyl ATCC12555,P.pneumotropicabiotypeJawetzATCC35149,P.multocidaATCC43137,B.bronchisepticaATCC19395,P.aerogenesATCC27883,P.aviumATCC29546,M.haemolyticaATCC33369,P.dagmatisATCC43325,M.granulomatisATCC49244,A.faecalisATCC8750,S.pullorumCMCC50047,P.mirabilisDJ150030,B.cereusCMCC63301,P.aeruginosaATCC47085,Y.pseudotuberculosisCMCC53518,S.moniliformisATCC14647,C.KutscheriCMCC65013,S.pneumoniaeCMCC36001,S.pyogenesCMCC32210,E.coliCMCC44110,C.piliformeStrain RJ,M.pulmonisStrainPG34,S.dysenteriaeCMCC51252,A.murisATCC49577.
图2 引物对HF1/HR8扩增参考菌株结果
Fig.2 Result of HF1/HR8 primers amplified reference strains

1. 100bp DNA Marker; 2-25. 引物对HF3/HR3扩增它属参考菌株结果,依次为嗜肺巴斯德杆菌ATCC12555、ATCC35149、多杀巴斯德杆菌ATCC43137、支气管鲍特杆菌ATCC19395、产气巴斯德杆菌ATCC27883、鸟巴斯德杆菌ATCC29546、溶血曼海姆杆菌ATCC33369、达科马巴斯德杆菌ATCC43325、肉芽肿巴斯德杆菌ATCC49244、粪产碱杆菌ATCC8750、鸡白痢沙门氏菌CMCC50047、奇异变形杆菌DJ150030、蜡样芽胞杆菌CMCC63301、绿脓杆菌ATCC47085、假结核耶尔森氏菌CMCC53518、念珠状链杆菌ATCC14647、鼠棒状杆菌CMCC65013、肺炎链球菌CMCC36001、乙型溶血性链球菌CMCC32210、大肠杆菌CMCC44110、泰泽氏菌RJ株、肺支原体PG34株、 痢疾志贺菌CMCC51252、小鼠放线杆菌ATCC49577.
1: 100 bp DNA Marker; 2-25: Results of primer pair HF3/HR3 amplified other species strains. From lanes 2 to 25, in order, isP.pneumotropicabiotypeHeylATCC12555,P.pneumotropicabiotypeJawetzATCC35149,P.multocidaATCC43137,B.bronchisepticaATCC19395,P.aerogenesATCC27883,P.aviumATCC29546,M.haemolyticaATCC33369,P.dagmatisATCC43325,M.granulomatisATCC49244,A.faecalisATCC8750,S.pullorumCMCC50047,P.mirabilisDJ150030,B.cereusCMCC63301,P.aeruginosaATCC47085,Y.pseudotuberculosisCMCC53518,S.moniliformisATCC14647,C.KutscheriCMCC65013,S.pneumoniaeCMCC36001,S.pyogenesCMCC32210, E.coli CMCC44110,C.piliformeStrain RJ,M.pulmonisStrain PG34,S.dysenteriaeCMCC51252, A.muris ATCC49577.
图3 引物对HF3/HR3扩增参考菌株结果
Fig.3 Result of HF3/HR3 primers amplified reference strains

表2 引物对HF1/HR8和HF3/HR3扩增参考菌株的结果Tab.2 Result of HF1/HR8 and HF3/HR3 primers amplified reference strains

表2(续表1)

表2(续表2)
注:“+”表示扩出目的片段,“—”表示未扩出目的片段。
Note: Symbols “+”, PCR result is positive; “—”, negative. b -- ATCC, American Type Culture Collection;CMCC, National Center for Medical Culture Collection.

1. 100 bp DNA marker;2-8. 由左至右为引物HF1/HR8扩增副鸡嗜血杆菌、溶血嗜血杆菌、流感嗜血杆菌和副流感嗜血杆菌1~10-6梯度DNA模板的敏感性;9. 空白对照。
1. 100bp DNA marker; 2-8. Sensitivity results of HF1/ HR8 primers amplified a series 10-fold dilutions of Haemophilus genomic DNA from 1 to 10-6in four picture from left to right, in order, areH.paragallinarum,H.haemolyticus,H.influenzaandH.parainfluenzae; 9. Blank control.
图4 HF1/HR8引物对扩增4株嗜血杆菌的敏感性
Fig.4 Sensitivity results of HF1/ HR8 primers amplified 4Haemophilusstrains

1. 100 bp DNA marker;2-8. 由左至右为引物HF3/HR3扩增副鸡嗜血杆菌、溶血嗜血杆菌、流感嗜血杆菌和副流感嗜血杆菌1~10-6梯度DNA模板的敏感性;9.空白对照。
1. 100 bp DNA marker; 2-8. Sensitivity results of HF3/ HR3 primers amplified a series 10-fold dilutions of Haemophilus genomic DNA from 1 to 10-6in four picture from left to right, in order, areH.paragallinarum,H.haemolyticus,H.influenzaandH.parainfluenzae; 9. Blank control.
图5 HF3/HR3引物对扩增4株嗜血杆菌的敏感性
Fig.5 Sensitivity results of HF3/ HR3 primers amplified 4Haemophilusstrains
2.3 敏感性 本研究中所提取的4株嗜血杆菌(副鸡嗜血杆菌、溶血嗜血杆菌、流感嗜血杆菌和副流感嗜血杆菌)的初始浓度不同,分别为56 ng/μL,210 ng/μL,73 ng/μL,67 ng/μL。由此所得HF1/HR8引物对扩增4株嗜血杆菌的检测限为0.56 ng/μL,2.1 pg/μL,0.73 ng/μL,67 pg/μL;HF3/HR3引物对扩增4株嗜血杆菌的检测限为0.56 ng/μL,0.21 ng/μL,0.73 ng/μL,0.67 ng/μL。
2.4 样品检测 利用所建立的CODEHOP简并引物PCR检测方法,对60份野生树鼩呼吸道样品DNA进行检测。结果显示HF1/HR8 单独扩增阳性的有46份,HF3/HR3单独扩增为阳性的有23份。HF1/HR8和HF3/HR3两组引物对扩增片段均为阳性的有20份,即在受试的60份树鼩呼吸道样品中,嗜血杆菌的阳性率为33.3%。
3 讨 论
本研究利用CODEHOP简并引物PCR方法,实现了对嗜血杆菌OMP基因的有效扩增。虽然所设计的简并引物中,没有单独的引物能实现对嗜血杆菌的检测。但将HF1/HR8和HF3/HR3两组引物相结合,能够区分巴斯德菌科中的交叉菌株,如多杀巴斯德杆菌、产气巴斯德杆菌、小鼠放线杆菌等,可有效的识别嗜血杆菌的存在。本方法的最低检测限根据嗜血杆菌菌株的不同而有所区别,HF1/HR8引物的检测限为2.1 pg/μL~0.73 ng/μL,HF3/HR3引物的检测限可达0.5 ng/μL。
所建立的CODEHOP PCR方法对60份野生树鼩呼吸道样品的检测结果阳性率为33.3%(20/60),此前对同一片样品,采用培养法检测出4株溶血嗜血杆菌,阳性率为6.7%(4/60)[11]。用SPSS19.0进行配对卡方检验,两种方法的结果差异有统计学意义(χ2=8.571,P=0.003<0.01)。目的片段进一步进行测序验证,发现能够获得Blast比对结果的均为流感嗜血杆菌,其它产物均未能获得比对结果,与标准菌株目的片段的比对结果相同。表明GenBank数据库中还未收录这几株嗜血杆菌的OMP编码序列。在受试的标准菌株中,除流感嗜血杆菌外,其它3株菌的OMP目的片段同样未获得序列比对结果,我们已将其基因序列提交至GenBank。本方法具有很好的特异性和稳定性,敏感性高,能够快速筛查样品中的嗜血杆菌。对树鼩这类新型实验动物的微生物质量控制方面具有良好的应用前景,在保障人类疾病动物模型的研究中具有重要的意义。
[1]Kilian M. Haemophilus[M]. In: Murray PR, Baron EJ, Jorgensen JH, et al. Manual of clinical microbiology 8th ed,(vol1). Washington DC: ASM, 2003, 623-634.
[2]Yang ZS, Fang H. Human and animal pathogenic bacteriology[M]. Shijiazhuang: Hebei Science and Technology Press, 2003:705-716.(in Chinese) 杨正时,房海. 人及动物病原细菌学[M]. 石家庄:河北科技出版社,2003:705-716.
[3]Bruant G,Gousset N,Quentin R, et al. Fimbrial ghf gene cluster of genital strains ofHaemophilusspp[J]. Infect Immun,2002, 70(10): 5438-5445. DOI: 10.1128/IAI.70.10.5438-5445.2002.
[4]Tian GZ, Zhang LJ, Wang XL, et al. Rapid detection ofHaemophilusinfluenzaeandHaemophilusparainfluenzaein nasopharyngeal swabs by multiplex PCR[J]. Biomed Environ Sci, 2012, 25(3): 367-371. DOI: 10.3967/0895 3988.2012.03.016
[5]Nedbalcova K, Satran P, Jaglic Z, et al.Haemophilusparasuisand Glasser’s disease in pigs: a review[J]. Veterinarni Medicina, 2006, 51(5): 168-179.
[6]Blackall P. Infectious coryza: overview of the disease and new diagnostic options[J]. Clin Microbiol Rev, 1999, 12(4): 627-632. DOI: 10.1016/S0006-8993(01)02897-9
[7]Rose TM, Schultz ER, Henikoff JG, et al. Consensus-degenerate hybrid oligonucleotide primers for amplification of distantly related sequences[J]. Nucleic Acids Res, 1998, 26(7): 1628-1635.
[8]Rose TM, Henikoff JG, Henikoff S. CODEHOP (COnsensus-DEgenerate Hybrid Oligonucleotide Primer) PCR primer design[J]. Nucleic Acids Res, 2003, 31(13): 3763-3766. DOI: 10.1093/nar/gkg524
[9]Huang XY, Xu J, Sun XM, et al. Development of application of tree shrew in human disease animal models research[J]. Lab Anim Sci, 2013, 30(2): 59-64. (in Chinese) 黄晓燕,徐娟,孙晓梅,等. 树鼩在人类疾病动物模型中应用研究进展[J]. 实验动物科学,2013,30(2):59-64.
[10]Staheli JP, Boyce R, Kovarik D, et al. CODEHOP PCR and CODEHOP PCR primer design[J]. Methods Mol Biol, 2011, 687: 57-73. DOI:10.1007/978-1-60761-944-4-5
[11]Xing J, Feng YF, Fu R, et al. The survey of culturable bacteria and fungi in wild tree shrews[J]. Lab Anim Sci, 2012, 29(3): 34-38. (in Chinese) 邢进,冯育芳,付瑞,等.野生树鼩可培养细菌和真菌携带情况的调查[J]. 实验动物科学,2012,29(3):34-38.
Establishment and application of CODEHOP PCR assay for detection ofHaemophilusspp. in tree shrews
XING Jin1,FENG Yu-fang1,YUE Bing-fei1,HE Zheng-ming1,SUN Xiao-mei2,DAI Jie-jie2
(1.NationalInstituteofFoodandDrugControl,InstituteofLaboratoryAnimalResources,Beijing100050,China;2.InstituteofMedicalBiology,ChineseAcademyofMedicineSciencesandPekingUnionMedicalCollege,Kunming650118,China)
We established a rapid detection method ofHaemophilusspp. in tree shrews, and provided a reference for tree shrews microbiological quality control. According to the outer membrane protein (OMP) multiple alignments of 6 differentHaemophilusspp. published in NCBI, a few pairs of degenerate primers were designed by CODEHOP designer online. Then, the specificity and sensitivity of designed CODEHOPs were evaluated by 4 standardHaemophilusstrains and 24 other genus strains. AndHaemophilusspp. in the tree shrews was detected by established CODEHOP PCR method. Results showed that degenerate primers HF-1/HR-8 and HF-3/HR3 could produce target fragments of 506 - 535 bp and 344 - 390 bp by amplifyingH.paragallinarum(ATCC29545),H.haemolyticus(ATCC33390),H.influenzae(ATCC33391),H.parainfluenzae(ATCC33392), respectively. Amplified nucleotides were sequenced and Blast in NCBI, which proved that results were accurate. It can effectively distinguishHaemophilusspp. and other genus together with two pairs of primers. The lowest detection limit of this method was 2.1 pg/uL. The 60 respiratory specimens of wild tree shrews were tested forHaemophilusspp. that positive rate was 20/60. In conclusion, the CODEHOP PCR performs well for detectionHaemophilusspp. in animal clinical specimens. It is easy to operate, with good specificity and sensitivity, and has practical application value.
Haemophilusspp.; CODEHOP PCR; tree shrew; degenerate primer; detection
国家科技支撑计划项目(No.2014BAI01B01)资助
贺争鸣,Email:zhengminghe57@163.com
1.中国食品药品检定研究院实验动物资源研究所,北京 100050; 2.中国医学科学院/北京协和医学院医学生物学研究所,昆明 650118
10.3969/j.issn.1002-2694.2015.11.002
R378
A
1002-2694(2015)11-0994-07
2015-05-22;
2015-07-16

