高羊茅在低氮胁迫下的蛋白组学分析
李小冬,舒健虹,于二汝,吴佳海,蔡一鸣,王小利*
(1.贵州省草业研究所,贵州 贵阳 550006;2.贵州省油料研究所,贵州 贵阳 550006)
高羊茅在低氮胁迫下的蛋白组学分析
李小冬1,舒健虹1,于二汝2,吴佳海1,蔡一鸣1,王小利1*
(1.贵州省草业研究所,贵州 贵阳 550006;2.贵州省油料研究所,贵州 贵阳 550006)
摘要:为了研究高羊茅在低氮胁迫条件下的蛋白水平变化,我们采用iTRAP技术分析了氮胁迫30 d的高羊茅叶片中蛋白组学的变化。一共检测到595个差异蛋白(295个上调,300个下调),分别参与了多个不同的代谢途径。在高严谨筛选标准下,氮代谢、氧化还原反应以及胁迫相关代谢等多个途径的基因被明显上调表达。通过生理生化测定发现,叶绿素、可溶性蛋白以及游离氨基酸的含量显著下降,而过氧化物酶(POD)、超氧化物歧化酶(SOD)以及谷胱甘肽合成酶(GS)等活性氧清除酶活性显著升高。采用荧光定量PCR的方法验证蛋白组学数据发现所有挑选的基因都具有相同的变化趋势。分析显著富集的14-3-3基因家族的表达,发现其都能被低氮胁迫诱导,因此胁迫相关的基因可能是调节高羊茅抵抗多种不同逆境的关键基因。本文首次在高羊茅中采用蛋白组学的方法分析低氮胁迫条件下基因的表达变化,获得重要候选基因,并对其应用进行了讨论。
关键词:高羊茅;低氮胁迫;蛋白组学;表达分析
非生物胁迫限制植物的生长发育与作物产量,植物主要通过两种方式来抵御这些伤害:一种是形成抵抗能力强生理活性弱的成熟种子等特殊形态以躲避不利环境;二是通过激活一些可逆的调节机制(如抗逆基因的表达)增强植物本身的抵抗力[1-2]。耐低氮胁迫新品种的培育是减少农作物生产投入成本的一个主要方面。研究植物在低氮胁迫条件下的分子响应机制将大大加快抗逆育种的进程。
通过转录组、蛋白组以及代谢组学等高通量生物信息学分析方法已经成功鉴定出许多参与逆境反应的功能基因,它们参与了碳、氮以及油脂代谢等多个过程,在模式植物以及农作物中,已经成功发现了逆境胁迫调节过程中许多关键酶的功能[3]。蛋白质是植物功能的体现者与执行者,其调节植物的抗逆性不仅体现在改变酶的催化活性,而且部分可以作为转录因子调节其他基因的表达。作为大分子物质,蛋白质能够调节胞内物质的组成与浓度,进而影响植物的渗透压等。因此,蛋白组的数据往往比转录组或者芯片分析的结果更可靠[4-5]。
前人对氮代谢的研究主要集中在对植物根系的影响。根系发育和根吸收氮元素的能力是影响植物对氮元素吸收能力的两个主要因素。氮元素对植物发育也有重要影响:随着氮素水平的增加,根系生物量和根系活力呈增加的趋势,根系总量增加主要表现在侧根数目明显增多[6];而在低氮条件下,植物也倾向于根系生长,但是在增加相同生物量的情况下,根茎比重值增大,而且不利于侧根生长[7-8],对大部分收获籽实和营养体的农作物不利。植物氮元素的代谢主要存在两种机制:一种叫HATS (high affinity transport system),是在低氮条件下植物启动组成型和诱导型表达的功能基因促进氮元素吸收;另一种是LATS (low affinity transport system),是在高氮条件下减缓植物吸收氮元素的机制[9-10],因此,这些不同机制导致了农作物不同品种对氮的吸收和利用具有显著的差异,相关的研究在玉米(Zeamays)、油菜(Brassicacampestris)和小麦(Triticumaestivum)等农作物中都有报道[10-13]。
氮元素吸收的分子模式在拟南芥(Arabidopsisthaliana)中已经有很好的研究成果,氮不仅是一种重要的矿物质元素,而且还作为一种重要的外部信号调节氮代谢基因的表达[14-17]。氮元素作为生物体主要元素之一,其代谢过程往往与其他信号途径互作。磷酸烯醇丙酮酸羧化酶(PEPC)是有机酸代谢途径的关键酶,有报道称PEPC在12 mmol/L硝酸盐处理2 h后表达显著下调,另外淀粉合成的关键基因腺苷二磷酸葡萄糖焦磷酸化酶(AGPS2)的表达同样受硝酸盐的抑制[18]。
在牧草类作物中,关于蛋白组学的研究在苜蓿(Medicagosativa)已经有开展,但是在禾本科牧草中的报道还很少。本研究通过蛋白组学的方法分析高羊茅(Festucaarundinacea)在低氮胁迫条件下表达谱的变化,共检测到595个差异蛋白,这些蛋白参与氮代谢、细胞氧化还原和逆境相关等多个不同的代谢途径。生理生化测定发现,叶绿素、可溶性蛋白以及游离氨基酸的含量显著下降,而过氧化物酶(POD)、超氧化物歧化酶(SOD)以及谷胱甘肽合成酶(GS)等酶活性在胁迫条件下显著升高,并采用荧光定量PCR的方法对随机挑选的候选基因进行了表达水平的分析。本研究为在禾本科牧草中研究氮代谢以及通过转基因技术培育抗性种质资源提供候选基因与技术储备。
1材料与方法
1.1材料
本研究采用2005年贵州省草业研究所育成的国家牧草新品种黔草1号高羊茅(登记号:299)为试验材料。本实验Hoagland水培液所用的无机盐均购自上海国药公司,RNA提取试剂盒购自Axygen公司,RNA反转录试剂盒购自Thermo Scientific公司,荧光定量PCR试剂盒购自Promega公司。
1.2方法
1.2.1试验材料的准备和非生物胁迫处理从新鲜收获的黔草1号高羊茅中选取300粒饱满的种子,用50℃温水浸泡过夜,加50 mL 75%酒精搅动、浸泡30 s进行表面消毒,用100 mL无菌水冲洗3次。在培养皿底铺垫2~3张灭菌滤纸,加入4~5 mL无菌蒸馏水,然后将消毒好的种子均匀点播于滤纸上,放入光照培养箱中发芽7 d,每天补加3~4 mL无菌水使滤纸保持湿润,萌发后将幼苗放置在装有Hoagland营养液的水培装置中培养30 d进行低氮胁迫处理:将幼苗分别转移到无氮水培液(低氮胁迫,营养液中NO3-被Cl-取代)和正常水培液中(对照,标准Hoagland营养液)进行生长处理,低氮胁迫15 d,每3 d更换1次营养液,同时为了缩小温室内微环境的影响,栽植盆进行定期旋转。剪取胁迫与对照的高羊茅叶片,用液氮速冻,保存在-80℃的冰箱中待用。植株培养条件参考文献[19],具体为光照16 h,黑暗8 h,生长温度22℃。
1.2.2蛋白酶解与iTRAQ标记蛋白酶解采用FASP程序,参照文献[20],打断后的多肽采用4-plex/8-plex iTRAQ (applied biosystems)试剂进行标记,实验程序参照试剂盒说明书。操作过程如下:将200 μg蛋白样品溶解到30 μL STD缓冲液中(4% SDS,100 mmol/L DTT,150 mmol/L Tris-HCl,pH 8.0)。采用UA缓冲液(8 mol/L Urea尿素,150 mmol/L Tris-HCl,pH 8.0)进行超滤透析去除DTT和小分子量蛋白(30 kD)。然后加入100 μL 0.05 mol/L 碘乙酰胺到UA缓冲液中,黑暗条件孵育20 min用来封闭多肽残基的C端。用100 μL UA缓冲液润洗3次,然后用100 μL DS缓冲液[50 mmol/L三乙基碳酸氢铵(triethylammonium bicarbonate), pH 8.5]润洗2次。用40 μL DS缓冲液溶解,加入2 μg胰蛋白酶(promega,美国)37℃暗光条件下酶解过夜后收集起来备用。采用紫外分光的方法在280 nm利用消光系数计算蛋白浓度。将iTRAQ试剂融入70 μL 乙醇,加入待标记的样品组织,标记完成后冷冻真空抽干备用。
1.2.3蛋白分离与色谱分析将iTRAQ标记好的蛋白利用通用电气医疗集团生命科学部(GE Healthcare,美国)的AKTA蛋白自动纯化系统进行纯化。将标记好的多肽冲洗溶解于2 mL A缓冲液中(25% ACN中加入10 mmol/L KH2PO4,pH 2.7),加入到 4.6×100 mm多聚磺乙基的分离柱中(5 μm,200 Å)(PolyLC,美国)。洗脱采用0%~10% B缓冲液(25% ACN中加入500 mmol/L KCl和10 mmol/L KH2PO4,pH 2.7)以1 mL/min 的流速洗脱2 min,10%~20% B 缓冲液洗脱25 min,20%~45% B 缓冲液洗脱5 min,50%~100% B 缓冲液洗脱5 min。每min的样品都单独收集,通过在214 nm条件下监测吸光值,将相似的样品混合最终形成10个样品池进行脱盐处理,每个样品通过浓缩再溶解到40 μL 0.1%(v/v)三氟乙酸中保存在-80℃备用。
1.2.4液相质谱分析将制备好的样品上样到nanoLC-MS/MS液相质谱联用分析仪中分析。具体过程如下:取10 μL(约5 μg)样品溶液注射到 C18反相色谱柱(15 cm长,内径75 μm),内部填充5 μm RP-C18 树脂混合液A(0.1%甲酸),洗脱时采用线性浓度的B缓冲液 (80% 氰化甲烷和0.1% 甲酸溶液),以流速250 mL/min分离超过140 min。质谱数据是根据扫描300~1800 m/z 范围丰度最高的高能诱导解离(HCD)片段。
1.2.5数据库比对分析质谱数据采用MASCOT和Proteome Discoverer 1.3软件进行分析,差异蛋白的序列根据UniProtKB数据库获得,通过NCBI BLAST与SwissProt数据库比对与功能注释,阈值小于10-3的序列进行Blast2GO与 GO分析,阈值设为10-6。BLAST无结果的未注释基因通过InterProScan3搜索EBI 数据库获取已知蛋白的模体,通过InterProScan GO进行注释。代谢途径分析通过比对KEGG数据库。
1.2.6生理生化指标的测定用0.8 cm打孔器取低氮胁迫处理组和对照组的高羊茅叶片各10片,称重后,加入5 mL 80% 丙酮溶液进行萃取,分别测定663,646以及470 nm下的吸光值,叶绿素a(Chl a)与叶绿素b(Chl b)的计算方法分别为:Chl a=12.21 OD663-2.81 OD646; Chl b=20.13 OD646-5.03 OD663,总叶绿素(T Chl)为Chl a+Chl b。取100 mg处理组和对照组新鲜叶片,加液氮磨碎,加入1 mL提取缓冲液(甲醇∶水=1∶1)超声处理30 min,13200 r/min,4℃离心10 min,上清液用来进行可溶性蛋白和游离氨基酸含量的测定,测定分析是由北京艾米诺医药研究公司分析完成,详细操作程序参考文献[21]。氧化还原酶活性采用试剂盒测定,称取100 mg新鲜处理组和对照组的叶片,加1 mL预冷的PBS提取缓冲液,4℃,用研钵磨碎,超氧化物歧化酶(SOD)和谷氨酰胺合成酶(GS)分别采用试剂盒S0102和S0055(碧云天,北京)测定,过氧化物酶(POD)活性采用试剂盒A084-3 (南京建成)测定,测定过程均按照试剂盒说明书进行。
1.2.7高羊茅总RNA提取与反转录PCR荧光定量分析RNA提取与反转录PCR参考文献[22],RNA提取采用TaKaRa RNAiso Reagent试剂盒,具体操作过程参照试剂盒说明书。RNA的反转录应用RevertAid First Strand cDNA Synthesis Kit完成,用1 μL DNase处理RNA,每样品取5 μg RNA反转录成cDNA,具体程序参照试剂盒说明书。应用荧光定量PCR技术验证筛选的候选基因的表达变化。具体方法如下,将合成的cDNA稀释20倍作为荧光定量PCR的模板。PCR的程序为:95℃ 5 min,95℃ 10 s,58℃ 10 s,72℃ 30 s,45个循环,72℃ 10 min,从65℃缓慢升温到95℃,每0.5℃收集一次荧光信号用于制作溶解曲线,荧光定量引物利用在线软件http://www.idtdna.com/Scitools/Applications/Primerquest设计,引物序列参照表1。试验方法和程序参照文献[20],采用2-ΔΔct算法,每个样品3个生物学重复,每个生物学重复3个技术重复。

表1 本实验中使用的引物
1.3数据分析
采用Excel与PowerPoint完成实验数据作图及统计分析。
2结果与分析
2.1高羊茅蛋白组数据总体描述
我们采用了iTRAQ技术分析了高羊茅叶片在低氮胁迫条件下蛋白组的变化,在差异基因阈值设为>1.2倍或者<0.83倍,并且P<0.05情况下,我们共检测到595个蛋白表达有显著变化,其中有295个蛋白上调表达,300个基因下调表达,对所有氮胁迫诱导差异表达基因进行GO功能聚类(Gene Ontology database,http://geneontology.org)。在细胞组分聚类分析中,蛋白主要定位于细胞质(77)、细胞器(61)、质膜(25)(图1A);在生物过程聚类中参与最多的依次为代谢过程(58)、细胞过程(40)、单有机体过程(18)、逆境反应(13)等过程(图1B);在分子功能聚类分析中发现具有结合功能(51)和催化功能(45)的基因占据大部分(图1C)。
2.2差异基因代谢途径分析
高羊茅在低氮胁迫条件下,有几个代谢途径的基因呈现明显的变化。其中氮代谢途径的基因,在高严谨筛选条件下(表达差异倍数>1.5,或者<0.67),共有10个蛋白(CL10359.Contig4_All、CL5776.Contig1_All、Unigene12843_All、CL5527.Contig1_All、CL8689.Contig1_All、CL8708.Contig2_All、Unigene9083_All、Unigene23702_All、CL11136.Contig1_All和CL340.Contig11_All)被上调1.52~2.24倍,另外5个蛋白(Unigene6516_All、Unigene23474_All、Unigene21261_All、Unigene42040_All和CL7688.Contig1_All)被下调了0.48~0.63倍(表2)。这些基因参与氮元素的吸收、固定、同化、转运等多个过程。
在低氮胁迫条件下,植株不仅会出现生长缓慢、叶片变黄、分蘖数减少等显著的表型变化,同时在生理生化上也有显著差异。在研究中发现,严格筛选条件下,共有5个蛋白(Unigene24111_All、Unigene39854_All、CL784.Contig2_All、CL6318.Contig1_All和Unigene16350_All)的表达上调,有一个蛋白CL224.Contig5_All下调表达,这6个基因都参与了细胞内氧化还原调节(表3)。
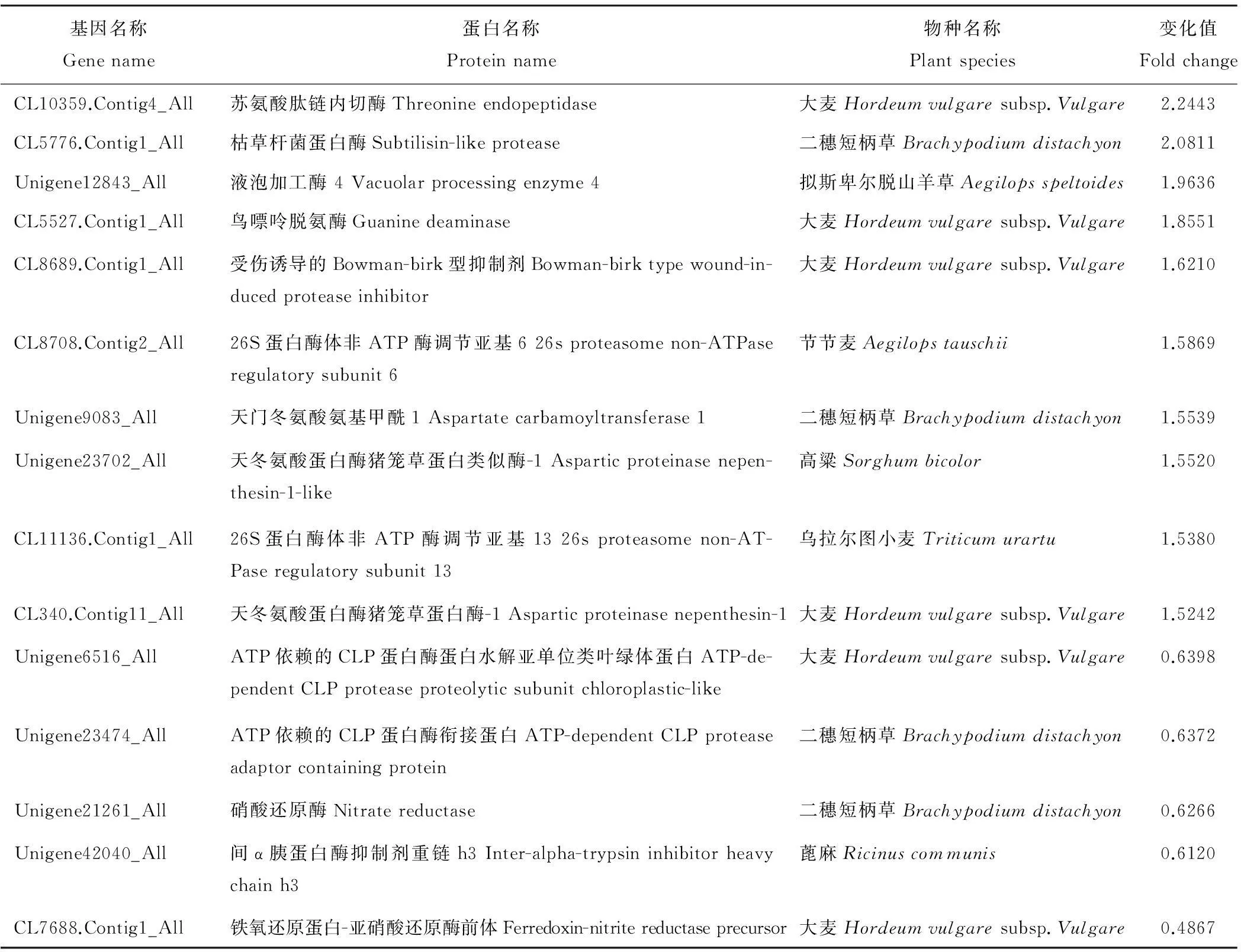
表2 氮代谢途径的基因表达变化
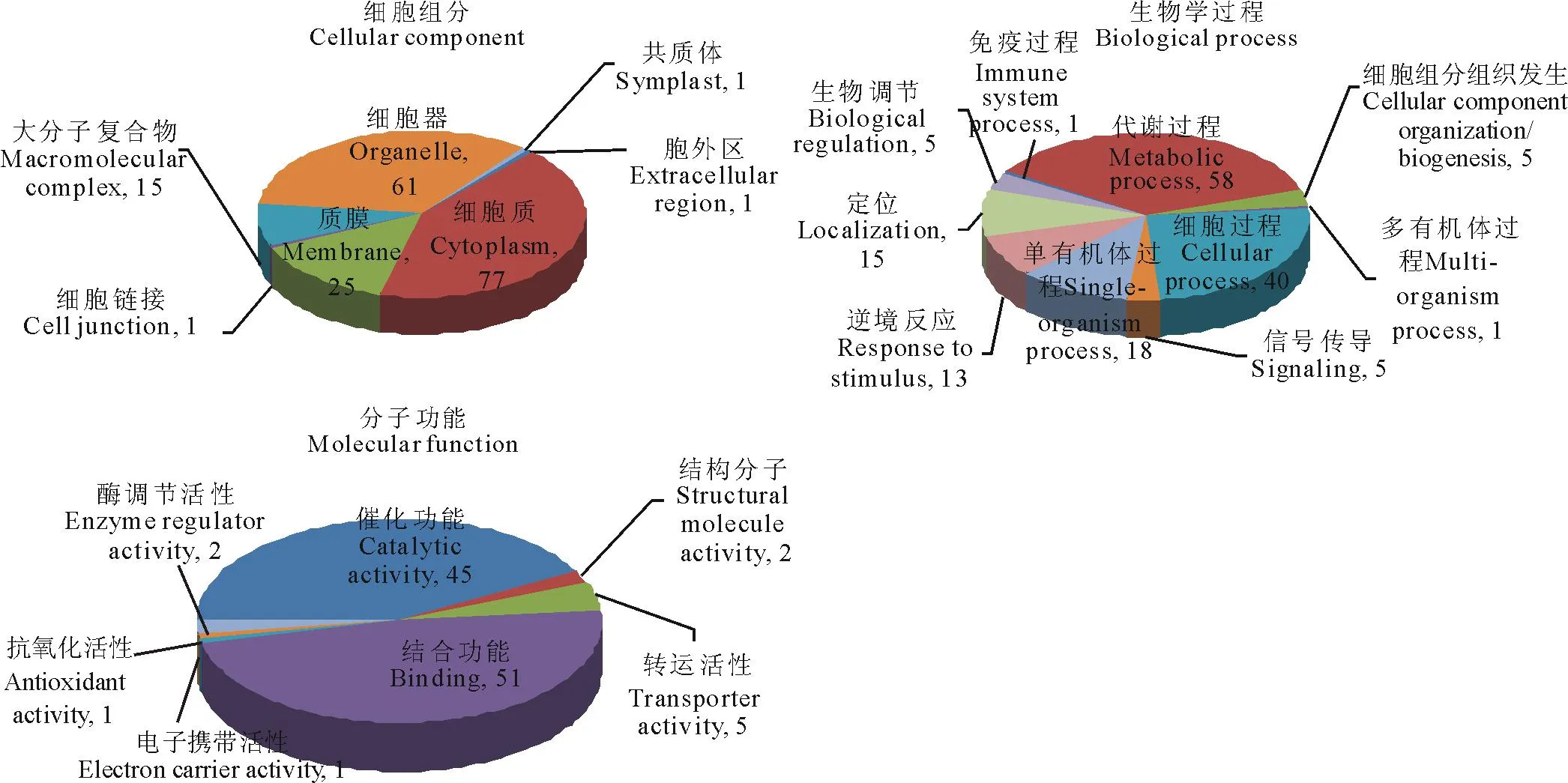
图1 高羊茅底单处理条件下差异蛋白的生物信息预测
图中数字表示每个类别中包含的蛋白数目。The figures stand for the number of proteins contained in each categories.

表3 氧化还原反应途径的基因表达变化
除了氮代谢以及氧化还原反应途径外,胁迫相关的基因也有明显的变化,在高严谨筛选条件下,共有15个蛋白显著被诱导,只有1个蛋白的表达被抑制(表4)。在这些上调表达的基因中,高羊茅14-3-3家族蛋白如CL210.Contig4_All、CL210.Contig7_All、CL210.Contig2_All、Unigene6925_All在低氮胁迫条件下,其表达呈现整体被诱导的趋势。具有一致变化趋势的除14-3-3蛋白外,还有3个谷胱甘肽转移酶(CL468.Contig2_All、CL468.Contig1_All 和CL4631.Contig2_All)以及2个胁迫相关蛋白(CL11641.Contig1_All和CL3913.Contig3_All)。这些关键基因的上调表达说明其在高羊茅氮代谢过程中起到重要的调节作用。
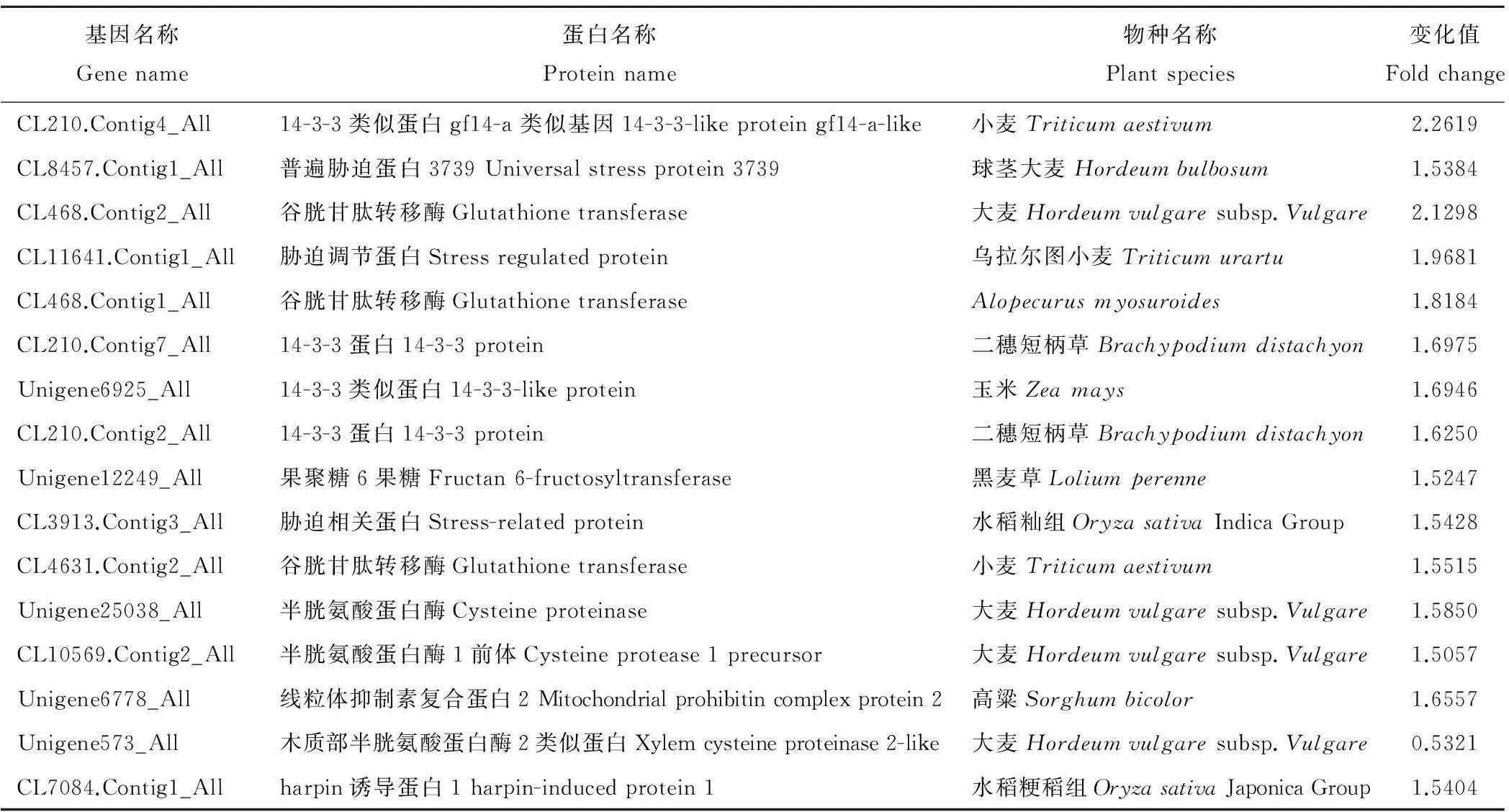
表4 胁迫相关途径的基因表达变化
2.3高羊茅在胁迫条件下生理生化的改变
为了验证蛋白组学的结果,我们测定了低氮胁迫条件下,高羊茅叶片中叶绿素、可溶性蛋白、氨基酸等的含量,以及POD、SOD、GS等酶的活性。氮胁迫显著降低了叶片中叶绿素的含量,在对照材料鲜叶中,叶绿素a、叶绿素b以及总叶绿素含量依次为(1.45±0.12),(0.67±0.11)和(2.12±0.22) mg/g(图2A)。低氮胁迫条件下其含量分别降低到(0.80±0.16),(0.48±0.08)和(1.28±0.22) mg/g,暗示低氮胁迫能够显著降低光合作用的效率(图2A)。
与叶绿素含量变化类似,在低氮胁迫下,可溶性蛋白的含量也显著降低了47%(图2B)。在检测的19种游离氨基酸中,只有色氨酸的含量增加4倍,而其余氨基酸含量下降了20%~80%(图2C)。另外,低氮胁迫打破了细胞内的氧化还原平衡,POD、T-SOD 和GS的活性被分别上调了1.43,2.07和1.78倍(图2D)。这些生理生化水平的变化与蛋白组学中检测到调节氮代谢以及氧化还原反应蛋白水平的变化可能存在密切的联系。
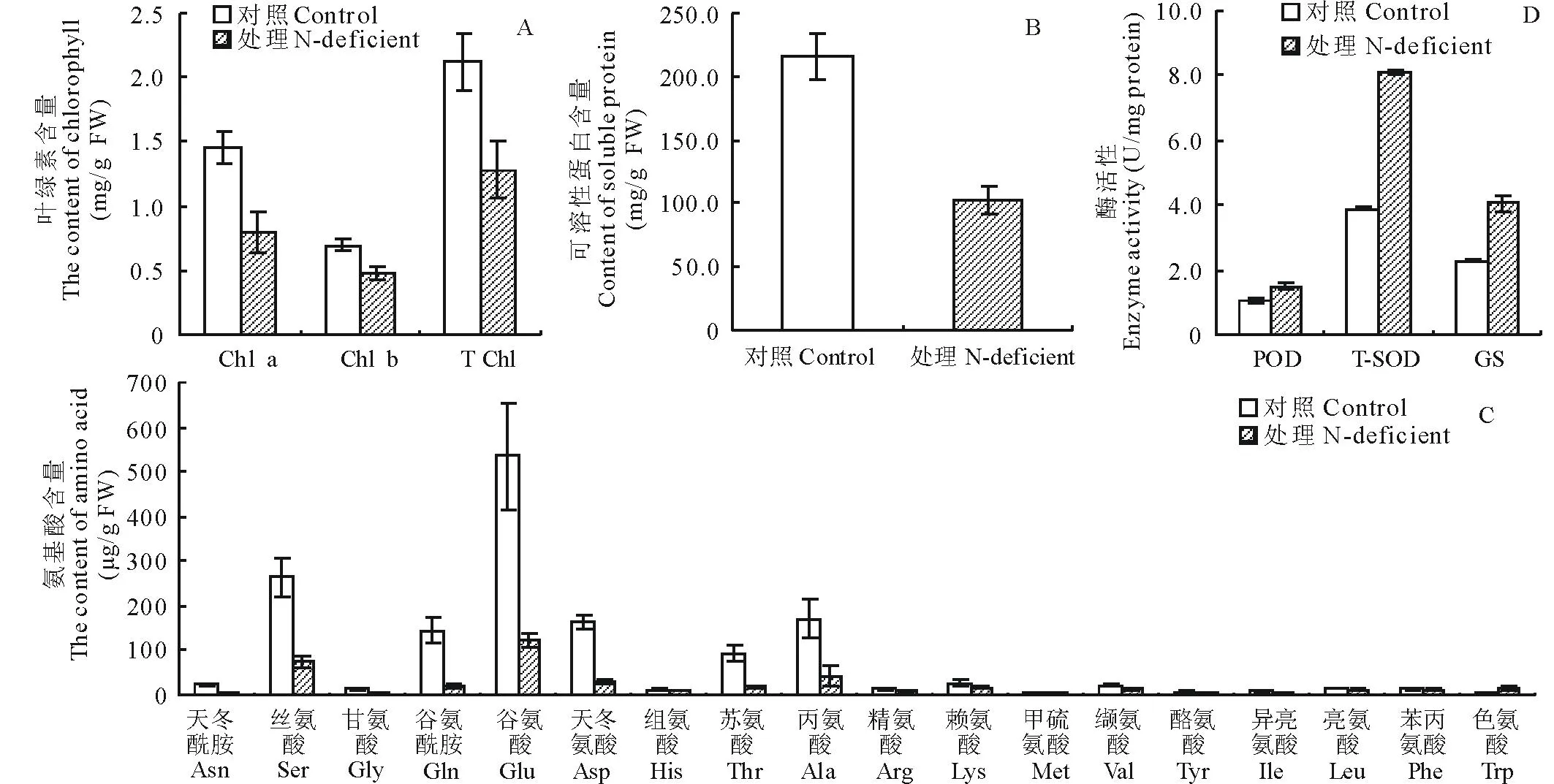
图2 氮胁迫条件下高羊茅生理生化变化Fig.2 Physiological and biochemical changes in tall fescue under low nitrogen condition Chl a:叶绿素a Chlorophyll a;Chl b:叶绿素b Chlorophyll b;T Chl:总叶绿素Total chlorophyll;POD:过氧化物酶Peroxidase;T-SOD:总超氧化物歧化酶Total superoxide dismutase;GS:谷胱甘肽合成酶Glutathione synthetase.
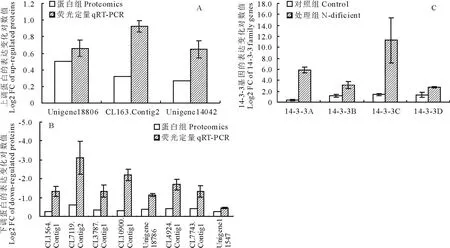
图3 荧光定量PCR验证蛋白组数据
2.4荧光定量验证蛋白组学数据及14-3-3基因表达分析
为了验证蛋白数据的可靠性,我们随机挑选了12个基因,采用荧光定量PCR方法分析其在低氮胁迫条件下的表达变化。包括3个上调表达的基因(Unigene18806、CL163.Contig2和Unigene14042)和8个下调的基因(CL1564.Contig1、CL7119.Contig2、CL3787.Contig1、CL10900.Contig1、Unigene18786、CL4924.Contig1、CL7743.Contig1和Unigene11547)。所有挑选的基因的表达变化趋势与蛋白组相同。然而,蛋白差异筛选标准为大于1.2或者小于0.8的前提下,有4个基因(Unigene18806、CL163.Contig2、Unigene14042和Unigene11547)的荧光定量变化倍数小于2。说明这些基因在低氮胁迫条件下的表达只受到轻微的激活或抑制,或者最终的蛋白含量受到了转录后翻译的影响。
上述挑选的上调表达基因定量PCR验证结果,虽然具有和蛋白组数据相同的变化趋势,但是其诱导的幅度并不大(小于2),为了检验上调表达数据的可靠性,我们对14-3-3基因家族在低氮胁迫条件下的表达变化也进行了验证。4个被检测的14-3-3基因CL210.Contig4_All、CL210.Contig7_All、CL210.Contig2_All和Unigene6925_All分别命名为14-3-3A、14-3-3B、14-3-3C和14-3-3D,在低氮胁迫后4个基因都显著被诱导2.7~11.3倍,说明蛋白组的数据是可信的。
3讨论与结论
氮元素是限制植物生长发育的关键因素之一,和20世纪70年代相比,植物的产量已经增长了一倍,其中氮肥是主要贡献因素之一。然而氮肥的大量使用直接导致了植物氮利用效率的下降[23]。通过改良氮利用效率是减少氮肥使用的主要方法之一,植物对氮元素的吸收利用受到其自身的精细调控,通过调节基因的表达、关键酶活性以及代谢物的含量,可以改变植物应对不同氮营养水平的能力[24-25]。牧草类作物相对于其他农作物受贫瘠土壤胁迫的危害更加严重,然而相应的理论研究还比较滞后。本文采用iTRAQ的方法鉴定高羊茅叶片在低氮胁迫条件下蛋白水平的变化,获得了氮高效利用的候选基因,该研究在禾本科牧草类植物中处于领先地位,为在其他牧草类作物中开展类似研究提供了很好的借鉴作用。
在高羊茅低氮处理的蛋白组数据中,氮代谢途径蛋白发生了明显变化(表1),相应的生理生化测定结果也表明低氮胁迫显著降低了叶绿素含量、可溶性蛋白和游离氨基酸的水平(图2A~C),说明在低氮胁迫条件下,光合作用速率、蛋白质的翻译与合成等都受到显著的影响。类似的研究在水稻(Oryzasativa)中也有报道,在低氮胁迫条件下,水稻气体交换、光合效率、光合色素的含量、可溶性蛋白等都不同程度受到抑制[26]。另外在小麦低氮诱导的蛋白谱中同样也发现了光合作用、氮代谢相关蛋白表达的变化[27]。
光合中心Ⅱ(PSⅡ)的活性在低氮条件下被减弱,进而会导致植物细胞体内的活性氧化物质的积累,出于对不利环境的应激反应,植物细胞会激活活性氧清除系统以保护自身不受伤害[28]。我们的研究发现低氮处理下,高羊茅体内氧化还原代谢途径的关键蛋白都明显上调(表3),通过测定相应酶的活性发现POD、SOD与GS活性都显著增强(图2D),说明低氮胁迫间接地导致了植物细胞受到活性氧伤害在植物界中具有一定的普遍性。与其他植物一样,在高羊茅中,低氮胁迫能够激活活性氧清除系统。
低氮条件下,胁迫相关蛋白明显诱导,他们分别具有不同的功能,定位于不同的亚细胞器,参与不同的代谢途径。因此,低氮胁迫可能与其他胁迫存在广泛的互作,而这些基因则可能具有调节植物多种抗性的功能。其中最显著的是14-3-3蛋白,在前人研究中,14-3-3能够调控植物对非生物胁迫(干旱、高温、高盐等)和生物胁迫(病虫害)的应答[29]。在本研究中,我们一共发现了4个高羊茅14-3-3蛋白受低氮胁迫条件诱导(表4),并且其表达水平也得到荧光定量PCR的验证(图3C),说明14-3-3蛋白在高羊茅应对低氮胁迫的反应中可能起到至关重要的作用,为我们创制氮高效高羊茅新品种提供了重要的基因资源。
References:
[1]Levitt J. Responses of Plants to Environmental Stresses. Volume I: Chilling, Freezing, and High Temperature Stresses[M]. Orlando, Florida: Academic Press, 1980.
[2]Larcher W. Physiological Plant Ecology: Ecophysiology and Stress Physiology of Functional Groups[M]. New York: Springer Science & Business Media, 2003.
[3]Hirai M Y, Yano M, Goodenowe D B,etal. Integration of transcriptomics and metabolomics for understanding of global responses to nutritional stresses inArabidopsisthaliana. Proceedings of the National Academy of Sciences of the United States of America, 2004, 101(27): 10205-10210.
[4]Gygi S P, Rochon Y, Franza B R,etal. Correlation between and mRNA in east. Molecular and Cellular Biology, 1999, 19(3): 1720-1730.
[5]Bogeat-Triboulot M-B, Brosché M, Renaut J,etal. Gradual soil water depletion results in reversible changes of gene expression, protein profiles, ecophysiology, and growth performance inPopuluseuphratica, a poplar growing in arid regions. Plant Physiology, 2007, 143(2): 876-892.
[6]Liu X J, Ye F, Zhang X L. Effects of exogenous nitrogen forms on root characteristics of alfalfa at different growth stages. Acta Prataculturae Sinica, 2015, 24(6): 53-63.
[7]Goss M, Miller M, Bailey L,etal. Root growth and distribution in relation to nutrient availability and uptake. European Journal of Agronomy, 1993, 2(2): 57-67.
[8]Drew M, Saker L, Ashley T. Nutrient supply and the growth of the seminal root system in barley I. The effect of nitrate concentration on the growth of axes and laterals. Journal of Experimental Botany, 1973, 24(6): 1189-1202.
[9]Rao K P, Rains D W. Nitrate absorption by barley II. Influence of nitrate reductase activity. Plant Physiology, 1976, 57(1): 59-62.
[10]Siddiqi M Y, Glass A D, Ruth T J,etal. Studies of the regulation of nitrate influx by barley seedlings using13NO3-1. Plant Physiology, 1989, 90(3): 806-813.
[11]Daniel-Vedele F, Filleur S, Caboche M. Nitrate transport: a key step in nitrate assimilation. Current Opinion in Plant Biology, 1998, 1(3): 235-239.
[12]Orsel M, Filleur S, Fraisier V,etal. Nitrate transport in plants: which gene and which control. Journal of Experimental Botany, 2002, 53: 825-833.
[13]Oscarson P. The strategy of the wheat plant in acclimating growth and grain production to nitrogen availability. Journal of Experimental Botany, 2000, 51: 1921-1929.
[14]Walch-Liu P, Filleur S, Gan Y,etal. Signaling mechanisms integrating root and shoot responses to changes in the nitrogen supply. Photosynthesis Research, 2005, 83(2): 239-250.
[15]Stitt M, Krapp A. The interaction between elevated carbon dioxide and nitrogen nutrition: the physiological and molecular background. Plant, Cell & Environment, 1999, 22(6): 583-621.
[16]Lejay L, Tillard P, Lepetit M,etal. Molecular and functional regulation of two NO3- uptake systems by N- and C-status ofArabidopsisplants. The Plant Journal, 1999, 18(5): 509-519.
[17]Wang R, Tischner R, Gutiérrez R A,etal. Genomic analysis of the nitrate response using a nitrate reductase-null mutant ofArabidopsis. Plant Physiology, 2004, 136(1): 2512-2522.
[18]Scheible W R, Gonzalez-Fontes A, Lauerer M,etal. Nitrate acts as a signal to induce organic acid metabolism and repress starch metabolism in tobacco. The Plant Cell, 1997, 9(5): 783-798.
[19]Yu E, Fan C, Yang Q,etal. Identification of heat responsive genes inBrassicanapussiliques at the seed-filling stage through transcriptional profiling. PloS One, 2014, 9(7): e101914.
[20]WiŜniewski J R, Zougman A, Nagaraj N,etal. Universal sample preparation method for proteome analysis. Nature Methods, 2009, 6(5): 359-362.
[21]Feng Z, Zhou X, Wu F,etal. Both dietary supplementation with monosodium L-glutamate and fat modify circulating and tissue amino acid pools in growing pigs, but with little interactive effect. PloS One, 2014, 9(1): e84533.
[22]Li X, Yu E, Fan C,etal. Developmental, cytological and transcriptional analysis of autotetraploidArabidopsis. Planta, 2012, 236(2): 579-596.
[23]Ju X T, Xing G X, Chen X P,etal. Reducing environmental risk by improving N management in intensive Chinese agricultural systems. Proceedings of the National Academy of Sciences, 2009, 106(9): 3041-3046.
[24]Sakakibara H. Nitrate-specific and cytokinin-mediated nitrogen signaling pathways in plants. Journal of Plant Research, 2003, 116(3): 253-257.
[25]Sakakibara H, Takei K, Hirose N. Interactions between nitrogen and cytokinin in the regulation of metabolism and development. Trends in Plant Science, 2006, 11(9): 440-448.
[26]Huang Z A, Jiang D A, Yang Y,etal. Effects of nitrogen deficiency on gas exchange, chlorophyll fluorescence, and antioxidant enzymes in leaves of rice plants. Photosynthetica, 2004, 42(3): 357-364.
[27]Chandna R, Ahmad A. Nitrogen stress-induced alterations in the leaf proteome of two wheat varieties grown at different nitrogen levels. Physiology and Molecular Biology of Plants, 2015, 21(1): 19-33.
[28]Li B L, Mei H S. Relationship between oat leaf senescence and activated oxygen metabolism. Acta Phytophysiologica Sinica, 1989, 15(1): 6-12.
[29]Keller C K, Radwan O. The functional role of 14-3-3 proteins in plant-stress interactions. i-ACES, 2015, 1(2): 100-110.
参考文献:
[6]刘晓静, 叶芳, 张晓玲. 外源氮素形态对紫花苜蓿不同生育期根系特性的影响. 草业学报, 2015, 24(6): 53-63.
[28]李柏林, 梅慧生. 燕麦叶片衰老与活性氧代谢的关系. 植物生理学报, 1989, 15(1): 6-12.
Proteomic analysis of nitrogen stress-responsive proteins in the leaves of tall fescue
LI Xiao-Dong1, SHU Jian-Hong1, YU Er-Ru2, WU Jia-Hai1, CAI Yi-Ming1, WANG Xiao-Li1*
1.GuizhouInstituteofPrataculture,Guiyang550006,China; 2.GuizhouInstituteofOilCrops,Guiyang550006,China
Abstract:In order to thoroughly investigate the protein level changes of tall fescue in low-nitrogen conditions, we analyzed the proteome of the leaves from 30 day old plants exposed to low-nitrogen stress using the iTRAP technique. In total, 595 proteins were differentially accumulated (295 up-regulated and 300 down-regulated), which participated in diverse metabolic pathways. According to a strict selection standard, we discovered that the genes related to redox reactions and stresses significantly increased. Physiological and biochemical analysis revealed that the contents of chlorophyll, soluble proteins and free amino acid dramatically decreased, while reactive oxygen-scavenging enzymes such as POD, SOD and GS were highly active. The expression pattern of affected genes, detected by real-time fluorescence quantitative RT-PCR, coincided with the proteomic data. Notably, most of the 14-3-3 family of proteins were enriched by nitrogen shortage, suggesting that these involve the key genes that take part in diverse stresses in tall fescue. In this study, we provide proteomic information for tall fescue subjected to low-nitrogen availability. We also highlight some of the key genes involved and discuss their potential uses.
Key words:tall fescue; nitrogen stress; proteomic; gene expression
*通信作者
Corresponding author. E-mail: wangxiaolizhenyuan@126.com
作者简介:李小冬(1984-),男,湖南邵阳人,副研究员,博士。 E-mail: lixiaodongzl@163.com
基金项目:国家自然基金项目“高羊茅光周期调控基因FaCONSTANS特异性表达与生物学功能分析”(31360576)资助。
收稿日期:2015-09-29;改回日期:2015-11-16
DOI:10.11686/cyxb2015470
http://cyxb.lzu.edu.cn
李小冬,舒健虹,于二汝,吴佳海,蔡一鸣,王小利. 高羊茅在低氮胁迫下的蛋白组学分析. 草业学报, 2016, 25(3): 67-76.
LI Xiao-Dong, SHU Jian-Hong, YU Er-Ru, WU Jia-Hai, CAI Yi-Ming, WANG Xiao-Li. Proteomic analysis of nitrogen stress-responsive proteins in the leaves of tall fescue. Acta Prataculturae Sinica, 2016, 25(3): 67-76.

