藜麦EST-SSR的开发及通用性分析
张体付 戚维聪 顾闽峰 张晓林 李 坦 赵 涵,*
1江苏省农业科学院/江苏省农业生物学重点实验室,江苏南京 210014;2盐城市新洋农业试验站,江苏盐城 224336
藜麦EST-SSR的开发及通用性分析
张体付1,**戚维聪1,**顾闽峰2张晓林1李坦1赵涵1,*
1江苏省农业科学院/江苏省农业生物学重点实验室,江苏南京 210014;2盐城市新洋农业试验站,江苏盐城 224336
摘要:藜麦因营养均衡受到越来越多的关注,但尚未深入开展其基础研究。开发微卫星序列重复SSR分子标记将为藜麦的遗传分析提供重要资源。本研究利用NCBI数据库中藜麦RNA测序RNA-Seq及表达序列标签EST数据挖掘、验证及评价藜麦EST-SSR,共发现1862个藜麦非单核苷酸EST-SSR。其中,二核苷酸重复最多(38.3%),六核苷酸重复最少(11.7%)。不同重复类型SSR的数量随着核苷酸数目的增加呈下降趋势。在随机选取验证的119个EST-SSR标记中,66 (55.9%)个能够扩增出清晰条带,39个在4份藜麦资源中具有多态性,且其多态性与重复序列长度不具有显著相关性。t测验显示,多态性EST-SSR在藜麦与其他藜科种质间不存在显著差异,说明其具有良好的通用性,可用于藜科物种的遗传关系分析。
关键词:藜麦;EST-SSR;分子标记;遗传关系;通用性
本研究由江苏省农业科技自主创新资金项目[CX(14)2044]和美国国际发展署Securing Water for Food Award-salt Tolerant Quinoa子项目(UR4443 B)资助。
This study was supported by Jiangsu Agriculture Science and Technology Innovation Fund [CX(14)2044]and the subproject of “Securing Water for Food Award-salt Tolerant Quinoa” from USAID (UR4443 B).
**同等贡献(Contributed equally to this work)
第一作者联系方式:张体付,E-mail:zhangtifu_82@jaas.ac.cn,Tel:025-84391957
藜麦(Chenopodium quinoa Willd.)原产于南美安第斯地区,属一年生草本植物,具有耐干旱、耐盐碱特性[1-2]。与主要作物玉米、水稻、小麦相比,藜麦种子含有更高的碳水化合物(64%)和蛋白质(平均为16%),特别是含有人体所需的所有必需氨基酸,是唯一一种被联合国粮农组织认可的单体即可满足人体基本营养需求的植物[3]。因此,开发和利用藜麦种质资源对于发展节水农业、提高盐碱、干旱土地利用率等方面具有重要意义。特别是当前中国种质资源没有新突破,藜麦作为有潜力的种质资源受到越来越多的关注。中国作物学会藜麦分会的成立及“首届中国藜麦产业(长春)高峰论坛”的召开标志着藜麦在我国已经开始了系统性的研究和产业推动工作。
为了进一步推广、研究藜麦资源,联合国将2013年设为国际藜麦年(http://www.fao.org/quinoa-2013/zh/),美国国际发展署也于2014年正式启动了藜麦耐盐项目(http://www.securingwaterforfood.org/wur/)的研究工作。然而与主要作物相比,人们还未对藜麦深入研究。由于长期缺少生物技术研发的投入,藜麦的遗传基础理论薄弱,育种水平没有明显改善。与玉米和小麦相比,其单产仍然很低,平均只有0.8 t hm-2。除提高栽培、管理水平外,利用生物技术快速培育高产品种也是提高藜麦产量的重要途径。因此,大规模开发藜麦分子标记,是进一步挖掘藜麦重要农艺性状的遗传位点及利用分子标记进行辅助选择的重要基础,对利用生物技术解决育种瓶颈具有重要科学意义和应用价值。但目前关于藜麦分子标记开发的研究报道相对较少,可用于藜麦遗传育种研究的工具和相关资源非常有限。
1993年Fairbanks等[4]首先报道了基于藜麦基因组的RAPD标记。这种分子标记可用于藜麦种间杂交种的鉴定以及藜麦和其它藜科物种的遗传变异分析。随后,通过对富含微卫星基元的克隆测序,Mason等[5]于2005年开发并验证了208个在藜麦种间具有较高多态性的共显性SSR分子标记。同年,Coles等[6]利用藜麦未成熟种子和花的EST文库发展了51个SNP标记,分析发现平均每462 bp就会出现一个SNP。之后Fuentes等[7]的研究表明,在藜麦中相比于二核苷酸重复的SSR分子标记,长度大于20 bp的三核苷酸基元SSR具有更高的多态性。2008 年Jarvis等[8]借助富含GA、CAA和AAT重复的文库以及BES,开发了216个新型多态性SSR以及6 个BES-SSR,并构建了藜麦首个基于SSR标记的遗传连锁图谱。该遗传连锁图谱包含200个SSR,由38个连锁群组成,覆盖了藜麦913 cM的遗传距离。2012年Maughan等[9]利用113个藜麦材料进一步将功能性SNP的分子标记数目扩展为511个,并利用新开发的SNP标记将藜麦的遗传连锁图谱缩小为29个连锁群,总的遗传距离扩大为1404 cM。
藜麦基因组研究发现其大小介于主要作物水稻基因组(430 Mb)和玉米基因组(2500 Mb)之间,约为967 Mb[10-12]。随着测序技术的快速发展,水稻、玉米等二倍体作物的全基因组测序相继完成。得益于此,水稻、玉米等作物在分子遗传领域的研究取得深入进展,且相关研究成果已广泛用于育种实践并取得巨大效益。然而,藜麦因其为四倍体物种(2n = 4x = 36)[13-14],基因组序列组装困难,至今尚无基因组测序相关报道。因此,很难大规模开发分子标记。近年来,公共数据库公布的物种表达谱序列信息越来越多,为大规模开发EST-SSR分子标记提供了丰富资源[15-17]。截止到目前,尚未见利用藜麦表达谱序列挖掘EST-SSR和开发分子标记的报道。为了弥补藜麦EST-SSR分子标记开发的空白、丰富藜麦分子标记资源,本研究利用公共数据库NCBI中所有藜麦表达序列数据,通过生物信息学手段大规模挖掘藜麦EST-SSR,并对藜麦EST-SSR核苷酸重复特点、多态性以及应用作出评价。
1 材料与方法
1.1试验材料与表达序列数据的获取
试验材料均为藜科物种,包括4份藜麦,2份苍白茎藜,1份台湾藜和1份杖藜共8份种质(表1)。藜麦表达序列数据来自NCBI的SRA数据库(http://www.ncbi.nlm.nih.gov/sra),包括11.9 G Illumina HiSeq 2000的 RNA-Seq数据(登录号为SRX257003 和SRX256971)以及2.1 G Roche 454的EST数据(登录号:SRX084791)。
1.2数据预处理与EST-SSR鉴定
利用Fastq-Dump程序将SRA格式数据转换成Fastq格式文件,并采用并行清理通道将转换的文件清理,控制数据质量,包括Q20 (1%的碱基错误率)清理及L40 (长度≥40 bp)过滤,并去除5¢、3¢端的polyT、polyA序列以及载体序列。随后,利用Trinity软件按照默认参数对质量控制后的序列组装拼接,去除冗余的序列获得unigene[18]。通过MISA软件(http://pgrc.ipk-gatersleben.de/misa/)对unigene序列进行SSR位点识别,其识别条件为单核苷酸重复不低于10次,二核苷酸重复不低于8次,三核苷酸重复不低于7次,四核苷酸重复不低于5次,五核苷酸及六核苷酸重复不低于4次,复合SSR的识别条件是2个SSR之间的距离不超过50 bp。

表1 8份供试材料信息Table 1 Information of eight Chenopodiaceous species tested in the experiment
1.3SSR引物的设计
利用Primer 3.0软件(http://primer3.sourceforge.net/)对SSR位点的侧翼序列进行引物设计,设置参数Tm为58℃±3℃,引物长度为20±3 bp,产物预期长度为100~450 bp,其他参数为默认。
1.4DNA提取、PCR扩增及电泳检测
利用Karroten DNA提取试剂盒提取8份试验材料的幼苗DNA。经过1%琼脂糖凝胶检测的DNA用于PCR扩增。PCR体系为25 μL,含2 mmol L-1MgCl2、100 μmol L-1dNTPs、0.2 μmol L-1引物、1 U Taq酶及50 μg DNA。PCR程序为94℃ 3 min;94℃30 s,58℃ 35 s,72℃ 50 s,38个循环;72℃ 3 min。PCR扩增产物经10%聚丙烯酰胺凝胶100 V电压电泳120 min,EB染色后在紫外透射仪上观察结果。
1.5多态性分析
EST-SSR的多态性信息含量PIC (polymorphism information content)的计算公式为式中Pi为第i个等位基因的频率,k为等位基因的数量[19]。根据Ott[20]的分析,PIC≥0.10为多态性SSR,PIC≥0.70为高多态性SSR。
1.6聚类分析
利用本研究发展的EST-SSR及42个通过基因组DNA文库发展的SSR标记对供试材料进行基因分型[8]。基于UPGMA法计算遗传相似性[19],采用NTSYS-pc 2.1软件聚类[21]。
2 结果与分析
2.1藜麦EST-SSR的信息分析
根据获取的14.0 G藜麦RNA-Seq数据以及EST数据,利用Trinity软件拼接得到19 571条unigene,总碱基数为80 448 006 bp,平均每条unigene长约4 kb。其中,16 854条序列含有SSR位点。通过MISA软件对unigene序列进行SSR位点搜索,发现藜麦EST-SSR重复类型丰富,从单核苷酸重复到六核苷酸重复均有出现。其中,单核苷酸重复SSR丰度最高,达到18 476个。与单核苷酸重复相比,非单核苷酸重复SSR数量明显降低,只有1862个。尽管对原始序列预处理时去除了5¢、3¢端的polyT、polyA序列,但A/T类型占单核苷酸SSR的比例高达97.9%说明假阳性A/T的存在。因此,本文不对单核苷酸SSR进行深入分析。非单核苷酸重复中,二核苷酸重复SSR最多,占非单核苷酸重复SSR总数的38.3% (713个),其次为三核苷酸重复,占非单核苷酸重复SSR总数的22.7% (423个),最少的为六核苷酸重复,占非单核苷酸重复SSR总数的11.7% (217 个)。结果显示,不同重复类型的SSR数量随着基元核苷酸数目的增加呈下降趋势。
不同重复类型的藜麦EST-SSR均有多种基元,二、三、四、五、六核苷酸重复分别有3、10、18、47、103种,共计181种基元。各重复类型SSR中优势基元如图1所示。二、四、五核苷酸重复的优势基元分别为AG/CT、AAAT/ATTT及AAAAT/ATTTT,分别占各自重复类型的49.8%、41.9%及28.9%。三核苷酸重复的优势基元有AAT/ATT、AAC/GTT和AAG/CTT三种,分别占全部三核苷酸重复的28.6%、20.3%和17.0%,共65.9%。六核苷酸重复的优势基元有AAAGAG/CTCTTT和ACTATC/AGTGAT两种,分别占全部六核苷酸重复SSR的5.5%和5.1%,共10.6%。
各重复类型的藜麦EST-SSR在不同重复数下的数量分布差异明显(表2)。二核苷酸重复呈主要分布的重复数有四种,分别为8、9、10和11。三、四、五、六核苷酸重复呈主要分布的重复数各自只有一种,分别为7、5、4和4。此外,重复数最多的基元为两碱基重复AC/GT,重复次数为171次,这也是长度最长的重复(342 bp)。
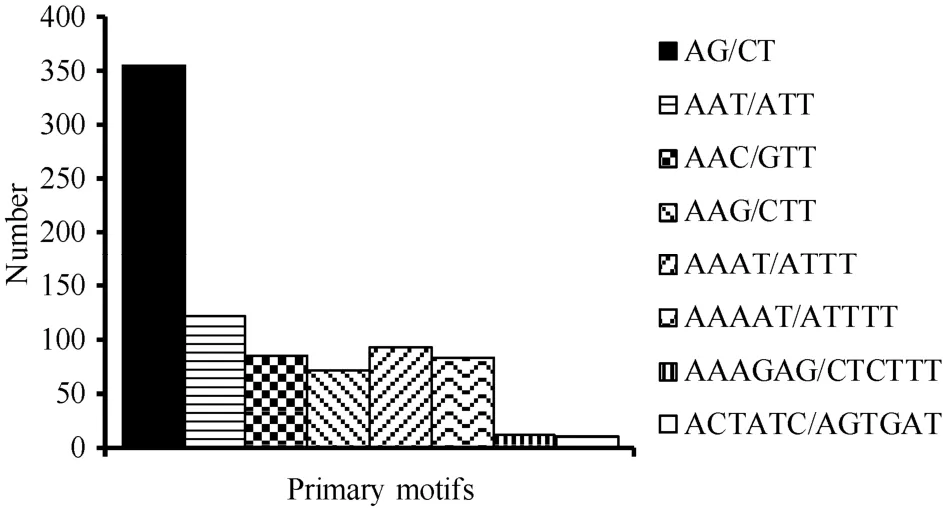
图1 不同重复类型EST-SSR的优势基元Fig.1 Primary motifs of EST-SSR with different repeat types
2.2藜麦EST-SSR的验证及多态性分析
利用Primer 3.0对侧翼序列大于200 bp且重复长度大于16 bp的非单核苷酸重复SSR位点设计引物,随机选取119个EST-SSR位点的引物对4份藜麦材料(表1) DNA扩增的结果显示,共有66对EST-SSR引物(表3)能够获得清晰的扩增条带,成功率为55.9%;六核苷酸重复SSR的成功率最高,为74.3%;五核苷酸重复SSR的成功率最低,为31.8%(图2)。多个引物出现了三等位基因、四等位基因带型(图3)。这66对EST-SSR引物共扩增出112种条带,平均每对引物扩增1.7种条带。其中39对扩增出两种以上类型的条带,2对扩增出4种类型的条带(表3)。三核苷酸重复及六核苷酸重复具有最高的平均扩增条带数,为2.3种;其次为二核苷酸重复,为2.0种;最低的是五核苷酸重复,为1.7种。通过PIC计算,39个为多态性EST-SSR (PIC≥0.10),占总数的59.1%,其中2个具有高多态性(PIC≥0.70)。相关性分析表明,EST-SSR多态性与基元的碱基数(r = -0.241,P = 0.051)或重复序列长度(r = -0.138,P = 0.270)的相关性均不显著,但与条带数呈极显著正相关(r = 0.536,P = 0.000)。同时本研究也利用前人报道的42对藜麦基因组SSR引物[8]对相同材料进行了扩增,共得到了66种条带,平均每对引物扩增1.6种条带;其中21个基因组SSR的PIC≥0.10。t测验表明EST-SSR与基因组SSR的PIC差异不显著(P = 0.316)。

表2 不同重复类型EST-SSR的分布规律Table 2 Distribution of EST-SSR with different repeat types
2.3藜麦EST-SSR的通用性分析
为验证藜麦EST-SSR分子标记的通用性,本研究将验证的66个EST-SSR及42个基因组SSR共计108个分子标记用于其他4份藜科种质(表1)的基因型分析。其中,49个EST-SSR及26个基因组SSR在该藜科种质中表现出多态性(PIC≥0.10)。统计分析发现,EST-SSR在其他4份藜科种质的PIC与藜麦中的PIC无显著差异(P = 0.121),但基因组SSR在藜麦中的PIC值显著高于其在另外4种藜科种质中的值(P = 0.044)。该结果表明,藜麦的EST-SSR在其他藜科种质中的稳定性和通用性高于基因组SSR。基于EST-SSR引物PCR结果的UPGMA聚类分析表明,来自南美的四份藜科种质被聚成一类,包括3份藜麦和1份苍白茎藜;来自北美的杖藜、欧洲的藜麦以及中国的台湾藜被明显地分开,各自分成三类;来自南美的另一份苍白茎藜并没有与南美的其他4份藜科种质聚为一类,提示该苍白茎藜与来自同一区域的其他4份藜科种质亲缘关系较远(图4)。该结果表明藜麦EST-SSR可用于藜科种质的遗传关系分析。

表3 验证的EST-SSR引物序列及其扩增条带种类Table 3 Sequence of validated EST-SSR primers and their amplified band type
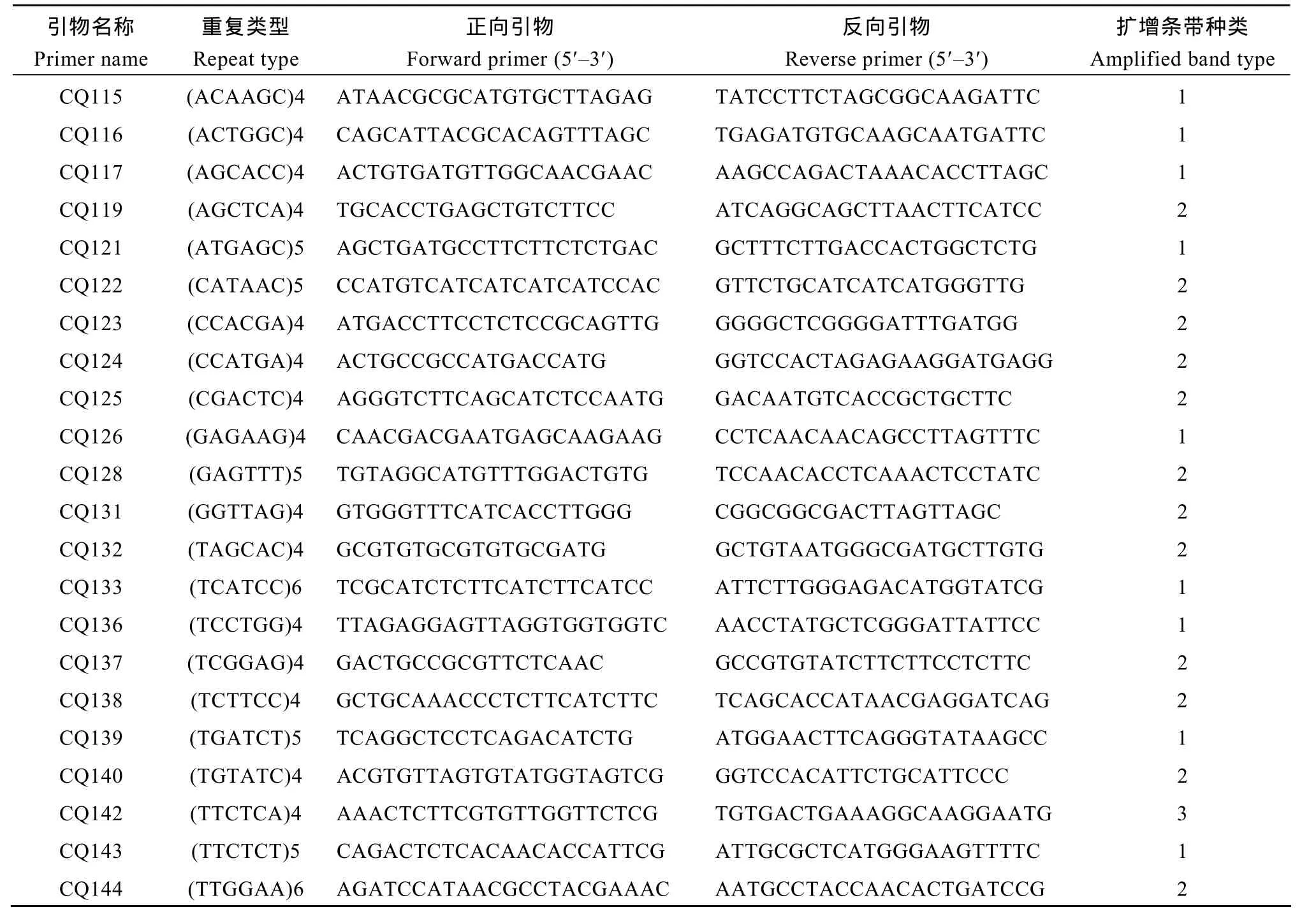
(续表3 )
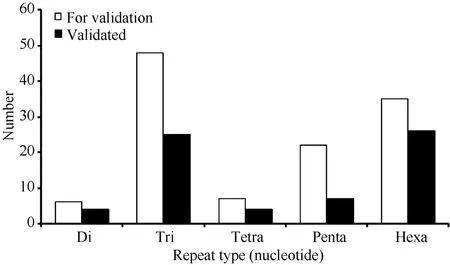
图2 EST-SSR的验证Fig.2 Validation of EST-SSR
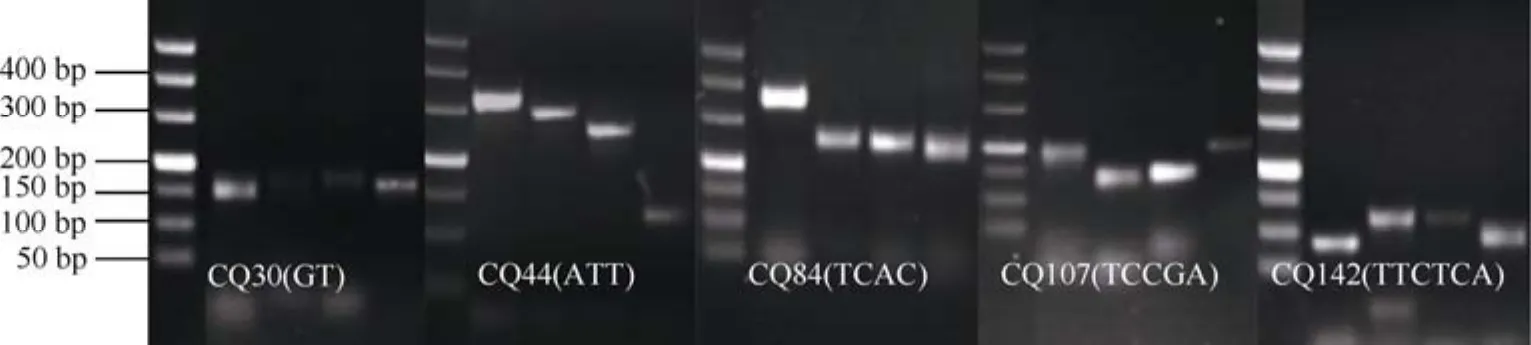
图3 多种EST-SSR扩增带型图Fig.3 Different amplified patterns of EST-SSR
3 讨论
SSR在真核生物中普遍存在,基于SSR开发的分子标记具有多态性高、稳定性好、共显性等优点[22],多用于遗传连锁图谱构建、遗传多样性分析、指纹图谱以及分子标记辅助选择等[23-26]。早期的SSR标记开发主要依赖基因组文库和cDNA文库,成本高且耗时长。近年来,通过生物信息学手段利用公共数据库中丰富的基因组和转录组序列发展分子标记成为开发SSR及其他类型分子标记的重要途径[27-29]。本研究通过NCBI数据库藜麦RNA-Seq及EST数据挖掘藜麦全基因组范围的SSR位点,共发现1862个非单核苷酸SSR位点。尽管EST-SSR重复类型丰富,但不同物种EST-SSR的主要重复类型不同。研究发现植物中多以二、三核苷酸重复为主[30-31]。主要作物玉米、水稻、小麦、大豆EST-SSR中三核苷酸重复类型占的比例最大[32]。与大多数植物一样,本研究发现藜麦EST-SSR中二、三核苷酸重复为主要重复类型。不同重复类型藜麦EST-SSR数量随基元碱基数量增加呈下降趋势,这种EST-SSR重复类型的偏好性可能与分析的EST数据量有关,也可能与其自身长度的稳定性有关。不同物种,同一重复类型的优势基元有所不同。本研究中,藜麦二核苷酸重复类型的优势基元为AG/CT,与玉米、水稻、小麦该重复类型优势基元一致,但与棉花的优势基元不同[32-33]。在双子叶模式植物拟南芥EST-SSR中,二核苷酸优势基元也是AG/CT,但是拟南芥EST-SSR中的三核苷酸优势基元为AAG/TTC,而且其含量高于AG/CT。另外拟南芥EST-SSR还富含ATC/TAG、GGA/CCT、AAC/TTG等三核苷酸基元,这些都是在藜麦EST-SSR中比较稀少的。藜麦EST-SSR中所富含的三核苷酸基元如AAT/TTA、AAC/GTT等在拟南芥中比较少见[30]。包括藜麦在内的多个物种中3个以上核苷酸重复类型的优势基元均不尽相同[28,32]。
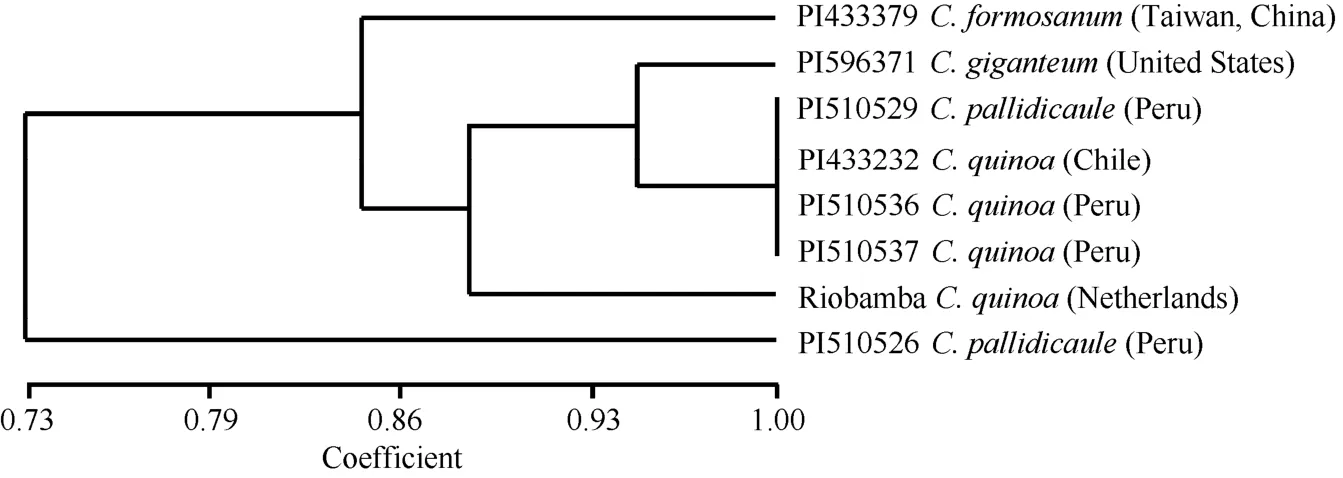
图4 基于相似性系数构建的8份藜科种质的UPGMA树状图Fig.4 UPGMA dendrogram of eight Chenopodiaceous species based on similarity coefficient
利用富含CA、ATT和ATG的基因组文库,Mason等[10]开发了208个藜麦多态性分子标记,但与基因组文库不同,SSR中的主要基元为GA和CAA。Jarvis等[8]利用相同的技术构建了富含GA、AAT和CAA的基因组文库,开发了216个藜麦多态性基因组SSR标记。新开发的基因组SSR的主要基元与基因组文库相一致。本研究开发的EST-SSR中二、三核苷酸重复类型优势基元与前人开发的藜麦基因组SSR及其他植物中报道的优势基元并不相同。最主要的原因可能是EST序列代表基因编码区,编码区只占基因组的很小部分,基因组大部分为基因间区。因此,通过EST开发的SSR与通过基因组手段开发的SSR的优势基元不同。通常情况下,由于EST编码功能基因,序列保守性高,EST-SSR的多态性低于基因组SSR[34-35]。Jarvis等[12]报道利用其开发的基因组SSR在22份藜麦种质中可平均扩增4种带型。在本研究中,新开发的66对EST-SSR引物在4份藜麦种质中最多可以扩增出4种带型,平均扩增1.7种带型。同时,我们以Jarvis等[12]报道的42对基因组SSR引物为对照,平均扩增1.6种带型。t测验显示两种类型SSR的PIC差异不显著。尽管有研究认为重复序列长度大于20 bp的SSR具有高多态性[5,36],但本研究分析EST-SSR重复序列长度与多态性的相关性发现二者并不存在显著的相关。EST-SSR广泛用于物种遗传关系分析[26]。本研究开发的藜麦EST-SSR可以将藜科种质的区域特点明显地划分出来,在不同藜科物种间具有良好的通用性。由于植物中直系同源基因存在的普遍性,且EST-SSR编码功能基因,因此,EST-SSR比基因组SSR具有更高的通用性。
4 结论
藜麦基因组约有1800个非单核苷酸EST-SSR。二、三核苷酸重复为主要重复类型。藜麦EST-SSR重复序列长度与多态性之间不具有显著相关性。藜麦EST-SSR在藜科物种间具有良好的通用性,可以用于藜科种质的遗传关系分析等研究。
References
[1]Razzaghi F,Plauborg F,Jacobsen S,Jensen C R,Andersen M N.Effect of nitrogen and water availability of three soil types on yield,radiation use efficiency and evapotranspiration in field-grown quinoa.Agric Water Manag,2012,109:20-29
[2]Adolf V I,Jacobsen S,Shabala S.Salt tolerance mechanisms in quinoa (Chenopodium quinoa Willd.).Environ Exp Bot,2013,92:43-54
[3]Ogungbenle H N.Nutritional evaluation and functional propertiesof quinoa (Chenopodium quinoa) flour.Intl J Food Sci Nutr,2003,54:153-158
[4]Fairbanks D J,Waldrigues A,Ruas C F,Ruas P M,Maughan P J,Robison L R,Andersen W R,Riede C R,Pauley C S,Caetano L G.Efficient characterization of biological diversity using field DNA extraction and random amplified polymorphic DNA markers.Rev Brazil Genet,1993,16:11-22
[5]Mason S L,Stevens M R,Jellen E N,Bonifacio A,Fairbanks D J,Coleman C E,Mccarty R R,Rasmussen A G,Maughan P J.Development and use of microsatellite markers for germplasm characterization in quinoa (Chenopodium quinoa Willd.).Crop Sci,2005,45:1618-1630
[6]Coles N D,Coleman C E,Christensen S A,Jellen E N,Stevens M R,Bonifacio A,Rojas-Beltran J A,Fairbanks D J,Maughan P J.Development and use of an expressed sequenced tag library in quinoa (Chenopodium quinoa Willd.) for the discovery of single nucleotide polymorphisms.Plant Sci,2005,168:439-447
[7]Fuentes F F,Bazile D,Bhargava A,Martínez E A.Implications of farmers’ seed exchanges for on-farm conservation of quinoa,as revealed by its genetic diversity in Chile.J Agric Sci,2012,150:702-716
[8]Jarvis D E,Maughan P J,Kopp O R,Jellen E N,Mallory M A,Pattee J,Bonifacio A,Coleman C E,Stevens M R,Fairbanks D J.Simple sequence repeat marker development and genetic mapping in quinoa (Chenopodium quinoa Willd.).J Genet,2008,87:39-51
[9]Maughan P J,Smith S M,Rojas-Beltrán J A,Elzinga D,Raney J A,Jellen E N,Bonifacio A,Udall J A,Fairbanks D J.Single nucleotide polymorphisms identification,characterization and linkage mapping in Chenopodium quinoa.Plant Genome,2012,5:1-7
[10]Eckardt N A.Sequencing the rice genome.Plant Cell,2000,12:2011-2017
[11]Martienssen R A,Rabinowicz P D,O’Shaughnessy A,McCombie W R.Sequencing the maize genome.Curr Opin Plant Biol,2004,7:102-107
[12]Stevens M R,Coleman C E,Parkinson S E,Maughan P J,Zhang H B,Balzotti M R,Kooyman D L,Arumuganathan K,Bonifacio A,Fairbanks D J,Jellen E N,Stevens J J.Construction of a quinoa (Chenopodium quinoa Willd.) BAC library and its use in identifying genes encoding seed storage proteins.Theor Appl Genet,2006,112:1593-1600
[13]Palomino G,Hernández L T,de la Cruz Torres E.Nuclear genome size and chromosome analysis in Chenopodium quinoa and C.berlandieri subsp.nuttalliae.Euphytica,2008,164:221-230
[14]Fuentes F,Bhargava A.Morphological analysis of quinoa germplasm grown under lowland desert conditions.J Agron Crop Sci,2011,197:124-134
[15]Xia H,Zheng X,Chen L,Gao H,Yang H,Long P,Rong J,Lu B,Li J,Luo L.Genetic differentiation revealed by selective loci of drought-responding EST-SSRs between upland and lowland rice in China.PloS One,2014,9:e106352
[16]Asadi A A,Monfared S R.Characterization of EST-SSR markers in durum wheat EST library and functional analysis of SSR-containing EST fragments.Mol Genet Genom,2014,289:625-640
[17]Chuang T,Yang M,Lin C,Hsieh P,Hung L.Comparative genomics of grass EST libraries reveals previously uncharacterized splicing events in crop plants.BMC Plant Biol,2015,15:39-53
[18]Grabherr M G,Haas B J,Yassour M,Levin J Z,Thompson D A,Amit I,Adiconis X,Fan L,Raychowdhury R,Zeng Q.Full-length transcriptome assembly from RNA-Seq data without a reference genome.Nat Biotechnol,2011,29:644-652
[19]Nei M.Estimation of average heterozygosity and genetic distance from a small number of individuals.Genetics,1978,89:583-590
[20]Ott J.Strategies for characterizing highly polymorphic markers in human gene mapping.Am J Human Genet,1992,51:283-290
[21]Rohlf F J.NTSYS-pc:Numerical taxonomy system.ver.2.1.Setauket,NY:Exeter Publishing Ltd.2002
[22]Tautz D,Renz M.Simple sequences are ubiquitous repetitive components of eukaryotic genomes.Nucl Acids Res,1984,12:4127-4138
[23]潘海涛,汪俊君,王盈盈,齐照良,李斯深.小麦EST-SSR的开发和遗传作图.中国农业科学,2010,24:452-461 Pan H T,Wang J J,Wang Y Y,Qi Z L,Li S S.Development and mapping of EST-SSR markersin wheat.Sci Agric Sin,2010,24:452-461 (in Chinese with English abstract)
[24]赖勇,王鹏喜,范贵强,司二静,王晋,杨轲,孟亚雄,李葆春,马小乐,尚勋武,王化俊.大麦SSR标记遗传多样性及其与农艺性状关联分析.中国农业科学,2013,27:233-242 Lai Y,Wang P X,Fan G Q,Si E J,Wang J,Yang K,Meng Y X,Li B C,Ma X L,Shang X W,Wang H J.Genetic diversity and association analysis using SSR markers in barley.Sci Agric Sin,2013,27:233-242 (in Chinese with English abstract)
[25]宋海斌,崔喜波,马鸿艳,朱子成,栾非时.基于SSR标记的甜瓜品种(系) DNA指纹图谱库的构建.中国农业科学,2012,26:2676-2689 Song H B,Cui X B,Ma H Y,Zhu Z C,Luan F S.Construction of DNA fingerprint database based on SSR marker for varieties (lines) of Cucumis melo L.Sci Agric Sin,2012,26:2676-2689 (in Chinese with English abstract)
[26]王西成,姜淑苓,上官凌飞,曹玉芬,乔玉山,章镇,房经贵.梨EST-SSR标记的开发及其在梨品种遗传多样性分析中的应用评价.中国农业科学,2010,24:5079-5087 Wang X C,Jiang S L,Shang-Guan L F,Cao Y F,Qiao Y S,Zhang Z,Fang J G.Development of EST-derived SSR markers for pear and evaluation of their application in pear genetic diversity analysis.Sci Agric Sin,2010,24:5079-5087 (in Chinese with English abstract)
[27]张体付,葛敏,韦玉才,赵涵.玉米功能性Insertion/Deletion (InDel)分子标记的挖掘及其在杂交种纯度鉴定中的应用.玉米科学,2012,20(2):64-68 Zhang T F,Ge M,Wei Y C,Zhao H.Discovery for maize function Insertion/Deletion (InDel) polymorphic marker and its implication in purity identification of maize hybrid seeds.J Maize Sci,2012,20(2):64-68 (in Chinese with English abstract)
[28]戚维聪,程计华,黄邦全,李坦,林峰.基于海甘蓝RNA-Seq序列开发EST-SSR分子标记.江苏农业学报,2014,30:997-1002 Qi W C,Cheng J H,Huang B Q,Li T,Lin F.Development and characterization of EST-SSR markers derived from RNA-Seq in Crambe abyssinica.Jiangsu J Agric Sci,2014,30:997-1002 (inChinese with English abstract)
[29]束永俊,李勇,吴娜拉胡,柏锡,才华,纪巍,朱延明.大豆EST-SNP的挖掘、鉴定及其CAPS标记的开发.作物学报,2010,36:574-579 Shu Y J,Li Y,Wu N L H,Bai X,Cai H,Ji W,Zhu Y M.Mining and identification of SNP from EST sequences and conversion of CAPS markers in soybean.Acta Agron Sin,2010,36:574-579 (in Chinese with English abstract)
[30]Cardle L,Ramsay L,Milbourne D,Macaulay M,Marshall D,Waugh R.Computational and experimental characterization of physically clustered simple sequence repeats in plants.Genetics,2000,156:847-854
[31]李淑娴,张新叶,王英亚,尹佟明.桉树EST序列中微卫星含量及相关特征.植物学报,2010,45:363-371 Li S X,Zhang X Y,Wang Y Y,Yin T M.Content and characteristics of microsatellites detected in expressed sequence tag sequences in Eucalyptus.Chin Bull Bot,2010,45:363-371 (in Chinese with English abstract)
[32]Gao L,Tang J,Li H,Jia J.Analysis of microsatellites in major crops assessed by computational and experimental approaches.Mol Breed,2003,12:245-261
[33]程海亮,陆才瑞,邹长松,余道乾,姜鹏飞,杨文翠,张友平,王巧连,宋国立.基于10个棉花腺体相关材料转录组的EST-SSR标记开发.棉花学报,2015,27:1-8 Cheng H L,Lu C R,Zou C S,Yu D Q,Jiang P F,Yang W C,Zhang Y P,Wang Q L,Song G L.Development of EST-SSR markers in cotton based on the transcriptome information of 10 gland related materials.Cotton Sci,2015,27:1-8 (in Chinese with English abstract)
[34]Chabane K,Ablett G A,Cordeiro G M,Valkoun J,Henry R J.EST versus genomic derived microsatellite markers for genotyping wild and cultivated barley.Genet Resourc Crop Evol,2005,52:903-909
[35]Cho Y G,Ishii T,Temnykh S,Chen X,Lipovich L,McCouch S R,Park W D,Ayres N,Cartinhour S.Diversity of microsatellites derived from genomic libraries and GenBank sequences in rice (Oryza sativa L.).Theor Appl Genet,2000,100:713-722
[36]Temnykh S,Declerck G,Lukashova A,Lipovich L,Cartinhour S,McCouch S.Computational and experimental analysis of microsatellites in rice (Oryza sativa L.):frequency,length variation,transposon associations,and genetic marker potential.Genome Res,2001,11:1441-1452
URL:http://www.cnki.net/kcms/detail/11.1809.S.20160125.1622.006.html
Exploration and Transferability Evaluation of EST-SSRs in Quinoa
ZHANG Ti-Fu1,**,QI Wi-Cong1,**,GU Min-Feng2,ZHANG Xiao-Lin1,LI Tan1,and ZHAO Han1,*1Provincial Key Laboratory of Agrobiology,Jiangsu Academy of Agricultural Sciences,Nanjing 210014,China;2Xinyang Agricultural Experiment Station of Yancheng City,Yancheng 224336,China
Abstract:Quinoa draws more and more attentions from people,since quinoa seed as grain has comprehensive nutrients.But the fundamental research on quinoa just starts and remains in a moderated level.Simple sequence repeat (SSR) development in quinoa will enhance the resource for its genetic analysis.Here,the available RNA based sequencing (RNA-Seq) and expressed sequence tag (EST) data of quinoa deposited in the National Center for Biotechnology Information (NCBI) were engaged in EST-SSR development.Totally,1862 non-mononucleotide EST-SSRs were identified.Among the EST-SSRs,the dinucleotide type was the most abundant (38.3%),and the hexanucleotide was the minimal (11.7%).The amount of the EST-SSR showed the declined trend along with the increase in its motif nucleotide length.Among 119 EST-SSR primers randomly chosen for validation,66 (55.9%) primers could give clear amplification bands and 39 showed polymorphisms in four quinoa accessions.Further analysis showed that the polymorphisms of EST-SSRs had no significant correlation with their motif nucleotide length.In addition,t-test demonstrated that the significant difference of EST-SSR polymorphisms was not occurred between quinoa accessions and other Chenopodiaceae germplasm.These results indicating EST-SSRs developed in quinoa could be transferable in Chenopodiaceous genus and applied in genetic relationship analysis.
Keywords:Quinoa;EST-SSR;Molecular marker;Genetic relationship;Transferability
收稿日期Received():2015-05-31;Accepted(接受日期):2016-01-11;Published online(网络出版日期):2016-01-25.
*通讯作者(
Corresponding author):赵涵,E-mail:zhaohan@jaas.ac.cn,Tel:025-84390751
DOI:10.3724/SP.J.1006.2016.00492

