鼠伤寒沙门菌rpoN基因缺失株构建及生物学特性研究
苏洋洋,黄 骏,吴 白,印云聪,刘 珍,陈素娟,彭大新,刘秀梵
(扬州大学兽医学院,江苏省动物重要疫病与人兽共患病防控协同创新中心,扬州 225009)
鼠伤寒沙门菌rpoN基因缺失株构建及生物学特性研究
苏洋洋,黄骏,吴白,印云聪,刘珍,陈素娟,彭大新*,刘秀梵
(扬州大学兽医学院,江苏省动物重要疫病与人兽共患病防控协同创新中心,扬州 225009)
摘要:为了探索σ因子RpoN在鼠伤寒沙门菌生物被膜形成过程中的作用,对两株生物被膜形成能力较强的鼠伤寒沙门菌利用Red同源重组系统构建rpoN基因缺失株,利用原核表达载体构建回复株;比较野生株、缺失株和回复株的生物被膜形成能力和对环境应激抵抗力的差异,并通过建立的csgA和bcsA基因实时定量PCR方法检测编码生物被膜成分基因的表达差异。结果显示,与野生株相比,△rpoN缺失株生物被膜形成能力增强,主要由卷曲菌毛表达量显著增加引起。回复株生物被膜形成能力与野生株相似。△rpoN缺失株在酸性应激和碱性应激条件下对外界环境应激的抵抗力均显著增强。RpoN为鼠伤寒沙门菌中生物被膜形成相关σ因子之一,为进一步研究沙门菌生物被膜形成的调控机制提供理论基础。
关键词:鼠伤寒沙门菌;生物被膜;rpoN基因;突变株;环境应激
沙门菌属是肠杆菌科中重要的病原菌属,人感染后常引起肠炎和急性胃肠炎等。全球每年感染沙门菌病例约有9千多万,其中死亡人数高达15万[1]。鼠伤寒沙门菌为泛嗜性的沙门菌,能引起家禽、哺乳动物和人的感染,是目前世界各国分离率最高的菌型之一,具有重要的公共卫生意义[2]。
σ因子是RNA聚合酶全酶的重要组成成分,专一性地识别特定的启动子并起始转录。肠杆菌科的细菌中存在多种σ因子[3],其中RpoS(由rpoS基因编码)能够在细菌的指数生长期和稳定生长期受到环境应激时调控特定基因的转录与表达。rpoS基因的缺失可影响沙门菌生物被膜形成,降低其对环境应激的抵抗力。除rpoS基因外,其他σ因子也参与了卷曲菌毛和纤维素的合成及生物被膜的形成。由于这种rpoS基因不依赖调控途径在鼠伤寒沙门菌占有一定的比例,有必要鉴定出沙门菌中参与卷曲菌毛和纤维素的合成及生物被膜形成的其他σ因子,并阐明其调控通路。前期研究发现,RpoN、RpoE、FliA、RpoH、RpoS、RpoD等6 个σ因子在沙门菌指数期和生物被膜形成期的基因表达存在差异[4]。本研究运用Red同源重组法构建出两株生物被膜的形成能力较强的鼠伤寒沙门菌的rpoN基因缺失株,通过结晶紫染色法测定其生物被膜的形成能力,利用实时荧光定量PCR(qRT-PCR)方法检测了编码卷曲菌毛合成的csgA基因和编码纤维素合成的bcsA基因的表达差异,最终确定rpoN基因缺失可导致鼠伤寒沙门菌生物被膜形成能力增强。
1材料与方法
1.1材料
1.1.1菌株及质粒鼠伤寒沙门菌S016、S025为本实验室临床病料分离株;大肠杆菌DH5α由本实验室保存;质粒pKD46含有温度敏感型复制子;质粒pKD3含氯霉素抗性基因和FRT位点;质粒pCP20含氨苄青霉素抗性和氯霉素抗性的温度敏感性复制子,为美国马萨诸塞大学医学院Murphy教授惠赠。
1.1.2仪器和试剂酶标仪(BIO-TEK),LightCycler®Nano实时荧光PCR仪(Roche)。结晶紫(Crystal Violet)、刚果红(Congo red)和考马斯亮蓝(Coomassie brilliant blue)购自上海生工生物工程有限公司;胰酪胨大豆肉汤培养基TSB购自Fluka公司;限制性核酸内切酶、Taq酶、dNTP、T4连接酶等购自TaKaRa公司;DNA凝胶回收试剂盒购自Axygen公司,质粒抽提试剂盒QIAprep Spin Miniprep Kit购自QIAGEN公司;反转录试剂盒PrimeScriptTMRT reagent Kit with gDNA Eraser,荧光定量试剂盒SYBR®Premix Ex TaqTM均购自TaKaRa生物工程公司(大连)。
1.2生物被膜的结晶紫染色测定
参照L.A.Pratt等[5]方法,利用96孔聚苯乙烯U型细胞培养板,进行沙门菌生物被膜结晶紫染色定量测定。每次试验重复2个孔,置于酶标仪测定OD550 nm值,重复三次取其平均值。
1.3生物被膜成分的检测
挑取单个菌落接种于液体TSB中,37 ℃、220 r·min-1过夜振荡培养,取10 μL培养液分别接种于含刚果红(40 mg·L-1)和考马斯亮蓝(20 mg·L-1)平板或含荧光增强剂(200 mg·L-1)的无盐LB平板,置于28 ℃静置培养,4 d后比较各菌落的形态,后者于紫外灯下观察各菌落的荧光强度[6]。
1.4rpoN因子基因缺失株的构建
缺失株构建方法按照K.C.Murphy等[7]的方法进行,λ Red同源重组所用引物由两部分组成,分别是5′端50 bp的序列与目的基因两侧序列同源,和3′端20 bp的序列与pKD3质粒中氯霉素抗性基因(cat)两端序列同源,打靶引物rpoN-D1/rpoN-D2序列见表1。所有引物由南京金斯瑞生物科技有限公司合成。以鼠伤寒沙门菌S016和S025为母本进行同源重组操作,煮沸裂解法提取缺失株的DNA,以引物rpoN-F/rpoN-R进行PCR扩增鉴定,野生株、cat基因插入株和无抗性缺失株PCR扩增预期的片段大小分别为1 434、1 192 和263 bp。鉴定正确的缺失株分别命名为S016ΔrpoN和S025ΔrpoN。
表1PCR扩增引物序列
Table 1Primers used for amplification of PCR

引物Primers序列(5'→3')PCRPrimesequences用途PurposerpoN-FrpoN-RATGAAGCAAGGTTTGCAACTCAGGCTCAAACCAGCTGTTTGCGTTGGTTTPrimersusedforamplificationofrpoNgenesrpoN-D1ATGACGCCTCAGCTACAACAGGCCATCCGTCTGTTGCAGTTGTCTACGCTGTGTAGGCTGGAGCTGCTTCrpoN-D2ATGGATAAAGACTCTCGGTACTTCGCAACAGTGCGGCGCGCCACCATGATCATATGAATATCCTCCTTAGPrimersusedforamplificationoffragmentswithcatgenerpoN-HFrpoN-HRCGGGATCCATGAAGCAAGGTTTGCAACTCAGGCCCGCTCGAGTCAAACCAGCTGTTTGCGTTGGTTTPrimersusedforgenecomplementationcsgA-FcsgA-RATGAAACTTTTAAAAGTGGCAGCTTAATACTGGTTAGCCGTGGPrimersusedforamplificationofcsgAgenesbcsA-FbcsA-RCATTTACGCTTCGCTTGGTCCCTTTGTGCGGATTGAPrimersusedforamplificationofbcsAgenescsgA-QFcsgA-QRbcsA-QFbcsA-QRTCGACCAGTGGAACGCTAAAAACCAACCTGACGCACCATTACCGGGCGTGAATCATTTCGTCTCAGGAACCAGCCCATTGTCqRT-PCR
1.5实时荧光定量PCR检测csgA基因和bcsA基因的表达
1.5.1引物设计根据csgA基因和bcsA基因的核苷酸序列,设计PCR扩增引物和qRT-PCR引物,引物序列见表1。
1.5.2标准品制备以细菌DNA为模板,csgA-F/R和bcsA-F/R为引物PCR扩增csgA基因和bcsA基因,克隆至pEASY-T3载体获得重组质粒。用EcoRⅠ酶切后,1.5%琼脂糖凝胶电泳进行鉴定,阳性质粒送测序。1.5.3细菌总RNA的提取与RNA反转录取37 ℃ 220 r ·min-1过夜培养的菌液用1∶10稀释的TSB培养基按1∶100稀释,接种到聚丙乙烯一次性细胞培养瓶,28 ℃静置培养4、8和24 h。使用RNApure超纯总RNA快速提取试剂盒提取样品菌液的RNA,按照PrimeScriptTMRT reagent Kit with gDNA Eraser 说明去除DNA进而反转录。以反转录cDNA和标准品质粒DNA作为模板,csgA-QF/QR和bcsA-QF/QR为引物进行荧光定量PCR扩增。1.5.4实时荧光定量RT-PCR荧光定量PCR反应体系:SYBR®Premix Ex TaqTM(2*)10.0 μL,PCR Forward Primer (10 μmol·L-1)、PCR Reverse Prime (10 μmol·L-1)各0.4 μL,ROX Reference Dye (50*)0.4 μL,DNA模板2.0 μL,dH2O(灭菌蒸馏水) 6.8 μL,共20.0 μL。反应条件:95 ℃预变性30 s;95 ℃变性5 s,60 ℃退火20 s,共40个循环;熔解曲线阶段:60 ℃ 15 s,95 ℃ 1 s。以扩增曲线、Ct值为标准,通过软件分析得到相应的标准曲线,计算目的基因的拷贝数。
1.6回复株的构建
根据GenBank中的rpoN基因序列设计扩增引物(rpoN-HF/rpoN-HR),并在上下游引物中分别加入BamHⅠ和XhoⅠ酶切位点,以细菌基因组DNA为模板扩增rpoN基因。克隆至pEASY T3载体后获得重组质粒pEASY-rpoN。用限制性内切酶BamHⅠ和XhoⅠ分别对重组质粒pEASY-rpoN和原核表达载体pGEX-6p-1进行双酶切,经DNA纯化试剂盒回收目的片段后连接过夜。连接产物转化JM109感受态细胞后,涂布于含氨苄青霉素的LB平板上,37 ℃过夜培养。鉴定阳性克隆,测序验证。将构建成功的重组表达质粒通过电击转化分别转入S016ΔrpoN和S025ΔrpoN中,并分别命名为S016ΔrpoNR和S025ΔrpoNR。
1.7生长曲线的测定
挑取野生株(S016、S025)、基因突变株(S016ΔrpoN、S025ΔrpoN)和回复株(S016ΔrpoNR、S025ΔrpoNR)的单个菌落接种于液体LB培养基,37 ℃过夜振荡培养。将过夜培养液接种于新鲜LB培养基,OD600 nm值调至0.1,37 ℃、220 r·min-1振荡培养8 h,每隔1 h测定菌液的OD600 nm值,绘制出各菌株的生长曲线。
1.8对环境应激抵抗力的测定
挑取单个菌落接种于液体LB培养基,37 ℃静置培养过夜后4 ℃、4 000 r·min-1离心10 min,用等量的PBS重悬菌体,并将其OD600 nm调至0.5。酸性应激条件(pH=5.0)下,37 ℃振荡培养20 min;碱性应激条件(pH=10.0)下,37 ℃振荡培养30 min;54 ℃热应激条件下,取100 μL重悬菌液热击3 min;氧化应激条件(10 μmol·L-1H2O2)下,37 ℃振摇培养30 min。取各应激条件下所得菌液按10倍倍比稀释,接种于麦康凯琼脂平板,过夜培养后计数[8]。
2结果
2.1基因缺失株的鉴定
以引物RpoN-F/RpoN-R PCR扩增2株鼠伤寒沙门菌rpoN基因,测序显示开放阅读框架全长1 434 bp,相似性99.9%。运用λ Red同源重组系统构建出S016ΔrpoN、S025ΔrpoN基因缺失株。PCR鉴定,所有缺失株均扩增出预期大小的条带(图1)。
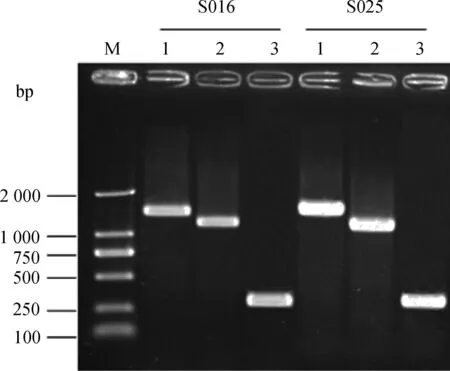
M.DL2000 DNA相对分子质量标准;1.野生型菌株扩增条带;2.cat基因插入株扩增条带;3.缺失株扩增条带M.DL2000 marker;1.The amplified fragments of wild-type strain;2.The amplified fragments of mutants with cat gene;3.The amplified fragments of mutants without cat gene图1 S016和S025突变株的PCR鉴定Fig.1 Identification of S016 and S025 mutants by PCR
2.2回复株的构建
重组表达质粒pGEX-6p-rpoN经BamHⅠ和XhoⅠ双酶切成约5 kb大小的pGEX-6p-1载体片段以及1.5 kb左右的rpoN目的片段,结果与预期相符,证明重组表达质粒构建成功(图2)。将表达质粒pGEX-6p-rpoN分别导入两株ΔrpoN缺失株中获得回复株S016ΔrpoNR和S025ΔrpoNR。
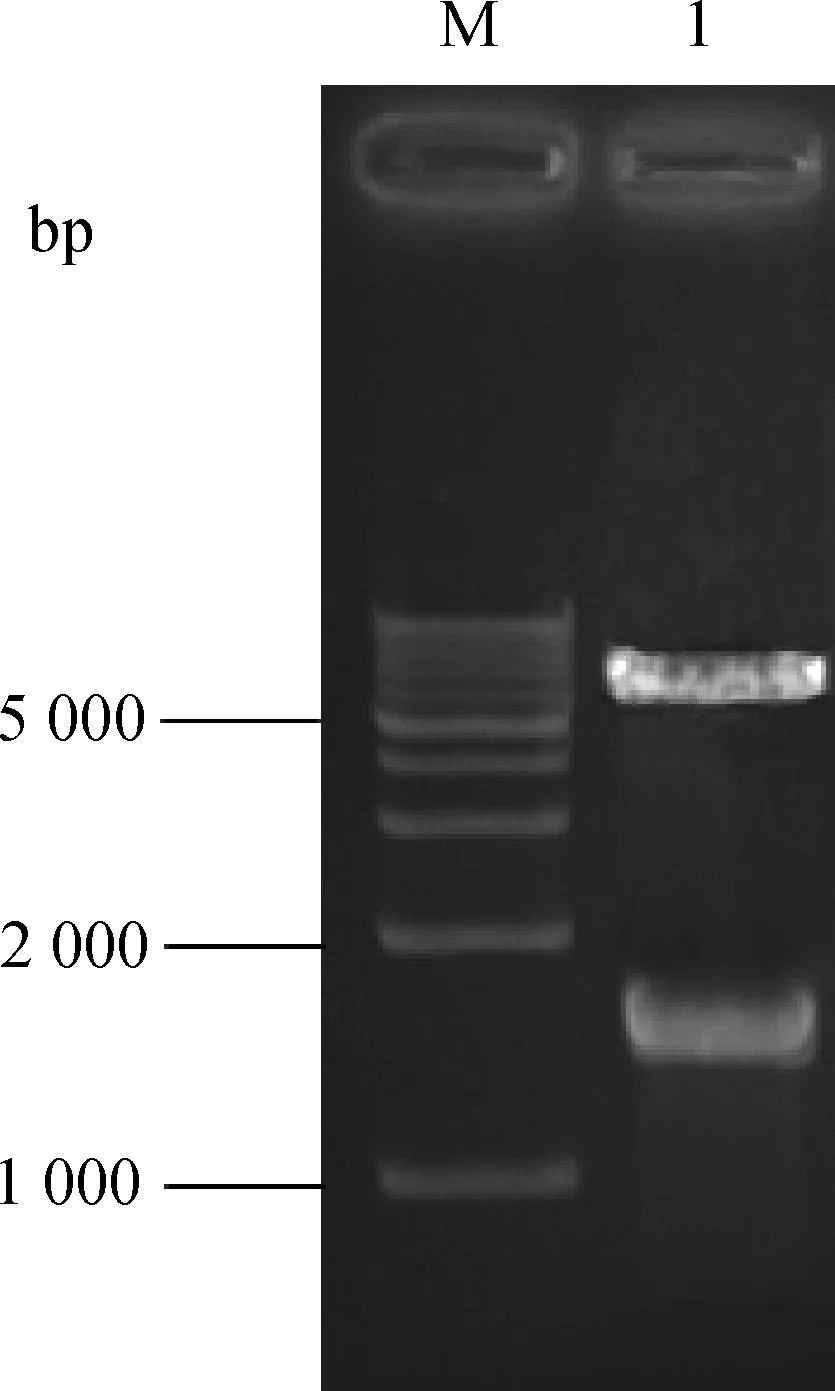
M.1 kb DNA相对分子质量标准; 1. pGEX-6p-rpoNM.1 kb marker; 1. pGEX-6p-rpoN图2 重组质粒的酶切鉴定图谱Fig.2 Restriction endonuclease digestion pattern of recombinant plasmid
2.3生长曲线的测定
对野生株(S016、S025)、基因突变株(S016ΔrpoN、S025ΔrpoN)和回复株(S016ΔrpoNR、S025ΔrpoNR)OD600 nm值的测定,绘制出生长曲线(图3)。由图可见,与野生株相比,两株rpoN基因缺失株的生长速度低于野生株。回复株的生长速度与野生株相似。
2.4生物被膜形成能力及成分的测定
以结晶紫染色定量法测定生物被膜形成能力,结果显示,野生株S016和S025的OD550 nm值分别为1.568±0.176和1.031±0.015,缺失株S016ΔrpoN和S025ΔrpoN的OD550 nm值分别为3.367±0.025和1.391±0.053。与野生株相比,S016ΔrpoN缺失株能形成较强的生物被膜,差异极显著(P<0.01);而S025ΔrpoN缺失株形成生物被膜的能力稍有增强(图4)。
刚果红和考马斯亮蓝平板法测定生物被膜成分结果显示,野生株S016和S025均呈红色干燥粗糙型菌落,紫外灯下荧光板的荧光强。与野生株相比,S016ΔrpoN缺失株呈更为明显的红色干燥粗糙型菌落,表面更为粗糙,同时紫外灯下荧光板荧光增强;而S025ΔrpoN缺失株表型变化与S016ΔrpoN缺失株类似,但程度弱于后者(图5)。

*.P<0.05图3 野生株S016(A)和S025(B)及各自缺失株和回复株的生长曲线Fig.3 Growth curves of wild-type strains S016(A)and S025(B) and their mutants and revertants

**.P<0.01图4 96孔细胞板上形成生物被膜后结晶紫染色结果(A)以及OD550 nm测定(B)Fig.4 Biofilm formed on 96-well plate and stained by CV (A) and determination of OD550 nm(B)
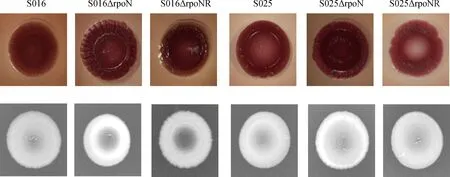
图5 野生株、缺失株及回复株的生物被膜表型及纤维素的表达测定Fig.5 The morphotype and expression of cellulose by wild-type strains,mutants and revertants
回复试验结果显示,回复株S016ΔrpoNR和S025ΔrpoNR的OD550 nm值分别为1.405±0.015和1.046±0.020,且菌落均呈现红色干燥粗糙型,荧光强度与野生株相似,说明rpoN回复株恢复了类似野生株的生物被膜形成能力。
2.5qRT-PCR检测csgA基因和bcsA基因的转录差异
以引物csgA-QF/QR和bcsA-QF/QR建立的qRT-PCR呈现单峰,说明特异性较好;扩增曲线显示重组质粒浓度在102~109可形成标准曲线。qRT-PCR检测结果显示,在生物被膜形成过程中,特别是在24 h,两株缺失株编码卷曲菌毛合成的csgA基因的转录量与野生株相比均显著增加,其中S016ΔrpoN缺失株csgA基因的转录量增加极显著;而缺失株编码纤维素合成的bcsA基因的转录较野生株相比没有显著差异(图6)。
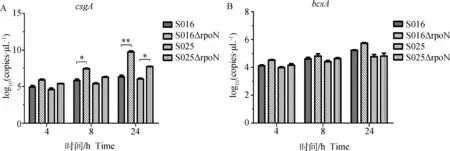
*.P<0.05;**.P<0.01图6 荧光定量PCR检测细菌在生物被膜形成过程中csgA基因(A)和bcsA基因(B)的转录量Fig.6 Transcription of csgA (A) and bcsA (B) genes in Salmonella strains during the period of biofilm formation detected by the quantitative real-time PCR

A.热应激;B.氧化应激;C.酸应激;D.碱应激。**.P<0.01A.Heat shock;B.Oxidative stress;C.Acid endurance;D.Alkali endurance.**.P<0.01图7 野生株、缺失株和回复株对于环境应激抵抗力的测定Fig.7 Determination of resistance to environmental stress for wild-type strains,mutants and revertants
2.6对环境应激抵抗力的检测
与野生株S016相比,S016ΔrpoN缺失株在酸性应激和碱性应激条件下对外界环境应激的抵抗力均显著增强(P<0.05);在热应激、氧化应激条件下抵抗力轻度增强。与野生株S025相比,S025ΔrpoN缺失株对各种外界应激的抵抗力均略有增强(图7)。
3讨论
鼠伤寒沙门菌是一种能引起人畜共患病的常见致病菌,能够在动物体内以及动物肉产品的包装和转运过程中形成生物被膜。从而起到抵御外界不利环境和宿主免疫系统的作用,最终导致疾病的发生和持续反复感染[9-11]。目前已发现多种与沙门菌生物被膜形成有关的基因,启动生物被膜形成的中心基因是csgD,rpoS基因可通过直接或间接激活csgD基因调控卷曲菌毛和纤维素的合成,进而影响生物被膜的形成[12-13];rpoE基因也能通过调控卷曲菌毛的表达影响生物被膜的形成[14]。在本实验室前期研究的基础上,作者运用Red同源重组法构建出两株生物被膜形成能力较强的鼠伤寒沙门菌的rpoN基因缺失株,发现其生物被膜形成能力增强,由此推断rpoN基因与鼠伤寒沙门菌生物被膜形成相关。
转录起始是细菌基因调控的关键一步,rpoN基因能够促进活化剂依赖的转录起始,从而调节各种代谢和细菌的发病有关的基因表达。V.S.Iyer等研究发现,rpoN基因缺失可使粪肠球菌对自体溶解产生耐受,生物被膜的形成能力增强[15]。A.S.Belik等研究也表明,在大肠杆菌K-12中,rpoN基因缺失能增强细菌生物被膜的形成[16]。本研究构建的两株rpoN基因缺失株的生物被膜形成能力较野生株相比均有增强,同时在刚果红和考马斯亮蓝平板上菌落更为粗糙,荧光定量PCR检测显示在生物被膜形成时期缺失株csgA基因的表达显著增加,说明其卷曲菌毛表达量增多;紫外灯下荧光板中rpoN基因缺失株的荧光强度与野生株相似,荧光定量PCR检测显示在生物被膜形成时期缺失株bcsA基因的表达量与野生株也无明显差异,说明纤维素表达量没有变化。因此,在鼠伤寒沙门菌中,rpoN基因的缺失也同样可以增强细菌生物被膜的形成,并通过影响卷曲菌毛的合成而导致其生物被膜形成能力增强。
细菌处于外界不利条件(如极端的营养条件及温度变化、氧化、抗菌素和消毒剂等)时,可产生胞外聚合物黏附在介质表面,以固着的方式形成生物被膜,从而更好地适应环境[17-19]。环境应激试验结果表明,rpoN基因缺失使细菌在酸应激和碱应激条件下对外界抵抗力增强,这可能与其生物被膜形成能力增强有关。因此,RpoN是一个重要的压力调控因子。本研究鉴定出rpoN基因为鼠伤寒沙门菌中生物被膜形成相关σ因子之一,同时rpoN基因能够通过影响卷曲菌毛的合成从而影响沙门菌生物被膜形成。这一发现不仅能丰富σ因子对生物被膜调控的机制,而且为新型药物的设计和新型疫苗的研制提供靶标。
4结论
成功构建了2株鼠伤寒沙门菌rpoN基因缺失株。rpoN基因缺失株生物被膜形成能力增强,对外界环境应激的抵抗力也显著增强。以上结果表明RpoN为鼠伤寒沙门菌中生物被膜形成相关σ因子之一。
参考文献(References):
[1]MAJOWICZ S E,MUSTO J,SCALLAN E,et al.The global burden of nontyphoidalSalmonellagastroenteritis[J].ClinInfectDis,2010,50(6):882-889.
[2]PATI N B,VISHWAKARMA V,JAISWAL S,et al.Deletion ofinvHgene inSalmonellaentericaserovarTyphimuriumlimits the secretion of Sip effector proteins[J].MicrobesInfect,2013,15(1):66-73.
[3]ISHIHAMA A.Functional modulation ofEscherichiacoliRNA polymerase[J].AnnuRevMicrobiol,2000,54:499-518.
[4]黄骏,陈素娟,黄凯,等.鸡白痢沙门氏菌生物被膜形成相关基因rpoE的鉴定[J].微生物学报,2015,55(2):156-163.
HUANG J,CHEN S J,HUANG K,et al.Identification ofrpoEgene associated with biofilm formation ofSalmonellapullorum[J].ActaMicrobiologicaSinica,2015,55(2):156-163.(in Chinese)
[5]PRATT L A,KOLTER R.Genetic analysis ofEscherichiacolibiofilm formation:roles of flagella,motility,chemotaxis and type I pili[J].MolMicrobiol,1998,30(2):285-293.
[6]ANRIANY Y,SAHU S N,WESSELS K R,et al.Alteration of the rugose phenotype inwaaGandddhCmutants ofSalmonellaentericaserovarTyphimuriumDT104 is associated with inverse production of curli and cellulose[J].ApplEnvironMicrobiol,2006,72(7):5002-5012.
[7]MURPHY K C.Use of bacteriophage lambda recombination functions to promote gene replacement inEscherichiacoli[J].JBacteriol, 1998,180(8):2063-2071.
[8]CHI F,WANG Y,GALLAHER T K,et al.Identification of IbeR as a stationary-phase regulator in meningiticEscherichiacoliK1 that carries a loss-of-function mutation inrpoS[J].JBiomedBiotechnol,2009,2009:520283.
[9]LEDEBOER N A,FRYE J G,MCCLELLAND M,et al.SalmonellaentericaserovarTyphimuriumrequires the Lpf,Pef,and Tafi fimbriae for biofilm formation on HEp-2 tissue culture cells and chicken intestinal epithelium[J].InfectImmun,2006,74(6):3156-3169.
[10]HAMILTON S,BONGAERTS R J,MULHOLLAND F,et al.The transcriptional programme ofSalmonellaentericaserovarTyphimuriumreveals a key role for tryptophan metabolism in biofilms[J].BMCGenomics,2009,10:599.
[11]CRULL K,ROHDE M,WESTPHAL K,et al.Biofilm formation bySalmonellaentericaserovarTyphimuriumcolonizing solid tumours[J].CellMicrobiol,2011,13(8):1223-1233.
[12]BROWN P K,DOZOIS C M,NICKERSON C A,et al.MlrA,a novel regulator of curli (AgF) and extracellular matrix synthesis byEscherichiacoliandSalmonellaentericaserovarTyphimurium[J].MolMicrobiol,2001,41(2):349-363.
[13]GRANTCHAROVA N,PETERS V,MONTEIRO C,et al.Bistable expression of CsgD in biofilm development ofSalmonellaentericaserovartyphimurium[J].JBacteriol,2010,192(2):456-466.
[14]YOO A Y,YU J E,YOO H,et al.Role of sigma factor E in regulation ofSalmonellaAgf expression[J].BiochemBiophysResCommun,2013,430(1):131-136.
[15]IYER V S,HANCOCK L E.Deletion of sigma(54) (rpoN) alters the rate of autolysis and biofilm formation inEnterococcusfaecalis[J].JBacteriol,2012,194(2):368-375.
[16]BELIK A S,TARASOVA N N,KHMEL’ I A.Regulation of biofilm formation inEscherichiacoliK12:effect of mutations in HNS,StpA,lon,andrpoNgenes[J].MolGenMikrobiolVirusol,2008(4):3-5.
[17]COSTERTON J W,STEWART P S.Battling biofilms[J].SciAm,2001,285(1):74-81.
[18]DE SILVA G D,KANTZANOU M,JUSTICE A.Theicaoperon and biofilm production in coagulase-negativeStaphylococciassociated with carriage and disease in a neonatal intensive care unit[J].JClinMicrobiol,2002,40(2):382-388.
[19]DONLAN R M.Biofilms:microbial life on surfaces[J].EmergInfectDis,2002,8(9):881-890.
(编辑白永平)
Construction and Biological Characterization of rpoN Gene Deletion Mutants ofSalmonellaTyphimurium
SU Yang-yang,HUANG Jun,WU Bai,YIN Yun-cong,LIU Zhen,CHEN Su-juan,PENG Da-xin*,LIU Xiu-fan
(JiangsuCo-InnovationCenterforthePreventionandControlofImportantAnimalInfectiousDiseaseandZoonoses,CollegeofVeterinaryMedicine,YangzhouUniversity,Yangzhou225009,China)
Abstract:The aim of this study was to understand the role ofrpoNgene on biofilm formation ofS.Typhimurium.TwoS.Typhimuriumstrains with strong biofilm-forming ability were selected for construction of deletion mutants with deficiency of generpoNusing the Red recombination system,and complemented strains were constructed by using prokaryotic expression vector.The biofilm-forming ability,and resistance to environmental stress of the wild-type,rpoNmutant andrpoNcomplemented strains were compared.A quantitative real-time PCR method based oncsgAandbcsAgenes was established to compare the expression of their biofilm components.The results showed that the biofilm formation,mainly contributed by increased expression of curli,was significantly enhanced in therpoNgene deletion mutants when compared with that of the wild-type strains.The biofilm formation of revertants was similar to that of the wild-type strains.Furthermore,deletion ofrpoNgene ofS.Typhimuriumstrains resulted in their increased resistance to acid and alkali environment.Therefore, RpoN was identified as one of the associated sigma factors involved in biofilm formation ofS.Typhimurium.These data may be helpful for elucidating the regulatory mechanism ofSalmonellabiofilm formation.
Key words:SalmonellaTyphimurium;biofilm formation;rpoNgene;mutants;environmental stress
doi:10.11843/j.issn.0366-6964.2016.04.017
收稿日期:2015-10-16
基金项目:国家自然科学基金项目(31572530);公益行业(农业)科研专项(201303044);江苏高校优势学科建设工程;江苏省家禽疫病防控工程技术研究中心(BM2013450)
作者简介:苏洋洋(1992-),江苏涟水人,硕士生,主要从事微生物学研究,E-mail:1106610036@qq.com *通信作者:彭大新,E-mail: pengdx@yzu.edu.cn
中图分类号:S852.612
文献标志码:A
文章编号:0366-6964(2016)04-0771-08

