PmrA-PmrB二元调控系统介导大肠杆菌对黏杆菌素耐药的机制研究
葛 琳,郭大伟,何 方,黄金虎,王丽平
(南京农业大学动物医学院,南京 210095)
PmrA-PmrB二元调控系统介导大肠杆菌对黏杆菌素耐药的机制研究
葛琳,郭大伟,何方,黄金虎,王丽平*
(南京农业大学动物医学院,南京 210095)
摘要:已报道PmrA-PmrB二元调控系统在革兰阴性菌对多黏菌素B耐药过程中起重要作用,基于此认识,作者拟从基因突变和mRNA表达两个方面探讨PmrA-PmrB介导大肠杆菌对黏杆菌素耐药的可能性。首先检测了临床分离的52株禽致病性大肠杆菌对黏杆菌素的敏感性,筛选出耐药菌株;然后采用step-wise方法对黏杆菌素敏感菌株进行诱导,获得人工诱导的耐药菌株;最后通过PCR扩增所有耐药菌株的pmrA-pmrB后采用Mega软件进行突变位点分析,并采用实时荧光定量PCR(qRT-PCR)技术检测所有耐药菌株中pmrA-pmrB的mRNA转录水平的变化,拟阐明PmrA-PmrB二元调控系统对禽致病性大肠杆菌黏杆菌素耐药性产生的作用。MIC结果显示,52株大肠杆菌中,虽然大多数菌株(88.5%,46/52)仍对黏杆菌素敏感,但也分离到少数耐药菌株(为11.5%)。突变位点分析表明人工诱导成功的5株耐药菌(9R、36R、53R、91R和107R)pmrA未发生突变,而pmrB均有不同程度的突变,其中耐药菌株9R、36R和53R各在G55A (G19R)、T500C(L167P)和T263A(V88E)发生点突变,而91R和107R在229位和478位各插入长为30和189 bp的序列。但临床分离的6株耐药菌株(MIC=4~8 μg·mL-1)的pmrA-pmrB均未发生突变。qRT-PCR结果显示发生点突变的三株人工诱导耐药菌pmrA-pmrB转录量均极显著(P<0.01)或显著(P<0.05)上升,发生插入突变的两株耐药菌pmrA-pmrB转录量虽有上升趋势,但变化不显著(P>0.05),而临床耐药菌株(n=6)的pmrA-pmrB转录量均无变化。进一步检测人工诱导不同MIC的耐药菌株中pmrA-pmrB的突变位点,发现菌株MIC达16 μg·mL-1时pmrA-pmrB才会发生突变。PmrA和PmrB介导了大肠杆菌对黏杆菌素的高度耐药,其中PmrB的点突变伴随PmrA-PmrB的高表达或者PmrB的组氨酸激酶-腺苷酰环化酶-甲基结合蛋白-磷酸化酶(HAMP)结构域的插入突变是导致禽致病性大肠杆菌对黏杆菌素高度耐药的机制之一。
关键词:黏杆菌素耐药;大肠杆菌;pmrA-pmrB;突变
多黏菌素 E (polymyxin E) 又称黏杆菌素(colistin),是Bacilluspolymyxinvar.colistinus代谢产生的碱性多肽类抗生素,含有酰胺键与脂肪酸相连组成的阳离子环[1]。黏杆菌素主要通过与细菌的外膜脂多糖结合,改变细胞膜稳定性,从而导致细菌破裂死亡,故对革兰阴性杆菌有强烈的抑菌作用[2]。2006—2009年,人类肠道菌群的流行病学检测已有证据显示鲍曼不动杆菌、铜绿假单胞菌和肺炎克雷伯杆菌对黏杆菌素的耐药率分别维持在0.9%、1.5%和0.4%,大肠杆菌耐药率维持在0.2%,其中大肠杆菌对头孢类、环丙沙星等药物耐药率均已达到20%以上[3]。由于多药耐药大肠杆菌感染广泛流行,后续又缺乏有效的抗生素,所以多黏菌素类抗生素作为革兰阴性杆菌感染治疗的最后一道防线已经开始应用于临床,但目前在临床中已分离到其耐药菌株[4-5]。
二元调控系统可作为原核生物应答外界环境的信号转导途径[6]。典型的二元调控系统由组氨酸激酶和相关的应答调控蛋白组成,组氨酸激酶感受外界环境变化后调节应答蛋白的磷酸化水平,而后对一系列应答基因进行调控[7]。目前发现PmrA-PmrB二元调控系统参与铜绿假单胞菌和克雷伯杆菌对多肽菌素B和抗菌肽的耐药性调节[8]。S.M.Moskowitz等[9]发现多黏菌素B耐药菌株中存在pmrA-pmrB基因的突变,该突变有利于pmrB感受器激酶磷酸化,进而激活pmrA,介导细菌对多黏菌素B的耐药。已经有研究证实二元调控系统元件发生突变可以使其过表达,导致arnBCADTEF基因激活,转录翻译出4-氨基-4-脱氧-L-阿拉伯糖(L-Ara4N)或烟酸季戊四醇酯(PEtN),添加到细菌外膜的脂质A结构上,降低细菌对多黏菌素的吸附[10-12]。K.Nishino等[13-14]研究发现大肠杆菌的二元调控系统过表达还可引起eygA等基因的表达变化,介导大肠杆菌对多种抗生素产生耐药性。pmr作为多黏菌素耐药相关基因,可以通过促进LPS共价修饰,减少药物与革兰阴性菌的接触,导致细菌耐药[15]。已有相关文献报道,在低Mg2+环境下,PhoP-PhoQ和PmrA-PmrB可以导致铜绿假单胞菌对黏杆菌素耐药[16]。
本试验借鉴鲍曼不动杆菌及铜绿假单胞菌等革兰阴性菌的研究成果,充分论证该研究的可行性后,以临床分离的52株禽致病性大肠杆菌为研究对象,通过MIC测定、诱导耐药菌、检测分析相关基因突变位点以及mRNA转录水平变化,明确PmrA-PmrB在禽致病性大肠杆菌对黏杆菌素耐药中的作用,进一步为大肠杆菌耐药性的分子流行病学调查和发掘新的药物靶位提供参考,进而探索阻遏细菌耐药性上升趋势的合理策略。
1材料与方法
1.1试剂
Solution I,pMD-19T载体;2×Prime STAR GXL DNA Polymerse Mix;DL2000 marker购自上海皓嘉生物科技有限公司;Trizol,2×Taq MasterMix,SYBY Green real-time PCR Master Mix购自南京钟鼎生物科技有限公司;焦碳酸二乙酯(DEPC),dNTP,RNAse inhibitor,Random primer,琼脂糖,M-MLV反转录酶购自南京生兴生物科技有限公司;无水乙醇,异丙醇,氯仿购自南京寿德有限公司;PCR产物回收试剂盒为AXYGEN生物公司提供。
1.2菌株
52株大肠杆菌,2014年1—12月分离自江苏5个不同的发病鸭场,经鉴定后保存于本实验室。所有相关菌株均挑取单菌落于LB肉汤,37 ℃ 150 r·min-1震荡培养16 h后用于试验。
1.3MIC测定
根据美国临床检验标准委员会推荐的肉汤二倍稀释法测定52株禽致病性大肠杆菌对黏杆菌素的最小抑菌浓度(minimum inhibitory concentration,MIC),并根据欧洲抗菌药物敏感性试验委员会(EUCAST)[17]规定的耐药性折点分析菌株的耐药性。
女人来得最晚,被介绍的时候,坐正首的一个黑脸男人笑着说知道她。女人很惊诧,说您是哪位呀,怎么知道我?女人以为这个男人曾经是她的患者也说不定,就没在意。可那人接着说我叫田连民,道外区的税收员。
1.4耐药株体外诱导
根据上述已测定的MIC值,选择9、36、53、91和107 号(表1)敏感菌株进行体外诱导试验。采用step-wise方法,以1/2×MIC,2×MIC,4×MIC,8×MIC依次类推的药物梯度逐步诱导敏感菌株,实时监测MIC变化情况直至其耐药。取各亲本菌株不同MIC值(8、16、64和≥256 μg·mL-1)的耐药菌株在无药平板上连续传代,直至其耐药性稳定,保存备用。
表1试验所用引物序列
Table 1Primers used in this study

相关引物Relatedprimer引物序列(5'-3')Sequence退火温度/℃Annealedtemperature产物长度/bpProductlengthPCR引物(PrimersforPCR)pmrA-FpmrA-RATGAAAATTCTGATTGTTGAAGACGTTAGTTTTCCTCATTCGCGACC64669pmrB-FpmrB-RATGCATTTTCTGCGCCGACTTATATCTGGTTTGCCACGTACTGA641092RT-PCR引物(PrimersforRT-PCR)pmrA-FpmrA-RCCTTTTGCGCTGGAAGAGTTCTTTGGGCGTCAGAATCAAC59158pmrB-FpmrB-RCTGCAAGAAGATGACGGAGCCTGTGTAATGCGGCTGACAA59156dnaE-FdnaE-RGGACATGAGCACCGAAGATTGGAAGCCCATCTGGTTGATA59161
1.5突变位点检测
PCR扩增所有耐药菌株的pmrA-pmrB(引物由苏州金唯智生物有限公司合成,见表1),产物纯化后连接pMD-19T载体并转化DH5α进行蓝白斑筛选,选择插入条带正确的菌液送样测序。所测得的基因序列均采用MEGA软件进行突变位点分析,并上传GenBank获取相应的基因序列号。
1.6qRT-PCR检测pmrA和pmrB转录水平
提取各临床分离耐药菌株、诱导耐药菌株及其相应亲本菌株的总RNA,Nanodrop 2000紫外分光光度计测定RNA浓度及纯度后反转录为cDNA,分别以pmrA和pmrB为模板设计qRT-PCR引物,以大肠杆菌看家基因dnaE为内参进行荧光定量PCR反应(引物见表1 所示)。
1.7统计学处理
2结果
2.1MIC测定及耐药菌体外诱导
临床分离各菌株的MIC见表2所示。经统计可知,本次所分离的禽致病性大肠杆菌对黏杆菌素大多数呈敏感(88.5%),但也分离到6株低度耐药菌株(1、3、12、38、54和94号),占分离菌株的11.5%。
根据已测定菌株的MIC值,选取5株敏感菌株(9、36、53、91和107号)采用黏杆菌素进行耐药性诱导,筛选到不同程度的黏杆菌素耐药菌株(表3),选取MIC分别为8、16、64和≥ 256μg·ml-1的耐药菌株进行后续研究。
表2黏杆菌素对临床分离大肠杆菌的最小抑菌浓度
Table 2MICs of colistin againstE.colistrains isolated from diseased ducks
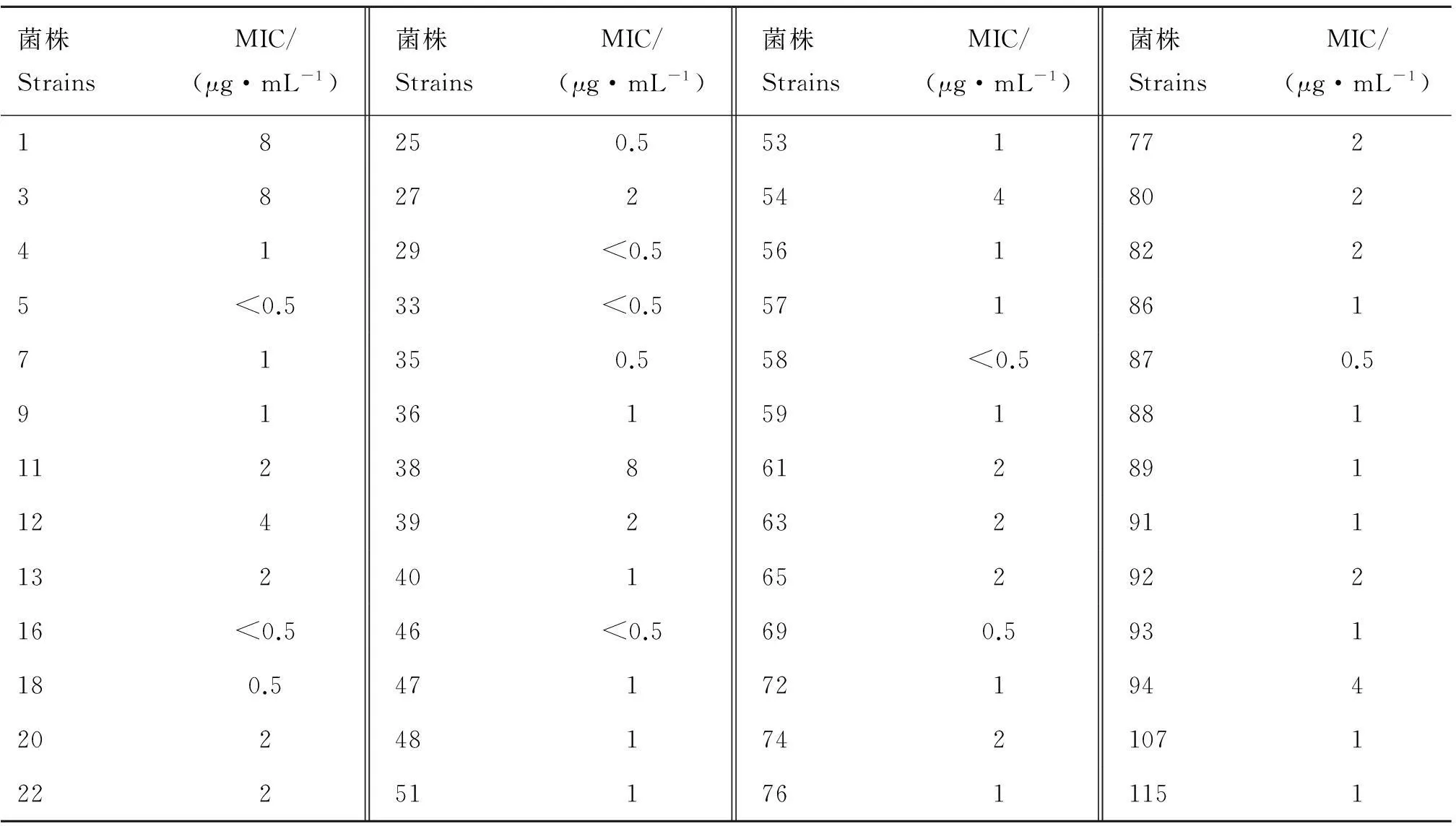
菌株StrainsMIC/(μg·mL-1)菌株StrainsMIC/(μg·mL-1)菌株StrainsMIC/(μg·mL-1)菌株StrainsMIC/(μg·mL-1)18250.5531772382725448024129<0.55618225<0.533<0.557186171350.558<0.5870.59136159188111238861289112439263291113240165292216<0.546<0.5690.5931180.547172194420248174210712225117611151
2.2pmrA和pmrB突变位点分析
通过PCR扩增所有耐药菌株和部分敏感菌株的二元调控系统pmrA和pmrB基因,并采用MEGA2.0软件分析了所有菌株的pmrA和pmrB基因序列。表3结果表明:临床分离的低度耐药菌株的pmrA和pmrB均未发生突变;所有人工诱导耐药菌株的pmrA无突变,但部分耐药菌株pmrB则发生不同位点突变。所检测到的耐药菌株pmrB突变情况分为两种:即3株敏感菌9、36和53菌株所产生的部分耐药菌分别在55位、500位和263位发生点突变使之氨基酸发生相应变化,而另2株敏感菌91和107菌株所产生的部分耐药菌则分别在229位和478位发生插入突变,91R插入序列重复了亲本株pmrB208—237位之间的序列,107R插入序列重复了亲本株pmrB289—477位之间的序列,并且结果显示,所有人工诱导耐药菌株的MIC值达到16 μg·mL-1以上时,pmrB基因才会发生突变。以上所有测序基因的序列均已上传至GenBank并获得序列号。
2.3诱导耐药菌及临床耐药菌pmrA和pmrBmRNA转录变化
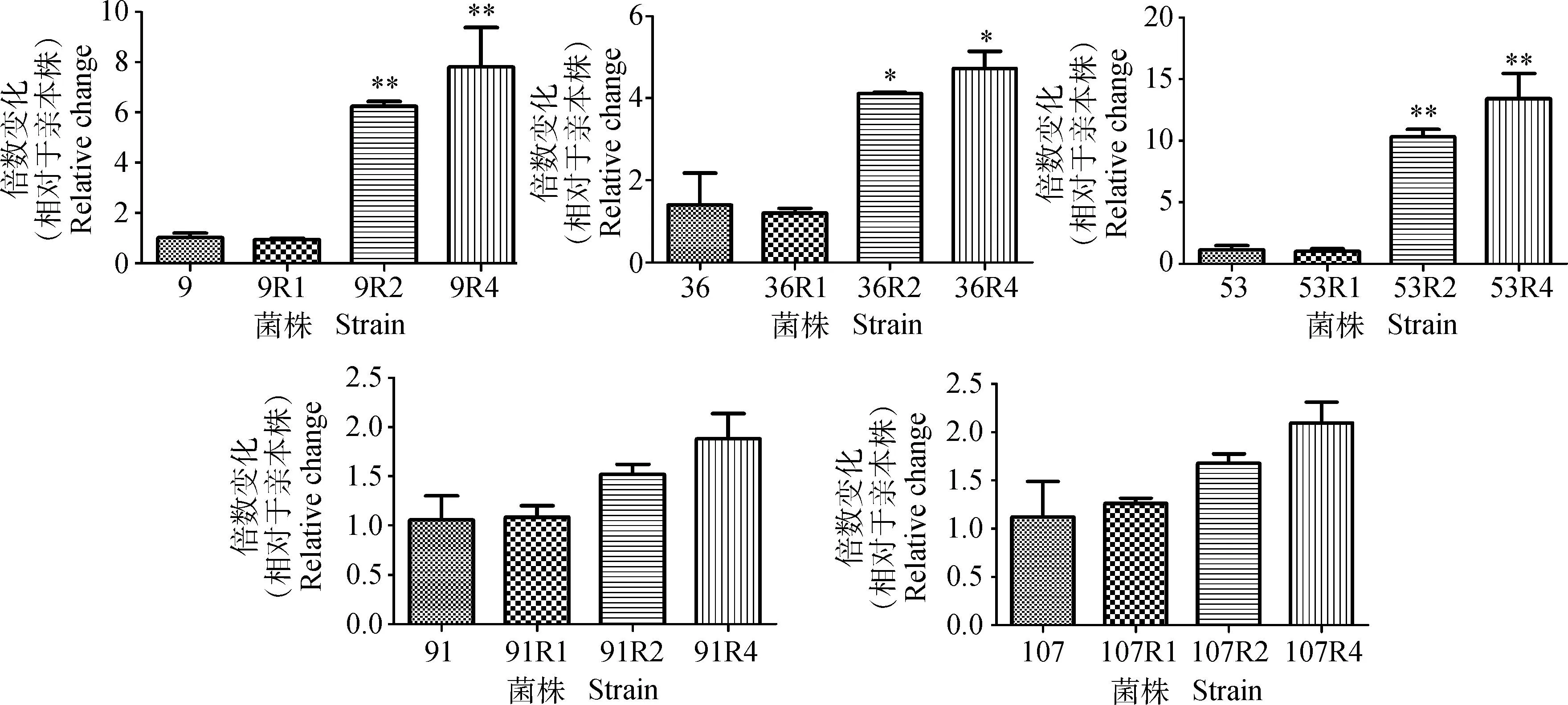
如图1所示,36、53和9号菌诱导产生的耐药菌株MIC达到16 μg·mL-1以上时,pmrAmRNA的转录量都呈极显著上升(P<0.01),并且各耐药菌株随MIC升高,其转录量也呈上升趋势。91和107号菌株所产生的耐药菌株即使MIC大于256 μg·mL-1,pmrA的mRNA转录量也未发生显著变化(P>0.05)。
图2结果显示,9、53 和36 号菌诱导所产生的耐药菌株在MIC达16 μg·mL-1以上时,pmrB转录量呈极显著(9R2、9R4和53R2、53R4,P<0.01)或显著(36R2、36R4,P<0.05) 上升;但是91号和107号菌株产生的所有耐药菌pmrB转录量虽有上升趋势,但均未发生显著变化(P>0.05)。
图3结果显示,与敏感株相比,临床分离耐药菌株的pmrA、pmrB转录量均无显著变化(P>0.05)。
表3耐药大肠杆菌的pmrB突变位点
Table 3pmrBmutations in colistin resistance strains
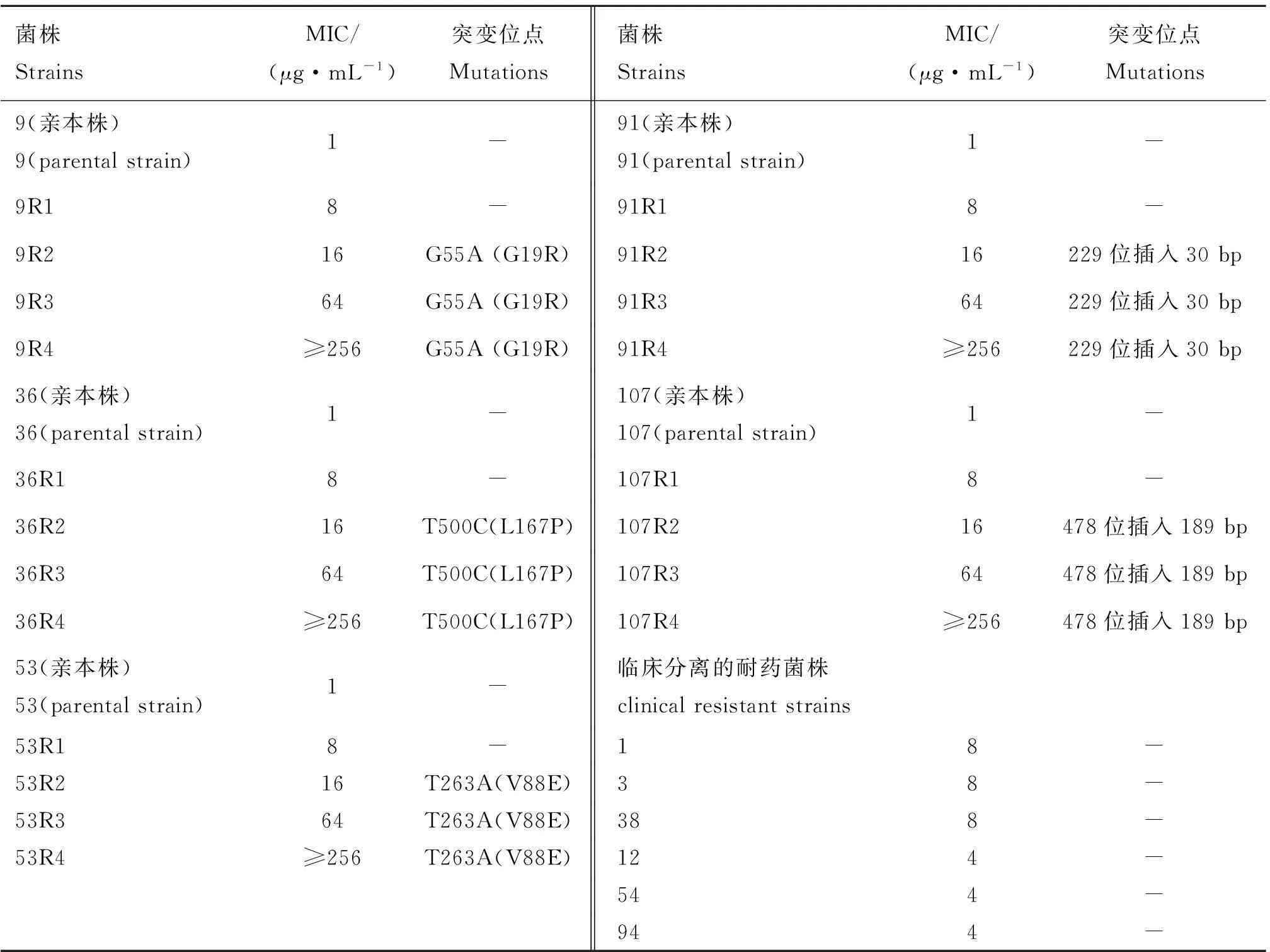
菌株StrainsMIC/(μg·mL-1)突变位点Mutations菌株StrainsMIC/(μg·mL-1)突变位点Mutations9(亲本株)9(parentalstrain)1-91(亲本株)91(parentalstrain)1-9R18-91R18-9R216G55A(G19R)91R216229位插入30bp9R364G55A(G19R)91R364229位插入30bp9R4≥256G55A(G19R)91R4≥256229位插入30bp36(亲本株)36(parentalstrain)1-107(亲本株)107(parentalstrain)1-36R18-107R18-36R216T500C(L167P)107R216478位插入189bp36R364T500C(L167P)107R364478位插入189bp36R4≥256T500C(L167P)107R4≥256478位插入189bp53(亲本株)53(parentalstrain)1-临床分离的耐药菌株clinicalresistantstrains53R18-18-53R216T263A(V88E)38-53R364T263A(V88E)388-53R4≥256T263A(V88E)124-544-944-
“-”表示无任何突变位点
“-” means no mutations

R1.MIC=8 μg·mL-1;R2.MIC=16 μg·mL-1;R4.MIC≥256 μg·mL-1;**.P<0.01图1 各耐药菌株pmrA转录量变化Fig.1 Changes of pmrA mRNA transcription levels in resistance strains
R1.MIC=8 μg·mL-1;R2.MIC=16 μg·mL-1;R4.MIC≥256 μg·mL-1;*.P<0.05;**.P<0.01图2 各耐药菌株pmrB转录量变化Fig.2 Changes of pmrB mRNA transcription levels in resistance strains
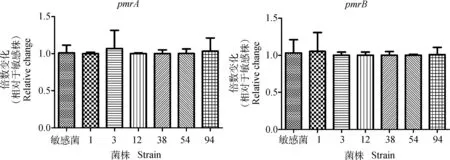
图3 临床耐药菌株pmrA-pmrB转录量变化Fig.3 Changes of pmrA-pmrB mRNA transcription levels in resistance strains
3讨论
多黏菌素对生长繁殖期与静止期细菌均有杀菌作用,最早在20世纪60年代用于革兰阴性菌感染,80年代后由于其肾毒性而逐渐被弃用[2]。近年来,由于多药耐药革兰阴性菌感染广泛流行,多黏菌素作为治疗的最后选择又被应用于临床[18]。黏杆菌素也是兽医临床常用的多肽类抗生素,本研究结果显示临床分离致病性大肠杆菌对黏杆菌素的耐药率相对较低,与A.C.Gales等[3]对革兰阴性菌的调查结果相一致,表明黏杆菌素在临床治疗大肠杆菌时仍有一定优势,尤其对其他抗菌药耐药的大肠杆菌仍有较好效果。虽然如此,本试验中也分离到对黏杆菌素低度耐药的禽致病性大肠杆菌,应引起重视。
现已报道鲍曼不动杆菌和铜绿假单胞菌的多黏菌素耐药菌株,其耐药机制分别与lpxA、lpxC、lpxD突变以及二元调控系统PhoP-PhoQ和PmrA-PmrB的调控有关[19-20]。A.Quesada等[21]初步研究猪源和禽源大肠杆菌与沙门菌对黏杆菌素的耐药机制,发现耐药菌的PmrA/PmrB发生突变,证实PmrA S39I、R81S和PmrB V161G突变与菌株对黏杆菌素的耐药存在相关性。除此以外未见有关大肠杆菌对黏杆菌素耐药机制的研究报道。
本试验参考已报道的有关黏杆菌素耐药的相关基因,首先检测了各诱导耐药菌株lpxA、lpxC、lpxD基因是否发生突变及其在转录水平的表达量变化,但未检测到该类基因的任何变化,因此推测二元调控系统PmrA-PmrB在大肠杆菌对黏杆菌素耐药中产生作用,故本试验从突变位点分析和转录水平检测这两个方面探讨二元调控系统PmrA-PmrB在大肠杆菌对黏杆菌素耐药中的作用。PmrA-PmrB二元调控系统中,pmrB基因的HATPase结构域(762—1 059位)提供能量将hisKA结构域(432—594位)上的组氨酸His (456位) 磷酸化,进而将信号传递给pmrA,从而起到调控基因表达的作用。此外,HAMP结构域(207—408位)编码组氨酸激酶及磷酸酶类,并含有甲基化结合位点。突变位点分析显示,人工诱导耐药株的pmrB序列均存在突变现象,如91R耐药菌的HAMP结构域(207—408位)的229位插入的30 bp序列;53R菌株的(T263A)突变,使得第89位氨基酸由缬氨酸变为谷氨酸,该突变位点也位于HAMP结构域(207—408);107R耐药菌在478位插入的189 bp序列,该位点处于hisKA结构域(432—594位)中,临近磷酸化位点(456位)和HAMP结构域(207—408位);36R耐药菌在500位发生点突变(T500C),该突变位点与J.Y.Lee等[20]在克雷伯杆菌与铜绿假单胞菌中证实的与黏杆菌素耐药相关的突变位点相同,同样位于pmrB的hisKA结构域(432—594位)内;9R(G55A)位点的突变导致第19位氨基酸由甘氨酸变为精氨酸,位于TM1结构域(39—105位),该结构域为跨膜转运区。已有研究表明pmrB上的HATPase结构域(762—1 059位)提供能量将hisKA结构域(432—594位)上的组氨酸His(456位) 磷酸化,进而将信号传递给pmrA从而起到调控基因表达的作用[22]。A.O.Olaitan等[23]的研究结果也证实HAMP结构域发生点突变、缺失或移码都能导致对多黏菌素耐药。还有结果证实多黏菌素B耐药菌株中存在pmrA-pmrB基因的突变,该突变有利于pmrB感受器激酶磷酸化,进而激活pmrA,使其表达量上升,介导细菌对黏杆菌素的耐药[9]。本试验发现发生点突变3株耐药菌的pmrA-pmrB转录量均极显著(P<0.01)或显著上升(P<0.05)上升,并且随着耐药菌MIC升高,转录量也逐渐升高,但是发生插入突变的两株耐药菌的pmrA-pmrB转录量虽有上升趋势,但变化不显著 (P>0.05),这两类耐药菌之间差异的原因值得进一步系统研究。同时人工诱导耐药大肠杆菌对多黏菌素产生高度耐药时(MIC至少高于16 μg·mL-1)其pmrB才会发生突变,进而导致pmrA-pmrBmRNA转录显著上升,该现象解释了临床分离的耐药菌株(MIC=8 μg·mL-1)为何未检测到pmrA-pmrB突变和mRNA转录量变化,由此可见大肠杆菌对黏杆菌素仍存在其他耐药机制,有待依次揭示。
综上所述,pmrA和pmrB在大肠杆菌对黏杆菌素的耐药中起一定作用,pmrB的点突变伴随pmrA-pmrB的高表达或者pmrBHAMP结构域的插入突变是导致大肠杆菌对黏杆菌素高度耐药的机制之一。
参考文献(References):
[1]LI J,NATION R L,TURNIDGE J D,et al.Colistin:the re-emerging antibiotic for multidrug-resistant Gram-negative bacterial infections[J].LancetInfectDis,2006,6(9):589-601.
[2]王旭,向文良,冯玮.多黏菌素E研究进展[J].现代农业科学,2009,16(1):24-25.
WANG X,XIANG W L,FENG W.The reserach progress of polymyxin E[J]ModernAgriculturalSciences,2009,16(1):24-25.(in Chinese)
[3]GALES A C,JONES R N,SADER H S.Contemporary activity of colistin and polymyxin B against a worldwide collection of Gram-negative pathogens:results from the SENTRY Antimicrobial Surveillance Program (2006-09)[J].JAntimicrobChemother,2011,66(9):2070-2074.
[4]H K S,T S S,GOEL B R,et al.Quantitative assessment of apical debris extrusion and intracanal debris in the apical third,using hand instrumentation and three rotary instrumentation systems[J].JClinDiagnRes,2014,8(2):206-210.
[5]GALLES A C,JONES R N,SADER H S.Antimicrobial susceptibility profile of contemporary clinical strains of Stenotrophomonas maltophilia isolates:can moxifloxacin activity be predicted by levofloxacin MIC results?[J].JChemother,2008,20(1):38-42.
[6]MIZUNO T.His-Asp phosphotransfer signal transduction[J].JBiochem,1998,123(4):555-563.
[7]HOCH J A.Two-component and phosphorelay signal transduction[J].CurrOpinMicrobiol,2000,3(2):165-170.
[8]LEWENZA S,FALSAFI R K,WINSOR G,et al.Construction of a mini-Tn5-luxCDABE mutant library in Pseudomonas aeruginosa PAO1:a tool for identifying differentially regulated genes[J].GenomeRes,2005,15(4):583-589.
[9]MOSKOWITZ S M,ERNST R K,MILLER S I.PmrAB,a two-component regulatory system of Pseudomonas aeruginosa that modulates resistance to cationic antimicrobial peptides and addition of aminoarabinose to lipid A[J].JBacteriol,2004,186(2):575-579.
[10]GUNN J S,LIM K B,KRUEGER J,et al.PmrA-PmrB-regulated genes necessary for 4-aminoarabinose lipid A modification and polymyxin resistance[J].MolMicrobiol,1998,27(6):1171-1182.
[11]GUO L,LIM K B,GUNN J S,et al.Regulation of lipid A modifications by Salmonella typhimurium virulence genes phoP-phoQ[J].Science,1997,276(5310):250-253.
[12]ROLAND K L,MARTIN L E,ESTHER C R,et al.Spontaneous pmrA mutants of Salmonella typhimurium LT2 define a new two-component regulatory system with a possible role in virulence[J].JBacteriol,1993,175(13):4154-4164.
[13]NISHINO K,YAMAGUCHI A.EvgA of the two-component signal transduction system modulates production of the yhiUV multidrug transporter inEscherichiacoli[J].JBacteriol,2002,184(8):2319-2323.
[14]NISHINO K,YAMAGUCHI A.Overexpression of the response regulator evgA of the two-component signal transduction system modulates multidrug resistance conferred by multidrug resistance transporters[J].JBacteriol,2001,183(4):1455-1458.
[15]KEMPF I,FLEURY M A,DRIDER D,et al.What do we know about resistance to colistin in Enterobacteriaceae in avian and pig production in Europe?[J].IntJAntimicrobAgents,2013,42(5):379-383.
[16]MCPHEE J B,BAINS M,WINSOR G,et al.Contribution of the PhoP-PhoQ and PmrA-PmrB two-component regulatory systems to Mg2+-induced gene regulation in Pseudomonas aeruginosa[J].JBacteriol,2006,188(11):3995-4006.
[17]European Committee for Antimicrobial Susceptibility Testing (EUCAST) of the European Society of Clinical Microbiology and Infectious Diseases (ESCMID).EUCAST Definitive Document E.DEF 2.1,August 2000:Determination of antimicrobial susceptibility test breakpoints[J].ClinMicrobiolInfect,2000,6(10):570-572.
[18]李然,王睿,李碧艳.多黏菌素对泛耐药革兰阴性杆菌严重感染临床应用新进展[J].中国药物应用与监测,2009,6(6):364-367
LI R,WANG R,LI B Y.New advances of polymyxins in the treatment of pan drug resistant gram-negative bacterial infection[J].ChineseJournalofDrugApplicationandMonitoring,2009,6(6):364-367.(in Chinese)
[19]MOFFATT J H,HARPER M,HARRISON P,et al.Colistin resistance in Acinetobacter baumannii is mediated by complete loss of lipopolysaccharide production[J].AntimicrobAgentsChemother,2010,54(12):4971-4977.
[20]LEE J Y,KO K S.Mutations and expression of PmrAB and PhoPQ related with colistin resistance in Pseudomonas aeruginosa clinical isolates[J].DiagnMicrobiolInfectDis,2014,78(3):271-276.
[21]QUESADA A,PORRERO M C,TÉLLEZ S,et al.Polymorphism of genes encoding PmrAB in colistin-resistant strains ofEscherichiacoliand Salmonella enterica isolated from poultry and swine[J].JAntimicrobChemother,2015,70(1):71-74.
[22]YU Z,QIN W,LIN J,et al.Antibacterial mechanisms of polymyxin and bacterial resistance[J].BiomedResInt,2015,2015:679109.
[23]OLAITAN A O,MORAND S,ROLAIN J M.Mechanisms of polymyxin resistance:acquired and intrinsic resistance in bacteria[J].FrontMicrobiol,2014,5:643.
(编辑白永平)
Resistance Mechanism of Escherichia coli to Colistin Mediated by PmrA-PmrB
GE Lin,GUO Da-wei,HE Fang,HUANG Jin-hu,WANG Li-ping*
(CollegeofVeterinaryMedicine,NanjingAgriculturalUniversity,Nanjing210095,China)
Abstract:It has been recently reported that PmrA-PmrB are associated with polymyxin B resistance.To determine whether PmrA-PmrB is contributing to colistin resistance inE.coli,we investigated amino acid alterations and transcription level ofpmrA-pmrBin colistin resistance strains.All clinical resistance isolated strains were screened for colistin resistance by using MIC determinations and step-wise method was used to induce colistin resistant strains.pmrA-pmrBgenes were amplified by PCR from resistant strain and their sequences were characterized by Mega software.Simultaneously,expression levels of PmrA-PmrB were detected by quantitive Real-time PCR.MIC (minimum inhibitory concentration) results showed that 88.5% of 52 clinic resistance strains tested in this study were susceptibility to colistin and 11.5% of the strains were resistant.Five mutations were identified inpmrBin induced colistin-resistant mutants 9R (G55A),36R (T500C),53R (T263A),91R (insertion of 30 bp at position of 229) and 107R (insertion of 189 bp at position of 478).However,six clinical resistance isolates (MIC=4-8 μg·mL-1) did not show any mutations inpmrA-pmrB.Further study showed thatpmrA-Bexpression levels were significantly activated in three lab-derived colistin resistant strains with point mutations ofpmrB.The expression level ofpmrA-pmrBgenes tended to increase but not significant changed in resistant strains with insertion mutations compared with its correspond parent strains.The transcription level ofpmrA-pmrBin 6 clinical resistance isolates remained unchanged compared with susceptible strains.In addition,when MICs were over than 16 μg·mL-1,mutations inpmrA-pmrBwould occur.Mutations inpmrBor high expression levels inpmrA-pmrBcontribute to high-level colistin resistance inE.colistrains.
Key words:colistin resistance;Escherichiacoli;pmrA-pmrB;mutations
doi:10.11843/j.issn.0366-6964.2016.04.022
收稿日期:2015-11-09
基金项目:江苏省自然基金(BK2012771);江苏省高校"青蓝工程"中青年学术带头人项目
作者简介:葛琳(1989-),女,安徽淮北人,硕士,主要从事细菌耐药性的研究, E-mail:760135099@qq.com *通信作者:王丽平,E-mail:wlp71@163.com
中图分类号:S859.7
文献标志码:A
文章编号:0366-6964(2016)04-0812-08

