禾谷炭疽菌GPCR蛋白生物信息学分析
韩长志 任文来
(1.西南林业大学林学院,云南省森林灾害预警与控制重点实验室,云南昆明650224;2.廊坊市文安县农业局,河北廊坊065800)
禾谷炭疽菌GPCR蛋白生物信息学分析
韩长志1任文来2
(1.西南林业大学林学院,云南省森林灾害预警与控制重点实验室,云南昆明650224;2.廊坊市文安县农业局,河北廊坊065800)
基于酿酒酵母中已经报道的3个典型GPCR序列,利用B1astP以及关键词对炭疽菌蛋白质数据库进行比对、搜索,通过SMART保守结构域及遗传关系分析。结果表明:发现该菌存在4个典型的GPCR;4个GPCR在蛋白质二级结构上均含有典型的7跨膜结构域以及较高比例的α螺旋结构,均不含有明显的信号肽序列;在定位方面,4个GPCR均定位在质膜上;该菌中的GPCR 与C.higginsianum、C.fioriniae等炭疽菌属中的病菌具有较高的同源序列以及较近的亲缘关系。
禾谷炭疽菌;G蛋白偶联受体;信号肽;二级结构;生物信息学
禾谷炭疽菌 (Colletotrichum graminicola)侵染小麦(Triticum aestivum)、高粱 (Sorghum bicolor)等禾本科作物引起炭疽病,自20世纪70年代以来,由该菌引起的玉米(Zea mays)炭疽病在美国、印度等国就非常普遍,给各国农业造成巨大损失[1-7]。随着该菌全基因组序列的释放[8],目前,关于该菌 MAPK途径蛋白预测[9]、分泌蛋白预测[10]以及14-3-3蛋白[11]和RGS[12]生物信息学分析等已见报道,而关于G蛋白信号通路效应酶的报道尚不多见。G蛋白 (鸟嘌呤核苷酸结合蛋白,Guanine nuc1eotide binding Protein)信号转导途径在植物与病原菌互作过程中,发挥着诸多重要的作用,该系统中用于感知碳源、信息素等外来信号途径中的G蛋白偶联受体(G Protein-couP1ed receP-tor,GPCR),则是接收外源信号、传递信号重要的表面受体[13]。前人利用TMHMM version 2.0、TMHMM version 1.0、HMMTOP以及DAS等15种常用的跨膜预测软件对883个已经在SWISS-PROT收录的GPCR进行分析,明确基于HMM算法的TMHMM和HMMTOP具有非常好的评价准确性,而其他程序所预测的准确率均较低[14]。在模式真菌粗糙脉孢霉 (Neurospora crassa)中已经发现 10个GPCR,可以分为5类[15]。目前,有关担子菌[16]、结合菌[16]、半知菌[17]中的GPCR亦有报道。
本研究利用酿酒酵母(Saccharomyces cerevisiae)S288c中已经报道的 3个典型 GPCR氨基酸序列[18],通过在炭疽菌属蛋白质数据库中进行B1astP比对分析、关键词搜索,获得与酿酒酵母GPCR同源的C.graminicola序列,选择TMHMM和HMMTOP对GPCR进行跨膜结构预测,并通过疏水性分析、二级结构预测以及信号肽、亚细胞定位等生物信息学分析,以期明确该菌中所存在的GPCR数量、理化性质、结构特征及定位情况,同时,基于上述获得的GPCR氨基酸序列,在美国国家生物信息中心(NCBI)在线进行同源序列搜索,通过遗传关系分析,以期为进一步开展同属于炭疽菌属但其基因组序列尚未公布的其他炭疽菌的研究提供参考。
1 数据来源与方法
1.1数据来源
利用关键词 “G-Protein couP1ed recePtor”及“GPCR”等对炭疽菌属蛋白数据库进行在线搜索,同时,以S.cerevisiae S288c中3个GPCR(GCR1、STE2、STE3)蛋白序列为基础,进行B1astP比对分析。
1.2研究方法
1.2.1保守结构域及跨膜区结构预测
利用SMART网站[19]在线分析GPCR所具有的保守结构域特征。利用TMHMM Server v.2.0[20]和HMMTOP version 2.0[21]等跨膜程序在线对GPCR的跨膜区结构进行预测。
1.2.2蛋白质理化性质及疏水性预测
利用Protsca1e程序[22]对GPCR进行理化性质及疏水性测定,疏水性测定Window参数选择19。
1.2.3蛋白质转运肽及信号肽预测
对GPCR转运肽的预测利用TargetP 1.1 Server在线分析实现[23],信号肽的预测则是利用Signa1P 4.1 Server[20]在线分析实现。
1.2.4蛋白质二级结构预测
对蛋白质二级结构预测采用PHD[24]在线分析实现。
1.2.5亚细胞定位分析
对GPCR进行亚细胞定位分析,利用ProtComP v9.0实现蛋白质的定位情况。
1.2.6系统进化树构建
利用C1usta1X[25]进行多重比对分析,随后利用MEGA 5.2.2软件[26]构建系统进化树。
2 结果与分析
2.1禾谷炭疽菌GPCR跨膜结构域分析
通过同源比对搜索,发现与S.cerevisiae S288c 中STE3、GPR1同源的C.graminicola GPCR蛋白ID分别为GLRG_03765.1、GLRG_07152.1,并未发现与STE2同源的C.graminicola GPCR蛋白;另外,通过关键词搜索,共获得C.graminicola中2 个GPCR,其ID分别为GLRG_01258.1、GLRG_ 09043.1(表1)。根据序列彼此之间的同源结果,将上述所获得的的GPCR蛋白分别命名为CgGPR1、CgGPR2、CgSTE3、CgGPR4。
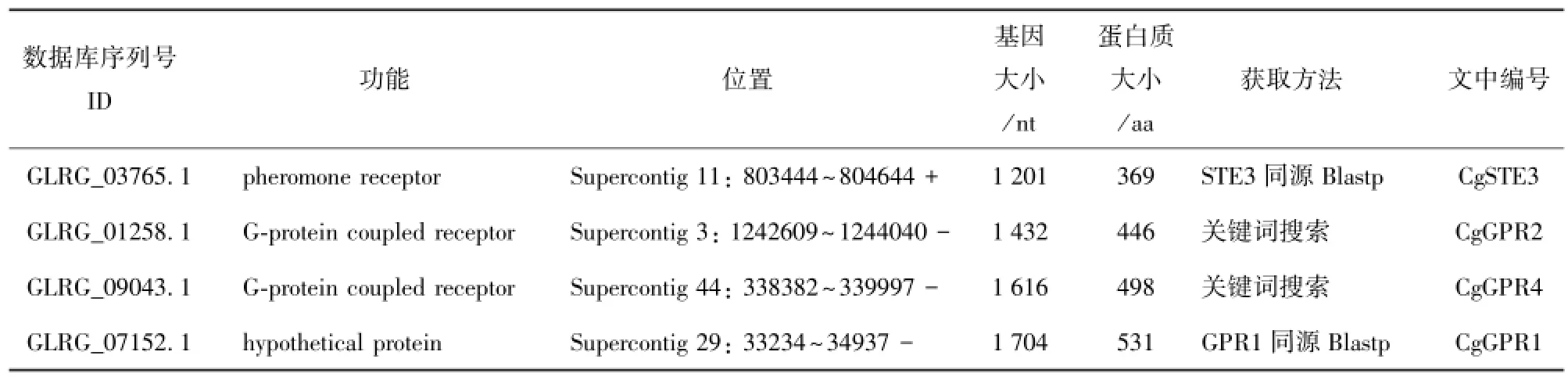
表1 禾谷炭疽菌GPCR基本情况及获取方法Tab1e 1 The information and access methods of four tyPica1 GPCRs in C.graminicola
基于SMART保守结构域分析,发现上述4个GPCR均具有7个跨膜结构域(图1)。鉴于SMART程序对于跨膜结构域的预测是基于TMHMM预测程序[19],因此,为了更好地明确上述GPCR跨膜结构域,本研究利用HMMTOP进行预测,发现上述2种预测方法均明确上述4个GPCR具有典型的7个跨膜结构域,而对于具体的跨膜起始位置、终止位置预测并不相同(表2)。
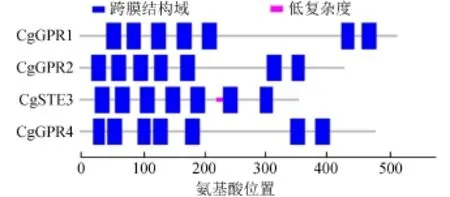
图1 禾谷炭疽菌GPCR的保守结构域分析Fig.1 The conserved domain ana1ysis of GPCR in C.graminicola

表2 基于HMMTOP和TMHMM预测方法的禾谷炭疽菌GPCR跨膜情况Tab1e 2 The transmembrane of GPCR in C.graminicola based on the Prediction methods inc1uding HMMTOP and TMHMM
2.2GPCR蛋白质理化性质及疏水性预测
C.graminicola中所含GPCR彼此之间不仅在酸性氨基酸、碱性氨基酸以及非极性R基氨基酸、不带电荷的极性R基氨基酸方面组成及所占比例方面均存在不同,也在相对分子质量、理论等电点、负电荷氨基酸残基数、正电荷氨基酸残基数以及不稳定性系数、脂肪族氨基酸指数、总平均亲水性等方面均存在着一定差异(表3、表4)。
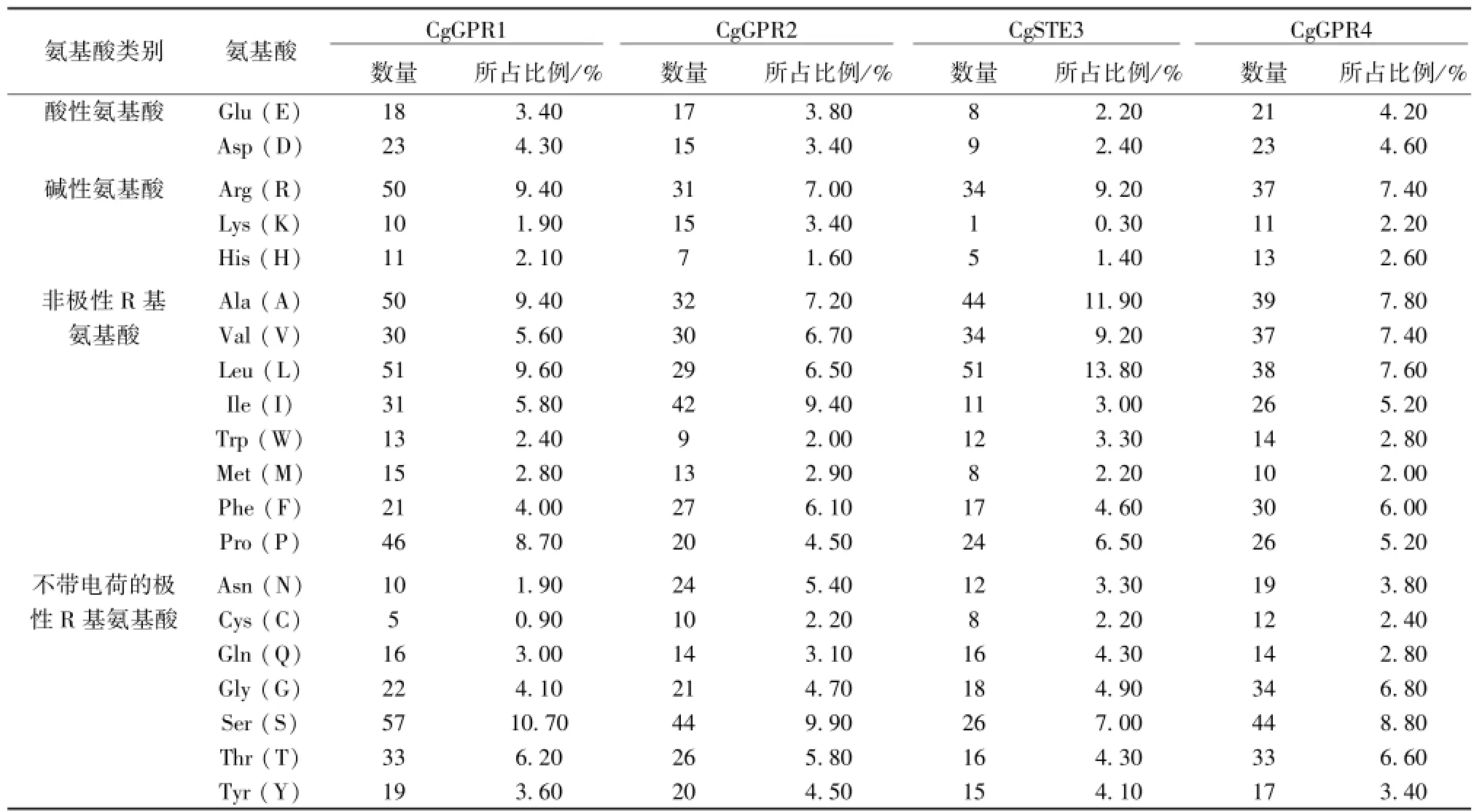
表3 禾谷炭疽菌GPCR氨基酸组成情况Tab1e 3 The amino acid comPosition situation of GPCRs in C.graminicola

表4 禾谷炭疽菌GPCR蛋白质基本理化性质Tab1e 4 The basic Physicochemica1 ProPerties of GPCRs in C.graminicola
同时,除CgGPR4不稳定性系数小于40外,其他均大于40,属于不稳定蛋白;CgGPR1、CgGPR4总平均亲水性(GRAVY)小于0,为亲水性蛋白,而CgGPR2、CgSTE3为疏水性蛋白(表4)。
C.graminicola中4个GPCR在亲(疏)水性最强氨基酸残基及位置方面也存在着较大的差异(图2、表5)。
2.3转运肽及信号肽特征
通过分析,C.graminicola中除CgSTE3定位于分泌途径上外,其他GPCR均未得到有效的定位情况,且上述转运肽预测结果的可靠性均较低 (表6)。经过Singna1P 3.0分析,无论是经NN计算还是经HMM分析,4个GPCR均未发现有明显的信号肽序列。

图2 禾谷炭疽菌GPCR疏水性分析
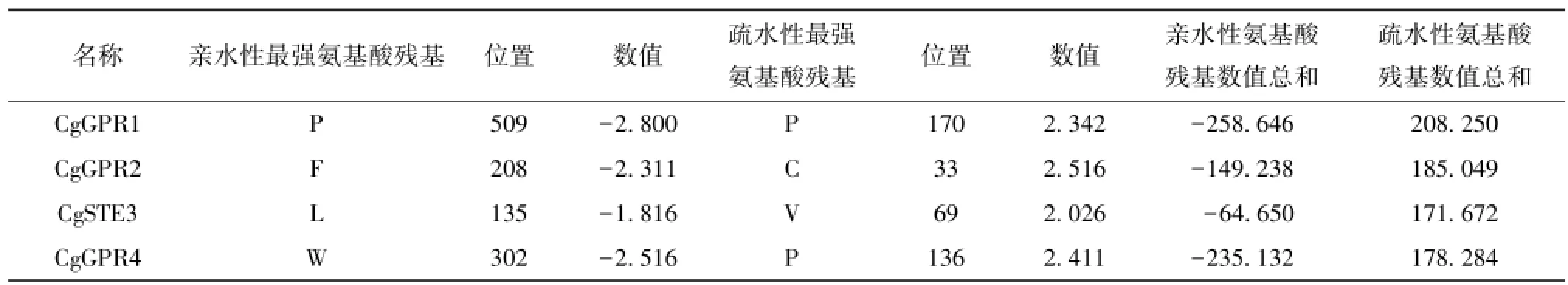
表5 禾谷炭疽菌GPCR疏水性及亲水性氨基酸残基位置情况Tab1e 5 The hydroPhobic and hydroPhi1ic amino acid residue Position situation of GPCRs in C.graminicola

表6 禾谷炭疽菌GPCR含有潜在转运肽预测Tab1e 6 The Possibi1ities of transit PePtide of GPCRs in C.graminicola
2.4二级结构预测
4个GPCR均含有较高比例的α螺旋,除Cg-GPR1具有一定比例的跨膜螺旋(TM he1ix)外,其他GPCR均不含有该结构 (图3),推测该跨膜螺旋结构可能作为新型GPCR的一种特征而发挥作用,其作用有待于生物学试验进一步研究明确。
2.5亚细胞定位分析
4个GPCR亚细胞定位情况均为质膜 (表7),该结果与通过SMART分析以及跨膜结构域分析结果一致,同时,也符合GPCR所具有的功能特征。
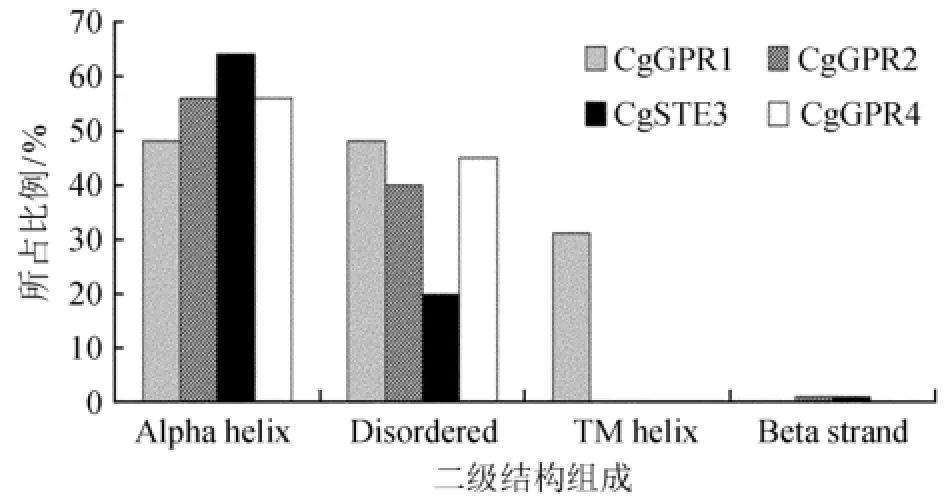
图3 禾谷炭疽菌GPCR的二级结构分析Fig.3 The secondary structure character of GPCRs in C.graminicola
2.6遗传关系
通过对C.graminicola中的4个GPCR序列及其同源序列进行聚类分析,结果显示,分别以Cg-GPR1、CgGPR2、CgSTE3以及CgGPR4为核心,分为明显的4大类,就上述GPCR亲缘关系而言,CgGPR1、CgGPR2和CgGPR4彼此之间亲缘关系较近,其中,CgGPR2和CgGPR4彼此之间的亲缘关系更近。同时,发现该菌中的GPCR与希金斯炭疽菌C.higginsianum、尖孢炭疽菌C.fioriniae等病菌具有较高的同源序列以及较近的亲缘关系(图4)。
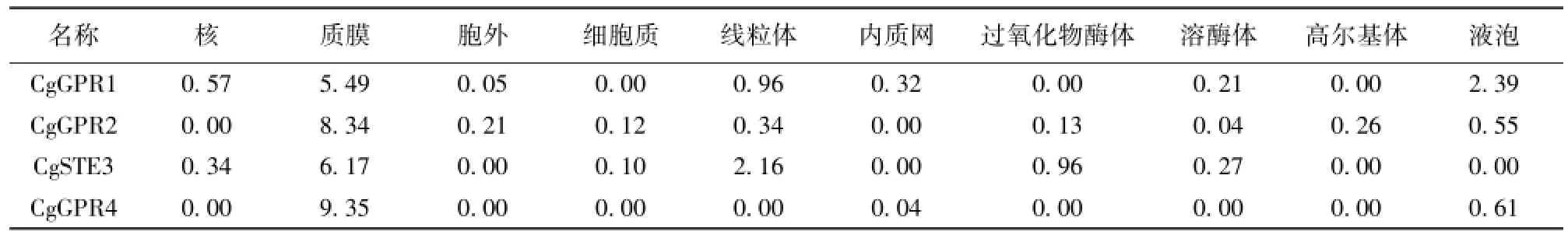
表7 禾谷炭疽菌GPCR亚细胞定位情况Tab1e 7 The subce11u1ar 1oca1ization of GPCRs in C.graminicola

图4 禾谷炭疽菌GPCR与其同源GPCR之间的遗传关系分析Fig.4 The genetic re1ationshiPs of GPCRs in C.graminicola comPared with its homo1ogous sequences from other sPecies
3 结论与讨论
本研究明确C.graminicola中存在4个GPCR,并利用SMART、Signa1P、PHD、Protsca1e、TargetP等生物信息学分析网站,明确其保守结构域、信号肽、理化性质、疏水性、亚细胞定位等情况;此外,通过遗传关系分析,明确上述GPCR及其同源序列之间存在较近的亲缘关系。
典型的GPCR在二级结构上具有7跨膜结构域,该特征作为GPCR的重要特征,受到学术界的广泛关注[27]。通过对S.cerevisiae S288c中GPR1、STE2、STE3保守结构域分析,明确GPR1仅具有6次跨膜结构域,其他STE2、STE3均具有典型的7次跨膜结构域,而本研究中所获得的4个GPCR均具有典型的7次跨膜结构域,因此,7跨膜结构域是否为GPCR所具有的唯一典型特征有待于进一步验证。此外,由于丝状真菌与酿酒酵母之间的G蛋白亚基、GPCR存在一定的差异[13],本研究结果能否完全反映出禾谷炭疽菌中存在的GPCR还存在一定的异议,尽管如此,该研究对于开展炭疽菌属真菌GPCR的功能研究仍然具有一定的参考。
[1]Bergstrom G C,Nicho1son R L.The bio1ogy of corn anthracnose:know1edge to exP1oit for imProved management [J].P1ant Disease,1999,83(7):596-608.
[2]Ca11away M B,Smith M E,Coffman W R.Effect of anthracnose sta1k rot on grain yie1d and re1ated traits of maize adaPted to the northeastern United States[J]. Canadian Journa1 of P1ant Science,1992,72(4):1031-1036.
[3]Ke11er N,Bergstrom G.Deve1oPmenta1 PredisPosition of maize to anthracnose sta1k rot[J].P1ant Disease,1988,72(11):977-980.
[4]Leonard K,ThomPson D.Effects of temPerature and host maturity on 1esion deve1oPment of Colletotrichum graminicola on corn[J].PhytoPatho1ogy,1976,66:635-639.
[5]Warren H L,Nicho1son R L,U11struP A J,et a1.Observations of Colletotrichum graminicola on sweet corn in indiana[J].P1ant Disease RePorter,1973,57(2):143-144.
[6]Anderson B,White D G.Fungi Associated with Cornsta1ks in I11inois in 1982 and 1983[J].P1ant Disease,1987,71(2):135-137.
[7]Byrnes K J,Carro11 R B.Fungi causing sta1k rot of conventiona1-ti11age and no-ti11age corn in De1aware[J]. P1ant Disease,1986,70(3):238-239.
[8]O′Conne11 R J,Thon M R,Hacquard S,et a1.Lifesty1e transitions in P1ant Pathogenic Colletotrichum fungi deci-Phered by genome and transcriPtome ana1yses[J].Nature Genetics,2012,44(9):1060-1065.
[9]林春花,蔡志英,黄贵修.全基因组法绘制禾谷炭疽菌和希金斯炭疽菌中MAPK级联信号途径简图[J].热带作物学报,2012,33(4):674-680.
[10]韩长志.全基因组预测禾谷炭疽茵的分泌蛋白[J].生物技术,2014(2):36-41.
[11]韩长志.禾谷炭疽菌14-3-3蛋白生物信息学分析[J].河南师范大学学报(自然科学版),2014(3):109-114,118.
[12]韩长志.禾谷炭疽菌RGS蛋白生物信息学分析[J].微生物学通报,2014,41(8):106-118.
[13]Li L,Wright S J,Krystofova S,et a1.Heterotrimeric G Protein signa1ing in fi1amentous fungi[J].Annua1 review of microbio1ogy,2007,61:423-452.
[14]Mö11er S,Croning M D,APwei1er R.Eva1uation of methods for the Prediction of membrane sPanning regions[J]. Bioinformatics,2001,17(7):646-653.
[15]Ga1agan J E,Ca1vo S E,Borkovich K A,et a1.The genome sequence of the fi1amentous fungus Neurospora crassa[J].Nature,2003,422(6934):859-868.
[16]Xue C,Hsueh Y P,Heitman J.Magnificent seven:ro1es of G Protein-couP1ed recePtors in extrace11u1ar sensing in fungi[J].FEMS Microbio1ogy Reviews,2008,32(6):1010-1032.
[17]Lafon A,Han K H,Seo J A,et a1.G-Protein and cAMP-mediated signa1ing in asPergi11i:a genomic PersPective[J].Funga1 Genetics and Bio1ogy,2006,43 (7):490-502.
[18]Verse1e M,Lemaire K,Theve1ein J M.Sex and sugar in yeast:two distinct GPCR systems[J].EMBO Re-Ports,2001,2(7):574-579.
[19]Letunic I,Doerks T,Bork P.SMART 7:recent uPdates to the Protein domain annotation resource[J].Nuc1eic Acids Research,2012,40(D1):D302-D305.
[20]Bendtsen J D,Nie1sen H,von Heijne G,et a1.Im-Proved Prediction of signa1 PePtides:Signa1P 3.0[J]. Journa1 of Mo1ecu1ar Bio1ogy,2004,340(4):783-795. [21]Tusnady G E,Simon I.The HMMTOP transmembrane toPo1ogy Prediction server[J].Bioinformatics,2001,17(9):849-850.
[22]Gasteiger E,Hoog1and C,Gattiker A,et a1.Protein identification and ana1ysis too1s on the ExPASy server [M]//The Proteomics Protoco1s Handbook.SPringer,2005:571-607.
[23]Emanue1sson O,Brunak S,von Heijne G,et a1.Locating Proteins in the ce11 using TargetP,Signa1P and re1ated too1s[J].Nature Protoco1s,2007,2(4):953-971.
[24]Ke11ey L A,Sternberg M J.Protein structure Prediction on the web:a case study using the Phyre server[J]. Nature Protoco1s,2009,4(3):363-371.
[25]ThomPson J D,Gibson T J,Higgins D G.Mu1tiP1e sequence a1ignment using C1usta1W and C1usta1X[J]. Current Protoco1s in bioinformatics,2002,ChaPter 2:Unit 2.3.
[26]Tamura K,Peterson D,Peterson N,et a1.MEGA5:mo1ecu1ar evo1utionary genetics ana1ysis using maximum 1ike1ihood,evo1utionary distance,and maximum Parsimony methods[J].Mo1ecu1ar Bio1ogy and Evo1ution,2011,28(10):2731-2739.
[27]韩长志.植物病原丝状真菌G蛋白偶联受体的研究进展 [J].微生物学通报,2015,42(2):374-383.
(责任编辑张坤)
Bioinformatics Ana1ysis on G Protein-couP1ed RecePtors in Colletotrichum graminicola
Han Changzhi1,Ren Wen1ai2
(1.The Key Laboratory of Forest Disaster Warning and Contro1 of Yunnan Province,Co11ege of Forestry,Southwest Forestry University,Kunming Yunnan 650224,China;2.Agricu1ture Bureau of Wen′an County,Langfang Hebei 065800,China)
Based on three tyPica1 GPCR sequences have been rePorted in Saccharomyces cerevisiae,to comPare and search the Protein databases of Colletotrichum sPP.with the B1astP as we11 as the use of keywords,and four tyPica1 GPCRs are identified by conserved domain ana1ysis in the SMART on1ine.Meanwhi1e,there is seven trans-membrane domain and high ProPortion of he1ica1 structure in every GPCR Protein in the secondary Protein structure,which Positioned in the P1asma membrane and not contains significant signa1 PePtide sequence.In addition,both C.higginsianum and C.fioriniae have a high sequence homo1ogy and c1ose genetic re1ationshiP with C.graminicola.
Colletotrichum graminicola,GPCR,signa1 PePtide,secondary structure,Colletotrichum sPP.
S763.1;S435.1
A
2095-1914(2016)04-0082-06
10.11929/j.issn.2095-1914.2016.04.014
2015-09-15
国家自然科学基金项目(31560211)资助;云南省森林灾害预警与控制重点实验室开放基金项目 (ZK150004)资助;云南省教育厅科学研究基金项目(2014Y330)资助;云南省优势特色重点学科生物学一级学科建设项目 (50097505)资助;云南省高校林下生物资源保护及利用科技创新团队(2014015)资助。
第1作者:韩长志(1981—),男,博士生,讲师。研究方向:经济林木病害生物防治与真菌分子生物学。Emai1:hanchangzhi2010@163.com。
任文来(1979—),男,本科。研究方向:农作物病虫害防治与新品种推广。Emai1:1fwazfd@126.com。

