大肠埃希菌耐药特征及质粒介导喹诺酮耐药基因分布
潘 玫, 陈 山, 李 稳, 刘臣彪, 石 媛
大肠埃希菌耐药特征及质粒介导喹诺酮耐药基因分布
潘 玫1, 陈 山2, 李 稳1, 刘臣彪1, 石 媛1
目的 调查大肠埃希菌分离株的耐药特征及质粒介导喹诺酮耐药基因流行状况。方法 纸片扩散(K-B)法进行药物敏感试验,采用双纸片法检测产超广谱β内酰胺酶(ESBL),聚合酶链反应(PCR)检测qnrA、qnrB、qnrC、qnrD、qnrS、aac(6' )-Ib、qepA基因,限制性酶切反应确定aac(6' )-Ib-cr基因型。结果 179株大肠埃希菌中95株ESBL表型确证试验阳性,检出率为53.1%;产ESBL菌株未见对美罗培南耐药,对亚胺培南耐药率极低(1.1%),对阿米卡星、哌拉西林-他唑巴坦、头孢哌酮-舒巴坦耐药率皆低于30%,对哌拉西林、头孢噻肟、头孢曲松、氨曲南、甲氧苄啶-磺胺甲唑耐药率皆高于70%,对庆大霉素、头孢他啶、环丙沙星、左氧氟沙星耐药率在60%~70%;PCR扩增结果显示产ESBL菌株,具有qnr基因9株(9.5%),其中qnrA 2株、qnrB 5株、qnrS 2株。非产ESBL菌株,具有qnrB基因3株(3.6%);酶切反应显示产和非产ESBL菌株具有aac(6' )-Ib-cr基因分别为21株(22.1%)和5株(6.0%)。所有菌株皆未检测到qepA基因。结论 产ESBL大肠埃希菌呈现多重耐药趋势,质粒介导喹诺酮耐药基因以aac(6')-Ib-cr基因型为主。
大肠埃希菌; 超广谱β内酰胺酶; 质粒介导喹诺酮耐药; 基因
氟喹诺酮类药物是临床广泛应用的抗感染药物,伴随这类药物应用,细菌对其耐药已迅速发展。在肠杆菌科细菌中,最初喹诺酮耐药机制为染色体介导,包括药物靶位改变、膜渗透性改变和(或)外排泵系统过度表达。至上个世纪90年代末,发现质粒介导喹诺酮耐药(PMQR)机制的存在,目前PMQR基因,如qnr,aac(6')-Ib-cr及qepA等在临床菌株中流行以及对细菌耐药影响已引起广泛的重视。
大肠埃希菌为临床常见的病原菌, PMQR基因在产超广谱β内酰胺酶(ESBL)的大肠埃希菌高度流行已在世界范围内得到认同。对大肠埃希菌耐药状况和氟喹诺酮耐药基因分布的调查,对临床更合理应用氟喹诺酮类抗菌药物,减少其耐药菌株的产生具有重要意义。本研究的目的是评价大肠埃希菌(包括产ESBL和非产ESBL菌株)耐药状况及PMQR基因流行分布特征。
1 材料与方法
1.1材料
1.1.1菌株 179株分离自山东中医药大学附属医院2012年10月—2013年12月临床标本,同一患者同一部位仅用首次分离菌株。标准菌株由山东省临检中心馈赠。聚合酶链反应(PCR)阳性对照株皆由山东省立医院王勇博士提供。
1.1.2培养基与药敏纸片 MH培养基、药敏纸片为OXOID产品。
1.1.3仪器与试剂 鉴定系统为法国生物梅里埃公司产品GN鉴定板条。细菌鉴定仪为VITEK2-Compact全自动快速微生物鉴定仪(法国生物梅里埃公司产品)。PCR扩增仪为 7 300 Real Time PCR System(美国Applied Biosystems公司产品)。电泳仪为Power Pac Basic 型电泳仪(美国Bio-Rad公司产品)、凝胶成像分析仪为美国UVPGDS-8000。PCR引物为博尚生物公司合成,反应体系为大连宝生物有限公司产品。BstF5I为立陶宛MBI公司产品。
1.2方法
1.2.1药物敏感试验及ESBL表型确证试验 采用美国临床和实验室标准化协会(CLSI) 2014年版推荐的纸片扩散(K-B)法进行药敏试验及双纸片确证法[1]。
1.2.2PCR模板制备 挑纯培养菌落置入0.5 mL离心管内(内预置200 ng/mL 蛋白酶K溶液200 μL) , 95 ℃水浴10 min ,离心(17 000×g) 30 s。上清液即为基因检测的模板液, -20 ℃冰箱保存备用。反应体系、反应条件、引物序列见表1。
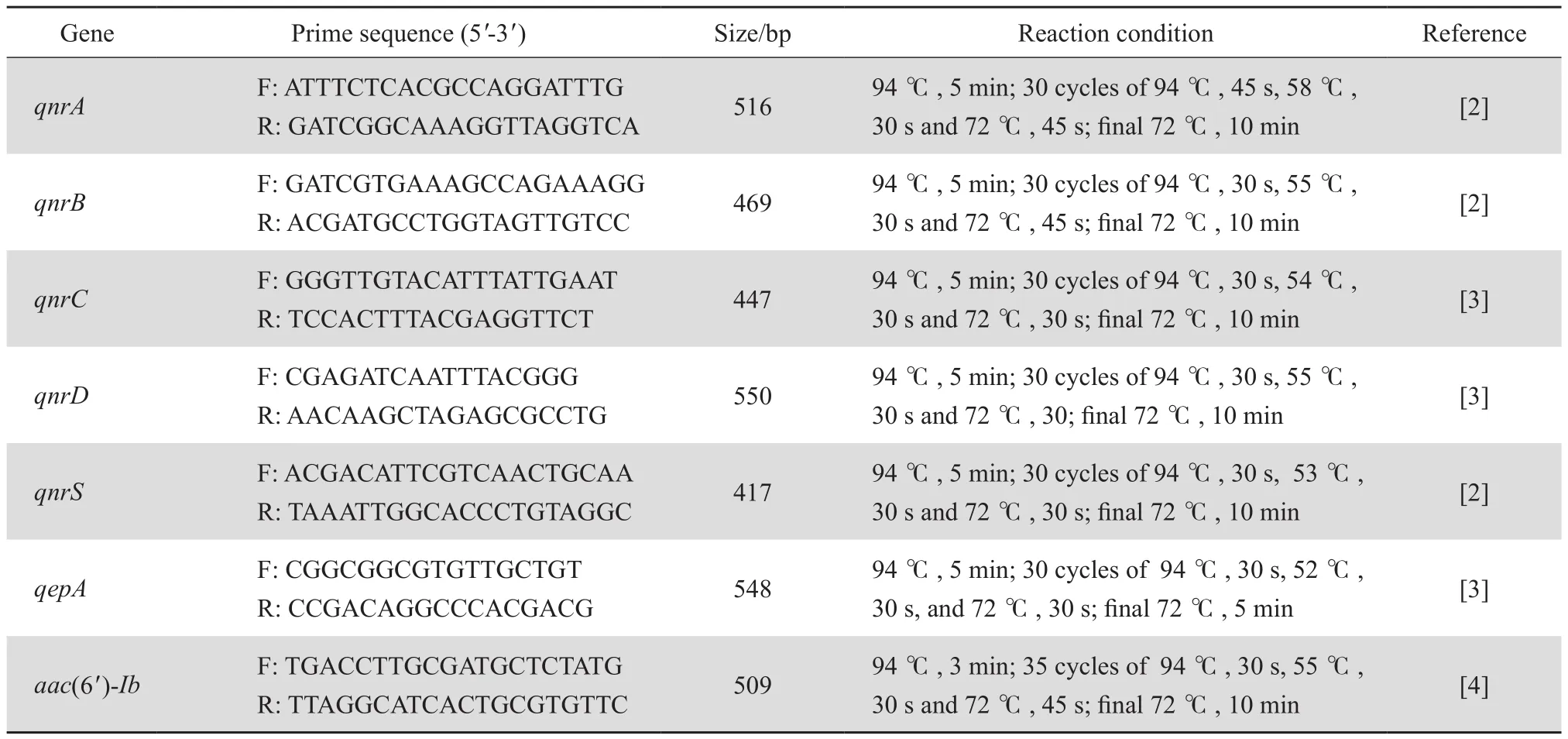
表1 PCR引物及反应条件Table 1 Primers used in PCR and references
1.2.3限制性酶切片段 应用BstF5I限制性酶消化所有aac(6')-Ib阳性的扩增产物,由于aac (6')-Ib-cr缺乏酶切位点,酶切产物进行电泳,缺乏272-bp和210-bp DNA酶切片段[5]。
1.2.4药敏数据分析 采用WHO 推荐的WHONET 5. 6软件对数据进行统计分析。
2.1大肠埃希菌ESBL表型检测及耐药状况
179株大肠埃希菌中,表型确证试验结果为产ESBL 95株,占53.1%,非产ESBL 84株,占46.9%,产与非产ESBL菌株耐药率见表2。
2 结果

表2 大肠埃希菌耐药率Table 2 Resistance profile of E. coli in terms of production of extended-spectrum beta-lactamases (%)
2.2大肠埃希菌中qnr,aac(6')-Ib-cr,qepA基因分布
179株大肠埃希菌中,检测出qnr 12株(6.7%);aac(6')-Ib 72株(40.2%),其中aac(6')-Ib-cr变体26株(14.5%);未见携带qepA基因菌株。具体基因分布见表3。PCR扩增结果见图1。

表3 大肠埃希菌株中qnr,aac(6')-Ib-cr,qepA基因分布Table 3 Distribution of qnr, aac(6')-Ib-cr, and qepA genes in E. coli isolates

图1 PCR扩增产物琼脂糖凝胶电泳Figure 1 Agarose gel electrophoresis of PCR products
3 讨论
大肠埃希菌是临床常见的感染病原菌,质粒介导的ESBL是其对β内酰胺类抗生素主要耐药机制, 因此ESBL检测对于耐药菌株流行病学调查及医院感染控制非常必要。本次检测大肠埃希菌产ESBL菌株占53.1%,低于同期卫生部全国细菌耐药检测网报告数据(68.2%)和上海市耐药监测网数据(61.3%),与同时期北京协和医院报道53.0%及CHINET细菌耐药监测数据54.0% 基本一致[6-9]。且高于国外报道的11.9%~49.4%[10-12]。
ESBL基因位于质粒上,而质粒可以通过接合、转化、转导、整合等形式在同种或异种细菌间传播,并可携带其他耐药基因, 故产ESBL细菌不仅对青霉素类、头孢菌素类耐药, 而且对氟喹诺酮类、氨基糖苷类及磺胺类药物耐药,从而限制临床抗感染治疗方案的选择,增加抗感染治疗失败的风险。本次研究结果显示,产ESBL菌株对哌拉西林、头孢噻肟、头孢曲松、氨曲南耐药率皆高于90%,对头孢他啶耐药率也高达68.4%,表明这些药物已经不适合作为抗大肠埃希菌感染的经验用药。而耐药率60%~75%的药物为:庆大霉素、环丙沙星、左氧氟沙星和甲氧苄啶-磺胺甲唑,结果与HU等[13]研究产酶菌株耐药数据相比(依次分别为36.7%,80%,83.3%,66.7%),本研究中菌株对庆大霉素耐药率更高,对环丙沙星、左氧氟沙星略低,甲氧苄啶-磺胺甲唑与之较为接近,从而表现出不同国家和地区的差异。本研究菌株对酶抑制剂复合制剂药物中头孢哌酮-舒巴坦和哌拉西林-他唑巴坦耐药率仅为29.5%和24.2%,体现复合制剂有较好的抑制酶活性及抗菌活性。同为氨基糖苷类药物,大肠埃希菌对阿米卡星和庆大霉素耐药率明显不同,国内外数据报道也显示出对阿米卡星耐药率远低于庆大霉素耐药率(5.0% 与 42.6%)[13],( 3.3% 与 36.7%)[14],(12.5% 与65.0%)[15],此可能与阿米卡星和庆大霉素对钝化酶稳定性不同相关。头孢西丁作为头霉素类相对头孢菌素对产ESBL菌株的抗菌活性较强,细菌对其耐药率为28.4%。本次研究出现大肠埃希菌对亚胺培南耐药的菌株,尽管只占0.6%(1/179),但仍要引起重视,需要进一步研究耐药机制,防止耐药株的扩散。
伴随喹诺酮类药物广泛应用,细菌对其耐药率呈现上升趋势,染色体上基因突变和膜渗透性改变导致喹诺酮类药物高水平的耐药,但众多学者对于细菌多重耐药及水平传播现象,尤其对于氟喹诺酮类耐药与产ESBL菌株相关性的方面给予密切关注。在1998年,发现质粒介导可水平传播的qnr基因导致喹诺酮耐药,其机制为qnr基因编码蛋白可结合在喹诺酮药物作用的靶位,从而保护菌体DNA。随后几个新的PMQR基因的耐药机制被发现,包括:aac(6')-Ib-cr基因和qepA基因等。aac(6')-Ib-cr基因编码乙酰转移酶,作用于药物哌嗪酸结构的仲胺,阻止药物乙酰化靶位,而qepA基因编码QepA蛋白为新外排泵,具有降低药物在菌体内积聚作用。虽然这些基因引起细菌对喹诺酮类药物低水平耐药,但其与染色体编码的旋转酶和拓扑异构酶Ⅳ突变具有相关性,而后者可造成高水平氟喹诺酮耐药。
本次研究结果显示:大肠埃希菌携带qnr基因占6.7%(12/179), aac(6')-Ib-cr占14.5%(26/179),与国内JIANG等[4]学者研究数据8.0% 和 9.9% 结果相近,但高于挪威和瑞典地区研究数据,0.85%(3/354),9.0%(32/354)[2],此可能与不同国家抗菌药物应用范围和程度有关。同时实验结果也表明:在产ESBL大肠埃希菌中qnr基因检出率为9.4%(9/95),高于FERJANI等[15]研究结果7.5%,墨西哥地区1.4%( 2/136)[16],西班牙报告的1.1%[17]。研究显示qnrB在qnr基因中占优势。同时本研究aac(6')-Ib-cr基因在产酶菌株中检出率为22.1%(21/95),与其他国家研究数据相比,较高检出率有:墨西哥61.7%(84/136)[16],韩国36.5%,(23/63)[18],巴西里约热内卢44.0%(11/25)[19],日本检出率较低为13.0%(6/46)[20],进而显示出不同地区基因流行特征,表明对本地区耐药基因流行病学调查的重要性。本次观察到产ESBL菌株与非产酶株间,qnr基因的检出结果(9.4%对3.6%)差异无统计学意义(P>0.05),此与其他学者报道不同[2,18,20-21],其原因可能与本次调查区域qnr流行水平较低及研究标本量较少有关; 而aac(6')-Ib-cr基因,两者具有明显的差异(22.1%对5.9%)差异具有统计学意义(P<0.01),表明产 ESBL菌株通过携带aac(6')-Ib-cr基因,在多种抗菌药物压力下,具有更大的进化优势,表现出多重耐药特征。
尽管PMQR基因自身促进低水平喹诺酮耐药,但PMQR基因的流行与拓扑异构酶突变及高水平氟喹诺酮耐药具有相关性[20- 21],可以促进最初敏感的菌株获得高水平的耐药,并由于PMQR基因可与产ESBL的某些基因位于同一质粒上,在菌株及菌种间水平传播[15-16],进而加速了菌株多重耐药的形成和传播。
[1]Clinical and Laboratory Standards Institute .Performance standards for antimicrobial susceptibility testing;twenty-second informational supplement [S]. 2014,M100-S24.
[2]KARAH N, POIREL L, BENGTSSON S, et al. Plasmidmediated quinolone resistance determinants qnr and aac(6' )-Ibcr in Escherichia coli and Klebsiella spp. from Norway and Sweden [J] . Diagn Microbiol Infect Dis, 2010, 66(4): 425-431.
[3]汤电,张小华,付晓平,等. 广东地区鱼源大肠埃希菌 ESBLs 和 PMQR流行分布调查[J]. 华南农业大学学报,2012,33(1):113-119.
[4]JIANG Y, ZHOU Z, QIAN Y, et at. Plasmid-mediated quinolone resistance determinants qnr and aac(6') - Ib-cr in extended- spectrum beta-lactamase-producing Escherichia coli and Klebsiella pneumoniae in China [J]. J Antimicrob Chemother, 2008,61(5):1003-1006.
[5]KIM HB, PARK CH , KIM CJ, et al. Prevalence of plasmidmediated quinolone resistance determinants over a 9-year period [J] . Antimicrob Agents Chemother, 2009,53(2):639-645.
[6]李耘,吕媛,薛峰,等. 卫生部全国细菌耐药监测网(Mohnarin)2011-2012年革兰阴性菌耐药监测报告[J]. 中国临床药理学杂志,2014,30(3):260-277.
[7]胡付品,朱德妹,汪复,等. 2013年上海市细菌耐药性监测[J].中国感染与化疗杂志,2014,14(6):461-473.
[8]张小江,张辉,窦红涛,等.2012年北京协和医院细菌耐药性监测[J].中国感染与化疗杂志,2014,14(2):104-111.
[9]胡付品,朱德妹,汪复,等. 2013年中国CHINET细菌耐药性监测[J].中国感染与化疗杂志,2014,14(5):365-374.
[10]POULOU A, GRIVAKOU E, VRIONI G, et al. Modified CLSI extended- spectrum β-lactamase( ESBL) confirmatory test for phenotypic detection of ESBLs among Enterobacteriaceae producing various β-lactamases [J]. J Clin Microbiol,2014,52 (5):1483-1489.
[11]ZOWAWI HM, BALKHY HH, WALSH TR, et al.β-Lactamase production in key gram-negative pathogen isolates from the Arabian Peninsula [J] . Clin Microbiol Rev, 2013,26(3) :361-380.
[12]FEGLO P, ADU-SARKODIE Y, AYISI L, et al. Emergence of a novel extended- spectrum-β-lactamase( ESBL)-producing,fluoroquinolone-resistant clone of extraintestinal pathogenic Escherichia coli in Kumasi, Ghana [J] . J Clin Microbiol, 2013,51(2):728-730.
[13]HU F, O' HARA JA, RIVERA JI, et al. Molecular features of community-associated extended-spectrum-β- lactamaseproducing Escherichia coli strains in the United States [J] . Antimicrob Agents Chemother,2014,58(11):6953-6957.
[14]MENDES RE, MENDOZA M, BANGA SINGH KK, et al. Regional resistance surveillance program results for 12 Asia-Pacific nations( 2011) [J]. Antimicrob Agents Chemother,2013,57(11):5721-5726.
[15]FERJANI S, SAIDANI M, AMINE FS, et al. Prevalence and characterization of plasmid-mediated quinolone resistance genes in extended-spectrum β-lactamase-producing Enterobacteriaceae in a Tunisian hospital [J] . Microb Drug Resist, 2015,21(2):158-166.
[16]SILVA-SANCHEZ J, BARRIOS H, REYNA-FLORES F, et al. Prevalence and characterization of plasmid-Mediated quinolone resistance genes in extended-spectrum β-lactamase-producing Enterobacteriaceae isolates in Mexico [J] . Microbial Drug Resist, 2011,17( 4): 497-507.
[17]BRIALES A, RODRíGUEZ- MARTíNEZ JM, VELASCO C, et al. Prevalence of plasmid-mediated quinolone resistance determinants qnr and aac(6')-Ib-cr in Escherichia coli and Klebsiella pneumoniae producing extended-spectrum-βlactamases in Spain[J]. Int J Antimicrob Agents, 2012,39(5):431-434.
[18]PARK KS, KIM MH, PARK TS, et al. Prevalence of the plasmid- mediated quinolone resistance genes, aac(6')-Ibcr, qepA, and oqxAB in clinical isolates of extended-spectrum ß-lactamase( ESBL)-producing Escherichia coli and Klebsiella pneumoniae in Korea [J]. Ann Clin Lab Sci, 2012, 42(2):191-197.
[19]PEIRANO G, ASENSI MD, PITONDO-SILVA A. et al. Molecular characteristics of extended- spectrum β-lactamaseproducing Escherichia coli from Rio de Janeiro, Brazil [J] . Clin Microbiol Infect,2011, 17( 7):1039-1043.
[20]ODE T, SAITO R, KUMITA W, et al. Analysis of plasmidmediated multidrug resistance in Escherichia coli and Klebsiella oxytocaisolates from clinical specimens in Japan [J]. Int J Antimicrob Agents, 2009, 34(4): 347-350.
[21]PASOM W, CHANAWONG A, LULITANOND A, et al. Plasmid- mediated quinolone resistance genes, aac(6')-Ib-cr,qnrS,qnrB,and qnrA, in urinary isolates of Escherichia coli and Klebsiella pneumoniae at a teaching hospital, Thailand[J]. Jpn J Infect Dis, 2013, 66( 5): 428-432.
Resistance profile and prevalence of plasmid-mediated quinolone resistance genes in Escherichia coli isolates
PAN Mei, CHEN Shan, LI Wen, LIU Chenbiao, SHI Yuan. (Department of Laboratory Medicine, Affiliated Hospital of Shandong University of Traditional Chinese Medicine, Jinan 250011, China)
Objective To investigate the resistance profile and the prevalence of plasmid-mediated quinolone resistance (PMQR)genes in Escherichia coli isolates. Methods Antimicrobial susceptibility testing was conducted by Kirby-Bauer method. Double disc method was used to detect extended-spectrum Beta-lactamases (ESBLs) phenotypically. The qnrA, qnrB, qnrC, qnrD, qnrS,aac(6' )-Ib, and qepA genes were identified by polymerase chain reaction (PCR). The aac(6' )-Ib-cr gene was confirmed by restriction endonuclease reaction. Results The overall prevalence of ESBLs-producing strains was 53.1% (95/179) in Escherichia coli isolates. The ESBLs-producing strains showed no resistance to meropenem and the lowest resistance rate (1.1%) to imipenem, followed by amikacin, piperacillin-tazobactam, and cefoperazone-sulbactam (16.8%, 24.2%, and 29.5% resistant respectively). More than 70% of the ESBLs-producing strains were resistant to piperacillin, cefotaxime, ceftriaxone, aztreonam, and trimethoprim-sulfamethoxazole,while 60%-70% were resistant to gentamicin, ceftazidime, ciprofloxacin, levofloxacin and levofloxacin. PCR results showed that qnr gene was identified in 9 (9.5%) of the ESBLs-producing E. coli strains, including qnrA in 2 strains, qnrB in 5 strains and qnrS in 2 strains. And qnrB gene was identified in 3 (3.6%) non-ESBLs-producing E. coli strains. Restriction endonuclease reaction revealed that aac(6' )-Ib-cr gene was positive in 21 (22.1%) ESBLspositive strains and 5 (6.0%) ESBLs-negative E. coli strains,respectively. qepA gene was not identified in any isolate. Conclusions ESBLs-producing E. coli showed resistance to multiple antimicrobial agents. aac(6' )-Ib-cr is the primary genotype of PMQR.
Escherichia coli; extended-spectrum betalactamase; plasmid-mediated quinolone resistance; genotype
·论著·
R 378.21
A
1009-7708(2016)01-0015-05
10.16718/j.1009-7708.2016.01.004
山东省科技发展计划项目(2012GGB14100);山东省临床重点专科建设项目(鲁卫医字[2013]26号)。
1. 山东中医药大学附属医院检验科,济南 250011;2. 山东省立医院检验科。
潘玫(1964—),女,医学硕士,副主任技师,主要从事临床病原微生物研究。
潘玫,E-mail: sdjnpm2006@126.com。
2015-06-11
2015-07-31

