猪流感病毒基因池中的新成员:新甲型H1N1流感病毒基因片段的存在及潜在威胁
高灵茜,崔梦一,樊晓晖
猪流感病毒基因池中的新成员:新甲型H1N1流感病毒基因片段的存在及潜在威胁
高灵茜,崔梦一,樊晓晖
新甲型H1N1流感病毒(2009 pandemic H1N1 virus,pdm/09)于2009年在人群中暴发以后,迅速在全球范围内传播,引起了21世纪的第一次流感大流行。pdm/09是由人的流感病毒、禽流感和猪流感病毒(swine influenza virus,SIV)经过重配后形成的病毒,它的基因片段已经进入了猪流感病毒当中并开始产生新的变异毒株,这些新的变异流感毒株在欧亚大陆、北美大陆及中国南部的各个地区被不断报道和发现,这表明猪源性pdm/09在人间流行后可返传给猪,成为猪流感病毒基因池中的固有组成,获得与SIV重组形成新的重配病毒的能力,并可能仍然具有感染人类的潜能。因此,必须关注新型重配病毒的进化:包括其在猪群中的生长适应、以及适应性感染人的进化过程。不仅如此,还必须加强对猪群及人群流感病毒的检测,了解重配病毒在人和猪两个种群中的进化过程。
新甲型H1N1流感病毒;猪流感病毒基因池;基因重配病毒;跨种属传播
流感病毒是流行性感冒的病原体,属于RNA病毒,分为甲乙丙3个型别,最容易发生变异的是甲型。甲型流感病毒的副链RNA有8个节段,每个节段为1个基因,这8个基因至少编码 10种以上蛋白肽: RNA1节段编码 PB2蛋白、RNA2编码 PB1蛋白、RNA3 编码 PA蛋白、RNA4 编码 HA蛋白、RNA5编码 NP蛋白、RNA6 编码 NA蛋白、RNA7编码 M1和M2蛋白、RNA8编码两个非结构蛋白—— NS1和 NS2[1]。甲型流感病毒依据其表面血凝素(HA)和神经氨酸酶(NA)的差异可分成不同的亚型,迄今为止,已有 17种HA亚型(H1~H17),10种NA亚型(N1~N10)被鉴定[1-3],任何一种HA与任何一种NA结合即为一种亚型。能够引起人类感染的亚型包括新甲型H1N1、H2N2、H3N2、H5N1、H5N2、H5N6、H7N2、H7N3、H7N7、H7N9、H9N2和H10N8。历史上H1N1、H2N2和H3N2引起几次流感世界大流行,最近出现的一次流感大流行发生在2009年,由新甲型H1N1流感病毒(2009 pandemic H1N1 virus,pdm/09)引起。
pdm/09在人群中暴发以后,迅速在全球范围内传播,引起了21世纪的第一次流感大流行。pdm/09是由人的流感病毒、禽流感和猪流感病毒(swine influenza virus,SIV)经过重配后形成的病毒[4-5],它不仅能感染人,而且在人群流行之后很快回传至猪群,与SIV进一步发生重配[6-8]。pdm/09的流行和肆虐提示了两个重要问题:流感病毒在人和猪之间的双向穿梭感染频繁的发生;猪作为中间宿主可以推动人源流感病毒和猪源流感病毒的基因片段发生重配并可能产生新的大流行株。而pdm/09的基因片段也已经进入了猪流感病毒当中并开始产生新的变异毒株,这些新的变异流感毒株在欧亚大陆和北美大陆各个地区被不断报道和发现[9-16],并有成为大流行株的可能性。本文拟对重配有pdm/09基因片段的SIV的流行情况和潜在危机做出讨论。
1 pdm/09基因组成及流行情况
在两次流感大范围流行爆发之后,pdm/09已经变为季节性流感毒株之一,(数据分析来源为WHO于2010年8月10日 的报到([http://www.who.int/mediacentre/news/statements/2010/h1n1_vpc_20100810/en/]).但是pdm/09对于流感病毒的流行学和生态学的影响却在逐渐加大。pdm/09是由北美猪流感病毒、北美禽流感病毒、人类甲型流感H1N1亚型病毒和常见于欧亚大陆的猪流感病毒重配产生[5],这些病毒已经在猪群中流行达到10年之久,是常见且稳定的SIV基因池来源[17]。在2009年pdm/09爆发之前,世界范围内猪群中存在稳定流通的SIV是 以基因重配H1N2亚型、三重重配H3N2亚型(triple reassortant,TR)和欧亚类禽H1N1亚型(European avian-like,ER)为主[18-22],即SIV基因池中三大供给亚型。而三重重配H3N2亚型病毒构成了pdm/09除了NA和M基因之外的其他6个基因的主要来源。1979年欧洲一株禽源H1N1病毒进入猪群中流通并成为了稳定的来源,被命名为欧亚类禽H1N1[23-25],而这株病毒是pdm/09的NA和M基因的主要来源。
可见pdm/09是一种典型的基因重配病毒,一直以来,基因重配学说是被流感界广泛接受的一个学说,该学说认为流感病毒新亚型大流行株是人、禽、猪流感病毒通过基因重配而来,而猪是发生基因重配的重要场所。猪气管存在能与禽流感病毒特异性结合的唾液酸α-2,3Gal受体,又存在能与人流感病毒特异性结合的唾液酸α-2,6Gal受体,禽流感病毒与人流感病毒均可感染猪,再加上SIV,3个不同来源的病毒易通过基因重配形成新病毒株,由于人群中普遍缺乏对新产生的重配病毒株的免疫力,一旦该病毒在猪中通过适应性的变化而具备有效的人传人能力,将有可能导致流感的大爆发,故猪被认为是“基因混合器“,而SIV的流行情况和基因组成更是需要得到关注和长时间监测。
基因重配的病毒也是成为大流行株的重要条件,大量证据证明,基因重配的流感病毒具有明显的选择优势,传播能力显著增强,从1957年的亚洲流感到1968年香港流感,再到2009年的pdm/09,都是由基因重配的病毒株所引发。在pdm/09爆发后的很短时间内已经可以检测到其从人传入猪[6-8,26-28],并在猪中不断穿梭重配,即与SIV重组形成新的重组病毒,并可能仍然具有感染人类的潜能。因此,必须关注这一重组病毒株的进化包括在猪群中的生长适应、以及适应感染人的进化过程。特别的是,这些基因重配都是以pdm/09内部基因的进入为主,而表面基因只短暂或偶尔在重配病毒株中发现,但这种趋势在逐步增大。对带有pdm/09内部基因的重配毒株遗传分析和系统进化研究表明pdm/09的基因片段已经开始取代现有欧亚大陆和北美SIV的基因来源(欧亚类禽H1N1、基因重配H1N2和三重重配H3N2病毒),从根本上改变了SIV的病毒池[18-19]。因此必须加强对猪群及人群流感病毒的检测,了解重组病毒在人和猪两个种群中的进化过程。而这种不同来源的内部基因和表面基因的重配组合是否意味着新一轮流感病毒重配的趋势,还值得我们进一步思考。
2 H1N1亚型重配毒株
在2009年4月pdm/09暴发后的不到一年时间内,D. Vijaykrishna等人[14]于2010年2月在对香港猪屠宰场的采样中发现了一株重配有pdm/09基因片段的H1N1亚型的SIV,通过对同一头猪进行血液,咽拭子和鼻腔拭子3份标本的采取,分离得到一株H1N1亚型的SIV—A/swine/Hong Kong/201/2010,遗传进化分析表明该毒株的一个表面基因NA来源于pdm/09,另一个表面基因HA则来源于欧亚类禽H1N1,其他6个内部基因的来源是三重重配H3N2,可见pdm/09进入到SIV和其他SIV发生基因重配的速度非常迅速,以先建立稳定传播,再进行机会性重配的模式寻找扩大化的感染机会。这也是最早的关于pdm/09和其他SIV发生重配新亚型毒株的报到。很快,在这之后3个月,Huachen Zhu等人[29]也在流感监测网络中筛查到了重配有pdm/09内部基因和欧亚类禽H1N1表面基因的重配病毒A/swine/Guangdong/1361/2010,且毒株来源达到了10个样品,说明有pdm/09基因片段的重配病毒有良好的猪群内部传播能力,事实也证明了这株病毒在猪和猪之间、猪和雪貂之间存在传播能力并可以在人肺组织进行有效复制,进一步提升重配有pdm/09基因片段的SIV感染人的能力和危险性。更重要的是,带有欧亚类禽H1N1亚型HA基因的病毒在近30年的时间内几乎很少感染到人,所以人群中普遍对于这个基因存在免疫缺陷或免疫空白,推测如果带有这个基因的重配病毒在获得pdm/09所赋予的传播能力进而感染到人,人群由于免疫缺陷就可能发生大规模的流行,可见猪群中出现带有欧亚类禽H1N1和pdm/09基因片段的重配病毒,并伴随大量多重病毒感染的情况不容小觑。在欧洲,Elke Starick[13]等人也于2010年在德国分离到一株重配病毒A/swine/Germany-NRW/R708/10,该病毒的HA和其他6个内部基因的来源是pdm/09,NA基因来源于欧洲类禽H1N1,进入2011年之后,全球各地关于重配有pdm/09基因片段的H1N1新亚型病毒的报道更加密集,地点也呈现散发状态,泰国、日本等地[11,13]均有出现,更加说明pdm/09在猪群中发生重配的范围已经有在全世界扩散甚至大量存在的可能性。
3 H1N2亚型重配病毒
H1N2亚型同样是SIV中常见的基因型,其来源有人源H1N2,基因重配H1N2和三重重配H3N2病毒,于1994年在英国最先被发现被开始逐步确立其在猪群中的传播[30,31],最终成为SIV基因池中固有的基因来源,自从pdm/09爆发之后,H1N2也在发生变化。 2010年在德国发现了可以确立种系稳定传播的H1N2亚型重配病毒,并且从第一株重配病毒A/sw/Papenburg/IDT12653/2010(NA来源于三重重配H3N2亚型,其余7个基因来源于pdm/09)的检出开始,一直持续到2012年12月,H1N2亚型重配病毒在德国北部的猪群中的检出率大大增长,Ana Moreno[12]等人在2010年5月通过对意大利境内某养猪场中出现有呼吸系统问题的猪的肺组织匀浆进行荧光定量PCR检测,而非传统的咽拭子和血清学鉴定,发现了呈甲型流感病毒和pdm/09双重阳性的流感病毒,进而筛选出一株H1N2亚型重配毒株A/Sw/It/116114/2010,该病毒除了表面基因NA之外其余的7个基因均来自pdm/09。两株新型重配病毒的发现遵循了相似的基因组成,可以认为是pdm/09在欧洲猪群中建立稳定传播后,与其他病毒的初步重配行为。几乎同时(2010年4月),同样在欧洲的英国,Howard[9]等发现了命名为A/swine/England/1382/10的H1N2重配毒株,其基因组成变化为除HA和NA之外所有6个内部基因均来源于pdm/09,说明pdm/09与其他病毒重配的多样性在逐步增加。在美洲,情况也不容乐观,美国发现的两株H1N2重配毒株[32](A/swine/Ohio/FAH10-1/2010 和A/swine/Ohio/FAH11-1/2010)的基因组成非常有潜在的威胁性,其HA和NA基因的来源更倾向于类人季节性H1N2流感病毒,特别是HA基因在系统进化重要氨基酸残基位点96A、133S和183P及抗原决定簇位点91Y、134A、180H、187N和 225G[33]上与人流感病毒保持了高度的一致性。相对保守稳定的NP和M基因则来源于pdm/09,导致这两株病毒获得了较高的关注度,直接把重配有pdm/09基因片段的猪流感病毒传播到人的可能性提升到较高程度。而且有趣的是与其他国家和地区的重配趋势不同,美国发现的至少6株H1N2和3株H3N2重配病毒更倾向于持有来源于pdm/09的M基因[10],而非其他地区的6个内部基因或者2个表面基因,其揭示的不同重配趋势是否有其他的可能性,值得继续关注。亚洲地区在中国、日本、韩国均有发现H1N2重配毒株[15,34-35],其中中国发现的2株H1N2重配毒株中分别来自广西及浙江,结合后面将阐述的H3N2亚型重配毒株的发现地区,可以说明重配病毒的存在出现了有倾向性的地域问题,而且两株病毒的发现地相距千里却显示出99.6%~100%的高度遗传进化相似性(PB2、 PB1、 PA、HA、NP、NA、 M和NS基因),提示它们在有共同祖先的前提下,某些陆禽鸟类(鹧鸪、鹌鹑、火鸡等)在流感病毒的传播和基因交流上发挥着某些不可忽视的重要作用[36-37],对于这些陆禽鸟类的流感病毒受体分布以及不同流感病毒感染后可以作为“放大器”的特点,都非常值得进一步关注。
4 H3N2亚型重配病毒
SIV基因池中的第3种供给是H3N2亚型流感病毒,2011年,在美国出现的带有pdm/09片段的重配毒株H3N2亚型至今已有超过340个人感染[38],即H3N2重配毒株感染人的情况已经发生。2009年8月—2012年9月,笔者所在课题组和香港大学联合的课题组在香港、广东及广西得到的37 213份猪呼吸道(鼻粘膜和气管)拭子标本中分离到了387株流感病毒,其中鉴定为H3N2亚型的有94株,广西地区的分离率为0.75%。值得注意的是,我们于2010年10月首次在国内分离到了重组有pdm/09内部基因片段的H3N2 SIV(SW/GX/NS2783/10,SW2783)[8],随后2011年7—8月间在香港和广东也分离到类似的重组有pdm/09内部基因片段的H3N2,后者在广东和香港两地的分离率达到24.3%[10]。通过对SW2783广西分离株等毒株的遗传进化分析,发现广西病毒株与一些越南H3N2三重重组病毒(triple reassortant,TR,是在2000—2007年间由人季节性H3N2病毒进入猪群后,经过三次重配形成的)具有同一宗支来源。更值得关注的是越南研究同行从一名两岁越南女孩体内分离得到一株H3N2毒株(Vietnam/302/11)(http://thanhniennews.com/health/vietnam-reports-first-human-case-of-h3n2-swine-flu-8302.html),其基因序列和中国广西分离到的H3N2 SIV高度相关。这提示在广西分离得到的重组有pdm/09基因片段的H3N2 SIV可能来自越南,而且已具备了感染人的能力。
如前文提到,禽流感病毒在作为“基因混合器”的猪体内长期不断的增殖复制,逐渐改变其对受体的特异性,即同时特异性结合唾液酸α-2,3Gal和α-2,6Gal受体到倾向于结合唾液酸α-2,6Gal受体(人以此受体为主)而能感染人,提示禽流感病毒在向逐渐适应人体的方向进化[39]。同样道理,重组有pdm/09基因片段的H3N2 SIV也可通过在作为潜在“基因混合器”的人体内长期不断的增殖复制,通过改变其对受体的特异性及其他特性,从而变得更加适应于人群。
5 pdm/09基因片段产生的威胁
2009年到2012年间,笔者所在课题组和香港大学合作分离得到的387份SIV中,有60%的病毒带有pdm/09基因片段,并形成了至少17种不同的重配基因型。对这些重配毒株进行分析后发现:pdm/09的PB2、PB1、PA、NP、M和NS等6个内部基因大量存在于重配毒株中(表1)。这一发现说明来自于人大流行毒株的pdm/09基因片段,可能有利于猪源流感病毒的生存、适应和传播。由于这些新基因型的毒株同时具有猪和人病毒基因,因此在两种宿主中都有可能复制、感染,甚至进一步传开。这对流感病毒的进化具有深远的影响,并有可能对人类构成更多样性的威胁。
表1广西和香港分离的3株带有新甲型H1N1流感病毒的H3N2重组型猪流感病毒
Tab.1Three Guangxi or Hong Kong reassortment H3N2 swine influenza viruses carrying genes from the pandemic H1N1/2009
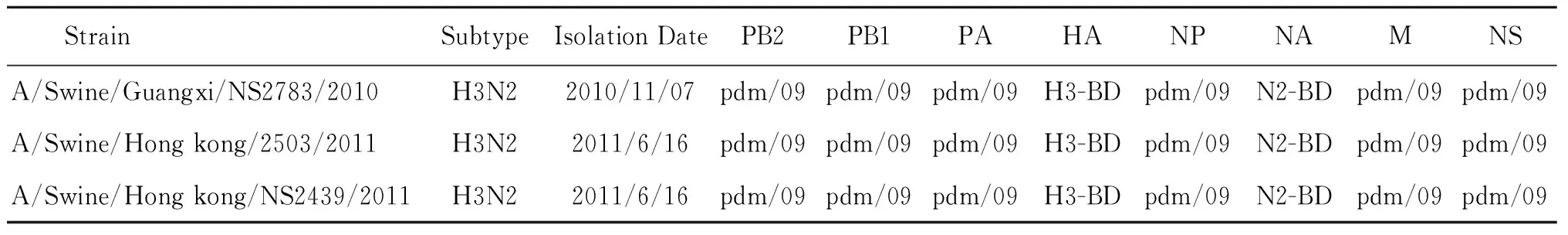
StrainSubtypeIsolationDatePB2PB1PAHANPNAMNSA/Swine/Guangxi/NS2783/2010H3N22010/11/07pdm/09pdm/09pdm/09H3-BDpdm/09N2-BDpdm/09pdm/09A/Swine/Hongkong/2503/2011H3N22011/6/16pdm/09pdm/09pdm/09H3-BDpdm/09N2-BDpdm/09pdm/09A/Swine/Hongkong/NS2439/2011H3N22011/6/16pdm/09pdm/09pdm/09H3-BDpdm/09N2-BDpdm/09pdm/09
注:pdm/09: pandemic H1N1/2009 lineage ,2009年引起世界大流行的新甲型H1N1流感病毒;H3-BD,N2-BD: A/Swine/Binh Duong/03_06/10-like H3N2 lineage,2010年在越南Binh Duong分离到的H3N2三重重组猪流感病毒
考虑到在亚洲(中国东南部)已经发现了H5N1和H9N2亚型流感病毒在猪群中存在流通的情况[40],这些禽源的流感病毒及其基因通过感染猪获得哺乳动物的适应性,那么出现禽源和pdm/09或带有pdm/09片段的重配毒株发生进一步重配的可能性就时刻存在。我们必须警惕、避免和干预重组有pdm/09基因片段的H3N2 SIV和禽流感病毒(H5N1、H7N9等能感染哺乳动物和人的病毒)的进一步重配,尤其是经过长期适应后的pdm/09重组病毒或H3N2 SIV,可能以一种更能适应人体生长的方式,为禽流感病毒提供有效的哺乳动物适应性基因。目前已经知道,禽流感病毒可以通过所编码蛋白的单个或多个氨基酸的突变,增强其对哺乳动物的致病性,例如HA基因的多个碱性氨基酸裂解位点的存在是禽流感病毒最重要的分子毒力基础。除此之外,HA影响病毒毒力的因素大多与宿主的唾液酸α-2,3Gal和α-2,6Gal受体有关,因为它们主要决定了病毒的宿主范围和组织亲嗜性。对于H5N1禽流感病毒来说,L129V/A134V、N182/KQ222L/G224S、S227N、182/192、G228S这些位点的突变,能增强病毒对α-2,6Gal受体的结合能力,而对于H1和H3亚型流感病毒,影响受体结合特性最重要的因素在190至225区域(H1 numbering)以及226和228区域(H3 numbering)。对于pdm/09而言,D222G的变化使其受体结合特性从α-2,6Gal受体转变为结合α-2,3Gal和α-2,6Gal两种受体。那么文中所总结的重配有pdm/09基因片段的新SIV病毒,其HA是否也遵循上述这些规律或者有新的变化可循,值得进一步探讨。
中国具有最为多样和复杂的SIV流感生态学和基因库,长期研究只揭示了欧洲类禽和三重重配病毒在猪中存在流通,是SIV基因池的重要供给病毒,直到2009年流感大暴发之后,才在中国猪群中发现了带pdm/09基因片段的重配毒株[29],这些重配毒株的发现引起了注意,前期对这些重配毒株分析无不证明pdm/09基因片段进入猪群并建立了稳定大量的传播(例如带pdm/09基因片段的重配毒株比带TR或者EA片段的重配病毒的基因多样性的水平低,说明其专注在猪群传播),以往的经验说明带有pdm/09片段的毒株都有成为大流行株的潜力,那么前期研究中发现的这些重配毒株是否具有传染人的威胁?或者pdm/09基因片段的出现是否意味着新的流感大暴发有可能在中国发生?均是考虑的重点,可以通过对这些重配毒株适应性感染实验后的遗传进化分析来探讨pdm/09在中国猪群基因池的地位及可能存在的威胁,还可以探讨感染后某些位点的变化是否具有标记性作用,进而为有成为大流行株潜力的毒株提供某些“分子证明”。
[1] Webster RG,Bean WJ,Gorman OT,et al. Evolution and ecology of influenza A viruses[J]. Curr Topics Microbiol Immunol,1992,385(9): 235-235. DOI: 10.1111/j.1751-0813.1992.tb09940.x
[2] Murphy BR. Orthomyxoviruses[J]. Fields Virol,1996: 1397-1445.
[3] Fouchier RAM,Vincent M,Anders W,et al. Characterization of a novel influenza A virus hemagglutinin subtype (H16) obtained from Black-Headed Gulls[J]. J Virol,2005,79(5): 2814-2822. DOI: 10.1128/jvi.79.5.2814-2822.2005
[4] Garten RJ,Davis CT,Russell CA,et al. Antigenic and genetic characteristics of swine-origin 2009 A(H1N1) influenza viruses circulating in humans[J]. Science,2009,325(5937): 197-201. DOI: 10.1126/science.1176225
[5] Smith GJD,Dhanasekaran V,Justin B,et al. Origins and evolutionary genomics of the 2009 swine-origin H1N1 influenza A epidemic[J]. Nature,2009,459(7250): 1122-1125. DOI: 10.1038/nature08182
[6] Howden KJ,Brockhoff EJ,Caya FD,et al. An investigation into human pandemic influenza virus (H1N1) 2009 on an Alberta swine farm[J]. Canad Vet J La Revue Veterinaire Canadienne,2009,50(11): 1153-1161.
[7] Ariel P,Javier C,Quiroga MA,et al. Pandemic (H1N1) 2009 outbreak on pig farm,Argentina[J]. Emerg Infect Dis,2010,16(2): 304-307. DOI: 10.3201/eid1602.091230
[8] Weingartl HM,Yohannes B,Tamiko H,et al. Genetic and pathobiologic characterization of pandemic H1N1 2009 influenza viruses from a naturally infected swine herd[J]. J Virol,2009,84(5): 2245-2256. DOI: 10.1128/JVI.02118-09
[9] Howard WA,Essen SC,Strugnell BW,et al. Reassortant pandemic (H1N1) 2009 virus in pigs,United Kingdom[J]. Emerg Infect Dis,2011,17(6): 1049-1052. DOI: 10.3201/eid/1706.101886
[10] Ducatez MF,Ben H,Evelyn SR,et al. Multiple reassortment between pandemic (H1N1) 2009 and endemic influenza viruses in pigs,United States[J]. Emerg Infect Dis,2011,17(9): 1624-1629. DOI: 10.3201/eid1709.110338
[11] Kitikoon P,Sreta D,Ayudhya SNN,et al. Brief report: Molecular characterization of a novel reassorted pandemic H1N1 2009 in Thai pigs[J]. Virus Genes,2011,43(1): 1-5. DOI: 10.1007/s11262-011-0597-5
[12] Moreno A,Di Trani L,Faccini S,et al. Novel H1N2 swine influenza reassortant strain in pigs derived from the pandemic H1N1/2009 virus[J]. Vet Microbiol,2011,149(3/4): 472-477. DOI: 10.1016/j.vetmic.2010.12.011
[13] Elke S,Elke L,Sasan F,et al. Reassorted pandemic (H1N1) 2009 influenza A virus discovered from pigs in Germany[J]. J General Virol,2011,92(3): 1184-1188. DOI: 10.1099/vir.0.028662-0
[14] Vijaykrishna D,Poon LLM,Zhu HC,et al. Reassortment of pandemic H1N1/2009 influenza a virus in swine[J]. Science,2010,328(5985): 1529. DOI: 10.1126/science.1189132
[15] Kirisawa R,Ogasawara Y,Yoshitake H,et al. Genomic reassortants of pandemic A (H1N1) 2009 virus and endemic porcine H1 and H3 viruses in swine in Japan[J]. J Vet Med Sci,2014,76: 1457-1470. DOI: 10.1292/jvms.14-0194
[16] Pravina K,Vincent AL,Gauger PC,et al. Pathogenicity and transmission in pigs of the novel A(H3N2)v influenza virus isolated from humans and characterization of swine H3N2 viruses isolated in 2010-2011[J]. J Virol,2012,86(12): 6804-6814. DOI: 10.1128/JVI.00197-12
[17] Zhu H,Webby R,Lam TTY,et al. History of swine influenza viruses in Asia[J]. Curr Topics Microbiol Immunol,2011,370: 57-68. DOI: 10.1007/82_2011_179
[18] Lorusso A,Vincent AL,Gramer MR,et al. Contemporary epidemiology of North American lineage triple reassortant influenza A viruses in pigs[J]. Curr Topics Microbiol Immunol,2011,370: 113-131. DOI: 10.1007/82_2011_196
[19] Webby RJ,Swenson SL,Krauss SL,et al. Evolution of swine H3N2 influenza viruses in the United States[J]. J Virol,2000,74(18): 8243-8251. DOI: 10.1128/jvi.74.18.8243-8251.2000
[20] Brown IH. The epidemiology and evolution of influenza viruses in pigs[J]. Vet Microbiol,2000,74(1): 29-46. DOI: 10.1016/s0378-1135(00)00164-4
[21] Naomi K,Jodie MV,Robert H,et al. A historical perspective of influenza A(H1N2) virus[J]. Emerg Infect Dis,2014,20(1): 6-12. DOI: 10.3201/eid2001.121848
[22] Karasin A,Olsen CG. Genetic characterization of an H1N2 influenza virus isolated from a pig in Indiana[J]. J Clin Microbiol,2000,38(6): 2453-2456.
[23] Brown IH. History and epidemiology of swine influenza in Europe. Swine Influenza[M]. Berlin: Springer. 2013:133-146. DOI: 10.1007/82_2011_194
[24] Castrucci MR,Donatelli I,Sidoli L,et al. Genetic reassortment between avian and human influenza A viruses in Italian pigs[J]. Virology,1993,193(1): 503-506. DOI: 10.1006/viro.1993.1155
[25] Ludwig S,Stitz L,Planz O,et al. European swine virus as a possible source for the next influenza pandemic?[J]. Virology,1995,212(2): 555-561. DOI: 10.1006/viro.1995.1513
[26] Welsh MD,Baird PM,Guelbenzu-Gonzalo MP,et al. Initial incursion of pandemic (H1N1) 2009 influenza A virus into European pigs[J]. Vet Rec,2010,166(21): 642-645. DOI: 10.1136/vr.4851
[27] Deng YM,Iannello P,Smith I,et al. Transmission of influenza A(H1N1) 2009 pandemic viruses in Australian swine[J]. Influenza & Other Respir Viruses,2012,6(3): e42-e47. DOI: 10.1111/j.1750-2659.2012.00337.x
[28] Min-Suk S,Jun Han L,Pascua PNQ,et al. Evidence of human-to-swine transmission of the pandemic (H1N1) 2009 influenza virus in South Korea[J]. J Clin Microbiol,2010,48(9): 3204-3211. DOI: 10.1128/jcm.00053-10
[29] Huachen Z,Boping Z,Xiaohui F,et al. Novel reassortment of Eurasian avian-like and pandemic/2009 influenza viruses in swine: infectious potential for human[J]. J Virol,2011,85(20): 10432-10439. DOI: 10.1128/jvi.05352-11
[30] Brown IH,Chakraverty P,Harris PA,et al. Disease outbreaks in pigs in Great Britain due to an influenza A virus of H1N2 subtype[J]. Vet Rec,1995,136(13): 328-329. DOI: 10.1136/vr.136.13.328
[31] Brown IH,Harris PA,Mccauley JW,et al. Multiple genetic reassortment of avian and human influenza A viruses in European pigs,resulting in the emergence of an H1N2 virus of novel genotype[J]. J General Virol,1998,79 (pt 12)(3): 2947-2955. DOI: 10.1099/0022-1317-79-12-2947
[32] Ali A,Khatri M,Wang L,et al. Identification of swine H1N2/pandemic H1N1 reassortant influenza virus in pigs,United States[J]. Vet Microbiol,2012,158(1/2): 6068. DOI: 10.1016/j.vetmic.2012.02.014
[33] Alessio L,Vincent AL,Harland ML,et al. Genetic and antigenic characterization of H1 influenza viruses from United States swine from 2008[J]. J General Virol,2011,92(4): 919-930. DOI: 10.1099/vir.0.027557-0
[34] Chuanling Q,Liping L,Huanliang Y,et al. Novel triple reassortant H1N2 influenza viruses bearing six internal genes of the pandemic 2009/H1N1 influenza virus were detected in pigs in China[J]. J Clin Virol,2014,61(4): 529-534. DOI: 10.1016/j.jcv.2014.10.014
[35] Han JY,Park SJ,Kim HK,et al. Identification of reassortant pandemic H1N1 influenza virus in Korean pigs[J]. J Microbiol Biotechnol,2012,22(5): 699-707. DOI: 10.4014/jmb.1106.05062
[36] Yang L,Zhang ZF,Fan XH,et al. Distribution characteristics of avian influenza virus and human influenza virus sialic acid receptors in chukar[J]. Chin J Zoonoses,2014,30(6): 545-550. DOI: 10.3969/cjz.j.issn.1002-2694.2014.06.001 (in Chinese)
杨利,张增峰,樊晓晖,等. 鹧鸪禽流感病毒和人流感病毒唾液酸受体分布特征[J]. 中国人兽共患病学报,2014,30(6): 545-550.
[37] Zhang ZF,Luo DZ,Fan XH,et al. Distribution characteristics and roles of sialic acid receptor of quail in the ecosystem of influenza A virus[J]. Guangxi Med J,2010,32(11): 1313-1317. DOI: 10.3969/j.issn.0253-4304.2010.11.001 (in Chinese)
张增峰,罗殿中,樊晓晖. 鹌鹑流感病毒SA受体的分布特点及其在流感病毒生态系统中的作用[J]. 广西医学,2010,32(11): 1313-1317.
[38] Jhung MA,Epperson S,Biggerstaff M,et al. Outbreak of variant influenza A(H3N2) virus in the United States[J]. Clin Infect Dis,2013,57(12): 1703-1712. DOI: 10.1093/cid/cit649
[39] Makarova NV,Ozaki HH,Webster RG,et al. Replication and transmission of influenza viruses in Japanese quail[J]. Virology,2003,310(1): 8-15. DOI: 10.1016/s0042-6822(03)00094-1[40] Peiris JS,Guan Y,Markwell D,et al. Cocirculation of avian H9N2 and contemporary "human" H3N2 influenza A viruses in pigs in southeastern China: potential for genetic reassortment?[J]. J Virol,2001,75(20): 9679-9686. DOI: 10.1128/jvi.75.20.9679-9686.2001
The new member of the swine influenza virus gene pool: the existence of the 2009 pandemic H1N1 virus gene fragments and potential threats
GAO Ling-xi,CUI Meng-yi,FAN Xiao-hui
(DepartmentofMicrobiology,SchoolofPreclinicalMedicine,GuangxiMedicalUniversity,Nanning530021,China)
The pandemic 2009 H1N1 influenza virus (pdm/09) has caused the 2009 pandemic and the first influenza pandemic of the 21st century. It was a swine-origin virus that was generated via multiple reassortments of the swine,human and avian influenza viruses. Pdm/09 can not only infect human but also transmit back into swine and further reassortments with the enzootic swine influenza virus (SIV). New gene reassortment viruses with gene segment of pdm/09 and other SIVs have repeated occurred in Eurasia,North America and southern China,which indicate swine-origin pdm/09 can transmit back into swine after human epidemic and become one of composition in the swine influenza virus gene pool. So the gene reassortment viruses may still have the potential to infect humans. Therefore,we must focus on the evolution of the new gene reassortment viruses,including swine epidemic and adaptable infect humans. Not only that,it need to strengthen the detection of influenza viruses in swine and human,to understand the evolution and cross-species infection of new reassortment viruses in both human and swine.
2009 pandemic H1N1 virus; swine influenza virus gene pool; gene ressortment viruses; cross-species infection
Fan Xiao-hui,Email: fanxiaohui63@163.com
10.3969/j.issn.1002-2694.2016.09.013
樊晓晖,Email:fanxiaohui63@163.com
广西医科大学微生物学教研室,南宁530021
R373.1
A
1002-2694(2016)09-0832-06
2015-12-21;
2016-04-19
广西科学研究与技术开发计划(No.桂科合1347004-27),广西研究生教育创新计划(No.YCBZ2014027),广西医科大学青年科学基金项目(No. GXMUYSF201525)以及“广西特聘专家”专项经费联合资助
Supported by the Guangxi Scientific Research and Technological Development Fund (No. 1347004-27),the Guangxi Graduate Education Innovation Fund (No. YCBZ2014027),the Guangxi Medical Youth Science Fund (No. GXMUYSF201525) and
"Guangxi Distinguished experts" Fund

