基于高通量测序技术的实验动物沙门氏菌检测方法的建立与评价
胡毅翔,张欢欢,余陈欢,金晓音,应华忠
(浙江省医学科学院 浙江省实验动物与安全性研究重点实验室, 杭州 310013)
技术方法
基于高通量测序技术的实验动物沙门氏菌检测方法的建立与评价
胡毅翔,张欢欢,余陈欢,金晓音,应华忠
(浙江省医学科学院 浙江省实验动物与安全性研究重点实验室, 杭州 310013)
目的 建立基于高通量测序技术的实验动物沙门氏菌检测方法,应用于实验动物沙门氏菌的检测。方法 提取小鼠粪便DNA样本,分别针对16S rDNA区域、23S rDNA区域、16S~23S rDNA IS、23S~5S rDNA IS、gyrB优选区域设计通用引物,对各个引物进行测试分析,优化扩增条件并建库,利用Illumina高通量测序技术检测区分42个样本中的沙门氏菌,评价该方法的特异性和稳定性。结果 筛选发现沙门氏菌的菌种分类优选区域为gyrB基因,gyrB基因引物序列为FP5’-AACCACCGCAATCAGACCTT3’,FP5’-AGCCACGAAACCTTCACYA-3’。对引物进行优化,确定最佳扩增条件及样品上样量并正式建库,通过高通量测序和序列分析能检测出42个样品中极微量的沙门氏菌,检测方法稳定,灵敏度高,检测限可达0~102的cfu。结论 本实验利用高通量测序技术,建立了一套完整检测实验动物沙门氏菌的生物体系,能检测出实验动物体内极微量的沙门氏菌,检测方法稳定性好,灵敏度高,可沿用至其他种类病原微生物的检测。
沙门氏菌;实验动物;高通量测序
沙门氏菌(Salmonellae)是一类可以引起沙门氏菌病的典型人畜共患病原菌,属肠杆菌科 (Enterobacteriaceae),兼性厌氧型革兰氏阴性菌[1,2]。自1885年Salmon等分离得到猪霍乱沙门氏菌以来,目前已检出沙门氏菌血清型约2 500余种,其中我国发现的100多种[3]。沙门氏菌在分类学上被分为两个种,分别为Salmonellaenterica和Salmonellabongori,可由多种途径向人类进行传播,进入人体后在肠道内寄生并繁殖,所产生的沙门氏菌内毒素能进入血液循环,引发肠道黏膜肿胀、脱落、甚至溃疡、坏死[4]。在实验室研究方面,携带或者潜在携带沙门氏菌的实验动物不仅对实验人员造成潜在的传染隐患,也会对后续科学实验产生较大的影响,因此对于实验动物的沙门氏菌的检测尤为重要[5]。
生化方法和免疫学方法是目前检测沙门氏菌的主要方法,但这类方法检验程序复杂繁琐,耗时长,部分检验过程及结果判定存在实验人员的主观判断,因此新的检测方法的引入势在必行[6]。分子生物学方法具有快速、准确的优势,现已开始被大量的应用于检测病原微生物,主要涉及到的分子生物学技术主要包括PCR技术、基因芯片和环介导等温扩增技术(LAMP)等[7,8]。近年来,高通量测序技术开始引入病原微生物检测领域,实现了更为高效的检测手段。本次研究主要采用的是Illumina的Miseq高通量测序平台,对于病原微生物检测该平台具有周期短、通量高、成本低、准确率高等优点[9,10]。本实验以分子生物学技术方法为基础,采用Miseq高通量测序技术检测实验室用小鼠所携带的沙门氏菌,通过本次研究,建立了一套完整检测实验动物沙门氏菌的技术体系。
1 材料和方法
1.1 动物
SPF级ICR小鼠42只,体重18~20 g,由浙江省医学科学院实验动物中心提供 (实验动物生产许可证号为 SCXK(浙) 2008-0034,使用许可证号为SYXK (浙) 2008-0114。
1.2 菌株
鼠伤寒沙门氏菌冻干株(编号: TA98),甲型副伤寒沙门氏菌冻干株(编号: CMCC(B)50093),肠炎沙门氏菌冻干株(编号: TDF918),志贺菌冻干株(编号: TG212)为本实验室冷冻保存。
1.3 药品和试剂
PremixExTaqTMHot Start Version(日本Takara公司,批号RR030A),DL2000 DNA Marker(日本Takara公司,批号3427),Axygen PCR Clean Up Kit(美国Axygen公司,批号AP-PCR-500),QubitdsDNA HS Assay Kit(美国Invitrogen公司,批号Q32851),PrimeScript® RT reagent Kit(日本Takara公司, 批号DRR037A)。高通量测序试剂盒(Illumina,美国),Trizol reagent(Invitrogen,美国,批号Q35438)。
1.4 主要仪器
Bioanalyzer 2100 生物分析仪(Agilent,美国);Hiseq 2500 高通量测序仪(Illumina,美国);荧光定量 PCR 仪(Bio-Rad,美国);Qubit 2.0核酸定量仪(Life Technologies,美国);组织研磨仪(TissueLyser,Qiagen,德国)。
1.5 实验方法
1.5.1 小鼠沙门氏菌模型的建立及样品收集: 小鼠适应性饲养3 d后,随机分为6组: 鼠伤寒沙门氏菌组(5只)、甲型副伤寒沙门氏菌组(5只)、志贺杆菌组(5只)、肠炎沙门氏菌和甲型副伤寒沙门氏菌(5只)、肠炎沙门氏菌组(18只)、阴性对照组(4只)。鼠伤寒沙门氏菌组每只小鼠灌服鼠伤寒沙门氏菌,甲型副伤寒沙门氏菌组每只小鼠灌服鼠伤寒沙门氏菌0.2 mL (5×103cfu/ mL),志贺杆菌组每只小鼠灌服鼠伤寒沙门氏菌0.2 mL (5×103cfu/ mL),肠炎沙门氏菌和甲型副伤寒沙门氏菌每只小鼠灌服肠炎沙门氏菌和甲型副伤寒沙门氏菌0.2 mL (5×103cfu/ mL),肠炎沙门氏菌组每只小鼠灌服鼠伤寒沙门氏菌0.2 mL(0 cfu/mL、5×101cfu/mL、5×102cfu/mL、5×103cfu/mL、5×104cfu/mL、5×105cfu/mL),每个浓度各3只,各组小鼠每隔2周灌胃1次,连续3次。取60 mg小鼠粪样置于2 mL离心管中,采用PowerFecal DNA Isolation Kit 试剂盒(MoBio,美国) 抽提100 mg小鼠粪便样本中微生物的总 DNA, 操作步骤按照说明书进行,完成基因组DNA抽提后,放-20℃保存备用,样品信息见表1。

表1 样品信息
1.5.2 菌种分类优选区域选择: 通过文献查找,针对沙门氏菌,寻找在研究范围内的几个菌种相互之间差异变化大的区域。NCBI数据库中查找沙门氏菌典型菌株,以鼠伤寒沙门氏菌(Salmonellaentericaserovar Typhimurium)典型菌株LT2、ST4/74等作为研究对象。通过序列比对,去除经比对菌种间同源关系极高的区域,最终筛选得到5大类区域: ①16S rDNA区域,②23S rDNA区域,③16S~23S rDNA ISR,④23S~5S rDNA ISR,⑤gyrB优选区域。
1.5.3 通用引物设计及优化: 针对①16S rDNA区域,②23S rDNA区域③16S~23S rDNA ISR,④23S~5S rDNA ISR,⑤gyrB优选区域设计通用引物,通用引物在多个菌种中都能PCR扩增得到产物。对设计的引物进行引物测试,淘汰不能扩增的引物。在原有的通用引物基础上加上建库和测序需用到的接头序列(Linker),区分样本使用的正反向Barcode序列(Index),增加终文库均一性和丰富度的连接碱基(heterogeneity spacer),如此设计的引物长度在90 bp~97 bp之间,该优化引物可直接用于建库,实际的建库工作需要一步PCR反应完成。优化引物设计图如图1所示:

图1 优化引物结构组成图Fig.1 Diagram of optimization of the primer structure
1.5.4 沙门氏菌建库PCR条件摸索: 建库PCR采用的是90~97 bp长度的长链引物,这与普通16~25 bp的引物PCR扩增条件会有很多不同,长链引物扩增条件比普通引物扩增的条件要苛刻。建库PCR条件摸索: 模板上样量、退火温度、循环、延伸时间,其中主要考查上样量和退火温度。(1)退火温度: 考察的退火温度为56℃、54℃、52℃。2) 模板上样量: 针对模板上样量设计11个模板浓度梯度实验,模板信息如表1所示。上样量共分为12个梯度,依次为500、200、100、50、25、10、1 ng、100、10、1,100,10 fg。
1.5.5 高通量测序体系的特异性和敏感性检测: 42个检测样本的建库后采用 Illumina Miseq 高通量测序平台进行高通量测序。随后对高质量序列进行提取, 提取方法: Miseq 测序得到的 PE reads 首先根据 overla关系进行拼接, 将成对的 reads 拼接成一条序列, 同时对reads 质量和 merge 的效果进行质量质控和过滤, 去除序列末端的后引物和接头序列、多碱基 N、polyA/T尾巴及低质量碱基; 去除所得序列的 barcode 标签序列、前引物序列;丢弃长度短于 200 bp、模糊碱基数>0、序列平均质量低于20的序列。高通量测序结果与样本信息进行对比,评价高通量测序体系检测沙门氏菌的特异性和敏感性。
2 结果
2.1 通用引物设计及引物测试
针对①16S rDNA区域,②23S rDNA区域, ③16S~23S rDNA ISR,④23S~5S rDNA ISR,⑤gyrB优选区域各设计3对通用引物。经过测试,16S rDNA、23S rDNA、gyrB各有一对引物通过测试。但分析后发现16S rDNA、23S rDNA的引物实际建库测序的结果与设计时的有偏差,它无法区分样品中的肠炎沙门氏菌。最后确定沙门氏菌的菌种分类优选区域为gyrB基因引物(表2)。gyrB基因通用引物引物测试电泳结果如图2所示,由于gyrB基因不仅存在于沙门氏菌中,在志贺杆菌、大肠杆菌等肠科杆菌中都存在该基因,因此样品中只要有以上任一菌种,都能扩增出目的条带。引物主要是优势扩增沙门氏菌,但是该引物对于其他沙门氏菌和肠科杆菌的模板DNA还是有微弱的结合能力,因此即使是沙门氏菌阴性样品,也能够扩增得到目的条带。针对gyrB基因设计的1对引物(测试了鼠伤寒沙门氏菌和志贺杆菌其中的两个样本),条带单一,条带大小约为456 bp,通过电泳图可以看到此对引物对沙门菌的gyrB基因优势扩增,条带较亮,对属于肠杆菌属的志贺杆菌的gyrB基因扩增能力有很大程度降低,条带较淡。

表2 gyrB基因通用引物基本信息
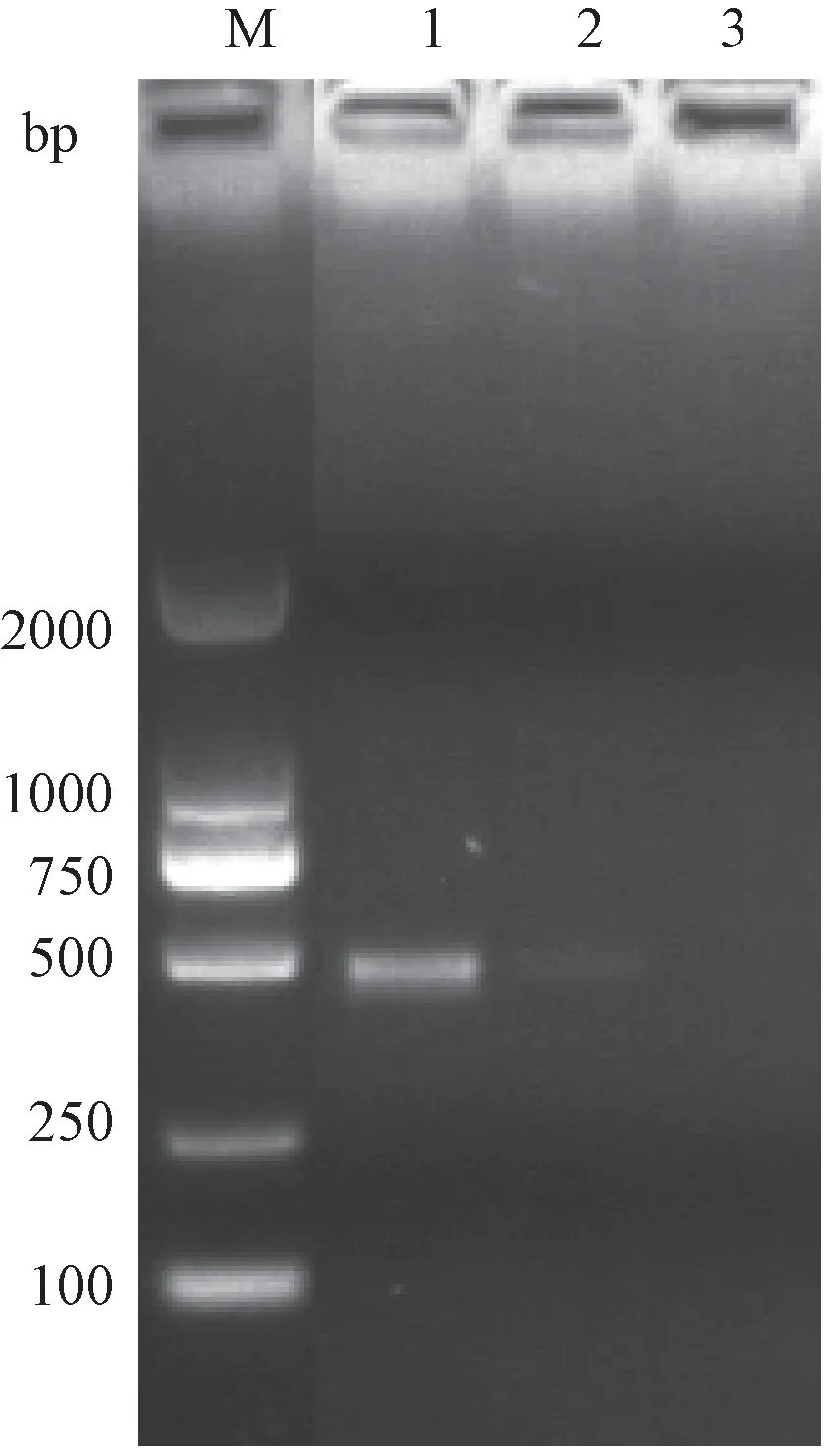
M-DL2000 marker 1-鼠伤寒沙门氏菌样本 2-志贺杆菌样本 3-无模板阴性对照图2 gyrB基因通用引物引物测试电泳图M-DL2000 marker. 1: Salmonella typhimurium sample; 2: Shigella sample; 3: Negative control.Fig.2 Electrophoregram of the universal primers for GyrB gene
2.2 沙门氏菌建库PCR条件优化
2.2.1 退火温度: 第1轮PCR,退火温度56℃,无扩增条带,未成功;第2轮PCR,退火温度54℃,无扩增条带,未成功;第3轮PCR,退火温度52℃,有扩增条带,成功。

M: DL2000 marker模板量为1: 500 ng;2: 200 ng;3: 100 ng;4: 50 ng;5: 25 ng;6: 10 ng;7: 1 ng;8: 100 pg;9: 10 pg;10: 1 pg;11: 100 fg;12: 10 fg。图3 沙门氏菌模板浓度梯度实验电泳结果图M: DL2000 Marker. Loading quantity of samples. 1: 500 ng; 2: 200 ng; 3: 100 ng; 4: 50 ng; 5: 25 ng; 6: 10 ng; 7: 1 ng; 8: 100 pg; 9: 10 pg; 10: 1 pg; 11: 100 fg; 12: 10 fg.Fig.3 Results of concentration gradient experiment of the Salmonella templates
2.2.2 模板上样量: 针对模板上样量设计一个模板浓度梯度实验,共分为12个梯度,依次为500、200、100、50、25、10、1 ng、100、10、1、100、10 fg,实验结果如下电泳图3。通过以上模板浓度梯度实验,可以发现模板在10 ng~200 ng范围内,都能扩增得到目的片段。为了保证最后的产物有足够的量,也就是要保证目的条带足够亮,选择较为合适的模板浓度150 ng作为体系最终的模板浓度。总结沙门氏菌gyrB基因建库PCR体系为DNA 150 ng、PremixExTaq12.5 μL,上下游优化引物 ( 10 pmol /μL) 各2.5 μL,补充ddH2O至20 μL;PCR最佳反应条件为: 98℃ 预变性 30 s; 98℃ 变性 10 s,52℃退火30s, 72℃延伸 10 min,共 40个循环; 72℃再延伸 5 min。
2.3 样本正式建库
采用以上摸索得到的建库PCR条件将所有样品进行建库,建库PCR结果如图4~6所示。结果显示,42个样本建库成功,均可得到456 bp大小的目的条带。
2.4 检测结果的准确性、特异性及重复性分析
42个样本的高通量测序检测结果如表3所示,各样本检测到的主体菌种与目的菌种基本上一致的。通过测序的方法在样品中存在的极微量的菌种也被检测到,因此在一些样品中除了要检测的主体菌种外,还存在极微量拷贝数的其他沙门氏菌菌种。在志贺杆菌中,特别设置了一个志贺杆菌的数据库用于19~23号样品的比对,发现无法检测到该菌种,说明设计的引物可以特异扩增沙门氏菌。在混合菌种中,该引物能将两种不同的菌种进行区分。针对肠炎沙门氏菌设置的cfu梯度,从检测的结果可以看出,在0~102的cfu范围内,也能检出,在其他cfu梯度样品中都能稳定检出,准确性及灵敏度高,各平行样本之间的检测结果一致,说明该方法具有较高重复性。通过高通量测序技术能够将检测出极微量的沙门氏菌,并能沙门氏菌很好的区分出来。

M: DL2000 marker 1-17 :1-17号样本图4 1-17号样本建库PCR电泳图M: DL2000 marker 1-17 : Sample 1-17Fig.4 PCR electrophoregram of the sequencing library of samples 1-17

M: DL2000 marker. 18-34:18-34号样本图5 18-34号样本建库PCR电泳图M: DL2000 marker. 18-34: Samples 18-34Fig.5 PCR electrophoregram of the sequencing library of samples 18-34

表3 沙门氏菌样本的数据分析结果
注: ST:Salmonellaentericaserovar Typhimurium;鼠伤寒沙门氏菌;SA:Salmonellaentericaserovar Paratyphi A;甲型副伤寒沙门氏菌;SE:Salmonellaentericaserovar Enteritidis;肠炎沙门氏菌;SH: Shigella;志贺菌

M: DL2000 marker. 35-42: 35-42号样本图6 35-42号样本建库PCR电泳图M: DL2000 marker. 35-42: Sample 35-42Fig.6 PCR electrophoregram of the sequencing library of samples 35-42
3 讨论
人畜共患病是指由同一种病原体引起,流行病学上相互关联,在人类和动物之间自然传播的疫病。其病原包括病毒、细菌、支原体、螺旋体、立克次氏体、衣原体、真菌、寄生虫等。这类疾病的特性在于很不容易消灭。因此这些病原微生物的存在会对人类健康、畜牧业安全生产、畜产品安全和公共卫生造成重大危害[11]。沙门氏菌是一类可以引起沙门氏菌病的典型人畜共患病原菌, 并且在实验动物中十分常见,可感染实验人员并对实验结果造成不可预估的影响,因此对于沙门氏菌的检测就变得尤为重要。
如今,分子生物学技术已越来越多的被应用于菌种鉴定,比如现在较为成熟的16S rDNA细菌菌种鉴定技术,ITS区真菌菌种鉴定技术等等,都体现出了简便、快速、高效的特点。菌种鉴定到属的级别还是相对较容易的,现在也有很多技术可以达成,但是想要鉴定到种、亚种甚至是菌株的水平,鉴定的困难程度就要提高很多。16S rDNA能很好的将菌种鉴定到属的层面, 但是由于16S rDNA的进化速度非常慢,即太保守,虽对种系有一定的区分能力,但是难以区分某些种系非常接近的菌种及同一菌种的不同菌株。23S rDNA也可用于菌种鉴定,有些学者认为其系统进化分析能力甚至比16S rDNA更强,但是由于23S rDNA的分子大小较大,因此它的使用也受到了限制[12,13]。核糖体基因间隔区(intergenic spacer region, ISR)为rDNA区域之间高度可变的区域,它的进化速度远远的高于rDNA区域,可以用于菌种间的鉴别。ISR区域主要有16S ~23S rDNA ISR区域和23S~5S rDNA ISR区域。ISR区域碱基变化程度与rDNA区域的程度相差不多,它优于rDNA区域的原因主要是该区域存在片段插入或缺失,导致片段大小的变化从而产生了种间和种内差异[14]。Morales等还发现23S~5S rDNA ISR区域比16S ~23S rDNA ISR区域具有更好的菌种区分能力[15]。然而,在某种程度上,ISR区域的种系鉴别能力也因其高度变异性和高度进化率而受到限制。
细菌中的一些基因也可以作为系统发育标记基因,比如gyrB基因,该基因编码DNA解旋酶(gyrase)的B亚单位,具有DNA依赖的ATP酶活性,催化ATP的水解。Fukushima应用gyrB基因对大肠埃希菌、沙门氏菌和志贺杆菌进行系统发育分析,证实了gyrB基因优于16S rDNA基因[14]。由于gyrB基因的进化速度比16S rDNA等其他基因快,因此更适用于亲缘关系较近的种内及种间的鉴定和分类。
在本研究中,针对16S rDNA、3S rDN、16S~23S rDNA IS、23S~5S rDNA IS、gyrB优选区域设计通用引物,对各个引物进行测试分析, 最终筛选发现沙门氏菌的菌种分类优选区域为gyrB基因,gyrB基因引物序列FP 5’-AACCACCGCAATCAGACCTT-3’,RP 5’-AGCCACGAAACCTTCACYA-3’。优化扩增条件并建库,利用Illumina高通量测序技术检测区分42个样本进行通过高通量测序和序列分析,能够检测出样本中极微量的沙门氏菌,检测方法稳定,检测限可达0~102的cfu,检测方法稳定性好,灵敏度高,可沿用至其他科学研究用的动物(如大鼠、豚鼠、家兔等)病原微生物的检测,同时还可将该实验体系应用于其他种类病原微生物的检测。
[1] 王军, 郑增忍, 王晶钰. 动物源性食品中沙门氏茵的风险评估 [J]. 中国动物检疫, 2007, 24(4): 23-25.
[2] 张嘉宁, 藏富妍, 顾为望. 应用PCR法检测小鼠沙门氏菌 [J]. 中国比较医学杂志, 1999, 9(1): 45-47.
[3] McGuinness S, McCabe E, O’Regan E, et al. Development and validation of a rapid real-time PCR based method for the specific detection of Salmonella on fresh meat [J]. Meat Sci, 2009, 83(3): 555-562.
[4] Liu FP, Tong HM.GyrAandParCgene mutation of clinically isolated fluoroquinolone-resistant strain of salmonella [J]. J Northeast Agr Univ, 2006,13(1): 47-50.
[5] Yan SS, Pendrak ML, Abela-Ridder B, et al. An overview of Salmonella typing — public health perspectives [J]. Clin Appl Immunol Rev, 2003, 4(3): 189-204.
[6] 范媛媛, 郑冰, 王树祥, 等. 一种新型培养基在沙门氏菌检测中的效果评价 [J]. 食品工业科技, 2008, 8: 415-417.
[7] 杨保伟, 盛敏, 席美丽, 等. 食源性沙门氏菌耐药性检测及相关质粒 [J]. 微生物学报, 2008, 48(8): 1006-1012.
[8] He LL, Sok D, Azadnia P, et al. Toward a more accurate view of human B-cell repertoire by next-generation sequencing, unbiased repertoire capture and single-molecule barcoding [J].Sci Rep, 2014, 4: 67-78.
[9] Weinstein JA, Jiang N, White III RA, et al. High-throughput sequencing of the Zebrafish antibody repertoire [J]. Science, 2009, 324(5928): 807-810.
[10] DeKosky BJ, Ippolito GC, Deschner RP, et al. High-throughput sequencing of the paired human immunoglobulin heavy and light chain repertoire [J]. Nat Biotech, 2013, 31(2):166-169.
[11] 王章云, 滕焕昭, 李柏桂, 等. 肠炎沙门氏菌引起的食物中毒细菌学调查 [J]. 中国人兽共患病杂志, 1999, 15(3): 115.
[12] Henrik C, Steen N, John EO. Phylogenetic relationships of Salmonella based on rRNA sequences [J]. Int J Systematic Bacteriol, 1998, 48: 605-610.
[13] Sara PL, Francisco R, Ruiting L. Variation of the ribosomal operon 16S-23S gene spacer region in representatives ofSalmonellaentericasubspecies [J]. J Bacteriol, 1998, 180(8) :2144-2151.
[14] Cesar AM, Richard G, Jean G. Linkage of avian and reproductive tract tropism with sequence divergence adjacent to the 5S ribosomal subunitrrfHofSalmonellaenterica[J]. FEMS Microbiol Lett, 2006, 264: 48-58.
[15] Fukushima M, Kakinuma R. Phylogenetic analysis of Salmonella, Shigella, and Escherichia coli strains on the basis of thegyrBgene sequence [J]. J Clin Microbiol, 2002, 40(8): 2779-2785.
Establishment and evaluation of a high throughput sequencing technology for detection ofSalmonellain laboratory animals
HU Yi-xiang, ZHANG Huan-huan, YU Chen-huan, JIN Xiao-yin, YING Hua-zhong
(Zhejiang Key Laboratory of Experimental Animal and Safety Evaluation, Zhejiang Academy of Medical Sciences, Hangzhou 310013, China)
Objective To establish a detection method ofSalmonellain laboratory animals based on a high-throughput sequencing technology, and to apply it in detection ofSalmonellain laboratory animals. Methods DNA samples were extracted from mouse feces. Universal primers for 16S rDNA, 23S rDNA, 16S-23S rDNA, 23S-5S rDNA region, gyrB preferred area were designed, respectively. Each primer was tested and analyzed to determine the best amplification conditions and build a database. Forty-two samples ofSalmonellawere assayed by Illumina high-throughput sequencing technology and evaluated the specificity and stability of this method. Results The species preferred region ofSalmonellawas gyrB gene region. The primers for gyrB gene were FP5’-AACCACCGCAATCAGACCTT3‘ and FP5’-AGCCACGAAACCTTCACYA-3’. The primers were optimized and determined, through a high-throughput sequencing, and the sequence analysis detected very small amount ofSalmonellain the 42 samples, indicating that this detection method is stable, highly sensitive, and the limit of detection reached to 0-102CFU. Conclusions We have established a complete detection system for detection ofSalmonellain laboratory animals based on a high-throughput sequencing technology, This system can detect trace amounts ofSalmonellain laboratory animals, and this detection method is stable and highly sensitive, which can be also used in detection of other kinds of pathogenic microorganism in laboratory animals.
Salmonella; Laboratory animal; High-throughput sequencing
浙江省卫生高层次人才(2012F30026),浙江省科技厅院所专项(2016F30001)。
胡毅翔(1989-),男,硕士研究生,研究方向为中药药理学。Email: aahyxaa@163.com。
应华忠(1968-),男,研究员,从事动物模型与比较医学研究。 Email: YHZ0101@126.com。
R-33
A
1671-7856(2016)10-0072-07
10.3969.j.issn.1671-7856.2016.10.014
2016-03-08

