青稞转录组SSR位点及其基因功能分析
徐金青,夏腾飞,王 蕾,王寒冬,张怀刚,刘登才,昌 西,沈裕虎
(1.中国科学院高原适应与进化重点实验室,中国科学院西北高原生物研究所,青海西宁 810001; 2.中国科学院大学,北京 100039; 3.四川农业大学小麦研究所,四川温江 611830; 4.西藏农牧学院,西藏林芝 860000;5.青海省作物分子育种重点实验室,青海西宁 810001)
青稞转录组SSR位点及其基因功能分析
徐金青1,2,夏腾飞1,2,王 蕾1,5,王寒冬1,张怀刚1,5,刘登才3,昌 西4,沈裕虎1,5
(1.中国科学院高原适应与进化重点实验室,中国科学院西北高原生物研究所,青海西宁 810001; 2.中国科学院大学,北京 100039; 3.四川农业大学小麦研究所,四川温江 611830; 4.西藏农牧学院,西藏林芝 860000;5.青海省作物分子育种重点实验室,青海西宁 810001)
为了探讨青稞转录中SSR位点信息及其所在基因的生物学功能,使用MISA软件分析青稞转录组中SSR的分布频率和重复基元的基本类型,通过BLASTX对含有SSR的Unigene与nr、COG、Swiss-Prot和KEGG等公共数据库进行比对和功能注释。结果表明,在青稞转录组拼接得到的58 065个Unigene中发现9 576条序列中含有11 930个SSR位点,SSR发生频率为16.49%,平均每6.63 kb出现1个SSR位点,共有119种重复基元(motif)。青稞转录组SSR出现频率最高的是三核苷酸重复基元(64.19%),其次是二核苷酸重复基元(24.05%)。AG/CT和AGG/CCT、CCG/CGG、AGC/CTG分别是二核苷酸重复和三核苷酸重复中的优势重复基元。在转录组中SSR重复次数以5~12次为主,基序长度主要集中在12~25 bp,平均长度为21.15 bp。9 576个含SSR的Unigene与nr、COG、Swiss-Prot和KEGG等公共数据库进行BLASTX比对,分别得到7 987、5 559、5 588和2 077个注释。通过基因功能注释发现青稞转录组中含SSR的序列主要与生物的基础代谢相关。
青稞;转录组;SSR;功能注释
简单重复序列(simple sequence repeat,SSR)又称简单序列长度多态性(simple sequence length polymorphism,SSLP)、短串联重复序列(short tandem repeats,STR)、微卫星(microsatellite)DNA,其串联重复的核心序列为1~6 bp[5]。SSR标记广泛分布在真核和原核生物基因组中,具有遗传共显性、扩增技术简单、基因组中广泛分布和多态性丰富等特点,已大量应用于遗传和物理图谱构建、基因定位、遗传多样性研究以及分子标记辅助育种中[5-6]。在青稞中,SSR标记已广泛应用于遗传多样性评估及其与农艺性状的关联分析研究中。杨 菁等[7]和吴昆仑[8]利用SSR标记分析,分别证实青海省栽培青稞和来自西藏、青海、四川和云南的青稞种质资源具有丰富的遗传多样性;潘志芬等[9]利用30对SSR引物研究青藏高原栽培青稞的遗传多样性,并发现其在淀粉性状、抗病性、麦芽性状以及裸粒性状等都存在丰富的遗传变异;孟亚雄[10]等利用92对SSR标记对108份青稞进行遗传多样性分析,并通过关联分析找到与株高、穗长、穗粒数和分蘖数等农艺性状相关联的标记。
传统的SSR(基因组SSR,genomic-SSR)标记的开发局限于已知基因组信息的物种。而以EST-SSR为代表的genetic-SSR来源于基因的转录区,直接与基因功能相关,进而与相关的重要性状关联。二代测序技术的诞生,使得转录组测序广泛得到应用,在此基础上开发的genetic-SSR相比EST-SSR提供更大的数据基础,从而加速了SSR标记、特别是与重要性状关联的SSR标记的开发[5]。由于不依赖于基因组信息,由转录组开发的分子标记是根据基因本身的差异而建立的标记,不仅信息量大,而且通用性好,在亲缘物种之间矫正连锁图谱和比较作图方面具有独特的优势[11]。本研究通过对青稞转录组数据进行SSR检测,并分析SSR的序列特征及其组成情况,同时注释其所在序列的基因功能,推测其可能相关联的性状,以期为今后开展青稞遗传多样性分析、遗传连锁图谱构建提供丰富的候选SSR标记,同时也为青稞分子标记辅助育种和功能基因定位研究提供理论基础。
1 材料与方法
1.1 试验材料与转录组数据
选取4个青稞栽培品种(表1)作为试验材料,种植于培养间。当植株长出4~5片叶子时,取单株叶片利用Trizol法提取总RNA后,送至北京百迈克生物公司(Biomarker Technologies),利用Illumina HiSeq 2000进行转录组测序。测序数据(raw reads)经去除rRNA、接头以及低质量的reads,得到clean reads。clean reads利用Trinity[12]软件进行从头组装,然后使用TGICL[13]去除冗余,得到共含79 122 598个核苷酸的58 065条Unigene。
1.2 转录组SSR搜索分析
通过MISA(MIcroSAtellite identification tool,http://pgrc.ipk-gatersleben.de/misa/)软件对转录组Unigene序列进行SSR位点识别,其识别条件为单核苷酸重复不低于15次,二核苷酸重复不低于6次,三核苷酸、四核苷酸、五核苷酸和六核苷酸重复不低于5次,复合SSR的识别条件是两个SSR位点间的距离不超过100 bp。将生成的文本文件导入到Excel中进行基本的统计分析。SSR发生频率=搜索到的含SSR的Unigene序列数量/总Unigene序列数量;SSR分布频率=SSR数量/总Unigene序列数量;SSR分布的平均距离=总Unigene长度/搜索到的SSR数量[6,14]。

表1 4份供试青稞材料的基本信息Table 1 Hulless barley materials used in this study
1.3 青稞转录组中含SSR的Unigene 的功能注释
通过BLASTX,分别将青稞转录组中含SSR的9 576条Unigene序列比对到nr(non-redundant)、COG(cluster of orthologous groups of proteins)、Swiss-Prot以及KEGG(kyoto encyclopedia of genes and genomes)等蛋白数据库,比对参数e值<10-5。将通过BLASTX与nr蛋白数据库比对生成的XML文件导入到Blast2GO[15]软件,得到转录组数据中含SSR的Unigene序列的基因本体(gene ontology,GO)注释信息,然后利用WEGO(http://wego.genomics.org.cn/cgi-bin/wego/index.pl)[16]在线分析软件对注释的Unigene序列进行GO功能分类统计,分析含有SSR的Unigene的功能分布特征;通过与COG库进行比对后,得到的Unigene注释结果按照COG数据库的23个类别进行分类统计;对含有SSR的Unigene序列所参与的代谢途径的分析则是根据其在KEGG数据库中的比对注释信息,得到其在KEGG本体(KEGG orthology,KO)系统中的相应K编号,然后利用K编号将Unigene注释到相应的代谢通路上。
TWEAK上游引物5′-ATCGCAGCCCATTATGAAGT-3′、下游引物 5′-GAAGAGTCCGAAGTAGGTGAGG-3′,p38MAPK 上游引物 5′-TCGAGACCGTTTCAGTCCAT-3′、下游引物5′-CCACGGACAAATATCCACT-3′,GAPDH上游引物5′-ATCACCATCTTCCAGGAGCGA-3′、下游引物5′-CCTTCTCCATGGTGGTGAAGA-3′均由生工生物工程(上海)股份有限公司合成。
2 结果与分析
2.1 青稞转录组中SSR的序列特征
根据获取的19.89 Gb青稞RNA-Seq数据,利用Trinity软件组装得到58 065条Unigene,总碱基数为79 122 598 bp,平均每条Unigene长约1.3 kb。使用MISA软件共搜索到11 930个SSR,分布于9 576条Unigene上,其中含有多个SSR(含复合SSR)的Unigene共1 875条,占含SSR的Unigene序列总数的19.58%。总体上,含SSR的Unigene序列占所有Unigene的16.49%,平均每6.63 kb出现1个SSR。
青稞转录组SSR重复类型丰富,从单核苷酸重复到六核苷酸重复均有出现,其中以三核苷酸重复为主(7 658个),占SSR总数的64.19%,分布频率为13.19%;其次是二核苷酸重复(2 869个),占SSR总数的24.05%,分布频率为4.94%;五核苷酸和六核苷酸重复SSR数量较少,二者加起来占SSR总数的1.19%,分布频率分别为0.19%和0.05%(表2)。二核苷酸重复的SSR平均长度最短,仅为14.97 bp,六核苷酸重复SSR平均长度最长,为31.45 bp(表2)。三核苷酸重复SSR的平均分布距离最短(10.33 kb),而六核苷酸重复SSR平均分布距离最长(2 728.37 kb)(表2)。
不同重复类型的青稞转录组SSR均有多种基元。在考虑碱基互补且包含复合SSR重复基元的情况下,单核苷酸、二核苷酸、三核苷酸、四核苷酸、五核苷酸和六核苷酸重复SSR出现的基元种类数分别为2、4、10、33、48和22种,共计119种基元(表2)。在筛选到的11 930个SSR中,单核苷酸重复的优势基元为C/G,占单核苷酸重复的68.42%,占SSR总数的3.05%,而A/T基元仅占单核苷酸重复的31.57%,占SSR总数的1.41%。说明对原始序列预处理时去除5′端polyT和3′端的polyA序列是有效的,基本可以排除假阳性A/T的存在。二核苷酸重复类型中,AG/CT基元最多,占该重复类型总数的59.25%。三核苷酸重复类型的优势基元有CCG/CGG、AGG/CCT和AGC/CTG三种,分别占三核苷酸重复类型总数的36.88%、20.34%和16.02%,共73.24%。四、五和六核苷酸重复类型中各基元的分布频率均较低,ATCC/ATGC、AGGGG/CCCCT和ACAGAG/CTCTGT为各自的优势基元,分别占各自重复类型的11.11%、10.62% 和13.79%(表2)。
青稞转录组SSR各重复类型在不同重复数下的数量存在明显差异(表3)。由于单核苷酸重复类型的识别条件设置为重复数≥15,故在表3中对单核苷酸重复未做统计。除单核苷酸外的其余各重复类型的重复数介于5~12次之间,随着重复次数的增加,各重复类型的出现频率逐步降低。各重复类型的重复数主要集中在5~7次,占SSR总数的70.49%。二核苷酸重复的主要重复数为6~11,三核苷酸重复的主要重复数为5~7次,四核苷酸重复的重复数主要为5~6,五、六核苷酸重复的主要重复数为5。另外,重复数最多的基元为单碱基重复C/G,重复次数为40次。三碱基重复AAC/GTT基元的重复数次之,为29次,这也是长度最长的重复(87 bp)。

表2 青稞转录组中SSR各重复类型的分布特征Table 2 Distribution characteristics of various SSR repeat types in hulless barley transcriptome

表3 青稞转录组SSR各重复类型在不同重复次数下的数量Table 3 Number of various SSR repeat types with different number of repeats in hulless barley transcriptome
-表示不符合鉴定条件。
- indicated that did not meet the analysis conditions.
青稞转录组SSR基元长度分布如图1所示。总体看来,青稞转录组的基元长度分布在12~87 bp。大部分青稞转录组SSR基元长度集中在12~25 bp(11 869个),占SSR总数的99.49%。基元长度为26~29 bp的SSR数量仅为12个,占SSR总数的0.10%。大于30 bp的有49个,仅占SSR总数的0.41%。其中基元长度为15 bp的SSR数量最多,有4 748个,占SSR总数的39.80%。基元长度为18 bp的SSR数量次之(2 315个),占SSR总数的19.40%。基元(AG/CT)长度为26 bp的SSR重复仅有1个。未出现基元长度为29 bp的SSR。

图1 青稞转录组SSR重复序列长度的分布频率
2.2 青稞转录组中含SSR序列的基因功能注释
通过MISA软件的搜索,共有9 576条Unigene含有SSR。为了解青稞转录组中含有SSR序列的基因功能,本研究通过与公共蛋白数据库进行比对,以期得到含有SSR序列的Unigene的功能注释及分类信息。结果发现,有7 987、5 559、5 588、2 077条Unigene分别与nr、COG、Swiss-Prot和KEGG比对得到注释信息,有1 683条Unigene在上述4个数据库中得到共同注释信息。
GO注释用于描绘基因及其基因产物的特点,将基因功能分为3个本体,即细胞组分(cellular component)、分子功能(molecular function)和生物进程(biological process)。其下又分了很多亚类,各类间互相关联,从不同角度对基因的功能进行分类注释。通过对含SSR的青稞Unigene进行GO注释,可以全面描述青稞中含SSR基因和基因产物的属性。将搜索到含有SSR的Unigene序列使用BLASTX比对到nr蛋白数据库,取比对分值最高的为序列的注释信息。其中7 987条Unigene序列得到了注释信息,1 589条Unigene序列无注释信息。再使用Blast2GO软件进行含SSR的Unigene的GO注释,有5 015条Unigene序列取得相应的GO分类号,占含SSR的Unigene序列总数的52.37%,其他2 972条不能成功注释。将含有SSR序列的5 015条Unigene编号及其对应的GO分类号导入到GO分类图形显示在线分析工具WEGO软件中,得到其基因功能分布(图2)。结果表明,有GO注释的5 015条Unigene序列被分配至细胞组分、分子功能和生物进程3个本体下的45个亚类中,3个本体分别包含12、11和22个亚类。在注释到生物进程类的Unigene中,分别有3 156条、2 627条被注释到代谢进程(metabolic process)和细胞进程(cellular process),分别占注释为该类的Unigene数量的62.93%和52.38%。在细胞组分中,分别有2 194和2 164条具有GO分类号的Unigene被注释到细胞(cell)和细胞成分(cell part)中,分别占注释为该类的43.75%和43.15%。而在分子功能中,结合活性(binding)和催化活性(catalytic activity)是两个最主要的功能分类,各有2 993条和2 275条,分别占注释为该类的59.68%和45.36%,其中多被注释为转移酶活性(transferase activity)、水解酶活性(hydrolase activity)、核苷酸结合(nucleotide binding)。综合以上信息,在青稞转录组中鉴定出的含有SSR的Unigene主要是参与细胞的基础代谢活动。
COG数据库是基于细菌、藻类和真核生物的系统进化关系构建得到的,可以对基因产物进行直系同源分类。对青稞转录组中含有SSR的Unigene进行COG分类(图3),共获得5 559个COG功能注释,涉及4个类别、23个功能亚类。总体来看,除缺乏注解(poorly characterized)的1 639条Unigene外(其中984条为一般功能预测,655条为功能未知),注释到细胞进程及信号传递(cellular processes and signaling)、信息存储及处理(information storage and processing)和代谢(metabolism)三大类别的分别有1 512条、1 235条和1 173条,分别占具COG功能注释Unigene数量的27.20%、22.22%和21.10%。在细胞进程及信号传递类中,有502条注释到细胞周期控制(cell cycle control)、细胞分裂(cell division)及染色体分隔(chromosome partitioning)亚类,有386条注释到翻译后修饰(posttranslational modification)、转运(protein turnover)及分子伴侣(chaperones)亚类,有297条注释到细胞壁(cell wall)、细胞膜(cell membrane)及质膜(envelope)的生物合成亚类中,分别占该类的33.20%、25.53%和19.64%。没有Unigene注释到细胞核结构(nuclear structure)和细胞外结构(extracellular structures)亚类。在信息存储及处理类中,有659条注释到翻译(translation)、核糖体结构(ribosomal structure)及生物合成(biogenesis)亚类,有292条注释到转录(transcription),有209条注释到复制、重组和修复(replication,recombination and repair)亚类,有75条注释到染色质结构和动力学亚类,分别占该类的53.36%、23.64%、16.92%和6.07%。在代谢类中,有330条注释到碳水化合物运输与代谢(carbohydrate transport and metabolism)亚类,占该类的28.13%,其次是氨基酸转运及代谢(amino acid transport and metabolism)、无机盐转运及代谢(inorganic ion transport and metabolism)和脂质转运及代谢(lipid transport and metabolism)。

1:细胞;2:细胞成分;3:细胞器;4:细胞器组分;5:大分子复合物;6:细胞膜;7:细胞被膜;8:细胞外区域;9:共质体;10:病毒体;11:细胞外区域部分;12:病毒粒子部分;13:代谢进程;14:细胞进程;15:生物调节;16:色素沉积;17:定位;18:建立定位;19:刺激反应;20:细胞成分组织;21:细胞成分的生物合成;22:多细胞生物进程;23:发育过程;24:废弃的生物进程;25:解剖结构形成;26:繁殖;27:生殖过程;28:多有机体过程;29:生长;30:免疫系统的过程;31:死亡;32:病毒繁殖;33:生物附着;34:节律过程;35:结合;36:催化活性;37:转运活性;38:转录调节活性;39:结构分子活性;40:翻译调节活性;41:分子传感活性;42:抗氧化活性;43:酶调节活性;44:电子载体活性;45:营养物储存活性。
1:Cell;2:Cell part;3:Organelle;4:Organelle part;5:Macromolecular complex;6:Membrane;7:Envelope;8:Extracellular region;9:Symplast;10:Virion;11:Extracellular region part;12:Virion part;13:Metabolic process;14:Cellular process;15:Biological regulation;16:Pigmentation;17:Localization;18:Establishment of localization;19:Response to stimulus;20:Cellular component organization;21:Cellular component biogenesis;22:Multicellular organismal process;23:Developmental process;24:Obsolete biological process;25:Anatomical structure formation;26:Reproduction;27:Reproductive process;28:Multi-organism process;29:Growth;30:Immune system process;31:Death;32:Viral reproduction;33:Biological adhesion;34:Rhythmic process;35:Binding;36:Catalytic activity;37:Transporter activity;38:Transcription regulator activity;39:Structural molecule activity;40:Translation regulator activity;41:Molecular transducer activity;42:Antioxidant activity;43:Enzyme regulator activity;44:Electron carrier activity;45:Nutrient reservoir activity.
图2 青稞转录组中含SSR的Unigene的GO注释
Fig.2 GO annotation of Unigenes containing SSR in hulless barley transcriptome

R:一般功能预测;S:未知功能;J:翻译、核糖体结构和生物合成;K:转录;L:复制、重组和修复;B:染色体结构与动力学;A:RNA加工与修改;D:细胞周期控制、细胞分裂、染色体分隔;O:翻译后修饰、蛋白质反转、分子伴侣;M:细胞壁、细胞膜、质膜生物合成;T:信号转导机制;U:细胞内运输、分泌和囊泡运输;V:防御机制;Z:细胞骨架;N:细胞运动;G:碳水化合物转运和代谢;E:氨基酸转运和代谢;P:无机盐转运和代谢;I:脂质转运和代谢;C:能量产生与转换;Q:次生代谢产物的合成、转运和代谢;H:辅酶转运和代谢;F:核苷酸转运和代谢。
R:General function prediction only; S:Function unknown; J:Translation,ribosomal structure and biogenesis; K:Transcription; L:Replication,recombination and repair; B:Chromatin structure and dynamics; A:RNA processing and modification; D:Cell cycle control,cell division and chromosome partitioning; O:Posttranslational modification,protein turnover and chaperones; M:Cell wall,membrane and envelope biogenesis; T:Signal transduction mechanisms; U:Intracellular trafficking,secretion and vesicular transport; V:Defense mechanisms; Z:Cytoskeleton; N:Cell motility; G:Carbohydrate transport and metabolism; E:Amino acid transport and metabolism; P:Inorganic ion transport and metabolism; I:Lipid transport and metabolism; C:Energy production and conversion; Q:Secondary metabolites biosynthesis,transport and metabolism; H:Coenzyme transport and metabolism; F:Nucleotide transport and metabolism.
图3 青稞基因组中含SSR的Unigene的COG功能注释
Fig.3 Functional classification of SSR Unigene in hulless barley transcriptome based on COG analysis
通过与KEGG数据库的比对,可以分析含SSR的Unigene 在青稞代谢途径中的富集情况。注释KEGG代谢通路时,会给每一个功能基因定一个K编号(K numbers),并注释到具有相应归类的代谢通路(pathway)中。分析发现,有2 077条(21.69%)含SSR的Unigene具有KEGG注释结果,共得到1 225个K编号并被注释到312个KEGG代谢通路中,平均1.70个Unigene具有相同的功能,说明Unigene中有许多功能相同。另外的7 499条(78.31%)未得到注释结果。在对注释到的312个通路图进行分析时,利用KEGG数据库的分类,将其归类到全部7大类代谢通路中,其中被注释到新陈代谢(metabolism)和遗传信息处理(genetic information processing)类的Unigene数量最多,分别有534和490条,占全部含SSR的Unigene的5.58%和5.12%,占能注释到通路的Unigene的25.71%和23.59%。对注释到新陈代谢通路中的Unigene作进一步分类分析,结果(图4)发现,除化学结构转化图(chemical structure transformation maps)亚类外,其他亚类中均有分布。注释到碳水化合物代谢、能量代谢、氨基酸代谢和脂质代谢途径中的Unigene占主导地位,分别有199、107、104和81条,各占新陈代谢通路的37.27%、20.04%、19.48%和15.17%。这与GO注释得到的基础代谢为主的注释结果一致。
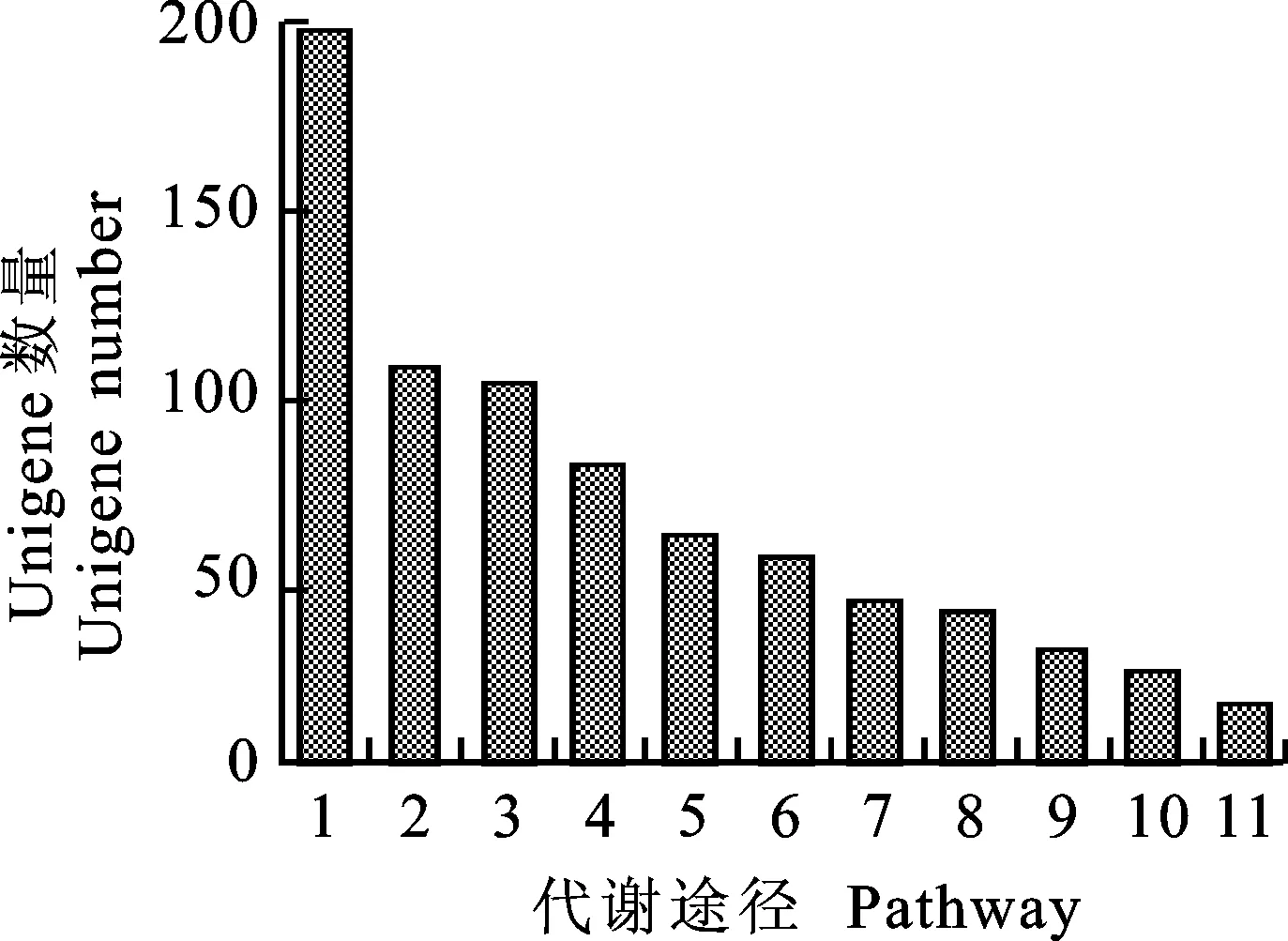
1:碳水化合物代谢;2:能量代谢;3:氨基酸代谢;4:脂质代谢;5:辅因子及维生素代谢;6:核苷酸代谢;7:萜类及聚酮代谢;8:其他氨基酸代谢;9:其他次生产物代谢;10:聚糖生物合成及代谢;11:异质物降解及代谢。
1:Carbon metabolism; 2:Energy metabolism; 3:Amino acid metabolism; 4:Lipid metabolism; 5:Metabolism of cofactors and vitamins; 6:Nucleotide metabolism; 7:Metabolism of terpenoids and polyketides; 8:Metabolism of other amino acids; 9:Biosynthesis of other secondary metabolites; 10:Glycan biosynthesis and metabolism; 11:Xenobiotics biodegradation and metabolism.
图4 青稞转录组中注释到新陈代谢通路中的含有SSR 的Unigene的代谢途径分析
Fig.4 Analysis of Unigenes containing SSR in hulless barley transcriptome annotated to metabolism pathway
有5 588条含SSR的Unigene在Swiss-Prot数据库中比对得到注释信息,其中有68条直接得到了在大麦(Hordeumvulgare)中的注释信息,2 946条是在拟南芥(Arabidopsisthaliana)中的注释信息,1 040条是在水稻(Oryzasativa)中的注释信息,84条是在玉米(Zeamays)中的注释信息,72条是在小麦(Triticumaestivum)中的注释信息。在Swiss-Prot数据库中得到注释的5 588条Unigene中有5 573条在nr库中也得到了注释信息,其功能涵盖了生物进程、细胞组分和分子功能三大类。因此GO注释的分类结果也可以大致用来解释在Swiss-Prot数据库得到注释的5 588条Unigene的分类信息。
3 讨 论
3.1 青稞转录组中SSR的序列特征
在本研究中,青稞转录组SSR分布频率为1/6.63 kb,与之前从大麦的EST数据中搜索到的SSR的频率基本一致(1/6.30 kb)[17]。与其他植物相比较,青稞转录组SSR出现频率高于小麦(1/15.60 kb)[18]、大豆(1/7.40 kb)[17]、番茄(1/11.10 kb)[17]、玉米(1/8.10 kb)[17]、拟南芥(1/13.83 kb)[17]、杨树(1/14.00 kb)[17]、棉花(1/20.00 kb)[17,19]和洋葱(1/14.10 kb)[20]。这表明,青稞转录组中SSR数量很丰富,出现频率比较高,考虑到在本文中搜索到的SSR基于转录组测序数据,因此这些SSR都有较高的利用潜能。
从目前已有的报道来看,大多数植物的EST-SSRs的重复单元类型以二核苷酸和三核苷酸为主。大麦[17,21]、燕麦[17]、黑麦[17]、水稻[21-22]、小麦[21]、高粱[21]、玉米[21]、藏茵陈川西獐牙菜[23]、洋葱[20]、云南松[14]等植物中以三核苷酸为主。而在党参[24]、野三七[25]、灯盏花[26]、茶树[27]等植物中则是以二核苷酸重复为主。此外南方红豆杉[28]、巴西橡胶树[29]的优势重复基元为六核苷酸。在本研究中,青稞转录组中的SSR序列以三核苷酸重复为主,其次是二核苷酸重复,不同重复类型的SSR数量随基元碱基数量增加呈下降趋势,这种SSR重复类型的偏好性可能与分析的数据量有关,也可能与其自身长度的稳定性有关。重复类型中三核苷酸重复居多,推测可能与三联体密码子选择作用有关,因为除三、六核苷酸重复之外,其他重复类型重复次数的改变,会导致阅读框的改变,容易造成移码突变,进而影响基因产物[23]。在青稞转录组SSR序列中二核苷酸重复以AG/CT为主,三核苷酸重复中CCG/CGG、AGG/CCT和AGC/CTG居多,这与藜麦(AG/CT)[30]、玉米(CCG/GGC和AGG/CCT)[17]基本一致。但是,不同的植物的优势重复基元存在差异,在藏茵陈川西獐牙菜中,二核苷酸重复中的优势基元类型是AT/TA[23],在辣椒中AAC/GTT[31]是三核苷酸重复中的优势重复基元,这可能与植物自身基因组的差异、数据量的大小以及分析数据的来源密切相关。
3.2 青稞转录组中含SSR序列的功能注释
通过对青稞转录组中含有SSR的Unigene在4个公共数据库中的比对和功能注释,发现在GO注释中主要归类于生物进程下的代谢进程和细胞进程、细胞组分下的细胞和细胞部分以及分子功能下的催化和结合活性。在COG注释中,大部分Unigene归类于细胞进程及信号传递类下的细胞周期控制、细胞分裂及染色体分隔亚类,信息存储及处理类下的翻译、核糖体结构及生物合成亚类,代谢类下的糖类运输与代谢亚类。另外,在KEGG代谢通路注释中,大部分Unigene注释到新陈代谢和遗传信息处理类,且在新陈代谢途径中,主要集中在碳水化合物代谢、能量代谢、氨基酸代谢和脂质代谢途径。综合以上注释信息,青稞转录组中含SSR的Unigene序列主要与生物的基础代谢相关。在注释过程中存在多个含SSR的Unigene共同注释到相同功能上,出现这种情况不仅因为在一个基因家族中多个基因行使相同的功能,而且也可能是转录本在后期加工过程中存在可变剪接造成的,此外也与软件拼接有关[32]。对转录组SSR的应用还需要进行相应的引物筛选等工作,同时,可以有针对性地选择与一定功能相关的基因作为SSR标记位点,从而利于目标性状的筛选[32]。
4 结 论
使用MISA软件分析了青稞转录组中SSR信息,发现青稞转录组中SSR出现频率比较高,平均每6.63 kb出现一个SSR位点,以三核苷酸重复为主要重复类型。对含有SSR的Unigene进行功能注释,表明青稞转录组中含SSR的Unigene主要与生物的基础代谢相关。总之,本文基于青稞转录组搜索到的SSR类型丰富,生物功能多样,具有很大的利用潜能。
[1] 张梅妞,张怀刚,蔡联炳,等.野生大麦与青稞高分子量谷蛋白亚基遗传变异研究[J].西北农业学报,2007,16(1):107.
ZHANG M N,ZHANG H G,CAI L B,etal.Genetic variation of high-molecular-weight glutenin subunits in wild barley and highland barley [J].ActaAgriculturaeBoreali-occidentalisSinica,2007,16(1):107.
[2] PURUGGANAN M D,FULLER D Q.The nature of selection during plant domestication [J].Nature,2009,457(7231):843.
[3] TAKETA S,AMANO S,TSUJINO Y,etal.Barley grain with adhering hulls is controlled by an ERF family transcription factor gene regulating a lipid biosynthesis pathway [J].ProceedingsoftheNationalAcademyofSciences,2008,105(10):4062.
[4] 吕远平,熊茉君,贾利蓉,等.青稞特性及在食品中的应用[J].食品科学,2005,26(7):267.
LÜ Y P,XIONG M J,JIA L R,etal.Characteristics of barley and application in food industry [J].FoodScience,2005,26(7):267.
[5] KALIA R K,RAI M K,KALIA S,etal.Microsatellite markers:An overview of the recent progress in plants [J].Euphytica,2011,177(3):309.
[6] 黄海燕,杜红岩,乌云塔娜,等.基于杜仲转录组序列的SSR分子标记的开发[J].林业科学,2013,49(5):176.
HUANG H Y,DU H Y,WUYUN T N,etal.Development of SSR molecular markers based on transcriptome sequencing ofEucommiaulmoides[J].ScientiaSilvaeSinicae,2013,49(5):176.
[7] 杨 菁,迟德钊,吴昆仑,等.青海省栽培青稞SSR标记遗传多样性研究[J].安徽农业科学,2010,38(8):4307.
YANG J,CHI D Z,WU K L,etal.Genetic diversity of SSR in cultivatedHordeumvulgareL.in Qinghai province [J].JournalofAnhuiAgriculturalSciences,2010,38(8):4307.
[8] 吴昆仑.青稞种质资源的SSR标记遗传多样性分析[J].麦类作物学报,2011,31(6):1030.
WU K L.Genetic diversity analysis of hulless barley germplasm by SSR markers [J].JournalofTriticeaeCrops,2011,31(6):1030.
[9] 潘志芬,邹弈星,邓光兵,等.青藏高原栽培青稞SSR标记遗传多样性研究[J].中山大学学报(自然科学版),2007,46(2):82.
PAN Z F,ZHOU Y X,DENG G B,etal.Genetic diversity of SSR markers in cultivated hulless barley from Qinghai-Tibet plateau in China [J].ActaScientiarumNaturaliumUniversitatisSunyatseni,2007,46(2):82.
[10] 孟亚雄,孟祎林,汪军成,等.青稞遗传多样性及其农艺性状与 SSR标记的关联分析[J].作物学报,2015,42(2):180.
MENG Y X,MENG Y L,WANG J C,etal.Genetic diversity and association analysis of agronomic characteristics with SSR markers in hulless barley [J].ActaAgronomicaSinica,2015,42(2):180.
[11] 李小白,向 林,罗 洁,等.转录组测序(RNA-seq)策略及其数据在分子标记开发上的应用[J].中国细胞生物学学报,2013,35(5):723.
LI X B,XIANG L,LUO J,etal.The strategy of RNA-seq,application and development of molecular marker derived from RNA-seq [J].ChineseJournalofCellBiology,2013,35(5):723.
[12] GRABHERR M G,HAAS B J,YASSOUR M,etal.Full-length transcriptome assembly from RNA-Seq data without a reference genome [J].NatureBiotechnology,2011,29(7):644.
[13] PERTEA G,HUANG X,LIANG F,etal.TIGR gene indices clustering tools (TGICL):A software system for fast clustering of large EST datasets [J].Bioinformatics,2003,19(5):651.
[14] 蔡年辉,许玉兰,徐 杨,等.云南松转录组SSR的分布及其序列特征[J].云南大学学报(自然科学版),2015,37(5):771.
CAI N H,XU Y L,XU Y,etal.The distribution and character of SSR sequences inPinusyunnanensisFranch [J].JournalofYunnanUniversity,2015,37(5):771.
[15] CONESA A,GOTZ S,GARCIA-GOMEZ J M,etal.Blast2GO:a universal tool for annotation,visualization and analysis in functional genomics research [J].Bioinformatics,2005,21(18):3674.
[16] YE J,FANG L,ZHENG H,etal.WEGO:a web tool for plotting GO annotations [J].Nucleicacidsresearch,2006,34(supply 2):W293.
[17] THIEL T,MICHALEK W,VARSHNEY R,etal.Exploiting EST databases for the development and characterization of gene-derived SSR-markers in barley (HordeumvulgareL.) [J].TheoreticalandAppliedGenetics,2003,106(3):414.
[18] KANTETY R V,LA ROTA M,MATTHEWS D E,etal.Data mining for simple sequence repeats in expressed sequence tags from barley,maize,rice,sorghum and wheat [J].PlantMolecularBiology,2002,48(5-6):504.
[19] CARDLE L,RAMSAY L,MILBOUME D,etal.Computational and experimental characterization of physically clustered simple sequence repeats in plants [J].Genetics,2000,156(2):850.
[20] 李满堂,张仕林,邓 鹏,等.洋葱转录组SSR信息分析及其多态性研究[J].园艺学报,2015,42(6):1103.
LI M T,ZHANG S L,DENG P,etal.Analysis on SSR information in transcriptome of onion and the polymorphism [J].ActaHorticulturaeSinica,2015,42(6):1103.
[21] LI L,WANG J,GUO Y,etal.Development of SSR markers from ESTs of gramineous species and their chromosome location on wheat [J].ProgressinNaturalScience,2008,18(12):1487.
[22] CHOY G,ISHII T,TEMNYKH S,etal.Diversity of microsatellites derived from genomic libraries and GenBank sequences in rice (OryzasativaL.) [J].TheoreticalandAppliedGenetics,2000,100(5):713.
[23] 刘 越,岳春江,王 翊,等.藏茵陈川西獐牙菜转录组SSR信息分析[J].中国中药杂志,2015,40(11):2068-2074.
LIU Y,YUE C J,WANG Y,etal.Data mining of simple sequence repeats in transcriptome sequences of Tibetan medicinal plant Zangyinchen Swertia mussotii [J].ChinaJournalofChineseMateriaMedica,2015,40(11):2068-2074.
[24] 王 东,曹玲亚,高建平.党参转录组中SSR位点信息分析[J].中草药,2014,45(16):2390.
WANG D,CAO L Y,GAO J P.Data mining of simple sequence repeats inCodonopsispilosulatranscriptome [J].ChineseTraditionalandHerbalDrugs,2014,45(16):2390.
[25] 李翠婷,张广辉,马春花,等.野三七转录组中SSR位点信息分析及其多态性研究[J].中草药,2014,45(10):1468.
LI C T,ZHANG G H,MA C H,etal.Analysis on SSR loci information in transcriptome ofPanaxvienamensisvar.fuscidiscusand its polymorphism [J].ChineseTraditionalandHerbalDrugs,2014,45(10):1468.
[26] 陈 茵,李翠婷,姜倪皓,等.灯盏花转录组中SSR位点信息分析及其多态性研究[J].中国中医药杂志,2014,39(7):1220.
CHEN Y,LI C T,JIANG N H,etal.Analysis on SSR loci information in transcriptome ofErigeronbreviscapus(Vant.) Hand.-Mazz.and its polymorphism [J].ChinaJournalofChineseMateriaMedica,2014,39(7):1220.
[27] 杨 华,陈 琪,韦朝领,等.茶树转录组中SSR位点的信息分析[J].安徽农业大学学报,2011,38(6):882.
YANG H,CHEN Q,WEI C L,etal.Analysis on SSR information inCamelliasinensistranscriptome [J].JournalofAnhuiAgriculturalUniversity,2011,38(6):882.
[28] 李炎林,杨星星,张家银,等.南方红豆杉转录组SSR挖掘及分子标记的研究[J].园艺学报,2014,41(4):735.
LI Y L,YANG X X,ZHANG J Y,etal.Studies on SSR molecular markers based on transcriptome ofTaxuschinensisvar.mairei [J].ActaHorticulturaeSinica,2014,41(4):735.
[29] 甘 霖,覃 碧,刘实忠,等.巴西橡胶树转录组中SSR位点的信息分析[J].广东农业科学,2014,41(16):142.
GAN L,QIN B,LIU S Z,etal.Bioinformatic analysis of SSR markers in transcriptome of rubber treeHeveabrasiliensisMuell.Arg.[J].GuangdongAgriculturalSciences,2014,41(16):142.
[30] 张体付,戚维聪,顾闽峰,等.藜麦EST-SSR的开发及通用性分析[J].作物学报,2016,42(4):495.
ZHANG T F,QI W C,GU M F,etal.Exploration and transferability evaluation of EST-SSRs in Quinoa [J].ActaAgronomicaSinica,2016,42(4):495.
[31] 刘 峰,王运生,田雪亮,等.辣椒转录组SSR挖掘及其多态性分析[J].园艺学报,2012,39(1):171.
LIU F,WANG Y S,TIAN X L,etal.SSR mining in pepper(CapsicumannuumL.)transcriptome and the polymorphism analysis [J].ActaHorticulturaeSinica,2012,39(1):171.
[32] 何 海,郭继云,马毅平,等.茯苓转录组SSR序列特征及其基因功能分析[J].中草药,2015,46(23):3563.
HE H,GUO J Y,MA Y P,etal.Characterization and gene function analysis of SSR sequences inPoriacocostranscriptome [J].ChineseTraditionalandHerbalDrugs,2015,46(23):3563.
Characterization and Gene Function Analysis of SSR Sequences in Hulless Barley Transcriptome
XU Jinqing1,2,XIA Tengfei1,2,WANG Lei1,5,WANG Handong1,
ZHANG Huaigang1,5,LIU Dengcai3,CHANG Xi4,SHEN Yuhu1,5
(1.Key Laboratory of Adaptation and Evolution of Plateau Biota,Northwest Plateau Institute of Biology,Chinese Academy of Sciences,Xining,Qinghai 810001,China; 2.University of Chinese Academy of Sciences,Beijing 100039,China; 3.Triticeae Research Institute,Sichuan Agricultural University,Wenjiang,Sichuan 611830,China; 4.Agricultural and Animal Husbandry College of Tibet University,Linzhi,Tibet 860000,China;5.Qinghai Province Key Laboratory of Crop Molecular Breeding,Xining,Qinghai 810001,China)
In order to characterize the SSRs in hulless barley (HordeumvulgareL. var.nudumHK. f.) transcriptome and annotate the SSR sequences based on bioinformatics analysis,the distribution frequency and basic repeat motifs of SSRs in hulless barley transcriptiome were screened by MISA software. The gene annotation of SSR sequences were obtained by BLASTX against nr,COG,Swiss-Prot and KEGG databases. A total of 11 930 SSRs with 119 kinds of repeat motifs were found in 58 065 Unigenes,distributed in 9 576 Unigenes,which accounted for 16.49% of all Unigenes,and the density of distribution was 6.63 kb per SSR. Among the SSRs in hulless barley transcriptome,the most abundant repeat motif was the tri-nucleotide (64.19%),followed by the di-nucleotide (24.05%). AG/CT and AGG/CCT,CCG/CGG and AGC/CTG were the superior type in di-,and tri-nucleotide repeat motif(s).Most of the repeat number of SSRs was from 5 to 12,and the length of the motif ranged from 12 to 25 bp,with an average of 21.15 bp. 9 576 SSR sequences were annotated with BLASTX against protein databases (nr,COG,Swiss-Prot and KEGG),of which,7 987,5 559,5 588 and 2 077 were annotated,respectively. The annotation of the SSR sequences in hulless barley suggested that they were mainly related to the basic biological metabolism.
Hulless barley; Transcriptome; SSR; Function annotation
时间:2017-01-16
2016-10-05
2016-12-22
青海省应用基础研究计划项目(2015-ZJ-702);中国科学院“西部之光”联合学者项目;西藏自治区西部提升计划“作物学学科建设”项目(XBTSZWXK-01)
E-mail:xjq1088@126.com
沈裕虎 (E-mail:shenyuhu@nwipb.cas.cn);昌 西 (E-mail:164281890@qq.com)
S512.3;S330
A
1009-1041(2017)02-0175-10
网络出版地址:http://www.cnki.net/kcms/detail/61.1359.S.20170116.1833.008.html

