基于COI基因片段的拟钉螺种类鉴定初探
柯雪梅,邱 旻,林陈鑫,葛 婧,郭志南,程明基,陈 清,陈国伟
基于COI基因片段的拟钉螺种类鉴定初探
柯雪梅1,邱 旻2,林陈鑫3,葛 婧2,郭志南1,程明基2,陈 清2,陈国伟1
目的探讨COI基因片段在拟钉螺种类鉴定中的作用。方法收集来自福建省内多个地区的疑似拟钉螺标本,用形态学方法进行物种鉴定后,取出螺肉提取基因组DNA,通过PCR的方式扩增COI基因序列片段并测序,通过序列比对及系统进化分析鉴定其物种。结果共采集到标本13份,通过形态学方法将样本鉴定为福建境拟钉螺属。所有标本经实验扩增到长度为515~598 bp的COI基因片段。13份标本中11株的COI基因片段与γ拟钉螺属最为相似(88.96%~97.82%),另外2株分别与拟钉螺属下的武鸣拟钉螺(87.08%)及新拟钉螺属下的开放新拟钉螺(88.55%)最为相似。在属水平上,13份标本中仅有1份的形态学鉴定结果与基因鉴定结果吻合,其余12份样本的形态学鉴定结果均与基因鉴定不同,两种方法的鉴定结果存在较大差异。结论通过COI基因检测可快速对拟钉螺亚科作出准确判断,但对属水平的鉴定的准确性还需要进一步的研究。
拟钉螺亚科;COI基因;物种鉴定
拟钉螺亚科(Triclulinae)隶属中腹足纲(Gastropoda)、前鳃亚纲(Prosobranch)、圆口螺科(Pomatiopsidae),目前主要分布于东南亚和中国南部[1]。已发现的拟钉螺亚科物种已达120种,其中在中国有近50 种,主要分布在长江流域地区及其以南的地区[1-2]。拟钉螺亚科的拟钉螺属(Tricula)、γ拟钉螺属(Gammatricula)以及新拟钉螺属(Neotricula)被认为是裂体属亚洲组血吸虫和(或)并殖吸虫的重要中间宿主[3-5],因此,掌握并了解拟钉螺亚科种类的分布情况对监控与预警上述两种寄生虫病有着重要的意义。传统的拟钉螺亚科物种鉴定方法采用形态学鉴定法,即按照螺个体的外壳特征和齿舌特征对其进行分类。但由于螺个体较小,部分形态学特征差异难以正确分辨,给分类工作带来很大的困难,容易错分。故近来许多学者开始使用分子标记即基因分类法作为分类研究的重要手段。细胞色素C氧化酶亚单位I(Cytochrome c oxidase subunit I,COI)基因是所有分子标记中使用较为广泛的一种。本文报告了一种基于COI基因的拟钉螺亚科物种分类方法,并对该方法在拟钉螺亚科物种鉴定中的应用价值做了初步探讨。
1 材料与方法
1.1 样本采集 从厦门市、福州市、漳州市、南平市等4市的淡水河(溪)流中采集疑似拟钉螺亚科物种共计13份,每份约100~150只螺。采集到的样本在经清水清洗后,进行初步的形态学鉴定。鉴定完毕后的螺体浸入75%乙醇溶液,置于-80 ℃冰箱冻存备用。
1.2 基因组DNA提取 从每份样本中取5~10枚拟钉螺,去除螺壳及其内脏,使用Earl’s平衡盐溶液洗净后一并置于研钵中研磨,将研磨后的螺体溶于Earl’s平衡盐溶液制成匀浆液。取200 μL匀浆液,使用Promega DNA extraction kit提取其中的总基因组DNA。提取到的DNA经检纯度、浓度合格后贮藏于-20 ℃冰箱备用。
1.3COI基因片段扩增及产物鉴定 采用Davis等[6]设计的针对拟钉螺COI基因设计的PCR引物对获得的基因组核酸进行扩增,正向引物序列为:5′-GGTCAACAAATCATAAAGATATTGG-3′,反向引物序列为:5′-TAAACTTCAGGGTGACCAAAAAAYCA-3′,扩增的目标片段的大小为598 bp。采用Promega公司的Master Green Mix试剂盒,参照使用说明构建如下25 μL反应体系:Master Green Mix 12.5 μL,正向引物1.0 μL,反向引物1.0 μL,DNAase Free Water 8.5 μL,基因组DNA2.0 μL。具体PCR反应流程如下:94 ℃预变性10 min,之后94 ℃ 1 min,51 ℃ 1 min 30 s,72 ℃ 1 min 45 s,循环40次, 最后72 ℃延伸10 min。扩增反应完成后,取5 μL产物进行1%琼脂糖凝胶电泳,于紫外灯下观察是否扩增成功。
1.4 扩增子测序及比对 扩增产物送至华大基因公司进行切胶纯化及回收后,使用前述引物进行双向序列测定(采用sanger法),使用Bioedit[7]软件对原始序列进行拼接,获得相应COI基因序列片段。之后使用Bioperl[8]软件将获得序列提交NCBI服务器进行BLAST比对,选取BLAST report中的最优结果对应的物种名称作为样品的参考。
1.5 基因序列分析 依据关飞等[1]的报道,选取我国境内5个省(福建、广西、湖北、浙江、四川)的12种拟钉螺相应的同源区段作为参考序列,并选择我国境内常见的湖北钉螺(Oncomelaniahupensis)作为外群。与本实验中获得的COI基因片段组成数据库进行序列分析:1)使用MEGA7.0[9]软件进行多序列比对;2)基于比对结果计算序列一致性情况;3)根据比对结果进行核苷酸替换模型测试,使用最适的替换模型计算参考序列与检出序列间的进化距离并构建距离矩阵;使用NJ(Neighbor-Joining)法构建系统进化树,并进行1 000次Bootsrap迭代检验进化树的可靠性。
2 结 果
2.1 标本采集及鉴定情况 本研究中采集的13份标本的具体采集地点见表2。
2.1.1 形态学鉴定 根据拟钉螺个体大小,外壳特征及齿舌情况等,将本次采集到的拟钉螺标本鉴定为此前在福建境内多有发现的6种拟钉螺属物种,见表1,图1。
2.1.2 分子鉴定 从13份标本的DNA中均扩增出符合预期大小的COI基因片段(515~598 bp)。经BLAST比对鉴定,11份样品的最优结果为γ拟钉螺属(Gammatricula)。其中采自厦门、福州以及漳州市的标本序列一致性较高,达94.17%~97.82%,而采自南平市的标本序列比对一致性只有87.08%~88.96%。另外,采自南平的NPQ-1的最优结果为武鸣拟钉螺(Triculawumingensis)序列一致性为87.08%,而同为南平的NPQ-2的最优结果为开放新拟钉螺(Neotriculaaperta)序列一致性为88.55%。13份标本中,仅有NPQ-2的2个鉴定结果在属水平一致,但种水平不一致。其它12份均在属水平上不一致,但未有亚科水平结果不一致的情况。
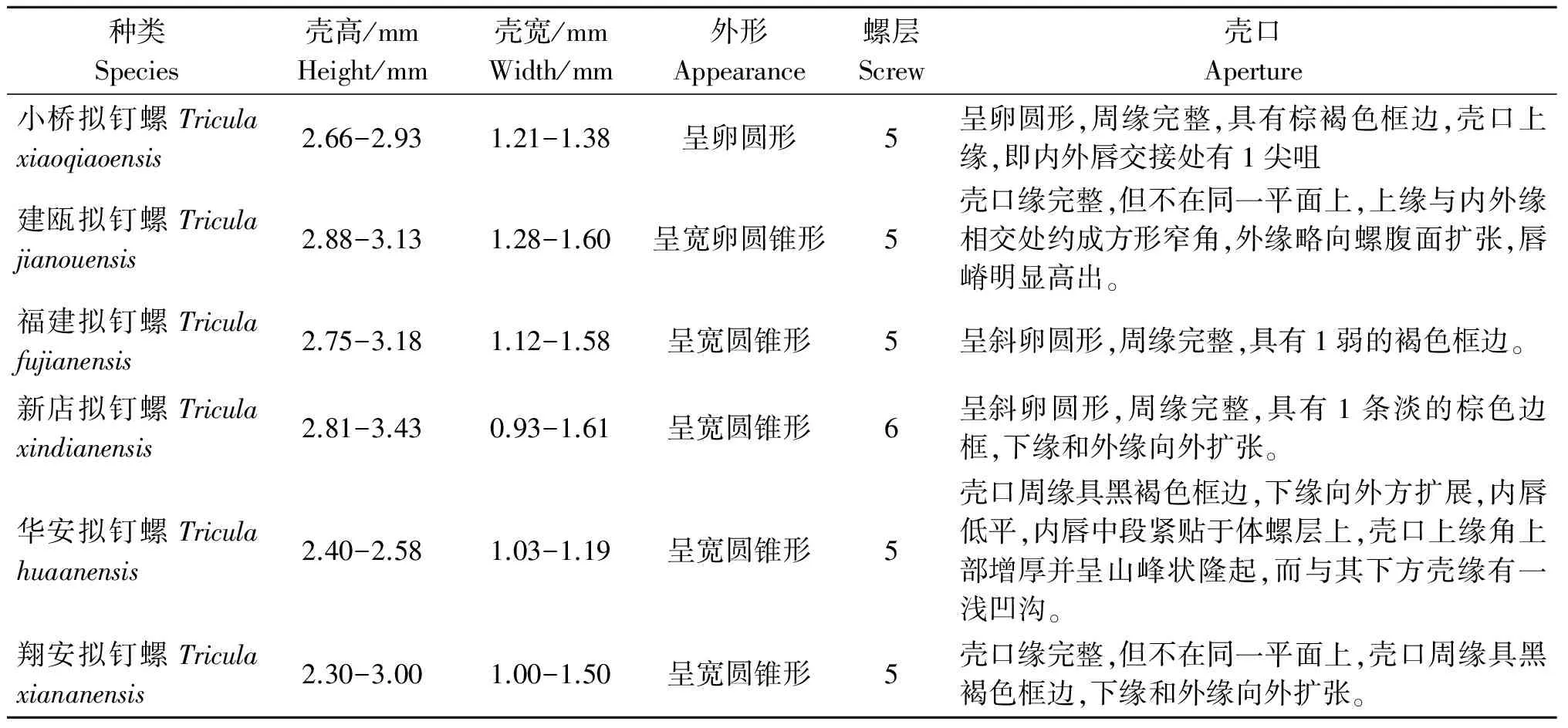
表1 福建省几种拟钉螺的形态特征比较Tab.1 Diagnosis features of Triculinae collected in Fujian Province

1-1小桥拟钉螺1-1 T.xiaoqiaoensis

1-2建瓯拟钉螺1-2 T.jianouensis

1-3福建拟钉螺1-3 T.fujianensis

1-4 翔安拟钉螺1-4 T.xiananensis

1-5新店拟钉螺1-5 T.xindianensis

1-6 华安拟钉螺1-6 T.huaanensis

1-7翔安拟钉螺齿舌带
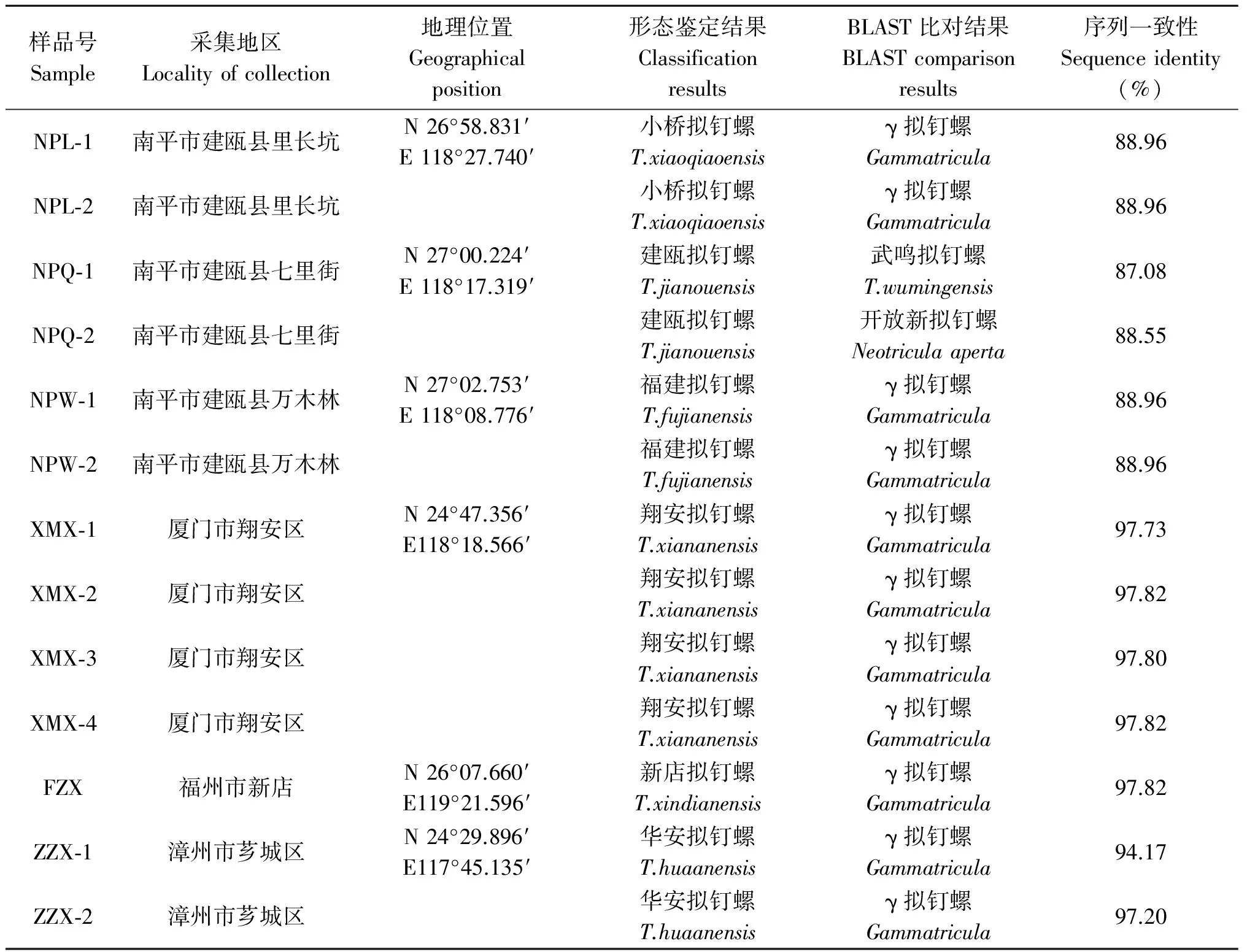
表2 福建境内采集疑似拟钉螺亚科物种标本的鉴定结果Tab.2 Classification of Triculinae collected in Fujian Province
2.2 系统进化分析 本研究中采用的参考序列的名称、来源地区以及GenBank登录号见表3。
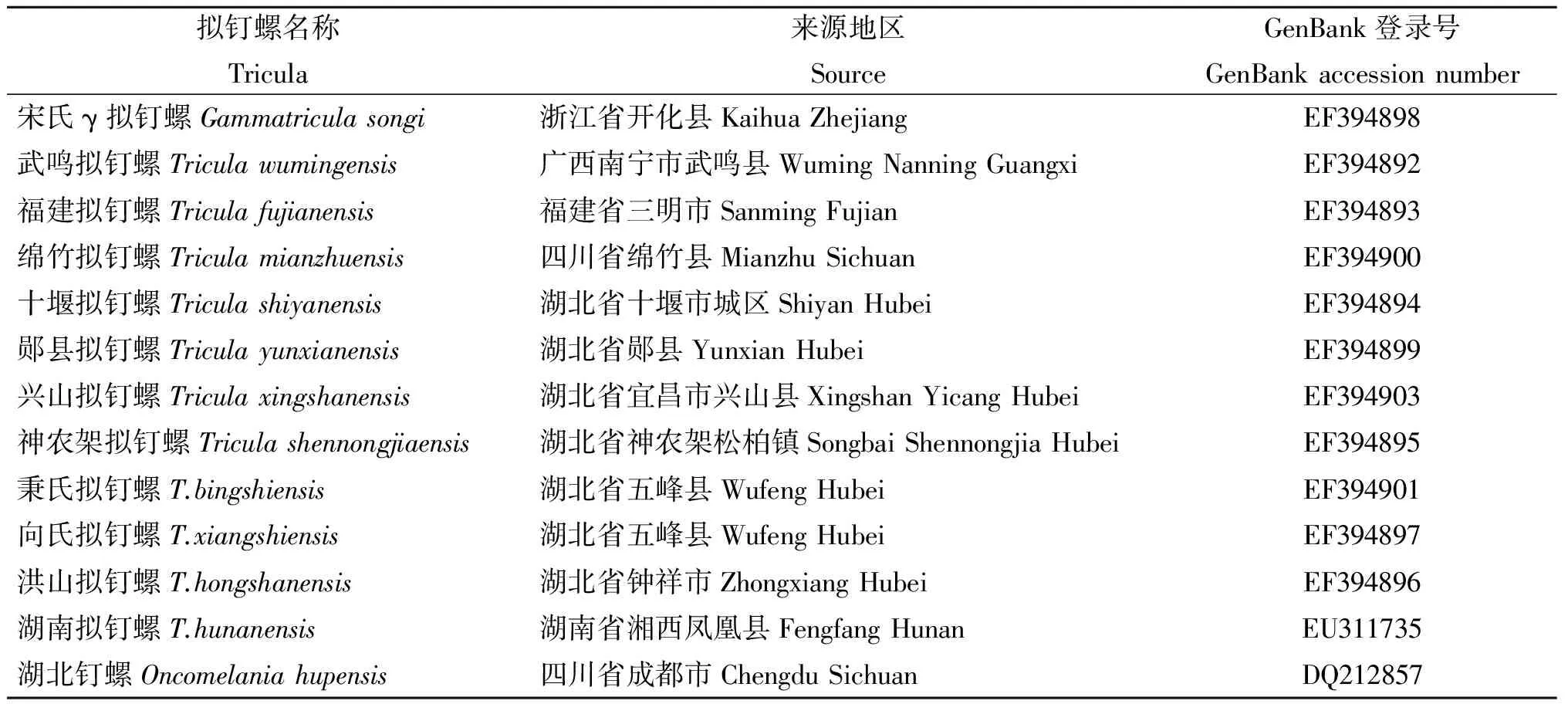
表3 研究中使用的拟钉螺参考序列信息Tab.3 Reference sequences used in this study
经计算,本次研究中的序列集最适用的替换模型为Tamura 3-parameter,故使用此模型估算各序列间的进化距离,并构建NJ树如图2。从计算结果看,本实验中采集的大部分拟钉螺与我国境内原先报道的拟钉螺在基因水平上有一定差异,COI基因序列一致性在85%~90%之间,进化距离为0.109~0.201不等。从进化树上可见,除南平七里街(NPQ-1 NPQ-2)的2份标本与武鸣拟钉螺聚为一支外,其它株均不与国内已知的任何一种拟钉螺聚为一支,而是独自成支,本研究中采集样本的序列相互之间相似度高,进化距离较近,相关分支的bootstrap值较高,表明该聚类结果较为可靠。
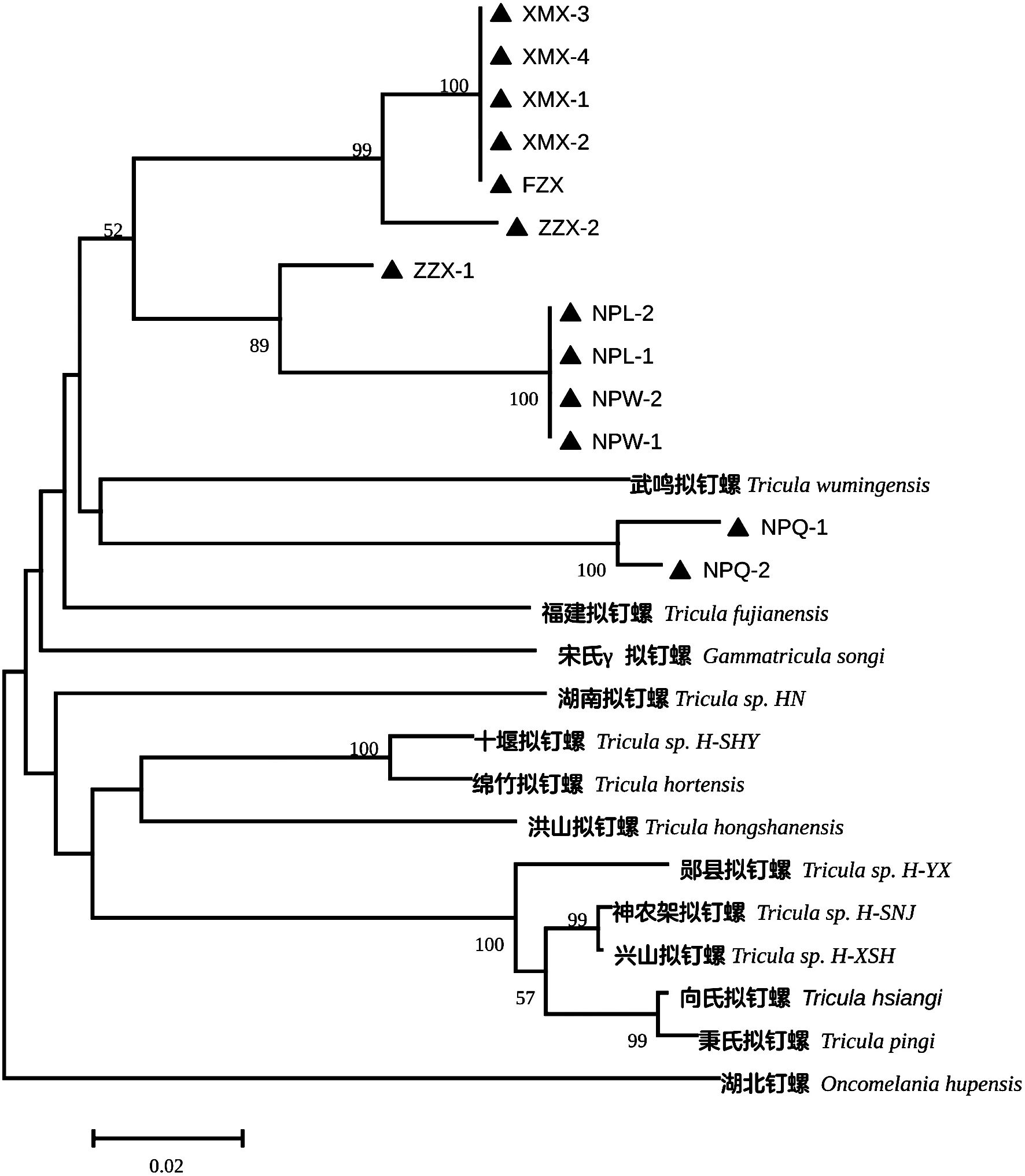
(带“▲”为本次研究中获得序列)(Sequences originally obtained in this study were marked with “▲”)图2 实验获得COI基因序列与参考序列通过邻接法构建的系统进化树Fig.2 NJ-Tree based on partial sequences of COI gene obtained in this study and reference sequences
3 讨 论
在本研究中,形态学鉴定和基因鉴定在拟钉螺亚科水平上鉴定结果完全一致,但是在属鉴定的结果间存在着较大的差异,这种差异的来源可能有如下几点:
首先,形态学的鉴定方法依赖物种间表观形态上的差异。对于拟钉螺这种个体特别小的淡水螺来说,其外壳、齿舌等特征差异并不明显,这无疑使鉴别工作变得十分困难。即使在李翠娥[10]等提出以细部解剖资料作为依据进行鉴别参考后,仅从形态及解剖学上对拟钉螺进行精细区分仍然困难重重。而小桥拟钉螺、建瓯拟钉螺、华安拟钉螺等在最初被记述、分类并命名时,往往仅以形态学与解剖学特征为基础[11-12],加之过去研究中确实多有发生仅依靠外观而错判为拟钉螺属的情况[10],因此,对于厦门、福州、漳州采集的7份标本,倾向于认为系最初分类的偏差及命名的错误导致了形态学鉴定上的偏差,这7份标本实际上应该属于γ拟钉螺。
另一方面,基于分子标记的分子鉴别方法虽然简单易行,但在实际应用中也需进一步评价和完善。该方法的本质是通过比对,寻找已知数据库内与被提交序列最为相似者,然后以这条序列的注释信息作为鉴定结果。这种方法的准确性依赖于两个重要因素:①提交序列本身的测序质量;②已知数据库的完备与准确。对于前者,由于实际样本情况的差异与实验条件的限制,难以保证所有样品的测序都能达到高质量且均一;至于后者,由于分子标记鉴定相关数据库仍在发展及完善中,难免出现库中数据不全或者不准确的情况。Buhay[13]等就曾提及现有的包含COI基因的数据库如NCBI Genbank及Barcode of Life(BOL)等在进行数据收录时并未收集数据的质量信息,因此研究者若想使用,只能自己进行相关的质控与筛选工作。综上,若相关方法及数据库在某一个物种上尚不成熟,则基因鉴定同样会出现偏差。比如本研究中所有采自南平的标本,虽然都能通过BLAST获得注释结果,但由于序列一致性并不算特别高,故不排除由于数据库不完备而发生了注释上的偏差的可能。因此相关的6份标本尚无法判明种属。
本研究表明,用COI基因片段对拟钉螺种类进行鉴定,在亚科水平上可得到与形态学鉴定完全一致的结果。但是在属鉴定水平上则差异较大。提示在基因鉴定方面仍需开展更多研究,例如选用多个分子标记联合判断、不断完善拟钉螺亚科相关的基因数据库信息等。
[1] Guan F. Molecular systematic study on some species of Triculine[D]. Wuhan: Huazhong University of Science & Technology, 2009. (in Chinese)
关飞. 拟钉螺亚科部分种类分子系统发育研究[D]. 武汉:华中科技大学,2009.
[2] Liu L, Huo G, He H, et al. A phylogeny for the pomatiopsidae (Gastropoda: Rissooidea): a resource for taxonomic, parasitological and biodiversity studies[J]. BMC Evolutionary Biol, 2014, 14(1): 29. DOI: 10.1186/1471-2148-14-29
[3] Li YS, Zen SP, Zhang SY, et al. Investigation on the first intermediate host of paragonimus and its geographic distribution and infection rate in Fujian, China[J]. Strait J Prevent Med, 2004, 10(06): 1-3. (in Chinese)
李友松,曾森平,张世阳,等. 福建省并殖吸虫第1中间宿主种类、分布及感染率的调查[J]. 海峡预防医学杂志,2004.10(06):1-3.
[4] Guo YH, Gu JR, Chen PL, et al. Study onTriculaand the related schistosomes[J]. Chin J Schistosomiasis Ctrl,1989, 1(1): 22-27. (in Chinese)
郭源华,顾金荣,陈溥林,等. 拟钉螺及其有关血吸虫的研究[J]. 中国血吸虫病防治杂志,1989.1(1):22-27.
[5] Guo YH, Ni CH, Xia F, et al. Tricula and its importance on medicine[Z]. Huanshan Anhui China: 1988.
[6] Davis GM, Wilke T, Zhang Y, et al. Snail-Schistosoma,Paragonimusinteractions in China: population ecology, genetic diversity, coevolution and emerging diseases[J]. Malacologia, 1999, 41(2): 355-377.
[7] Hall TA. BioEdit: a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT[C]. 1999.
[8] Stajich JE, Block D, Boulez K, et al. The Bioperl toolkit: Perl modules for the life sciences[J]. Genome Res, 2002, 12(10): 1611-1618. DOI: 10.1101/gr.361602
[9] Tamura K, Dudley J, Nei M, et al. MEGA4: molecular evolutionary genetics analysis (MEGA) software version 4.0[J]. Mol Biol Evolut, 2007, 24(8): 1596-1599. DOI: 10.1093/molbev/msm092
[10] Chen CE, Kang ZB, Liu YY. Snail hosts ofParagonimusin Asia and America[J]. Chin J Parasitol Parasot Dis,1994, 4(12): 279-284. (in Chinese)
陈翠娥,康在彬,刘月英. 亚洲和美洲的并殖吸虫的螺类宿主[J]. 中国寄生虫学与寄生虫病杂志,1994.4(12):279-284.
[11] Li YS, Lin CX, Zhou XM, et al. A new species of the Triculinae of freshwater snails that harbours cercariae of lung flukes from Fujian, China.[J]. Chin J Zoonoses, 2004, 10(20): 883-884. (in Chinese)
李友松,林陈鑫,周宪民,等. 携带肺吸虫尾蚴的淡水螺类拟钉螺亚科一新种(前鳃亚纲;中腹足目;圆口螺科;拟钉螺亚科)[J]. 中国人兽共患病杂志,2004.10(20):883-884.
[12] Cheng YZ, Wu XP, Li LS. Two new species ofTricula(Mesogastroppda, Pomatiopsidae) from China[J]. Zoologic Systematics, 2010, 4(35): 871-875.
[13] Buhay JE. “COI-like” sequences are becoming problematic in molecular systematic and DNA barcoding dtudies[J]. J Crustacean Biol, 2009, 29(1): 96-110. DOI: 10.1651/08-3020
Chen Guo-wei, Email: xmcdcxsk@163.com
SpeciesidentificationofTriculinaebasedonCOIgenesegment,FujianProvince
KE Xue-mei1, QIU Min2, LIN Chen-xin3, GE Jing2, GUO Zhi-nan1, CHENG Ming-ji2, CHEN Qing2, CHEN Guo-wei1
(1.XiamenCenterforDiseaseControlandPrevention,Xiamen361021,China; 2.PublicHealthCollegeofSouthernMedicalUniversity,Guangzhou510515,China; 3.FujianCenterforDiseaseControlandPrevention,Fuzhou350001,China)
The aim of the study is to evaluate the utility value of species identification of Triculinae based onCOIgene. Triculinae-like samples were collected from different places in Fujian Province. After classification based on morphology characters, genomic DNA was extracted from collected samples. Then the segments ofCOIgene were amplified and sequenced for taxonomy annotation and phylogenetic analysis. Eleven samples were annotated asGammatricula(identities: 88.96%-97.82%), the other two were annotated asTriculawumingensis(identity: 87.08%) andNeotriculaapart(identity: 88.55%), respectively. In most cases (12 out of 13), there was a difference between results based on different classification methods on a genus level. The alignment ofCOIgene segment is sufficient for preliminary identification of Triclulinae at family level. However, it is need further study in species identification of Triclulinae at genus level.
Triclulinae;COI; species classification phylogenetic
10.3969/j.issn.1002-2694.2017.08.012
福建省科技厅项目(No.2011D015)
陈国伟,xmcdcxsk@163.com
1.厦门市疾病预防控制中心,厦门 361021; 2.南方医科大学公共卫生学院,广州 510515; 3.福建省疾病预防控制中心,福州 350001
Supported by the Science and Technology Agency og Fujian Province (No. 2011D015)
R38
:A
:1002-2694(2017)08-0724-06
2016-11-03编辑:刘岱伟

