CRF与词典相结合的疾病命名实体识别*
龙光宇,徐 云,3
(1.中国科学技术大学 计算机科学与技术学院,安徽 合肥 230027;2.中国科学技术大学 安徽省高性能计算重点实验室,安徽 合肥 230027;3.国防科学技术大学 高性能计算协同创新中心,湖南 长沙 410073)
CRF与词典相结合的疾病命名实体识别*
龙光宇1,2,徐 云1,2,3
(1.中国科学技术大学 计算机科学与技术学院,安徽 合肥230027;2.中国科学技术大学 安徽省高性能计算重点实验室,安徽 合肥230027;3.国防科学技术大学 高性能计算协同创新中心,湖南 长沙410073)
生物医学文献中的疾病命名实体识别问题是疾病相关的生物信息学分析基础,疾病命名实体中的医学术语识别和边界确定是该问题的难点和关键。文中提出了一种CRF(ConditionalRandomField)与词典相结合的疾病命名实体识别方法。该方法利用网络资源来构建含有语义信息的医学术语词典,并使用该词典对医学术语进行识别,获得医学术语的语义信息,然后CRF结合这些信息对疾病命名实体进行识别。实验结果表明该方法有效。
疾病命名实体识别;医学术语词典;条件随机场
0 引言
随着基因技术的飞速发展,人们已经越来越清楚地认识到人类疾病的发生发展几乎都直接或间接地与基因有着密切的联系[1]。从生物医学文献中挖掘疾病与基因之间的关联对于疾病预防以及新药研制都有着重要的意义。伴随着生物医学文献爆炸性的增长,通过人工的方式来获取疾病-基因之间的联系明显是不可能的。随着自然语言处理及大数据等技术的发展使得自动从文献中发现疾病-基因之间的关系成为可能,其中疾病命名实体识别完成了这个任务的第一步。
目前解决疾病命名实体识别问题的主流方法是机器学习,但是识别效果还未达到基因、蛋白质的效果[2]。在对PharmGKB(Pharmacogenetics Knowledge Base)药理学知识库[3]中的大量疾病名称进行了详细的分析之后,发现绝大部分疾病名称都是由一些具有相似语义的医学术语组合而成,比如很多疾病命名实体都是由描述身体部分的词语和基本疾病术语组合而成,例如“lung cancer”和“breast cancer”都是由描述身体部分的“lung”和“breast”结合基本疾病术语“cancer”构成。疾病名称中的这些医学术语对疾病名称的识别具有很强的指示作用。
为了利用疾病命名实体中的医学术语信息进一步提高对疾病命名实体的识别效果,本文提出了一种与词典相结合的CRF(Conditional Random Field)模型去识别疾病实体。首先利用网络资源构造了一个包含疾病相关医学术语及其语义类别信息的词典,再利用词典获取文本中词语的语义类别信息,并把这些语义类别信息作为特征加入到CRF模型中。实验表明,医学术语词典提升了CRF模型的识别效果。
1 相关工作
1.1命名实体识别
解决命名实体识别有三种基本方法:基于词典的方法、基于规则的方法和基于机器学习的方法。
基于字典的方法是最简单的命名实体识别方法,该方法使用词典来匹配文献中的实体名称,其性能取决于词典的质量以及选取的匹配算法的性能。由于词典的容量有限,且该方法并不能识别未登录词,加上疾病命名不规范、变体多,单独使用基于词典的命名实体识别方法难以取得较好的结果。
基于规则的方法是利用正则表达式描述出命名实体中的词形、词性、语法等方面的特征,形成规则。基于规则的NER系统通常依赖于由领域专家设计的规则,耗时耗力,移植性很差。
基于机器学习的方法是目前解决命名实体识别问题的主流方法。目前常用的机器学习模型有很多,如支持向量机(SVM)、隐马尔可夫模型(HMM)、最大熵模型(ME)、条件随机场(CRF)等。近年来CRF模型被广泛地应用于生物体医学命名实体识别任务中,并取得了良好的效果。例如杨娅[4]等人提出了一个结合疾病词典特征的CRF模型去识别疾病实体,再利用全称-缩写词对进行后处理,在NCBI[5]语料库上取得了83.82%的F值。
1.2CRF模型
CRF是由Laffetry等人在最大熵模型和隐马尔科夫模型的基础上提出的统计序列标注算法。CRF具有很多优点,不仅放松了隐马尔科夫模型的条件独立性,而且能在一定程度上解决标记偏置问题。目前已经成功应用于生物文本挖掘的诸多课题,例如生物命名实体识别、关系抽取等。
一阶链式CRF是最简单的CRF模型,本文使用的就是一阶链式CRF。将CRF模型应用于生物医学命名实体识别任务中,就是把输入的句子中的单词序列作为观察序列o,标注的过程就是根据已知的单词序列来推断最有可能的标记序列s,即求解出p(s|o)取得最大值时的状态序列s。一阶链式CRF模型中的状态序列s的条件概率如下式:

其中,z是归一化因子,它是为了使所有状态序列的概率和为1。fk(si-1,si,o,i)是二值特征函数,λk是特征的权重,通过训练可以得到。通常采用L-BFGS迭代算法对CRF模型进行参数估计,使用维特比算法解码。
2 CRF与词典相结合的疾病实体识别
2.1CRF与词典相结合的疾病实体识别思路
CRF与词典相结合的疾病命名实体识别方法的具体思路如下:把疾病实体中的医学术语按照语义分为:身体部分术语、临床表现术语、疾病基本术语以及其他,并利用网络资源构造一个包含这些医学术语及其语义类别的词典。先利用词典获得文中词语的语义信息,并把语义信息传递给CRF模型作为特征去识别文中疾病实体。
2.2医学术语词典构造
通过对PharmGKB药理学知识库中大量疾病实体的详细分析,本文把疾病实体中的医学术语按语义分为四类:描述身体部分的术语(记为“BB”)、描述临床表现的术语(记为“BS”)、疾病基本术语(记为“BD”)以及其他(记为“BO”)。医学术语的具体分类信息见表1。

表1 医学术语分类说明
本文使用PharmGKB药理学知识库中的疾病资源文件来构造包含身体部分术语、临床表现术语和疾病基本术语的医学术语词典。下面介绍医学术语词典的构造步骤:
(1)把PharmGKB药理学知识库中的3 204个疾病实体名称(不包括每个疾病实体所对应的同义词)所对应的字符串,分割成单词,得到一个词汇表;
(2)对词汇表去除重复项、根据PubMed停用词表去除停用词;
(3)人工把剩余的词汇根据语义分为身体部分术语、临床表现术语、基本疾病术语以及其他。把前三类的医学术语词汇连同它们的类别标记一起存入医学术语词典。
传统的基于词典的命名实体识别方法中的词典存储的是实体名称及其同义词,本文中构造的医学术语词典存储的是医学术语单词及其语义类别。词典里每个条目都包括医学术语和它的类别,例如“lung,BB”、“ataxia,BS”、“nephritis,BD”等。
在医学术语词典构造完成之后,利用该词典去匹配待处理文本中的医学术语,结果返回医学术语的语义类别。
2.3CRF与词典相结合的疾病命名实体识别
一个好的特征集合是生物医学命名实体识别任务取得成功的关键,本文中的特征集合主要包括词法特征、领域特征以及医学词典特征三大类。
(1)词法特征:包括单词特征、词块特征、词性特征。
(2)领域特征:包括构词特征、词形特征、词缀特征、边界词特征、上下文特征、关键词特征。
(3)医学术语词典特征
除了以上这些特征,本文还定义了一个医学术语词典特征,该特征就是医学术语词典对当前单词的识别结果,包括“BB”、“BS”、“BD”和“BO”。因为绝大多数疾病命名实体的长度都是1~5,因此设定大小为5的上下文窗口,并设计了一元、二元、三元特征模板。
3 实验与分析
3.1实验语料
3.2实验结果及分析
本文在NCBI语料上进行了两组对比实验。
第一组是利用词法特征和领域特征作为特征集合的CRF模型,训练集上训练,测试集测试,以LEAMAN R[6]等人的疾病命名实体识别模型DNorm对比,实验结果如表2所示。

表2 CRF与DNorm实验结果对比
第二组是在第一组的CRF模型上加入医学术语词典特征,在训练集上训练,测试集测试。以第一组中的CRF模型实验结果作为对比,实验结果如表3所示。
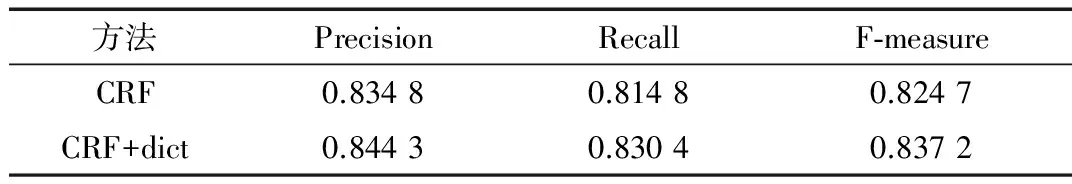
表3 ours与DNorm实验结果对比
第一组对比实验结果表明本文中的CRF模型的实验结果和DNorm相当,说明了利用本文中的词法和领域特征集合建立的CRF模型是有效的。
第二组对比试验表明结合了医学术语词典的CRF模型的识别效果有所提升。分析实验结果发现,导致识别效果提升的原因是一些不具有明显特征的疾病名称也被结合医学术语词典的CRF准确地识别了出来,例如“demyelination of the cerebral white matter”。单一CRF模型是通过特征来区分实体和非实体,因此难以捕捉到那些特征不明显的实体。本文中的医学术语词典能准确地识别出这些医学术语(“demyelination of the cerebral white matter”中“demyelination”(脱髓鞘)、“cerebral”(脑)都是表示身体部分的医学术语),并利用这些医学术语的语义信息为CRF模型提供支持,这正是本文模型的优势。
相比于未结合医学术语词典的CRF模型,医学术语词典提供了更多的语义信息,提升了CRF模型的识别效果。
4 结论
针对疾病命名实体识别问题,本文提出了CRF与词典相结合的疾病实体识别方法。在NCBI语料上进行的实验表明,含有语义信息的词典提升了CRF的识别效果。本文的研究还可以进一步完善,比如可以考虑更多特征继续提高疾病命名实体的识别效果。有效的疾病命名实体识别模型的建立,为后续从生物医学文献中自动抽取疾病-基因之间的关系打下了良好的基础。
[1] BOTSTEIN D,RISCH N.Discovering genotypes underlying human phenotypes: past successes for mendelian disease,future approaches for complex disease[J].Nature genetics,2003,33: 228-237.
[2] JIMENO A,JIMENEZ-RUIZ E,LEE V,et al.Assessment of disease named entity recognition on a corpus of annotated sentences[C].BMC Bioinformatics.BioMed Central Ltd,2008,9(Suppl 3): S3.
[3] HEWETT M,OLIVER D E,RUBIN D L,et al.PharmGKB: the pharmacogenetics knowledge base[J].Nucleic Acids Research,2002,30(1): 163-165.
[4] 杨娅.生物医学文本中的疾病实体识别和标准化研究[D].大连:大连理工大学,2015.

Combining CRF and dictionary based disease named entity recognition
Long Guangyu1,2,Xu Yun1,2,3
(1.School of Computer Science and Technology,University of Science and Technology of China,Hefei 230027,China;2.Key Laboratory of High Performance Computing of Anhui Province, University of Science and Technology of China, Hefei 230027, China;3.Collaborative Innovation Center of High Performance Computing, National University of Defense Technology, Changsha 410073,China)
The problem of disease name entity recognition in biomedical literature is the basis of bioinformatics analysis related to disease.The identification of the medical terms in disease named entity and determining the boundary of the disease named entity are the key and difficult points of the problem.This paper presents a method that compining CRF (Conditional Random Field) and a dictionary to solve the problem of disease named entity recognition.This method constructs a medical terminology dictionary which contains semantic information using network resources.Firstly,it uses the dictionary to identify the medical terminology,then obtains the semantic information of medical terminology.Then CRF combined with these semantic information to identify the disease named entity.The experimental results verify the effectiveness of the proposed algorithm.
disease named entity recognition; medical terminology dictionary; conditional random field
TP181
A
10.19358/j.issn.1674-7720.2017.21.016
龙光宇,徐云.CRF与词典相结合的疾病命名实体识别J.微型机与应用,2017,36(21):51-53.
国家自然科学基金(61672480)
2017-04-14)
龙光宇(1991-),男,硕士研究生,主要研究方向:生物文本挖掘。
徐云(1960-),男,博士,教授,博士生导师,主要研究方向:大数据挖掘,生物信息学应用等。

