茶树Copine家族基因CsBON3的克隆与表达分析
姚利娜,郝心愿,王璐,李娜娜,曾建明,杨亚军,王新超
中国农业科学院茶叶研究所/国家茶树改良中心,农业部茶树生物学与资源利用重点实验室,浙江 杭州 310008
茶树Copine家族基因CsBON3的克隆与表达分析
姚利娜,郝心愿,王璐,李娜娜,曾建明,杨亚军*,王新超*
中国农业科学院茶叶研究所/国家茶树改良中心,农业部茶树生物学与资源利用重点实验室,浙江 杭州 310008
Copine蛋白是一类包含2个C2(N端)和1个vWA(C端)保守域的Ca2+依赖蛋白或磷脂结合蛋白,在胞内信号转导中发挥重要作用。基于序列相似性分析,从茶树转录组数据库中筛选出1条与Copine家族基因高度同源的EST序列。经测序验证该序列包含1 746 bp的完整ORF,编码581个氨基酸。同源比对显示该基因与拟南芥AtBON3序列相似度最高(65%),将其命名为CsBON3(GenBank登录号为 KY435900)。生物信息学分析显示,CsBON3蛋白分子量为63.66 kD,理论等电点为5.48;具有Copine家族蛋白特有的保守结构域;属亲水性蛋白,无信号肽位点,非分泌性蛋白,无跨膜结构域。表达分析表明,CsBON3在茶树花和根系中表达量最高,茎干和成熟叶中表达量最低。低温(4℃)处理茶树 1 d后,其表达被显著上调;在生长阶段,该基因表达量高于休眠阶段;同时在接种炭疽菌的茶树叶片中,该基因也被快速上调,表明该基因可能与茶树低温、生长发育及抗病相关。
茶树;Copine家族;BON基因;逆境胁迫响应;休眠;表达分析
Copine蛋白在1998年首次从草履虫中分离得到,是一种高度保守具有重要功能的蛋白[1]。随后,该蛋白在哺乳动物、植物、线虫中也相继被发现。Copine蛋白家族共有两种保守结构域,氨基端2个C2结构域(C2A和C2B),羧基端1个von Willebrand factor A(vWA)结构域[1]。C2结构域具有钙依赖性磷脂结合蛋白的功能,即在 Ca2+的作用下,和磷脂结合使该蛋白定位在膜上[2],如蛋白激酶 C、磷脂酶 C[2-3]等具有囊泡运输功能的蛋白中均有C2结构域。vWA结构域与整合蛋白的结构域同源,整合蛋白具有介导细胞和细胞之间,以及细胞和细胞外基质之间的相互识别作用[4-6]。
Copine蛋白家族在哺乳动物中研究较深入,发现该蛋白家族具有许多重要的功能,如信号转导、膜转运、离子通道等。至目前研究为止,在哺乳动物中Copine蛋白共有8个家族成员,被命名为Copine I~Copine VIII,每个家族都具有不同的功能。然而,植物中Copine蛋白研究较少,植物中已经发现的Copine基因成员只有 1~3个,如拟南芥、杨树、棉花等物种中仅有3个基因成员(BON1、BON2、BON3),马铃薯、大豆、水稻、玉米等物种中仅有2个基因成员(BON1和BON3),而北美云杉、互叶梅、卷柏等物种中仅发现1个基因成员(BON1)[7]。目前只有拟南芥中的 Copine家族基因的功能被鉴定,认为具有调节植物生长发育和抗病的作用[8]。
茶树作为多年生常绿经济作物,其生长周期内会受多种逆境的胁迫。近年来,随着全球气候变化的影响,各种生物与非生物逆境如病害、低温胁迫等对茶树生长和品质的影响越来越频繁,严重影响了茶叶生产的经济效益。如何提高茶树的抗性也成为茶学研究的主要研究课题。笔者在实验室前期的茶树转录组数据库中筛选到了 1条与拟南芥AtBON3同源的EST片段,该基因在不同冷驯化样品之间表达有差异[9]。为进一步了解茶树BON基因的功能,笔者克隆了CsBON3基因,并对该基因进行了全面的生物信息学分析和表达检测,以期深入揭示BON基因在茶树抗性和生长发育调控中的作用。
1 材料与方法
1.1 试验材料及处理
试验材料为国家级茶树品种龙井 43,种植于中国农业科学院茶叶研究所内试验基地。组织表达特异性取样方法参照岳川等[10]所述,低温胁迫取样方法参照曹红利[11]等所述。在越冬龙井43茶树进入休眠时期,即从2015年10月初到2016年3月底,每隔1个月左右取 1次顶芽进行CsBON3的表达分析。以 3年生茶树品种龙井43为材料,人工气候室正常培养(25℃,16 h光照/8 h黑暗,湿度65%),对照组用喷壶均匀向整株茶苗喷清水,处理组用喷壶均匀向整株茶苗喷炭疽菌;在接种0、12、24、48、72、96 h后取茶树叶片进行CsBON3基因表达分析。以上取样及处理均设3个生物学重复,取样后立即放入液氮,置于-80℃冰箱中保存备用。
1.2 总RNA的提取及cDNA的合成
按照 CTAB法提取不同处理样品的总RNA,用NanoDrop ND2000微量核酸蛋白测定仪测定RNA浓度,1%变性琼脂糖凝胶电泳检测总 RNA质量,参照 Prime Script RT Reagent反转录试剂盒说明书反转录合成cDNA用于基因克隆和表达分析。
1.3 CsBON3基因克隆
以龙井43芽的cDNA为模板,依据EST序列设计引物进行RT-PCR验证。PCR反应体系采用 KOD-Plus-Neo酶(TOYOBO, Osaka,Japan),退火温度为 55℃。PCR反应后,再加入rTaq 聚合酶(TakaRa, 大连)于72℃反应20 min进行加尾。终产物利用1%琼脂糖凝胶电泳检测,目标条带回收纯化后,连接到pMD18-T载体,经大肠杆菌(DH5α)转化获得阳性单克隆并测序。引物合成和基因测序均由上海华津生物有限公司完成,本研究所涉及到的引物序列见表1。
1.4 CsBON3的表达分析
以茶树PTB为内参基因[12],荧光定量反应体系SYBR Mix (Roche, Basel, Switzerland)5.0 μL、cDNA 2 μL、上下游引物(10 μmol·L-1)各0.4 μL,加水至终体积10 μL。充分混匀,短暂离心后,于LightCycler 480 II实时荧光定量PCR仪上进行PCR扩增,反应程序为95℃5 min;95℃ 10 s,56℃ 15 s,12℃ 12 s进行45个循环后增加溶解曲线。每个反应设3次重复,最后采用 2–ΔΔCT或2-ΔCT法[13]分析结果。
1.5 生物信息学分析
利用 DNASTAR 软件包中的 Editseq 进行ORF查询;在NCBI数据库中分别用Blastn和 Blastx进行核酸和蛋白质序列的同源性比对分析;在 ClustalX2.0中进行多序列比对和排序后,利用DNAMAN和MEGA6.0输出同源比对和进化树构建结果[14]。用ExPASy网站的ProtParam分析程序预测蛋白分子量、等电点、氨基酸组分等[15],ProtScale程序分析该蛋白的疏水和亲水性,SignalP分析程序进行信号肽的预测[16],TargetP分析程序预测CsBON3蛋白的亚细胞定位,NetPhos分析程序预测蛋白磷酸化位点[17],GOR分析程序预测该蛋白二级结构组成[18]。

表1 RT-PCR和qRT-PCR引物信息Table 1 RT-PCR and qRT-PCR primer sequences
2 结果与分析
2.1 CsBON3的cDNA克隆
通过1%琼脂糖凝胶电泳对RT-PCR引物进行序列验证,获得1.9 kbp左右的单一条带(图 1)。
测序结果发现,该条带长1 987 bp。利用NCBI数据库中的 ORF Find查找该序列的开放阅读框(ORF),然后进行Blastx比对,发现该序列包含完整的ORF,全长为1 746 bp,编码581个氨基酸。与拟南芥的AtBON3序列相似度最高(65%),命名为CsBON3,提交GenBank,登录号为KY435900。

图1 茶树Copines蛋白家族基因CsBON3 RT-PCR电泳结果Fig. 1 Electrophoresis results of Copines family CsBON3 in tea plant
2.2 同源性和进化树分析
在NCBI数据库中对CsBON3的氨基酸序列进行Blastx比对,结果显示它与葡萄中预测的VvBON1(XP_002280913.1)氨基酸序列相似性最高,达80%,与黄胡萝卜(XP_017214747.1)、大豆(XP_003553742.1)的BON相似性分别是78%、77%。用相邻连接法构建进化树显示与葡萄的 VvBON1亲缘关系最近(图2)。
结合聚合进化树分析,从中挑选与CsBON3相似性较高的氨基酸序列进行同源性比对(图3)。所有的 BON氨基酸序列长度为 576~596个氨基酸,都有 Copine家族特有的结构域C2A、C2B、vWA。所有的 BON氨基酸序列在C2结构域中具有高度保守的与钙依赖磷脂结合作用有关的天冬氨酸残基;在vWA结构域中具有如蛋白激酶相似的 ATP结合甘氨酸环激酶序列和推测的赖氨酸催化位点(图3)。

图2 CsBON3的进化树分析Fig. 2 Phylogenetic analysis of the deduced amino acid sequence of CsBON3

图3 茶树与其他植物BON氨基酸序列比对Fig. 3 Multiple alignment of deduced amino acid sequences of CsBON with BON proteins from other plants
2.3 蛋白质序列基本性质分析
蛋白质理化性质分析表明,CsBON3蛋白分子量为63.66 kD,理论等电点(Theoretical pI)为 5.48,不稳定指数(Instability index, II)为30.22,根据Guruprasad法判断该蛋白稳定。CsBON3亲水性和疏水性值在-2.167~2.600之间,表明该蛋白为亲水性蛋白。SignalP信号肽组成分析程序预测 CsBON3蛋白不包含信号肽,属于非分泌蛋白;亚细胞定位预测结果表示该蛋白定位在质膜、细胞质可能性最大。NetPhos分析程序对CsBON3蛋白的磷酸化位点预测显示,该蛋白在翻译后可能存在48个磷酸化位点,其中包含18个丝氨酸(Ser,S)位点,7个苏氨酸(Thr, T)位点和23个络氨酸(Tyr, Y)位点。二级结构预测结果显示,CsBON3蛋白由 25.3% α-螺旋、22.55%的延伸链和 52.15%的无规则卷曲组成。蛋白结构功能预测结果显示,CsBON3蛋白氨基酸序列在 53~162 位、200~299 位、343~540 位之间高度保守,具有 Copine蛋白家族中共有的典型结构域——C2A、C2B、VWA结构域。
2.4 CsBON3基因的表达模式分析
2.4.1 组织表达差异性
CsBON3基因在花和根系中的表达要明显高于茎和成熟叶中的表达,芽中的表达量较花和根系中的表达量都要低,但明显比茎和成熟叶中高(图 4),这表明CsBON3基因具有组织表达特异性。
2.4.2 低温胁迫下的表达模式
低温条件下,CsBON3基因在茶树成熟叶片中的表达与低温胁迫时间相关,低温处理前期表达量变化不显著,但在处理 24 h后,表达量迅速升高,以后随着处理时间的延长表达量维持较高的水平,处理4 d后表达量为处理前的 2.98倍,表明CsBON3基因受低温诱导(图 5)。
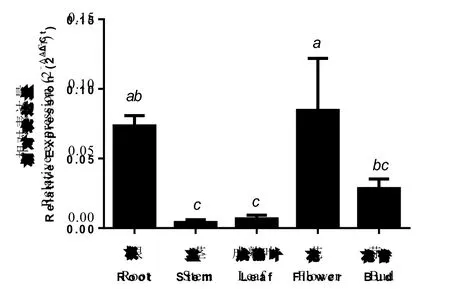
图4 同一品种不同组织器官中CsBON3的表达分析Fig. 4 Expression analysis of CsBON3 in different organs of a tea plant
2.4.3 CsBON3在茶树越冬芽休眠中的表达分析
利用qRT-PCR技术分析了CsBON3基因从秋季茶树顶芽开始休眠到春季萌发期间不同时间点的相对表达情况。可以看出,10月至 11月初,茶芽仍处于发育阶段,CsBON3基因表达上调,而进入11月以后,随着外界光照和温度的变化,茶芽进入休眠阶段,生长相对停滞,该基因表达量降低,在整个休眠阶段,该基因维持在较低表达水平。进入春季以后,日照时间增加,温度提高,茶芽逐渐打破休眠,恢复生长,该基因又上调表达,至 3月初茶芽萌动时表达量最高(图6)。
2.4.4 炭疽菌接种后CsBON3的表达分析
茶树炭疽菌接种 12 h后CsBON3基因表达量达到最大值,其表达量为未处理前的 2.3倍。随后表达逐渐下调,接种 48 h后表达量和未处理前一致,表明接种炭疽菌后短期内能诱导CsBON3基因的表达(图7)。

图5 CsBON3在低温胁迫下的表达分析Fig. 5 Expression analysis of CsBON3 under cold stress

图7 CsBON3接种炭疽菌后的表达分析Fig. 7 Expression analysis of CsBON3 inoculated with colletotrichum spores
3 讨论
3.1 CsBON3 蛋白质结构的特征
本研究从龙井43茶树中获得了1条BON基因,具有已报道的该基因的特征。CsBON3的序列长度、分子质量及等电点等均与其他物种的 BON相近;预测的二级结构与其他的BON高度相似,具有C2结构域特有的氨基酸残基(天冬氨酸)和vWA结构域特有的ATP结合甘氨酸环及推测的赖氨酸催化位点[19];所有的氨基酸序列的第2个氨基酸为甘氨酸,具有独特的肉豆蔻酰化修饰,与BON蛋白定位在质膜上密切相关[20]。
研究表明,BON蛋白氨基酸序列上C2结构域上有3~4个保守的天冬氨酸,是结合钙必不可少的残基位点。氨基酸序列比对发现,与钙离子结合作用相关的天冬氨酸,在CsBON3序列中大部分都存在,相当于AtBON1在C2A结构域中的D63、D69、D122和D124位点,C2B结构域的D209、D215和D269位点(图3中箭头所示)。CsBON3氨基酸序列的 C2B结构域中的第3个天冬氨酸D发生突变,被N取代,由此推测 CsBON3蛋白都具有同AtBON1的钙依赖磷脂结合作用,具体的结合特性可能略有不同。Copine蛋白的von Willebrand factor A (vWA)结构域功能与蛋白激酶相似,蛋白激酶具有保守的激酶基序包括 ATP结合甘氨酸环(AtBON1在 Ala350,Gly353)和推测的赖氨酸催化位点(AtBON1在Lys391)[21](图3中方框所示)。氨基酸序列比对发现CsBON3在vWA结构域具有保守的“ASNG”基序和赖氨酸催化位点,可以推测 CsBON3蛋白具有同 AtBON1蛋白相同的磷酸化的活性和 ATP酶活性。CsBON3氨基酸序列的vWA结构域中具有如整合蛋白中结合镁相关活性的保守残基,同AtBON1的vWA结构域D347、T349、S351、T445和D447位点(图3中加号所示)。vWA结构域的生化活性及功能调节需要我们后期进行更深入的研究才能确定。目前植物中拟南芥已验证有 3条BON序列,水稻已验证有2条BON序列,因此茶树BON基因很可能是多基因家族,新的BON基因有待继续发掘。
3.2 CsBON3 基因的表达
目前哺乳动物中的 Copine蛋白相关家族研究较多,植物中除拟南芥外,Copine蛋白在其他植物上的研究结果较少。拟南芥中有3条Copine基因,分别命名为AtBON1、AtBON2、AtBON3,它们在功能上具有冗余且具有组织表达差异性,过表达BON3基因可以回复bon1-1单突变体的表型[8]。GUS染色表明AtBON1、AtBON2、AtBON3在拟南芥生长发育过程中具有组织表达特异性,AtBON1主要在幼嫩的组织中表达如根尖、伸展的叶[22];AtBON2的表达模式与AtBON1相似,与AtBON1不同的是AtBON2在保卫细胞中表达量很高;而AtBON3在不同的组织中表达量都极低[8]。Zou等[7]研究表明,半定量 RT-PCR显示水稻OsBON1的各个组织表达都比OsBON3要低,如根系、茎、嫩叶、成熟叶、节点和花序;且OsBON1在叶、节点和花序中表达相对较高,在茎和根系中表达较低,而OsBON3在茎和节点中表达较高,在成熟叶中表达较低。本研究结果表明CsBON3具有明显的组织表达特异性,CsBON3基因在花和根系中的表达要明显高于茎和成熟叶中的表达,芽中的表达量较花和根系中表达量都要低,但明显比茎和成熟叶中高(图4)。
拟南芥在较大的温度范围内都会维持相似的大小和形态,Hua等[22]研究发现,bon1-1突变体在22℃时生长的形态与野生型(Col-0)表型有很大的差异,bon1-1突变体植株具有矮小、叶片卷曲、茎细且短等表型;bon1-1突变体在28℃时的表型与野生型(Col-0)基本一致,表明BON1基因和BAP1在低温下可能与细胞分裂和细胞扩增有关。本研究表明在4℃处理1 d后CsBON3表达量在成熟叶中显著升高,且随着处理时间的延长表达量一直维持较高的水平,表明在低温处理下CsBON3基因可能参与茶树叶片的低温响应。
Yang等[8]通过双突变和三突变的组合发现拟南芥的AtBON1/2/3基因在植物的生长发育过程中具有功能重叠的作用,双突变bon1-1bon2-2和bon1-1bon3-3还会引起细胞发生程序性死亡。本研究发现茶树越冬芽休眠过程中,CsBON3在深休眠期间(次年1月)表达量最低,随着休眠解除,CsBON3表达量逐渐上调,并在茶芽萌动期达到最大值(图6),表明CsBON3基因可能参与茶树芽的生长发育。
在拟南芥中,AtBON1在植物免疫中具有重要作用,研究发现AtBON1可以负调节抗病性基因SNC1[23];AtBON2、AtBON3单突变后对植物的免疫性作用并未有较大作用,但可以和AtBON1单突变体协同提高植物的免疫性[8]。Zou等[7]研究表明,在接种使水稻可产生稻瘟病的病菌后,OsBON1在接种12 h后显著上调持续到48 h,OsBON3表达量并未明显变化,可以推测OsBON1主要与植物抗病性相关,OsBON3主要参与植物的生长发育。本研究表明在龙井 43茶树接种炭疽菌 12 h后,CsBON3的表达量达到最大值,随后表达逐渐下调并和未处理前表达量一致,说明接种炭疽菌能短期内诱导CsBON3基因的表达,推测该基因可能与OsBON1一样,参与茶树的抗病反应。
鉴于BON基因在植物上的研究较少,且大多数的功能未知,而笔者在茶树上初步的研究结果发现,CsBON3基因对低温、病菌侵染具有一定程度的响应,而且还参与茶树芽的休眠与萌发过程,因此,推测与其他植物一样,茶树的BON基因可能也参与茶树的生长发育及抗逆过程。其具体的功能有待下一步通过功能基因组学的方法进行鉴定明确。
[1]Creutz C E, Tomsig J L, Snyder S L, et a1. The copines, a novel class of C2 domain-containing, calcium-dependent,phospholipid-binding proteins conserved from Paramecium to humans [J]. Journal of Biological Chemistry, 1998, 273(3):1393-1402.
[2]Rizo J, Südhof T C. C2-domains, structure and function of a universal Ca2+-binding domain [J]. Journal of Biological Chemistry, 1998, 273(26): 15879-15882.
[3]Nalefski E A, Falke J J. The C2 domain calciumbinding motif: structural and functional diversity [J]. Protein Science,1996, 5(12): 2375-2390.
[4]Tomsig J L, Creutz C E. Copines: a ubiquitous family of Ca2+-dependent phospholipid-binding proteins [J]. Cellular and Molecular Life Sciences, 2002, 59(9): 1467-1477.
[5]Tomsig J L, Snyder S L, Creutz C E. Identification of targets for calcium signaling through the copine family of proteins.Characterization of a coiled-coil copine-binding motif [J].Journal of Biological Chemistry, 2003, 278(12):10048-10054.
[6]Williams S C, Hinshelwood J, Perkins S J, et al. Production and functional activity of a recombinant von Willebrand factor-A domain from human complement factor B [J].Biochemical Journal, 1999, 342(3): 625-632.
[7]Zou B, Hong X, Ding Y, et al. Identification and analysis of copine/BONZAI proteins among evolutionarily diverse plant specie [J]. Genome, 2016, 59(8): 565-573.
[8]Yang S,Yang H, Grisafi P, et al. TheBON/CPNgene family represses cell death and promotes cell growth inArabidopsis[J]. Plant Journal, 2006, 45(2): 166-179.
[9]Wang X, Zhao Q, Ma C, et al. Global transcriptome profiles ofCamellia sinensisduring cold acclimation [J]. BMC Genomics, 2013, 14(1): 415.
[10]岳川, 曹红利, 周艳华, 等. 茶树谷胱甘肽还原酶基因CsGRs的克隆与表达分析[J]. 中国农业科学, 2014, 47(16):3277-3289.
[11]曹红利, 岳川, 郝心愿, 等. 茶树胆碱单加氧酶CsCMO的克隆及甜菜碱合成关键基因的表达分析[J]. 中国农业科学, 2013, 46(15): 3087-3096.
[12]Hao X, Horvath D, Chao W, et al. Identification and evaluation of reliable reference genes for quantitative real-time PCR analysis in tea plant (Camellia sinensis(L.) O.Kuntze) [J]. International Journal of Molecular Sciences,2014, 15(12): 22155-22172.
[13]Livak K J, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-delta delta C(T)) method [J]. Methods, 2001, 25(4):402-408.
[14]Tamura K, Peterson D, Peterson N, et al. MEGA5: molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance, and maximum parsimony methods [J].Molecular Biology and Evolution, 2011, 28(10): 2731-2739.
[15]Gastegier E, Hoogland C, Gattuker A, et al. The proteomics protocols handbook [M]. Biochemistry, 2005: 571-607.
[16]Petersen T N, Brunak S, von Heijne G, et al. SignalP 4.0:discriminating signal peptides from transmembrane regions[J]. Nature Methods, 2011, 8(10): 785-786.
[17]Zhou X, Lu P, Wulf G, et al. Phosphorylation-dependent prolyl isomerization: a novel signaling regulatory mechanism [J]. Cellular and Molecular Life Sciences , 1999,56(9/10): 788-806.
[18]Emanuelsson O, Nielsen H, Brunak S, et al. Predicting subcellular localization of proteins based on their N-terminal amino acid sequence [J]. Journal of Molecular Biology, 2000,300(4): 1005-1016.
[19]Hardie G, Hanks S. The protein kinase factsbook [M].London: Academic, 1995: 402-404
[20]Thompson G A, Okuyama H. Lipid-linked proteins of plants[J]. Progress in Lipid Research, 2000, 39(1): 19-39.
[21]Li Y, Gou M, Sun Q, et al. Requirement of calcium binding,myristoylation, and protein–protein interaction for the CopineBON1function inArabidopsis[J]. Journal of Biological Chemistry, 2010, 285(39): 29884-29891.
[22]Hua J, Grisafi P, Cheng S H, et al. Plant growth homeostasis is controlled by theArabidopsisBON1andBAP1genes [J].Genes Development, 2001,17(15): 2263-2272.
[23]Yang S, Hua J. A haplotype-specific resistance gene regulated byBONZAI1mediates temperature-dependent growth control inArabidopsis[J]. Plant Cell, 2004, 16(4): 1060-1071.
Cloning and Expression Analysis of CsBON3 from Copine Gene Family in Tea Plant (Camellia sinensis)
YAO Lina, HAO Xinyuan, WANG Lu, LI Nana, ZENG Jianming, YANG Yajun*, WANG Xinchao*
Tea Research Institute of Chinese Academy of Agricultural Sciences/National Center for Tea Improvement, Key Laboratory of Tea Biology and Resource Utilization, Ministry of Agriculture, Hangzhou 310008, China
Copine protein, a kind of Ca2+-dependent protein or phospholipid-binding protein containing two C2(N-terminal) and one vWA (C-terminal) conserved domain, plays an important role in intracellular signal transduction. Based on the sequence identity analysis, an EST sequence showing high homology to Copine family genes was selected from our previous tea plant transcriptome database. The sequence was verified to contain 1 746 bp complete ORF, encoding 581 amino acids. Homologous alignment showed that the gene had the highest similarity withArabidopsisAtBON3(65%) and was named asCsBON3(GenBank accession number: KY435900).Bioinformatic analysis showed that the molecular weight of CsBON3 protein was 63.66 kD and the theoretical isoelectric point was 5.48. It possesses the conserved domain unique to Copine family protein, which is a hydrophilic protein, no signal peptide site, non-secretory protein and no transmembrane domain. Expression analysis showed thatCsBON3had the highest expression levels in tea flowers and roots, and the lowest in stems and mature leaves. It was significantly up-regulated in tea plants treated with low temperature (4℃) for one day, and its expression was suppressed at dormant stage and up-regulated at growth stage. It was also up-regulated rapidly in the leaves with inoculation ofColletotrichum fructicola. The results indicated that theCsBON3plays important roles in tea plant growth in response to low temperature and disease resistance.
tea plant, Copine family, BON gene, stress response, dormancy, expression analysis
S571.1;Q52
A
1000-369X(2017)06-565-10
2017-01-18
2017-04-23
国家自然科学基金(31370690)、国家茶叶产业技术体系(CARS-19)资助
姚利娜,女,硕士研究生,主要从事茶树种质资源和育种研究。*通讯作者

