常规PCR法扩增口蹄疫病毒基因组5’端序列的探索
朱 研,苗书魁,马文戈,李金娜,魏玉荣,汪 萍,魏 婕,米晓云,黄 炯
(1新疆农业大学动物医学学院,乌鲁木齐 830052;2.新疆畜牧科学院兽医研究所/新疆畜牧科学院动物临床医学研究中心,乌鲁木齐 830011)
0 引 言
【研究意义】口蹄疫(foot-and-mouth disease,FMD)被世界动物卫生组织(Office International Des Epizooties,OIE)列为法定上报的动物疫病,被我国农业部列为一类疫病[1]。口蹄疫病毒(foot-and-mouth disease virus,FMDV)分为O、A、C、SAT1、SAT2、SAT3、AsiaⅠ共7个血清型,目前全球主要流行O型和A型FMD,其次为AsiaⅠ型和SAT2型,其中O型危害最为严重[2]。我国O型FMD主要流行3个拓扑型,为Cathay 拓扑型(古典中国型)、SEA拓扑型(东南亚拓扑型)和ME-SA拓扑型(中东-南亚拓扑型)。FMDV RNA约由8 500个核苷酸( nucleotides,nt)的无囊膜单股线状正链RNA组成,包括约1 300 nt的5’非编码区(5’untranslatedregion,5’UTR) 、约6.5 kb的1个大的开放阅读框(open reading frame,ORF) 、约90 nt的3’非编码区(3’untranslatedregion,3’UTR)及poly(A)组成[3]。在口蹄疫RNA基因组5’末端有一个特殊的小蛋白(VPg),通过磷酸二酯键与尿嘧啶残基相连[4]。紧接着的5’UTR由1个约360 nt可折叠成茎环结构的S片段、1个约100-420 nt的聚胞嘧啶poly(C)片段和1个可形成许多二级结构的L片段及其它部分组成[5],与启动多聚蛋白的翻译和病毒的复制有重要作用[6]。FMDV 5’端二级结构复杂,且有VPg(3B)蛋白、S片段、poly(C)等的存在,PCR时很难扩增出5’端的序列。试验用常规的PCR法扩增5’端的序列,为FMDV反向遗传学研究奠定基础。【前人研究进展】对于FMDV 5’端序列的扩增,研究者采用了不同的方法。有人先提取FMDV的RNA,再将其反转录成cDNA,最后扩增出5’端序列[7-9]。自1988年Frohman等报道cDNA末端快速扩增(Rapid Amplification of cDNA Ends,RACE)以来[10],有学者[11-12]用5’RACE(Rapid Amplification of cDNA Ends)法成功扩增出FMDV的5’端序列。袁子文等[13]采用融合PCR得到FMDV 5’端序列;仝燕许等[14]以特异性引物反转录扩增获得FMDV 5’端的S片段和E片段,再通过融合PCR得到SE片段。【本研究切入点】通过设计不同的反转录引物及扩增引物,采用常规PCR方法,以不同的扩增酶对FMDV O/Akesu/58 CE39A株的5’末端序列进行扩增。【拟解决的关键问题】探索以更简便有效的常规PCR法,扩增出FMDV 5’端序列所需的条件,为RNA病毒 5’端序列的扩增提供参考。
1 材料与方法
1.1 材 料
1.1.1 病毒及细胞
新疆畜牧科学院兽医研究所将1958年采自新疆阿克苏牦牛的一株O型口蹄疫病毒,经鸡胚传代驯化39代致弱为安全的毒株,命名为O/Akesu/58 CE39,筛选的单克隆毒株命名为O/Akesu/58 CE39A。O/Akesu/58 CE39A基因组RNA自1990年代由新疆畜牧科学院兽医研究所保存于液氮中。
BHK-21由该实验室保存,大肠杆菌感受态细胞DH5α购自天根生化科技(北京)有限公司,pMD19-T Vector购自TaKaRa公司。
1.1.2 主要试剂
LATaqDNA polymerase、EXTaqDNA polymerase、PrimeSTAR®HS DNA polymerase,cDNA Synthesis Kit购自Roche公司。
1.2 方 法
1.2.1 引物设计
根据GenBank中O型FMDV全基因组序列(GenBank 登录号:AJ539138),设计5’端反转录引物(a、b、c)及扩增引物(Ⅰ、Ⅱ、Ⅲ),由新疆昆泰锐生物技术有限公司合成。表1
表1 O/Akesu/58 CE39A株5’端序列的反转录引物和扩增引物
Table 1 Primers for reverse and amplification of the 5’ terminal sequences of the O/Akesu/58 CE39A strain
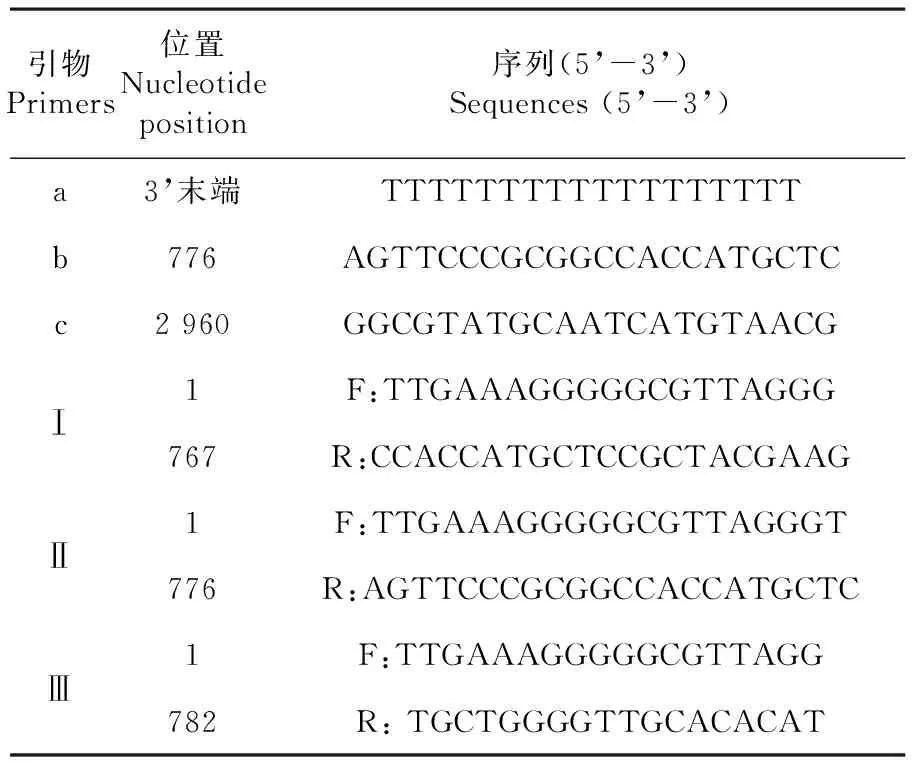
引物Primers位置Nucleotideposition序列(5’-3’)Sequences(5’-3’)a3’末端TTTTTTTTTTTTTTTTTTb776AGTTCCCGCGGCCACCATGCTCc2960GGCGTATGCAATCATGTAACGⅠ1F:TTGAAAGGGGGCGTTAGGG767R:CCACCATGCTCCGCTACGAAGⅡ1F:TTGAAAGGGGGCGTTAGGGT776R:AGTTCCCGCGGCCACCATGCTCⅢ1F:TTGAAAGGGGGCGTTAGG782R:TGCTGGGGTTGCACACAT
注:F:上游引物,R:下游引物
Note:
1.2.2 RNA纯度测定
取液氮中保存的FMDV O/Akesu/58 CE39A RNA 10 μL,加990 μL ddH2O,混匀,于紫外分光光度计测定其核酸纯度。
1.2.3 反转录
以反转录引物a、b、c分别进行反转录得到扩增模板cDNA1、cDNA2、cDNA3。
1.2.4 5’端序列的扩增
1.2.4.1 采用不同扩增引物及酶对5’端扩增
以cDNA1、cDNA2、cDNA3为模板,分别以扩增引物Ⅰ、Ⅱ、Ⅲ及不同的DNA聚合酶LATaq、 EXTaq、PrimeSTAR对5’端序列进行扩增,列出试验设计方案。表2
1.2.4.2 常规PCR退火温度的优化
参照引物合成的建议Tm值,拟设计退火温度55、60和65℃对表2试验分别进行扩增,确定最佳Tm。各扩增条件如下:
①94℃预变性2 min;94℃ 30 s,55℃ 20 s,72℃ 55 s进行30个循环;最后72℃延伸10 min。
②94℃预变性2 min;94℃ 30 s,60℃ 20 s,72℃ 55 s进行30个循环;最后72℃延伸10 min。
③94℃预变性2 min;94℃ 30 s,65℃ 20 s,72℃ 55 s进行30个循环;最后72℃延伸10 min。
表2 O/Akesu/58 CE39A株5’末端的扩增
Table 2 Amplification of the 5’ terminal of the O/Akesu/58 CE39A strain
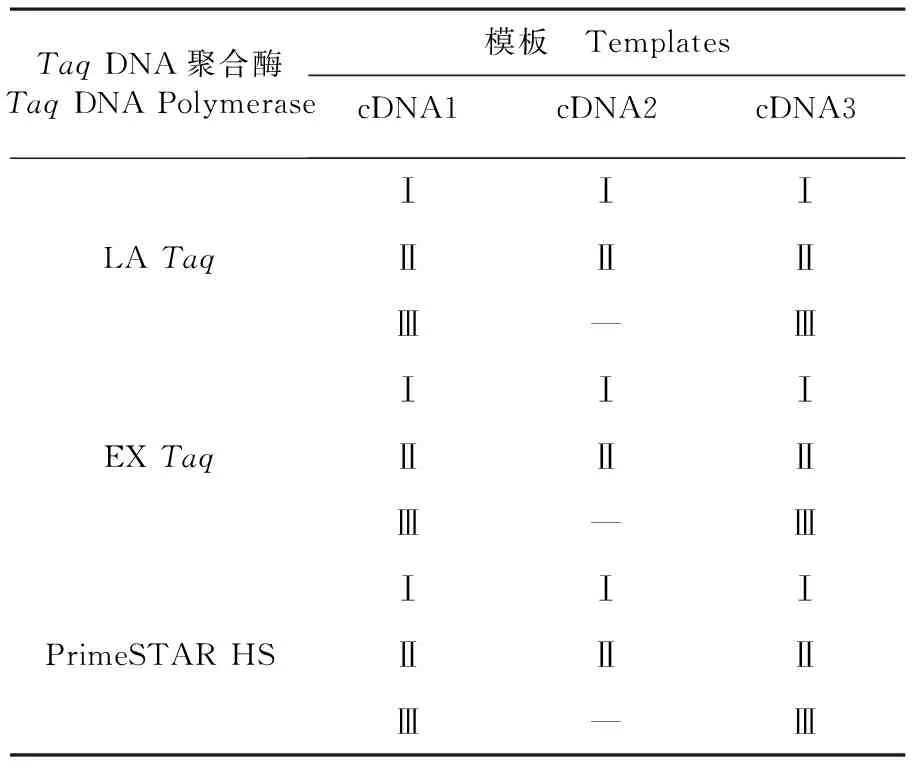
TaqDNA聚合酶TaqDNAPolymerase模板 TemplatescDNA1cDNA2cDNA3LATaqⅠⅠⅠⅡⅡⅡⅢ—ⅢEXTaqⅠⅠⅠⅡⅡⅡⅢ—ⅢPrimeSTARHSⅠⅠⅠⅡⅡⅡⅢ—Ⅲ
1.2.5 目的基因克隆及鉴定
将PCR产物克隆至pMD19-T载体,转化至感受态细胞DH5α,在LB培养基中过夜培养,提取质粒,通过酶切及测序鉴定,得到阳性质粒。
1.2.6 PCR灵敏度的检测
对能成功扩增出5’端序列的阳性cDNA以10倍梯度稀释,稀释范围为100~10-6,以这7个浓度的cDNA为模板,取相应扩增成功的引物及扩增酶进行三组重复的常规 PCR,同时以无菌去离子水为模板做阴性对照。将扩增产物进行1%琼脂糖凝胶电泳并拍照,确定模板的最大稀释度。
2 结果与分析
2.1 RNA纯度测定
以分光光度计测得O/Akesu/58 CE39A株基因组RNA A260/A280为1.871,表明此RNA中可能有部分蛋白质或其它有机物的污染,或是完整RNA降解导致纯度降低,但在允许范围1.8~2.0之内。
2.2 以不同的模板和酶扩增5’端序列
2.2.1 cDNA1模板扩增结果
用不同酶及扩增引物对5’端序列进行扩增,结果显示,以cDNA1为模板,用各扩增酶LATaq、EXTaq、PrimeSTAR及扩增引物Ⅰ、Ⅱ、Ⅲ在55℃、60℃、65℃退火温度下进行PCR,均未扩增出预期大小条带。表3
表3 以cDNA1为模板的扩增结果
Table 3 The results of amplification of cDNA1 template
引物PrimersTaqDNA聚合酶TaqDNAPolymerasePCR程序PCRprocedure是否扩增成功ResultsLATaq①、②、③①否、②否、③否ⅠEXTaq①、②、③①否、②否、③否PrimeSTAR①、②、③①否、②否、③否LATaq①、②、③①否、②否、③否ⅡEXTaq①、②、③①否、②否、③否PrimeSTAR①、②、③①否、②否、③否LATaq①、②、③①否、②否、③否ⅢEXTaq①、②、③①否、②否、③否PrimeSTAR①、②、③①否、②否、③否
2.2.2 cDNA2模板扩增结果
用不同酶及扩增引物对5’端序列进行扩增,结果表明,以cDNA2为模板,用EXTaq、PrimeSTAR扩增酶及扩增引物Ⅰ、Ⅱ在各退火温度下进行PCR,均未扩增出目的条带。但在LA酶作用下,两对引物均扩增出预期大小条带。在55及60℃退火温度下,扩增出的目的条带与弥散带混杂,将退火温度提高至65℃,可扩增出清晰的目的条带,表明在LA酶下,以cDNA2为模板扩增FMDV 5’端序列的最佳退火温度为65℃。表4,图1~3
2.2.3 cDNA3模板扩增结果
用不同酶及扩增引物对5’端序列进行扩增,结果显示,以cDNA3为模板,用3种扩增酶及3对扩增引物对5’端进行扩增,均未扩增出目的条带。表5
2.3 重组质粒的酶切鉴定
以cDNA2为模板,将LATaqDNA polymerase及扩增引物Ⅰ、Ⅱ下的PCR产物克隆至pMD19-T载体,转化至DH5α感受态细胞,提取质粒pMD19-T-Ⅰ、pMD19-T-Ⅱ,将质粒用BamH Ⅰ和SalⅠ进行双酶切鉴定,设无插入片段空质粒pMD19作为对照,以1%琼脂糖凝胶电泳分析,均得到预期大小的条带,表明该质粒为阳性质粒。图4
表4 以cDNA2为模板的扩增结果
Table 4 The results of amplification of cDNA2 template
引物PrimersTaqDNA聚合酶TaqDNAPolymerasePCR程序PCRprocedure是否扩增成功ResultsLATaq①、②、③①是、②是、③是ⅠEXTaq①、②、③①否、②否、③否PrimeSTAR①、②、③①否、②否、③否LATaq①、②、③①是、②是、③是ⅡEXTaq①、②、③①否、②否、③否PrimeSTAR①、②、③①否、②否、③否

注:M:GeneRuler 1Kb Plus DNA Ladder;1:引物Ⅰ的PCR产物; 2:引物Ⅱ的PCR产物
Note: M: GeneRuler 1Kb Plus DNA Ladder; 1: The PCR products by primer Ⅰ; 2: The PCR products by primer Ⅱ
图1 引物Ⅰ、Ⅱ在55℃退火温度下PCR结果
Fig.1 The PCR results of amplification at 55℃ annealing temperature with primer Ⅰ, primer Ⅱ

注:M:GeneRuler 1Kb Plus DNA Ladder;1:引物Ⅰ的PCR产物; 2:引物Ⅱ的PCR产物
Note: M: GeneRuler 1Kb Plus DNA Ladder; 1: The PCR products by primer Ⅰ; 2: The PCR products by primer Ⅱ
图2 引物Ⅰ、Ⅱ在60℃退火温度下PCR结果
Fig.2 The PCR results of amplification at 60℃ nnealing temperature with primer Ⅰ, primer Ⅱ

注:M:GeneRuler 1Kb Plus DNA Ladder;1:引物Ⅰ的PCR产物;2:引物Ⅱ的PCR产物
Note: M: GeneRuler 1Kb Plus DNA Ladder; 1: The PCR products by primer Ⅰ; 2: The PCR products by primer Ⅱ
图3 引物Ⅰ、Ⅱ在65℃退火温度下PCR结果
Fig.3 The PCR results of amplification at 65℃ annealing temperature with primer Ⅰ, primer Ⅱ
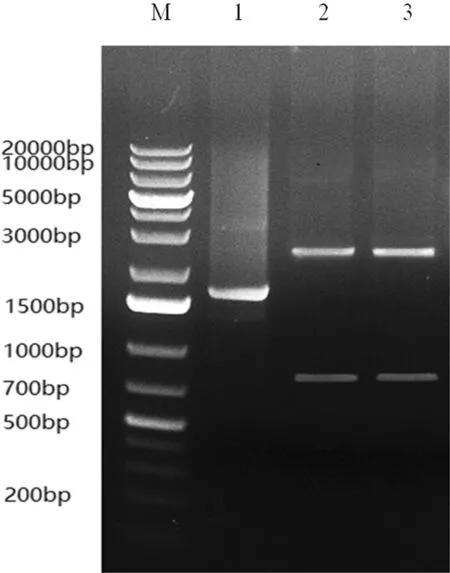
注:M: GeneRuler 1Kb Plus DNA Ladder;1:空质粒pMD19-T(2692 bp);2:pMD19-T-Ⅰ的酶切产物(2692 bp+787 bp);3:pMD19-T-Ⅱ的酶切产物(2692 bp+797 bp)
Note: M: GeneRuler 1Kb Plus DNA Ladder; 1: Empty plasmid pMD19-T(2692 bp); 2: Digestion of pMD19-T-Ⅰ positive clone(2692 bp+787 bp); 3: Digestion of pMD19-T-Ⅱ positive clone(2692 bp+797 bp)
图4 O/Akesu/58 CE39A株5’末端重组质粒的酶切鉴定
Fig.4 Restriction endonuclease digestion of recombinant plasmid of 5’ termini, O/Akesu/58 CE39A strain表5 以cDNA3为模板的扩增结果
Table 5 The results of amplification of cDNA3 template

引物PrimersTaqDNA聚合酶TaqDNAPolymerasePCR程序PCRprocedure是否扩增成功ResultsLATaq①、②、③①否、②否、③否ⅠEXTaq①、②、③①否、②否、③否PrimeSTAR①、②、③①否、②否、③否LATaq①、②、③①否、②否、③否ⅡEXTaq①、②、③①否、②否、③否PrimeSTAR①、②、③①否、②否、③否LATaq①、②、③①否、②否、③否ⅢEXTaq①、②、③①否、②否、③否PrimeSTAR①、②、③①否、②否、③否
2.4 重组质粒的测序
将酶切鉴定的阳性质粒进行测序,测序结果在DNAMAN软件中进行比对,结果显示,分别以引物Ⅰ、Ⅱ对O/Akesu/58 CE39A株5’末端进行扩增,所测得的序列787 bp、797 bp与参考序列(GenBank 登录号:AJ539138)5’末端的核苷酸同源性为86.2%和86.3%,再次证明得到的质粒为阳性质粒。
2.5 常规PCR灵敏度的检测
以LATaqDNA polymerase为扩增酶,以7个浓度的cDNA2为模板,分别用扩增引物Ⅰ、Ⅱ做常规PCR。扩增条件如下:94℃预变性2 min;94℃ 30 s,65℃ 20 s,72℃ 55 s进行30个循环;最后72℃延伸10 min。结果表明,在引物Ⅰ的电泳图中,稀释度≤10-3时能观测到目的条带,稀释度≥10-4时没有条带,阴性对照没有条带;在引物Ⅱ的电泳图中观察到相同的结果。因此,引物Ⅰ、Ⅱ可检测到的模板最大稀释度为10-3。图5,图6,表6

注:M: GeneRuler 1Kb Plus DNA Ladder;1:100稀释度PCR产物;1~7:100~10-6稀释度PCR产物;8:阴性对照的PCR产物
Note: M: GeneRuler 1Kb Plus DNA Ladder; 1: The PCR product by dilution 100; 1-7: The PCR products by dilution 100-10-6; 8: The PCR products of negative control
图5 引物Ⅰ在各浓度cDNA2模板下的PCR结果
Fig.5 The PCR results of each concentration of cDNA2 template with primer Ⅰ

注:M: GeneRuler 1Kb Plus DNA Ladder;1:100稀释度PCR产物;1~7:100-10-6稀释度PCR产物;8:阴性对照的PCR产物
M: GeneRuler 1Kb Plus DNA Ladder; 1: The PCR product by dilution 100; 1-7: The PCR products by dilution 100-10-6; 8: The PCR products of negative control
图6 引物Ⅱ在各浓度cDNA2模板下的PCR结果
Fig.6 The PCR results of each concentration of cDNA2 template with primer Ⅱ表6 常规PCR灵敏度测定结果
Table 6 Results of general PCR for the sensitivity determination
模板浓度Concentrationoftemplate10010-110-210-310-410-510-6引物Ⅰ PrimerⅠ++++---引物Ⅱ PrimerⅡ++++---
3 讨 论
口蹄疫病毒分为七个血清型,其中O型以拓扑型众多、免疫原性弱著称[2]。新疆畜牧科学院兽医研究所于1958年自阿克苏牦牛分离到一株O型口蹄疫病毒,该毒株被国家口蹄疫参考实验室命名为O/Akesu/58[15](GenBank 登录号:AF511039),新疆畜牧科学院兽医研究所将该毒株经鸡胚传代驯化39代致弱为安全的毒株,命名为O/Akesu/58 CE39A,并曾经于上世纪90年代作为弱毒疫苗应用于口蹄疫预防与控制。O/Akesu/58 CE39A基因组与O/Akesu/58核苷酸同源性仅为86.4%,反而与O1/manisa iso87的核苷酸同源性达到了90.2%。
FMDV基因组5’末端结构复杂,其5’UTR 360 nt的S片段可形成发卡结构,距5’末端约350-500 nt处,有100-420 nt的poly(C)结构,其长度随毒株的不同而大小不一。在S片段和poly(C)下游可以形成许多高度保守的二级结构,该二级结构由串联重复的假结(PKs)结构、“发卡状”的RNA复制相关顺式作用元件(cre)结构和内部含“三叶草”状的内部核糖体进入位点(IRES)构成[16]。尽管其它病毒5’端也含有复杂的二级结构,但只有在FMDV 5’端会同时出现各种结构[17],导致很难扩增出FMDV 5’端的序列。
5’端序列的扩增,有多种方法可供选用,如5’RACE法和染色体步移法。5’RACE试剂盒包括“去帽法”原理的环化技术模板跳转反转录的SMART RACE技术、末端脱氧核苷酸转移酶(TdT)加尾的锚定RACE等[18],但这些试剂盒价格昂贵,操作过程繁琐,对模板的结构要求高[19],只有对很难扩增的序列,才使用上述RACE试剂盒。染色体步移法包括反向PCR法、连接介导PCR法及特异引物PCR法[20-22],这些方法操作复杂,对操作人员的技术要求较高,成功率较低。近年来,有的学者通过融合PCR 技术得到FMDV全长cDNA的克隆,从而获得5’端的序列[13-14]。试验选用常规PCR法,通过不同的扩增酶及不同的反转录模板、特异性PCR引物对5’端序列进行了扩增,该方法不需要耗费大量的时间与精力,操作简便,价格便宜,适宜条件下扩增成功率较高,可针对性的对5’端的序列进行扩增。
DNA聚合酶有多种,每种酶各有其特性。在PCR时,酶的选择至关重要,选用不同的酶进行扩增,结果可能截然不同,其最终结果需要经过具体试验来验证。由表4可知,在相同模板、引物、PCR条件下,以3种不同的Taq酶进行PCR,结果只有LATaq酶能扩增出目的片段,说明试验中该酶的扩增能力大于EXTaq和PrimeSTAR。
试验在参考序列的不同位置(3’末端、776~797、2 960~2 979)设计了三条反转录引物,分别反转录后得到三种模板(cDNA1、cDNA2、cDNA3),以这三种模板分别进行PCR,结果发现,只有以cDNA2为模板,才能扩增出目的片段787 bp和797 bp,787 bp下游引物的位置,与反转录引物重合了一部分,797 bp下游引物的位置,与反转录引物位置完全重合。以cDNA1和cDNA3为模板均未扩增出目的片段,表明模板质量不够理想,或者模板长度对其结构有重要影响。
以cDNA为模板的常规PCR灵敏度检测表明,两对引物在模板稀释度≤10-3时均能扩增出5’端序列,但≥10-4时不能扩增出5’端序列。
试验在灵敏度检测时,未能计算出模板的最小拷贝数,仅确定了模板的最大稀释度,原因在于,病毒基因组RNA的反转录产物中,全长cDNA的拷贝数未知,且其中含有大量的非全长cDNA和其它残留物质(如残留的RNA、基因组等)。因此,仅通过反转录产物浓度的测定和计算稀释度的方法,并不能确定最大模板稀释度的PCR反应中全长cDNA拷贝数。试验使用到的病毒基因组反转录产物质量,影响因素复杂:第一,FMDV 5’末端结构复杂,存在茎环结构、假结结构等,难于反转录。第二,RNA质量的影响。FMDV RNA为单链结构,容易断裂,所提取的RNA中有断裂的RNA,直接影响了反转录产物的质量;不同批次间RNA质量差异也导致反转录产物的质量的不同。第三,反转录酶活性高时反转录效率高,反之低;第四,反转录体系的影响。如dNTP易降解,buffer中各成分浓度的影响等。第五,温度的影响。低温时会产生非特异条带,高温时可能会破坏RNA的二级结构。第六,FMDV RNA长约8 500 nt,基因组长,反转录引物有可能与模板上其它部位结合,产生大量非全长cDNA。
试验采用常规PCR法扩增FMDV 5’末端序列,通过不同模板、不同扩增酶、不同引物、不同PCR程序之间的比较,确定了扩增5’端序列的最佳条件,为FMDV复制机理、序列的扩增、结构与功能的关系、致病机理及宿主嗜性提供理论依据和技术支持。
4 结 论
常规PCR法扩增5’端序列较其它技术具有价格便宜、操作简便等优点。试验共进行了72次PCR,成功6次,成功率为6/72,在成功的6次中,2次扩增出了清晰的目的条带,其它4次都是目的条带与弥散带混杂,表明FMDV 5’端序列的扩增存在很大难度。常规PCR灵敏度检测表明,引物Ⅰ、Ⅱ可检测到的模板最大稀释度为10-3。
对类似FMDV 5’端难扩增的序列,为了保证模板的质量及扩增成功率,设计反转录引物时,可尽量在靠近该目的序列的3’端设计;设计PCR引物的下游引物时,可与反转录引物重合,或在靠近反转录引物的上游设计。
试验为FMDV 5’端序列的扩增奠定了基础,但对FMDV其它血清型及O型血清型其它株5’端序列的扩增,有待进一步的探索。
参考文献(References)
[1] 曹丽娟,倪伟,史慧君,等.O型口蹄疫病毒3D基因荧光定量PCR方法的建立及应用[J].中国畜牧兽医,2014,41(12):34-39.
CAO Li-juan, NI Wei, SHI Hui-jun, et al. (2014). Establishment and Application of Fluorescence Quantitative PCR of Type O Foot-and-mouth Disease Virus 3D Gene [J].ChineseAnimalHusbandryandVeterinaryMedicine, 41(12): 34-39. (in Chinese)
[2]刘在新.全球口蹄疫防控技术及病原特性研究概观[J].中国农业科学,2015,48(17):3 547-3 564.
LIU Zai-xin. (2015). Progress and Prospect of the Technologies to Control Foot-and-Mouth Disease and Its Pathogen Characteristics Worldwide [J].XinjiangAgriculturalSciences, 48(17): 3,547-3,564. (in Chinese)
[3]孙芳芳,董英,薛强,等.口蹄疫病毒分子生物学检测方法研究进展[J].中国畜牧兽医,2013,40(6):190-194.
SUN Fang-fang, DONG Ying, XUE Qiang, et al. (2013). Research Progress on the Molecular Biology Techniques in Detection of Foot and Mouth Disease Virus [J].ChineseAnimalHusbandryandVeterinaryMedicine, 40(6): 190-194. (in Chinese)
[4] Forss, S., & Schaller, H. (1982). A tandem repeat gene in a picornavirus.NucleicAcidsResearch, 10(20): 6,441-6,450.
[5] Mahy, B. W. (2005). Introduction and history of foot-and-mouth disease virus.CurrentTopicsinMicrobiology&Immunology, 288, 1.
[6] Quinto, S. L. D., & Martínezsalas, E. (1997). Conserved structural motifs located in distal loops of aphthovirus internal ribosome entry site domain 3 are required for internal initiation of translation.JournalofVirology,71(5):4,171-4,175.
[7] Li, D., Shang, Y. J., Liu, Z. X., Liu, X. T., & Cai, X. P. (2007). Comparisons of the complete genomes of two chinese isolates of a recent foot-and-mouth disease type asia 1 virus.ArchivesofVirology,152(9):1,699-1,708.
[8] Rajasekhar, R., Hosamani, M., Basagoudanavar, S. H., Sreenivasa, B. P., Tamil Selvan, R. P., & Saravanan, P., et al. (2013). Rescue of infective virus from a genome-length cdna clone of the fmdv serotype o (ind-r2/75) vaccine strain and its characterization.ResearchinVeterinaryScience, 95(1):291-297.
[9] Li, D., Liu, Z. X., Bao, H. F., Lu, Z. J., Guo, J. H., & Cao, Y. M., et al. (2007). The complete genome sequence of foot-and-mouth disease virus o/akesu/58 strain and its some molecular characteristics.ArchivesofVirology, 152(11): 2,079-2,085.
[10] Frohman, M. A., Dush, M. K., & Martin, G. R. (1988). Rapid production of full-length cdnas from rare transcripts: amplification using a single gene-specific oligonucleotide primer.ProceedingsoftheNationalAcademyofSciencesoftheUnitedStatesofAmerica, 85(23): 8,998-9,002.
[11] Oem, J. K., Lee, K. N., Cho, I. S., Kye, S. J., Park, J. H., & Yi, S. J. (2004). Comparison and analysis of the complete nucleotide sequence of foot-and-mouth disease viruses from animals in korea and other panasia strains.VirusGenes, 29(1): 63-71.
[12] Kanno, T., Yamakawa, M., Yoshida, K., & Sakamoto, K. (2002). The complete nucleotide sequence of the panasia strain of foot-and-mouth disease virus isolated in japan.VirusGenes, 25(2): 119-125.
[13]袁子文,李平花,孙普,等.一株A型口蹄疫流行毒株全序列的测定及其全长感染性克隆的构建[J].微生物学通报,2016,43(9):2 019-2 027.
YUAN Zi-wen, LI Ping-hua, SUN Pu, et al. (2016). Complete genome sequence analysis and construction full-length infectious cDNA clone of an epidemic Foot-and-mouth disease virus of serotype A [J].Microbiology, 43(9): 2,019-2,027. (in Chinese)
[14]仝燕许,陈豪泰,潘丽,等.口蹄疫病毒Asial/JSWX株基因组全长感染性克隆的体外拯救与序列分析[J].中国兽医科学,2014,44(8):771-780.
TONG Yan-xu, CHEN Hao-tai, PAN Li, et al. (2014).Invitrorescue and sequence analysis of an infectious full-length Cdna clone of foot-and-mouth disease virus Asia 1/JSWX strain [J].ChineseveterinaryScience, 44(8): 771-780. (in Chinese)
[15] Li, D., Liu, Z. X., Bao, H. F., Lu, Z. J., Guo, J. H., & Cao, Y. M., et al. (2007). The complete genome sequence of foot-and-mouth disease virus o/akesu/58 strain and its some molecular characteristics.ArchivesofVirology, 152(11):2,079-2,085.
[16]马晓霞.口蹄疫病毒结构蛋白的分段表达及免疫试验研究[D]. 兰州:兰州大学硕士学位论文,2009.
MA Xiao-xia. (2009).SegmentedexpressionofstructuralproteinofFoot-and-MouthDiseasevirusandImmunizationtest[D]. Master Dissertation. Lanzhou University, Lanzhou. (in Chinese)
[17]许保疆,游一,付仁一,等.口蹄疫病毒5'端非翻译区致病机理的分子基础[J].中国农学通报,2009,25(19):1-5.
XU Bao-jiang, YOU Yi, FU Ren-yi, et al. (2009). Molecular Basis of Pathogenesis of FMDV 5'Untranslated Region [J].ChineseAgriculturalScienceBulletin, 25(19): 1-5. (in Chinese)
[18]罗聪,何新华,陈虎,等.一种高效获取基因5'末端的RACE方法[J].植物生理学报,2011,47(4):409-414.
LUO Cong, HE Xin-hua, CHEN Hu, et al. (2011). A High-Efficient Method of RACE Technique for Obtaining the Gene 5' End [J].PlantPhysiologyJournal, 47(4): 409-414. (in Chinese)
[19]徐烨,刘雅婷,代文琼,等. 几种主要的 RACE 技术及应用[J].中国农业科技导报,2012,14(2):81-87.
XU Ye, LIU Ya-ting, DAI Wen-qiong, et al. (2012). Several Major RACE Technologies and their Application [J].JournalofAgriculturalScienceandTechnology, 14(2): 81-87. (in Chinese)
[20] Tsaftaris, A., Pasentzis, K., & Argiriou, A. (2010). Rolling circle amplification of genomic templates for inverse pcr (rca-gip): a method for 5′- and 3′-genome walking without anchoring.BiotechnologyLetters,32(1): 157-161.
[21] Jones, D. H., & Winistorfer, S. C. (1992). Sequence specific generation of a dna panhandle permits pcr amplification of unknown flanking dna.NucleicAcidsResearch, 20(3): 595-600.
[22] Gomma, A. E., Jin, M. K., Hwanyang, S., & Chung, G. (2015). An easy PCR-based genome-walking method for getting the unknown 5' flanking region of a Scenedesmus sp.JournalofCoastalLifeMedicine, 3(5): 348-351.

