基于PPI网络与机器学习的蛋白质功能预测方法
唐家琪,吴璟莉,2,3
(1.广西师范大学 计算机科学与信息工程学院,广西 桂林 541004; 2.广西师范大学 广西多源信息挖掘与安全重点实验室,广西 桂林 541004;3.广西区域多源信息集成与智能处理协同创新中心,广西 桂林 541004)
0 引言
蛋白质是执行生物体内各种重要生物活动的大分子,认识其功能对推动生命科学、农业、医疗等领域的发展意义重大。1961年,Anfinsen等[1]提出蛋白质一级序列决定其三维结构、蛋白质三维结构决定其功能的论断。相对于蛋白质三维结构,一级序列更容易通过生物实验测得,故早期的蛋白质功能预测方法大都基于序列相似性原理,利用BLAST(Basic Local Alignment Search Tool)[2]和PSI-BLAST(Position-Specific Iterated BLAST)[3]等工具计算功能未知的蛋白质与功能已知的蛋白质之间的序列相似度,若相似度较高则认为其具有相同的功能。然而,近年来的研究表明,序列相似的蛋白质能够形成不同的三维结构,故其功能不一定相同,而且序列差异较大的蛋白质也可能具有相同的功能[4];因此,基于序列相似性的蛋白质功能预测方法是不可靠的。
随着越来越多的蛋白质三维结构数据的产生,FATCAT(Functional And Tractographic Connectivity Analysis Toolbox)[5]和PAST(Polypeptide Angle Suffix Tree)[6]等蛋白质三维结构数据库相继建立,研究者提出了基于蛋白质三维结构的功能预测方法[7],这类方法通过计算功能未知的蛋白质与功能已知的蛋白质的三维结构相似度来判断其是否具有相同功能。相对于蛋白质的氨基酸序列,其三维结构更保守稳定[8],故基于三维结构的方法通常比基于序列的方法更准确,但由于已知三维结构的蛋白质数量较少,其应用范围较窄。
随着高通量生物实验技术与蛋白质相互作用(Protein-Protein Interaction, PPI)预测方法[9-10]的发展,产生了海量的、可用于大规模蛋白质功能注释的PPI数据,基于蛋白质相互作用网络(简称PPI网络)的功能预测方法深受关注。根据Oliver[11]提出的关联效应(Guilt-By-Association, GBA),相互作用的蛋白质具有相同或相似的功能,可以通过分析PPI网络的拓扑结构,根据网络中已经注释功能的蛋白质来推测网络中未注释功能的蛋白质的功能。Chi等[12]提出余弦迭代算法(Cosine Iterative Algorithm, CIA),其基于蛋白质之间动态相互作用,迭代更新邻居蛋白质的注释术语集,估计它们与未注释蛋白质之间的功能相似性来完成预测。Xiong等[13]采用谱聚类算法将PPI网络中的蛋白质划分为若干功能模块,根据顶点度、紧密度和介数三种中心性指标标注各模块中的重要蛋白质,再利用基于 Gibbs抽样的协同分类算法预测蛋白质功能。Wang等[14]针对注释术语间的功能关联性,设计了一种基于多标签学习的蛋白质功能预测算法。Teng等[15]根据相互作用的蛋白质在PPI网络中的主被动关系将无向的PPI网络转化成有向网络,并通过在有向PPI网络中传播基因本体术语(Diffusing GO Terms in the Directed PPI Network, GoDIN)的方法预测蛋白质的功能。Yu等[16]提出一种在混合图上随机游走的蛋白质功能预测方法,该方法不仅综合考虑了直接和间接相互作用信息,还利用功能相似性权重来减少噪声相互作用的影响。
基于PPI网络的功能预测效果依赖于网络的可靠程度。由于生物实验技术的制约,大多数PPI数据均存在一定程度的噪声,从而降低了这类方法的预测精度。本文将蛋白质家族(Family)、结构域(Domain)和重要位点(Important Site)信息作为顶点属性,整合到PPI网络中以减轻网络中数据噪声的影响,并提出了一种基于层次聚类(Hierarchical Clustering, HC)、主成分分析(Principal Component Analysis, PCA)与多层感知器(Multi-Layer Perceptron, MLP)的蛋白质功能预测方法(HC, PCA and MLP based Method, HPMM)。HPMM将蛋白质功能预测转化成多标签二分类问题,首先从PPI网络、蛋白质家族、结构域和重要位点中提取蛋白质的特征,再训练MLP模型用于功能预测。采用人类(Homo sapiens)数据集对蛋白质功能预测方法CIA[12]、GoDIN[15]和HPMM进行测试。实验结果表明,相比CIA和GoDIN,HPMM的精确度与F值更高。
1 问题与符号定义
PPI网络通常表示为无向图G(V,E),其中V={v1,v2,…,vn}为顶点集,E={eij|eij=(vi,vj),vi,vj∈V}为边集。顶点vi(i=1,2,…,n)表示蛋白质,边eij∈E表示其两端的蛋白质vi与蛋白质vj之间存在相互作用,di(i=1,2,…,n)表示顶点vi的度,即与蛋白质vi存在相互作用的蛋白质种类数。PPI网络中,假设v1,v2,…,vn1为功能已知的蛋白质,vn1+1,vn1+2,…,vn1+n2为功能未知的蛋白质,n=n1+n2。用邻接矩阵An×n表示图G,其中每个元素aij(i=1,2,…,n,j=1,2,…,n)的取值定义如下:
(1)
蛋白质家族、结构域和重要位点信息对蛋白质的功能有重要影响,故可以看成蛋白质的属性。用矩阵Pn×m记录蛋白质属性,每行表示一个蛋白质顶点,每列表示一个属性,元素pij(i=1,2,…,n,j=1,2,…,m)的取值定义如下:
(2)
将邻接矩阵An×n与属性矩阵Pn×m横向合并,得到蛋白质的特征矩阵Xn×(n+m),其中xi=(xi1,xi2,…,xi(n+m))为蛋白质样本vi(i=1,2,…,n)的特征向量,元素xij(i=1,2,…,n,j=1,2,…,n+m)的取值定义如下:
(3)
令Yn×w为记录蛋白质的功能注释信息的标签矩阵,其中w为数据集中功能注释的类别总数,Yn×w中每一行yi=(yi1,yi2,…,yiw)为蛋白质样本vi(i=1,2,…,n)的标签向量,其中的元素yij(i=1,2,…,n,j=1,2,…,w)的取值定义如下:
(4)
根据上述定义,以蛋白质为样本、功能术语为样本标签的蛋白质功能预测问题可转化为多标签二分类问题:将n1个功能已知的蛋白质用于训练预测模型,试图得到映射函数h:X→Y,使给定功能未知的n2个蛋白质的特征向量xi(i=n1+1,n1+2,…,n),预测其标签向量(即功能注释向量)h(xi)⊆Y。
2 功能预测方法
本章提出一种基于机器学习的蛋白质功能预测方法HPMM,输入为PPI网络中功能已知的蛋白质的功能注释术语,PPI网络及其每个蛋白质的属性信息(家族、结构域和重要位点);输出为PPI网络中功能未知的蛋白质的功能注释术语。如图1所示,HPMM主要分为特征提取、训练模型和功能预测3个阶段。

图1 HPMM流程 Fig. 1 Flow chart of HPMM
首先基于层次聚类和主成分分析进行特征提取,将提取的功能模块(Function Module)、属性(家族、结构域和重要位点)主成分(Principal Component)及顶点度 (Degree) 作为特征,对其归一化后用于训练多层感知器,从而得到一个多标签的二分类模型。然后用该模型预测PPI网络中功能未知的蛋白质。下面详细介绍预测方法HPMM的主要步骤。
2.1 功能模块特征提取
研究表明细胞功能是通过生物大分子之间相互作用形成的功能模块实现的[17],故同一功能模块中的蛋白质往往具有相似的功能,因此可先通过聚类算法从PPI网络中挖掘出若干功能模块,并将其作为蛋白质的特征以用于功能预测。由于功能模块特征取决于网络的整体拓扑结构,其受局部噪声相互作用的影响较小,鲁棒性较强。
层次聚类算法常被用于从PPI网络中挖掘功能模块,以确定模块中蛋白质的功能[18-19]。本文采用Clauset等[20]提出的一种针对复杂网络的凝聚层次聚类算法提取功能模块特征。算法输入为邻接矩阵An×n,输出为功能模块矩阵Fn×k1(k1为功能模块数),其中的元素fij(i=1,2,…,n,j=1,2,…,k1)取值为1(0),表示蛋白质vi属于(不属于)功能模块j。
算法利用NG(Newman and Girvan)模块度[21]来评价聚类效果,其定义如式(5)所示:
(5)
其中:fi=(fi1,fi2,…,fik)代表蛋白质vi的功能模块特征向量,函数δ(fi,fj)指示向量fi和fj取值是否相同:相同返回1,表示蛋白质vi和vj属于相同功能模块;反之返回0,表示蛋白质vi和vj属于不同的功能模块。模块度取值越大,则表示图的模块划分效果越好。
2.2 属性主成分特征提取
如前所述,由于蛋白质的家族、结构域和重要位点这些属性信息均对其功能起到重要的影响,可将其作为特征以减少预测结果对带数据噪声的PPI网络的依赖,从而提高预测精度;但是,在训练多层感知器时,若蛋白质样本维数过多则会使预测模型失去泛化能力,从而影响预测效果。本文考虑到蛋白质属性之间的相关性,使用一种基于奇异值分解(Singular Value Decomposition, SVD)的主成分分析方法[22],对属性矩阵Pn×m降维处理。
首先,对Pn×m进行z-score标准化处理,得到矩阵Zn×m,其中每个元素zij(i=1,2,…,n,j=1,2,…,m)取值如下:
(6)
其中:
(7)
然后,对矩阵Zn×m进行奇异值分解,使得Z=UΛΜ,其中U为n阶正交矩阵,Λ为n×m的半正定对角矩阵,Μ为m阶正交矩阵,U中每列uj(j=1,2,…,n)代表蛋白质属性的一个主成分,Λ中对角元素λj(j=1,2,…,n)代表对应主成分的方差,该值反映了主成分的重要程度。
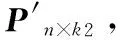
2.3 特征合并与标准化
本文将蛋白质在PPI网络中的顶点度数作为特征,并将其与功能模块特征和属性主成分特征合并,得到特征矩阵Γn×(k1+k2+1),其中元素γij(i=1,2,…,n,j=1,2,…,k1+k2+1)取值如下:
(8)
其中:di表示蛋白质vi的顶点度。该矩阵中包含了功能模块、属性主成分、和顶点度三类特征。功能模块特征反映了蛋白质在宏观的相互作用网络中所在的功能模块。属性主成分特征则反映了蛋白质微观层面的信息。顶点度是一种常用的中心性度量,反映了蛋白质在PPI网络中的重要程度,同时也代表了蛋白质参与生命活动的多少,即功能多样性[23]。这三类特征从不同层面表征了蛋白质,并且不容易被PPI网络中数据噪声干扰。
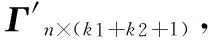
(9)
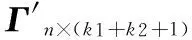
2.4 基于多层感知器的功能预测
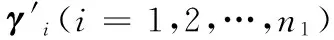
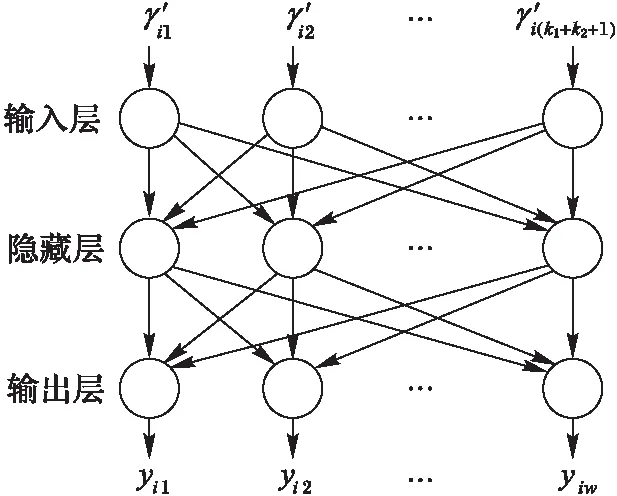
图2 多层感知器示意图 Fig. 2 Diagram of MLP
2.4.1 参数设置
输入层节点数等于特征向量的维数k1+k2+1,输出层的节点数等于数据集中的所有蛋白质拥有的功能注释数,即功能注释向量的维数w。
输出层使用Sigmoid激活函数,定义如下:
(10)
使用交叉熵(cross entropy)作为输出层的损失函数,对于样本vi,其交叉熵定义如下:
(11)


ReLu (x) = max(0,x)
(12)
训练该神经网络时,采用批量学习的方式[26],批量大小为训练集中蛋白质数的10%,迭代次数为400次,学习率(Learning Rate)为0.1,动量(Momentum)为0.9。
2.4.2 功能选择
3 实验与结果分析
本文用人类数据集对蛋白质功能预测方法HPMM、CIA[12]和GoDIN[15]进行比较分析。实验在一台4核8线程的微型计算机上进行,CPU型号为Intel@Core i7-3630QM 2.4 GHz×8,内存为8 GB,操作系统为Ubuntu 16.04 LTS 64位,编程工具为R 3.3.2。训练MLP时采用GPU加速技术和MXNet深度学习框架,GPU型号为GeForce GT 650M/PCIe/SSE2。
3.1 实验数据
本文的实验数据来自于DIP[27]、基因本体(Gene Ontology, GO)[28]和InterPro[29]数据库。DIP数据库提供了人类PPI网络,GO提供了功能注释, InterPro数据库提供了蛋白质家族、结构域和重要位点信息。
首先从DIP数据库下载人类PPI网络数据,并用UniProtKB/Swiss-Prot[30]对PPI网络中的蛋白质进行ID转换,然后去除网络中自相互作用、重复相互作用及无法转换的蛋白质;再通过biomaRt包[31]根据每个蛋白质的UniProtKB/Swiss-Prot编号获取对应的GO术语编号和InterPro编号。
GO包括分子功能(Molecular Function, MF)、生物过程(Biological Process, BP)和细胞组件(Cellular Component, CC)三个独立的子本体。为保证注释术语的可靠性,实验剔除了获取手段为IEA(Inferred from Electronic Annotation)、ND(No biological Data Available)和IC(Inferred by Curator)的功能注释。如上所述,由于MF、BP和CC三个子本体相互独立,分别为每个子本体建立PPI网络,分别称为MF、BP和CC网络,并删除了没有被GO术语注释的蛋白质。此外,为确保每个蛋白质均有与其相互作用的蛋白质,本文仅取PPI网络中的极大连通子图作为测试数据。
InterPro数据库是一个整合了蛋白质家族、结构域和重要位点信息的综合数据库,每个InterPro编号对应一条蛋白质的家族、结构域或重要位点信息。本文根据每个InterPro编号对应的信息存在与否将其编码成二元变量作为蛋白质顶点的属性。最终得到三个整合了多元生物信息的PPI网络,如表1所示。
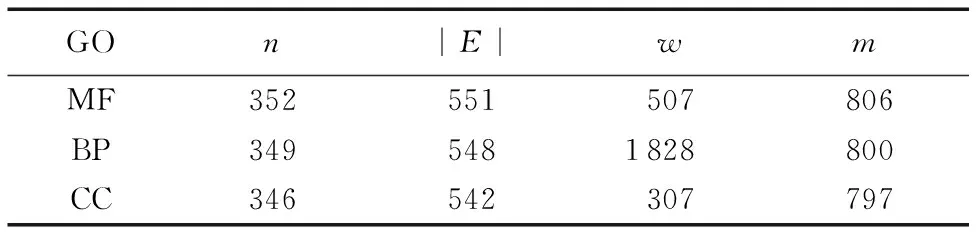
表1 人类数据集详情Tab. 1 Details of human data set
3.2 评价指标
本文将精确度(Precision)、召回率(Recall)和F值(F-Measure)作为评价指标来衡量算法的预测效果[32],其定义如式(13)~(15):
(13)
(14)
(15)
其中:TP表示预测的功能术语正确的个数,FP表示预测的功能术语错误的个数,FN表示实际的功能术语没有被预测到的个数。
3.3 性能评价
本节首先给出HPMM在MF、BP和CC三个PPI网络的功能模块特征和属性主成分特征提取情况及MLP节点设置情况,然后对其与CIA[12]和GoDIN[15]的预测效果进行比较分析。
从表2中可以看出,HPMM在三个PPI网络上提取的功能模块数和NG模块度差异不大。
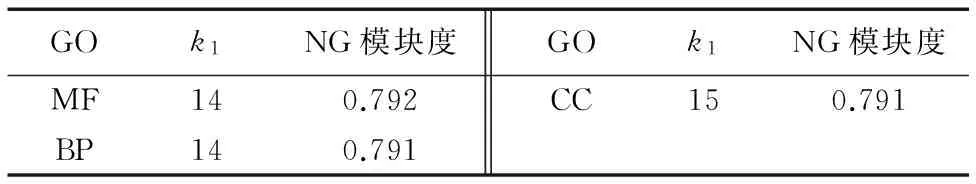
表2 不同PPI网络的功能模块特征Tab. 2 Functional module features of different PPI networks
在表3中,三个PPI网络的蛋白质属性主成分特征提取结果均接近70%的降维率。例如在MF网络中,蛋白质属性特征的个数由806降至243,降维率为69.9%。

表3 不同PPI网络的属性主成分特征Tab. 3 Attribute features of different PPI networks
从表4中可以看出,对三个PPI网络建立的MLP的输入层的节点数相近。这是因为三个PPI网络上的蛋白质功能模块特征数k1和属性主成分特征数k2接近。此外,由于MLP的输出层节点数和隐藏层节点数取决于GO术语数,故对BP网络建立的MLP的输出层节点数和隐藏层节点数最多,对MF网络建立的MLP的输出层节点数和隐藏层节点数介于BP网络和CC网络之间,对CC网络建立的MLP的输出层节点数和隐藏层节点数最少。

表4 MLP节点设置Tab. 4 MLP nodes setting
采用10折交叉验证(Ten-fold Cross Validation)来测试HPMM的预测效果。如3.1节所述,MF、BP和CC网络中的蛋白质均为经过注释的,为测试功能预测方法的性能,实验中分别将每个网络中的蛋白质平均分成10份,轮流将其中9份作为功能已知的蛋白质用于训练模型,其中1份作为功能未知的蛋白质用于预测,合并10次预测的结果,将预测概率最高的l个GO术语作为蛋白质的功能,对于MF、BP和CC网络,l分别设为6,15,5。最后将其与真实的功能注释情况比较。表5给出了HPMM、CIA和GoDIN这3种基于PPI网络的功能预测方法在MF、BP和CC网络上的精确度、召回率和F值。从精确度看,HPMM在3个网络上均高于CIA和GoDIN,并且在MF和CC网络上优势明显。从召回率看,HPMM在3个网络上均高于CIA,但不如GoDIN。这可能是由于GoDIN方法预测的GO术语较多,以降低精确度为代价提高了召回率。从F值看, HPMM在3个网络上均高于CIA和GoDIN,并且在MF和CC网络上优势明显。总的来看,HPMM能够有效预测蛋白质的功能,并在精确度和F值上优于CIA和GoDIN,其优势在MF和CC网络上尤为显著,这可能是由于这可能是由于HPMM考虑的蛋白质属性(家族、结构域和重要位点)与MF和CC的联系更紧密,故对其预测效果的提升幅度较大。
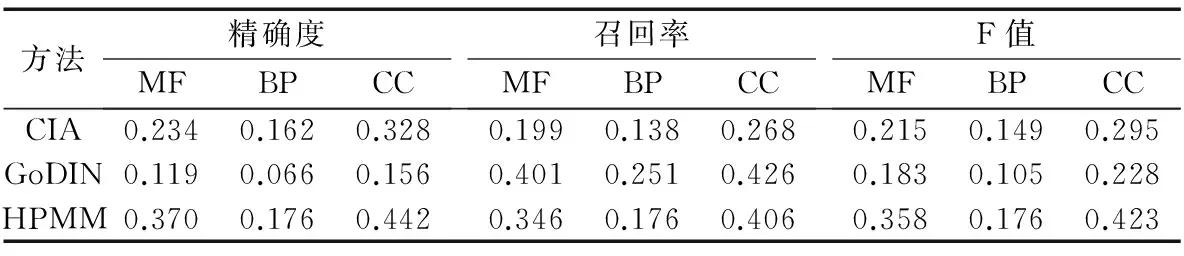
表5 蛋白质功能预测方法性能比较Tab. 5 Performance comparison of protein function prediction methods
4 结语
基于PPI网络的方法是近年来较为流行的一类蛋白质功能预测方法。这类方法预测成本较低,但其效果容易受PPI网络中数据噪声的影响。针对该问题,本文提出了一种基于机器学习的蛋白质功能预测方法HPMM。该方法将蛋白质家族、结构域和重要位点信息整合到PPI网络中,综合考虑蛋白质的微观信息和宏观相互作用以减轻网络中数据噪声的影响,并结合了层次聚类、主成分分析和多层感知器三种机器学习技术来预测蛋白质的功能。为验证HPMM的有效性,从DIP数据库下载人类PPI网络,从InterPro数据库获取蛋白质家族、结构域和重要位点对应的编号,并采用了GO功能注释方案。实验结果证明该方法能有效预测蛋白质的功能,并且在精确度与F值上优于CIA和GoDIN这两种完全基于PPI网络的方法。对于蛋白质功能预测今后的研究, 我们认为可以从以下几个方面入手:1)通过链路预测与图重构技术建立更具有生物统计特性的PPI网络以降低数据噪声的影响。2)深入研究PPI网络拓扑结构,综合考虑网络的局部特性与全局特性用于功能预测。3)研究整合多元生物数据的方法以提升预测效果。
参考文献(References)
[1] ANFINSEN C B, HABER E, SELA M, et al. The kinetics of formation of native ribonuclease during oxidation of the reduced polypeptide chain [J]. Proceedings of the National Academy of Sciences of the United States of America, 1961, 47(9):1309-1314.
[2] ALTSCHUL S F, GISH W, MILLER W, et al. Basic local alignment search tool [J]. Journal of Molecular Biology, 1990, 215(3): 403-410.
[3] ALTSCHUL S F, MADDEN T L, SCHFFER A A, et al. Gapped BLAST and PSI-BLAST: a new generation of protein database search programs [J]. Nucleic Acids Research, 1997, 25(17): 3389-3402.
[4] GILKS W R, AUDIT B, de ANGELIS D, et al. Percolation of annotation errors through hierarchically structured protein sequence databases [J]. Mathematical Biosciences, 2005, 193(2): 223-234.
[5] YE Y, GODZIK A. FATCAT: a Web server for flexible structure comparison and structure similarity searching [J]. Nucleic Acids Research, 2004, 32(Web Server issue):W582-W585.
[7] LASKOWSKI R A, WATSON J D, THORNTON J M. From protein structure to biochemical function? [J]. Journal of Structural & Functional Genomics, 2003, 4(2/3):167-177.
[8] WATSON J D, LASKOWSKI R A, THORNTON J M. Predicting protein function from sequence and structural data [J]. Current Opinion in Structural Biology, 2005, 15(3): 275-284.
[9] YOU Z H, LEI Y K, ZHU L, et al. Prediction of protein-protein interactions from amino acid sequences with ensemble extreme learning machines and principal component analysis [J]. BMC Bioinformatics, 2013, 14(S8): 1-11.
[10] WEI L, XING P, ZENG J, et al. Improved prediction of protein-protein interactions using novel negative samples, features, and an ensemble classifier [J]. Artificial Intelligence in Medicine, 2017,83: 67-74.
[11] OLIVER S. Proteomics: guilt-by-association goes global [J]. Nature, 2000, 403(6770): 601-603.
[12] CHI X, HOU J. An iterative approach of protein function prediction [J]. BMC Bioinformatics, 2011, 12(1): 437-445.
[13] XIONG W, XIE L, GUAN J, et al. Active learning for protein function prediction in protein-protein interaction networks [C]// Proceedings of the 8th IAPR International Conference on Pattern Recognition in Bioinformatics. Berlin: Springer, 2014: 172-183.
[14] WANG H, HUANG H, DING C. Function-function correlated multi-label protein function prediction over interaction networks [C]// Proceedings of the 16th Annual International Conference on Research in Computational Molecular Biology. Berlin: Springer, 2012: 302-313.
[15] TENG Z, GUO M, LIU X, et al. Revealing protein functions based on relationships of interacting proteins and GO terms [J]. Journal of Computational Biology, 2013,20(4): 322-343.
[16] YU G, WANG J, LIU J. Protein function prediction by random walks on a hybrid graph [J]. Current Proteomics, 2016, 13(2): 130-142.
[17] HARTWELL L H, HOPFIELD J J, LEIBLER S, et al. From molecular to modular cell biology [J]. Nature, 1999, 402(6761 Suppl):47-52.
[18] RIVES A W, GALITSKI T. Modular organization of cellular networks [J]. Proceedings of the National Academy of Sciences of the United States of America, 2003, 100(3): 1128-1133.
[20] CLAUSET A, NEWMAN M E J, MOORE C. Finding community structure in very large networks [J]. Physical Review E: Statistical, Nonlinear, and Soft Matter Physics, 2004, 70(6): 066111.
[21] NEWMAN M E J, GIRVAN M. Finding and evaluating community structure in networks [J]. Physical Review E: Statistical, Nonlinear, and Soft Matter Physics, 2004, 69(2): 026113.
[22] ABDI H, WILLIAMS L J. Principal component analysis [J]. Wiley Interdisciplinary Reviews Computational Statistics, 2010, 2(4): 433-459.
[23] GILLIS J, PAVLIDIS P. The impact of multifunctional genes on “guilt by association” analysis [J]. PLOS ONE, 2011, 6(2): e17258.
[24] CARPENTER G A, GROSSBERG S. Self-organizing neural networks for supervised and unsupervised learning and prediction [M]// From Statistics to Neural Networks, NATO ASI Series 136. Berlin: Springer, 1994: 319-348.
[25] GLOROT X, BORDES A, BENGIO Y. Deep sparse rectifier neural networks [EB/OL]. [2017- 03- 01]. http://proceedings.mlr.press/v15/glorot11a/glorot11a.pdf.
[26] 刘威,刘尚,周璇.BP神经网络子批量学习方法研究[J].智能
系统学报,2016,11(2):226-232.(LIU W, LIU S, ZHOU X. Subbatch learning method for BP neural networks [J]. CAAI Transactions on Intelligent Systems, 2016, 11(2):226-232.)
[27] XENARIOS I, RICE D W, SALWINSKI L, et al. DIP: the database of interacting proteins [J]. Nucleic Acids Research, 2000, 28(1): 289-291.
[28] ASHBURNER M, BALL C A, BLAKE J A, et al. Gene ontology: tool for the unification of biology [J]. Nature Genetics, 2000, 25(1): 25-29.
[29] MULDER N J, APWEILER R, ATTWOOD T K, et al. InterPro, progress and status in 2005 [J]. Nucleic Acids Research, 2005, 33(Database issue): D201-D205.
[30] CONSORTIUM U P. The Universal Protein resource (UniProt) in 2010 [J]. Nucleic Acids Research, 2010, 38(Database issue): 142-148.
[31] DURINCK S, SPELLMAN P T, BIRNEY E, et al. Mapping identifiers for the integration of genomic datasets with the R/Bioconductor package biomaRt [J]. Nature Protocols, 2009, 4(8):1184-1191.
[32] RADIVOJAC P, CLARK W T, ORON T R, et al. A large-scale evaluation of computational protein function prediction [J]. Nature Methods, 2013, 10(3):221-227.
This work is partially supported by the National Natural Science Foundation of China (61363035, 61762015), the Natural Science Foundation of Guangxi (2015GXNSFAA139288), the “Bagui Scholars” Project, the Systematic Research Foundation of Guangxi Key Laboratory of Multi-source Information Mining and Safety (14-A-03-02, 15-A-03-02), the Guangxi Graduate Education Innovation Program (XYCSZ2017067).
TANGJiaqi, born in 1992, M. S. candidate. His research interests include bioinformatics, machine learning.
WUJingli, born in 1978, Ph. D., professor. Her research interests include bioinformatics, algorithm design and analysis.

