莲草直胸跳甲小分子热激蛋白sHsp20.8基因的克隆及生物信息学分析
,,,,*
(1.山西农业大学 生命科学学院,山西 太谷 030801;2.山西农业大学 农学院,山西 太谷 030801)
热激蛋白(heat shock protein, Hsps)又称应激蛋白或分子伴侣,是细胞或生物体在遭受生物或非生物胁迫后,表达量增加或新合成的蛋白质[1],其结构高度保守,主要参与蛋白的转运、折叠、维持蛋白质自身稳定及免疫应答[2]。Hsps根据分子量、氨基酸序列和功能可以划分为五大家族,即小分子热激蛋白(small heat shock proteins, sHsps)、Hsp40、Hsp60、Hsp70和Hsp90家族[3]。在HSPs家族中,小分子热激蛋白是最不保守、极具多样性的,表现出广泛的序列变异和进化趋异。尽管sHsps在序列和大小上具有多样化,但绝大数拥有一些共同的特征。其单体分子量范围为12~42 kDa之间,主要结构由N末端区域、C末端区域及C端延伸区构成,高度保守的C末端有一个约90个氨基酸的结构域,被称为α晶体状结构域(alpha-crystallins domain, ACD)。ACD域的两侧是由平均55个氨基酸组成的N端和一个C末端(通常<20个氨基酸)构成[4,5]。sHsps不仅在胁迫条件下能够诱导表达,并且部分sHsps也是生物体中特定的组成成分,其以寡聚体天然状态发挥功能作用,并参与一些重要的细胞生理活动。作为非依赖ATP的分子伴侣,sHsps是细胞防御的第一道防线,防止细胞基质中的蛋白质发生不可逆的变性,尤其是胁迫细胞的蛋白变性[6,7]。近年来,与其它类型的热激蛋白相比,对sHsps的结构、功能和运行机制的研究相对较少。有关昆虫sHsps的研究,也主要集中在几种模式昆虫,这些小分子热激蛋白在昆虫的生长发育、生殖以及滞育等重要生命活动中发挥重要的作用,同时在抵御胁迫环境以及适应性进化中也具有重要意义[8]。
莲草直胸跳甲(Agasicleshygrophila),属鞘翅目Coleoptera,叶甲科Chrysomelidae,原产于南美洲阿根廷,是20世纪30年代引入我国的全球性恶性杂草—喜旱莲子草(Alternantheraphiloxeroides)的专食性天敌昆虫[9~12]。最新研究表明,随着全球气候变暖,喜旱莲子草和莲草直胸跳甲在我国均已向高纬度地区逐渐扩散,但喜旱莲子草的扩散面积远大于莲草直胸跳甲,主要原因是莲草直胸跳甲对环境的适应能力低于喜旱莲子草[13]。在高纬地区,低温限制了莲草直胸跳甲的分布北界。在低纬度地区,莲草直胸跳甲种群随季节变化,消长明显。温度是影响昆虫种群数量和地理分布的重要因素[14]。在自然界中昆虫经常面临冷或热的温度胁迫,经过长期的进化适应,昆虫自身也发展出了多种生理和生化上的应对策略。热激蛋白基因是与昆虫温度适应最为密切的基因之一,热激蛋白基因的诱导表达也是昆虫在极端温度下适应、生存所必须的[14,15]。近年来研究也发现,sHsps的表达可以提高昆虫的温度耐受性[14],但关于莲草直胸跳甲sHsps的研究还未见报道。
本研究基于实验室已完成的莲草直胸跳甲PacBio SMRT测序数据[16],克隆鉴定了一条带有完整开放阅读框的sHsp基因,根据其蛋白质的分子量大小命名为sHsp20.8,然后对其序列特征、理化性质及系统发育等进行生物信息学分析。为深入研究小分子热激蛋白基因的功能,探讨莲草直胸跳甲对环境适应的分子机理提供理论依据。
1 材料和方法
1.1 研究材料
莲草直胸跳甲:采集于华南农业大学(中国,广州),饲养于山西农业大学养虫室(山西,太谷),标准化条件(25 ℃±1℃,L∶D=14∶10,RH=85%±5%)下多代饲养,使之成为遗传背景较一致的试验种群。
喜旱莲子草:采自浙江省玉环县,现种植于山西农业大学生物安全与生物防治研究基地温室中。
1.2 sHsp20.8基因的克隆鉴定及测序
通过莲草直胸跳甲PacBio SMRT测序数据筛选sHsp20.8相关序列,然后使用Vector NTI软件对sHsp20.8的EST序列群进行拼接组装,获得完整的sHsp20.8基因序列。根据完整序列利用Primer3(http://bioinfo.ut.ee/primer3-0.4.0/)设计引物:sHsp20.8-F(TCCACAGATGGTGTGCTGTC)和sHsp20.8-R(CCGATTTAGCTGGTTCTCCA)。莲草直胸跳甲的总RNA的提取,通过PT-PCR扩增sHsp20.8序列,PCR反应程序:94 ℃预变性3 min;94 ℃变性30 s,56 ℃退火30 s,72 ℃延伸2 min;共35个循环;之后72 ℃延伸5 min,扩增产物回收后连接到pMD20-T载体,挑取阳性克隆送上海生工测序验证。
1.3 sHsp20.8基因及编码蛋白的生物信息学分析
采用NCBI ORF finder(https://www.ncbi.nlm.nih.gov/orffinder/)预测sHsp20.8基因的开放阅读框ORF。再经BLASTp相似性搜索,采用Clustal X 2.1软件进行多序列比对。通过SMART(http://smart.embl-heidelberg.de/)对sHsp20.8基因编码蛋白序列的保守结构域进行分析。并用MOTIF(http://www.genome.jp/tools/motif/)预测其motif。再通过ExPASy-ProtParam tool程序(http://web.expasy.org/protparam/)预测分析该基因编码蛋白序列的基本理化性质。同时还采用ProtScale(http://web.expasy.org/protscale/)在线预测sHsp20.8蛋白的亲/疏水性。SignalP 4.1 Setver(http://www.cbs.dtu.dk/services/SignalP/)分析sHsp20.8蛋白的信号肽[17]。亚细胞定位采用PSORT II(https://psort.hgc.jp/form2.html)。再用TMHMM Server v.2.0(http://www.cbs.dtu.dk/services/TMHMM/)预测sHsp20.8蛋白跨膜区。
蛋白的二级结构预测,采用在线SOMPA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)。用ProtFun 2.2 Server(http://www.cbs.dtu.dk/services/ProtFun/)预测其蛋白的功能。使用Phyre2(http://www.sbg.bio.ic.ac.uk/~phyre2/html/page.cgi?id=index)在线软件对莲草直胸跳甲sHsp20.8蛋白的三级结构预测。再使用MEGA7.0软件,选择邻接法(neighbor-joining method, NJ),构建系统发育树并分析其系统发育。
2 结果与分析
2.1 sHsp20.8基因及编码的蛋白序列
通过序列拼接及进一步试验验证,得到莲草直胸跳甲一条完整CDS,经ORF finder分析,该序列包含一个543 bp的完整ORF,编码180个氨基酸(图1)。序列分析发现,该蛋白在C端含有小分子热激蛋白典型的α晶体蛋白结构域(alpha-crystallin domain, ACD),该结构域是小分子热激蛋白家族进化上保守的标志,也是行使其功能所必要的结构。另外在C-末端延伸的保守区存在1个“I/L-X-I/L”的保守特征基序(图2)。通过BLASTp相似性搜索和同源性比较发现,莲草直胸跳甲sHsp20.8氨基酸序列与其它昆虫有着较高的相似性,尤其在α晶体蛋白结域。
2.2 sHsp20.8的保守结构域分析及motif预测
通过SMATR分析莲草直胸跳甲sHsp20.8蛋白质序列的结构功能域,结果显示该蛋白在第67~162区域,包含Hsp20家族的保守结构域(图3)。使用MOTIF在线程序(选择Pfam和NCBI-CDD数据库)预测显示,所有比对上的motif在65~162区域内均含有Hsp20/alpha crystallin family保守结构域。以上结果均表明,本研究所克隆的sHsp20.8属于Hsp20家族,并且获得该基因的完整序列。

图3 莲草直胸跳甲sHsp20.8保守结构域分析Fig.3 The analysis of conserved domain of A. hygophila sHsp20.8
2.3 sHsp20.8的理化性质及疏水性分析
ProParam对sHsp20.8基因所编码的180个氨基酸预测结果显示,sHsp20.8的分子量为20.8 kDa,理论等电点(pI)为6.45,分子式为C920H1460N262O283S5,总共包括2 930个原子。在组成sHsp20.8蛋白的20种氨基酸中,相对含量较高的氨基酸有Glu(17个,9.4%)、Lys(16个,8.9%)、Leu(14个,7.8%)、Thr(13个,7.2%),而半胱氨酸(Cys)所占比例最低,仅为0.6%,不含有色氨酸Trp。带正电荷残基总数(Arg + Lys)为28个,负电荷残基数(Asp + Glu)为30个。不稳定指数为45.35,根据不稳定指数小于40为稳定蛋白,推测sHsp20.8蛋白是不稳定的。脂肪系数为74.17,表明莲草直胸跳甲sHsp20.8蛋白热稳定性良好。ProtScale(Hphob./Kyte & Doolittle)疏水性结果显示(图4),该蛋白亲水性最大值为-3.522,疏水性最大值为1.289,其中亲水氨基酸含量较多,约占84.88%。sHsp20.8蛋白的总亲水平均值为-0.846,结果表明该蛋白是可溶性蛋白质。

图4 莲草直胸跳甲sHsp20.8氨基酸疏水性/亲水性预测Fig.4 Hydrophobicity/hydrophilicity prediction of A. hygrophila sHsp20.8
2.4 sHsp20.8的信号肽、亚细胞定位及跨膜分析
使用SignalP 4.1 Setver在线预测莲草直胸跳甲sHsp20.8蛋白的信号肽,结果显示mean S -Value为0.112,依据其大于0.5则判断为分泌蛋白,说明sHsp20.8蛋白不具有信号肽,不是分泌蛋白。PSORT II亚细胞定位预测也表明sHsp20.8蛋白定位于细胞核中。TMHMM跨膜预测显示sHsp20.8蛋白不含跨膜区。以上预测结果均表明,该蛋白可能主要在细胞核中防止蛋白发生不可逆的变性,从而使细胞免受不可逆的蛋白聚集导致损伤。
2.5 sHsp20.8的二级结构预测与分析
使用SOPMA软件对莲草直胸跳甲sHsp20.8蛋白二级结构分析结果(图5)显示,该蛋白质是由56.11%的无规则卷曲(Cc),23.89%的α螺旋(Hh),16.11%的β折叠(Ee)和3.89%的β转角(Tt)组成的。虽然4种结构类型贯穿于整条氨基酸链,但其中α螺旋和无规则卷曲是其主要的构成元件,β转角含量相对较少,仅零散分布于其中。ProtFun软件对sHsp20.8蛋白的功能分类预测结果显示,该蛋白具有酶活性。功能类别(functional category)显示该蛋白可能有运输和结合功能,与小分子热激蛋白抑制蛋白变性、促进蛋白折叠等特性较为符合。

图5 sHsp20.8的二级结构预测Fig.5 Secondary structure prediction of sHsp20.8注:A. α螺旋;B. 无规卷曲;C. β折叠;D. β转角。 Note:A. Alpha helix;B. Random coil;C. Extend strand;D. Beta turn.
2.6 sHsp20.8的三级结构预测
采用在线工具Phyre2进行sHsp20.8蛋白三级结构的同源建模,使用PyMOL三级结构显示软件分析sHsp20.8蛋白同源建模结果(图6)。可以清楚的区分出该蛋白N端结构域和C端结构域,其C末端有一个α晶体状结构域(ACD),6个反向平行β链形成的β夹层结构(β-sandwich)也主要集中于此。尽管sHsps序列间的保守性较低,但ACD的β夹层仍然是sHsps家族进化上保守的标志[5]。N端主要为α螺旋结构,能调节小分子热激蛋白形成多聚复合物。N端保守性较低,仅在密切相关的物种之间发现N末端序列具有保守性。
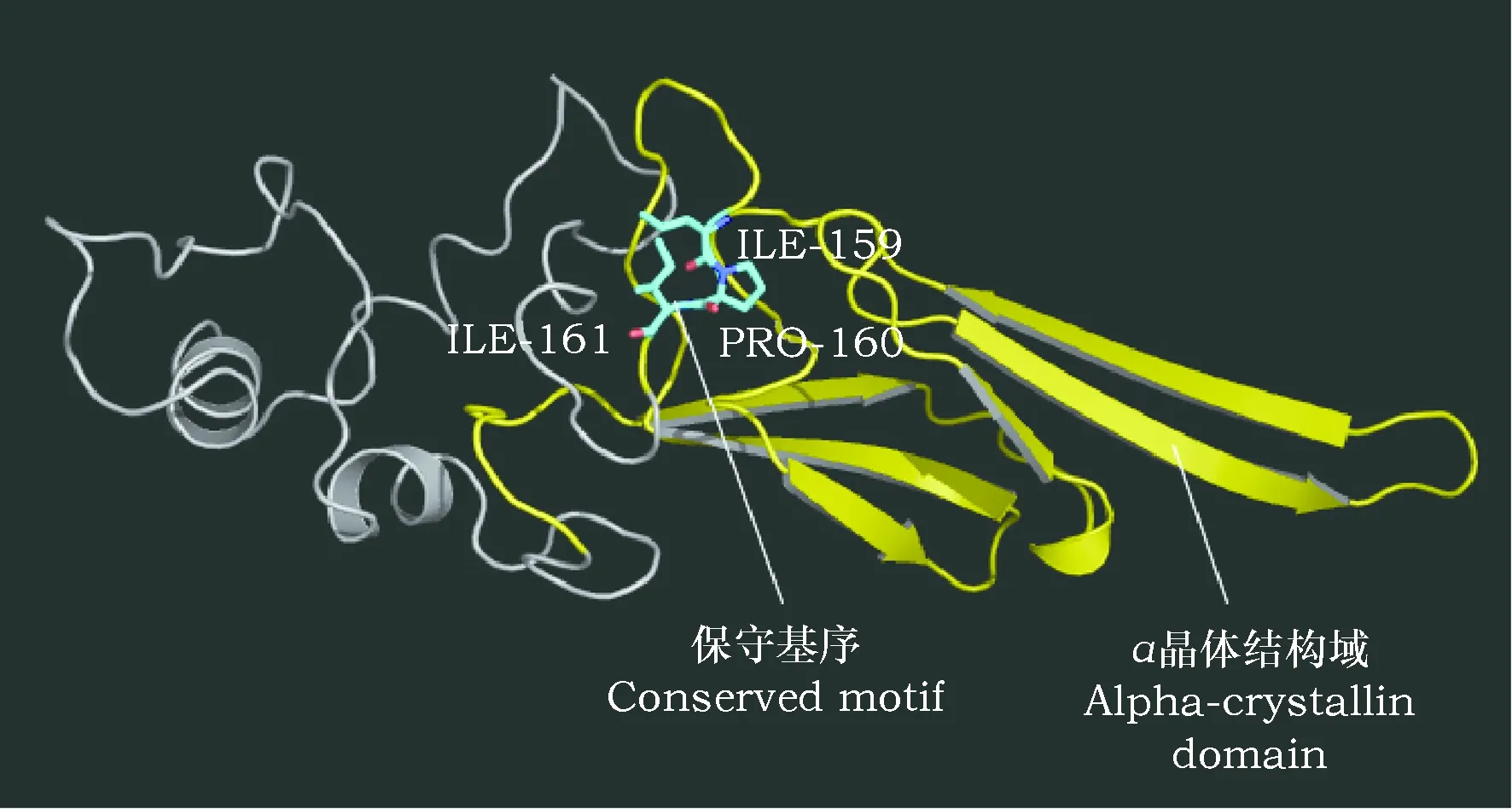
图6 莲草直胸跳甲sHsp20.8的三级结构Fig.6 Tertiary structure of A. hygrophila sHsp20.8 注:黄色表示莲草直胸跳甲sHsp20.8的α晶体状结构域;蓝色表示其特征基序(数字表示氨基酸位置)。 Note:The yellow color indicates the alpha-crystallin domain of A. hygrophila sHsp20.8;the blue color indicates the conserved motif(the number indicates the position of amino acids).
2.7 系统发育分析
利用MEGA7.0软件对5个目28种昆虫的33个sHsps氨基酸序列进行多序列比对。使用邻接法,重复1 000次,构建Bootstrap验证的系统进化树,图中框出的为莲草直胸跳甲的sHsp20.8。图7结果显示,莲草直胸跳甲sHsp20.8与花绒坚甲(Dastarcushelophoroides)、稻象甲(Lissorhoptrusoryzophilus)的sHsps亲缘关系最近聚为一支,与其他鞘翅目昆虫也共同聚为一支。5个目的昆虫都能各自分开聚类,符合昆虫分类学上的亲缘关系,表明sHsps蛋白也具有物种特异性,可以用于昆虫的系统发育分析。

图7 莲草直胸跳甲sHsp20.8和其他昆虫同源蛋白的系统进化树Fig.7 Phylogenetic tree of A. hygrophila sHsp20.8 and homologous proteins from other insect species based on the amino acid sequences 注:GenBank登录号。 Note:GenBank accession number: 1) A. pernyi (Antheraea pernyi) ApsHSP21: AFQ02692.1; 2) S.litura (Spodoptera litura) SlsHSP: ADK55522.1; 3) S. nonagrioides (Sesamia nonagrioides) SnsHSP20.8: ABC68342.2; 4) B.mori (Bombyx mori) BmsHSP20.4: NP_001037038.1; 5) C. suppressalis (Chilo suppressalis) CssHSP19.7: BAE94664.1; 6) C. pomonella (Cydia pomonella) CpsHSP22.2: ADX96002.1; 7) P. xylostella (Plutella xylostella) PxsHSP19.5: BAE48744.1; 8) D. plexippus (Danaus plexippus) DpsHSP22.2: OWR51030.1; 9) A. yamamai (Antheraea yamamai) AysHSP: AGY56063.1; 10) C. pomonella (Cydia pomonella) CpsHSP19.9: ADX96001.1; 11)M. brassicae (Mamestra brassicae) MbsHSP20.7: BAF03557.1; 12) B.tabaci (Bemisia tabaci) BtsHSP20: ADG03467.1; 13)T.vaporariorum (Trialeurodes vaporariorum) TvsHSP20: ACH85200.1; 14)T. vaporariorum (Trialeurodes vaporariorum) TvsHSP: ACI15853.1; 15) L. sativae (Liriomyza sativae) LssHSP20.8: ABE57141.1; 16) L. huidobrensis (Liriomyza huidobrensis) LhsHSP20: ABE57137.1; 17) L. sativae (Liriomyza sativae) LssHSP21.3: ABE57138.1; 18) D. antiqua (Delia antiqua) DasHSP23: ADX36150.1; 19) D. melanogaster (Drosophila melanogaster) DmsHSP23: AAL28629.1; 20) G. morsitans morsitans (Glossina morsitans morsitans) GmmsHSP23: ADD18977.1; 21) D. triauraria (Drosophila triauraria) DtsHSP27: AEB54706.1; 22) G. morsitans morsitans (Glossina morsitans morsitans) GmmsHSP: ADD18487.1; 23) D. ponderosae (Dendroctonus ponderosae) DpsHSP27-like: XP_019759317.1; 24) T. castaneum (Tribolium castaneum) TcsHSP21: XP_974367.1; 25) T. castaneum (Tribolium castaneum) TcsHSP23: XP_973344.1; 26) C. bowringi (Colaphellus bowringi) CbsHSP21: AMK38872.1; 27) G. atrocyanea (Gastrophysa atrocyanea) GasHSP21: BAD91164.1; 28) G. daurica (Galeruca daurica) GdsHSP20.6: ATY47553.1; 29) A. hygrophila (Agasicles hygrophila) AhsHSP20.8: 30) D. helophoroides (Dastarcus helophoroides) DhsHSP: ANA11235.1; 31) L. oryzophilus (Lissorhoptrus oryzophilus) LosHSP: AHE77375.1; 32) A. gambiae (Anopheles gambiae) AgsHSP: AAG15376.1; 33) L. migratoria (Locusta migratoria) LmsHSP20.6: ABC84493.1.
3 讨论与结论
本研究从莲草直胸跳甲中克隆获得了一条包含完整开放阅读框的小分子热激蛋白sHsp20.8基因。发现该基因编码的氨基酸序列含有小分子热激蛋白保守的α晶体蛋白结构域,含有典型的“I/L-X-I/L”保守特征基序,有研究表明该特征基序有助于驱动大寡聚体的装配[18]。当细胞受到外界环境的胁迫时,部分变性的蛋白质结合至sHsps的寡聚体上,使细胞免受不可逆蛋白聚集而导致的损伤;胁迫消失时,复合物解体,在ATP需能伴侣如Hsp70的帮助下重新折叠成正确的空间构象,从而发挥其正常的生物学功能[19,20]。与大分子热激蛋白相比,尽管小分子热激蛋白表现出较大的序列变异和进化趋异,但本研究对5个目28种昆虫的33个sHsps系统进化分析表明,各目昆虫的sHsps可以单独聚类,进一步说明小分子热激蛋白基因也可以作为昆虫系统进化分析的标志基因。
小分子热激蛋白(sHsps)的分布较广泛,在微生物、植物和动物中都有分布,同时也是分子量最小,保守性最低的一类热激蛋白。目前对于sHsps虽然研究不是特别深入,但已引起研究者的广泛关注。sHsps生物功能多样,大多数时候以寡聚体天然状态存在和发挥功能作用,当其作为分子伴侣时,通过磷酸化或去磷酸化的修饰来调节寡聚体的大小和状态,进而形成不同的寡聚体结合不同的底物蛋白[21,22]。研究表明,sHsps可以增强生物体对环境胁迫的耐受和恢复能力,并辅助变性蛋白质的结构重新组装,保持蛋白质的正常结构和生理功能[23]。如通过RNAi敲除果蝇sHsp22和sHsp23则中断了果蝇在寒冷伤害后的恢复进程[24];水稻大螟Sesamiainferens经过高温胁迫,sHsp19.6和sHsp20.6的mRNA水平显著上调,它们在其适应环境变化中发挥重要作用[25]。在昆虫中也鉴定出了多种功能不同的sHsps,这些sHsps在昆虫的生长发育、滞育及温度适应性中可能发挥重要作用[8]。有研究发现,家蚕Bombyxmori在滞育阶段代谢活动明显下降,但在其胚胎滞育期sHsp20.8表达上调[26];蛀茎夜蛾Sesamianonagrioides滞育幼虫中sHsp19.5表达稳定,而sHsp20.8先下调,在滞育终止时上调,参与了其滞育调控[27]。此外,sHsps调控发育的现象,在植物和动物的生殖和胚胎组织中都有所发现[28]。总之,sHsps可以参与生物体生长发育的调控和胁迫等应激反应。但这些研究主要集中在基因的表达模式水平,因此要想全面认识sHsps的功能作用,还需有效结合多种试验技术并更新试验方法。
本研究成功克隆了莲草直胸跳甲sHsp20.8基因,为深入研究sHsps在莲草直胸跳甲生长发育、温度适应性中的功能作用及分子机制奠定理论基础,同时也对莲草直胸跳甲生物防治喜旱莲子草的方针策略具有重要指导意义。

