宏基因组学及其在鱼类肠道微生物中的研究进展
陈泓宇,闫海亚,赵胤丞,肖 蘅,陈善元
( 云南大学 生命科学学院,云南 昆明 650091 )
传统的微生物研究方法主要利用微生物培养技术,但自然界中只有不到1%的微生物可以通过传统培养方法获得[1],极大的限制了微生物相关研究。宏基因组学最早由Handelsman等提出,自环境样品中提取出含有的总微生物DNA构建微生物宏基因组文库[2]。宏基因组学方法共包括全宏基因组技术及16S rRNA片段扩增技术,通过高通量测序,打破了传统以培养为主的微生物研究方式,又比变性梯度凝胶电泳和末端片段长度多态性等早期用于微生物研究的分子生物学技术效率更高,花费更少。利用宏基因组学方法可以准确获得样品微生物物种组成及丰度等信息[3],进入数据库预测微生物功能,还能分析微生物代谢网络等多方面的信息[4],为微生物的研究提供了便利的工具。
鱼类肠道微生物被认为对宿主生命活动有重要影响,在宿主免疫、宿主适应性及宿主营养代谢等方面均有一定作用。鱼类作为食品中重要的蛋白质来源,也是主要的饲养观赏动物。利用宏基因组学手段研究鱼类肠道微生物组成并分析鱼类免疫、食性等,对了解鱼类肠道微生物生理生化功能,水产经济鱼类的养殖繁育、病害防治及观赏鱼类的培育有重要意义。基于已完成宏基因组测序的部分鱼类肠道微生物及其相关食性研究信息,笔者从鱼类肠道微生物组成、鱼类肠道微生物与免疫、鱼类肠道微生物与食性等3个方面进行综述,并对之后的鱼类肠道宏基因组测序工作作出展望,以期了解鱼类肠道微生物的结构组成模式,分析鱼类肠道微生物与宿主免疫及食性间的关系,为今后鱼类肠道微生物相关研究提供参考。
1 宏基因组学
1.1 样品总DNA提取
宏基因组学以微生物生态群落中所有微生物基因组为研究对象,以基因组学技术为依托[5],其主要研究程序包括:(1)自样品中提取DNA;(2)DNA克隆到合适载体;(3)载体转化并构建基因组文库;(4)对基因组文库进行分析和筛选。
获取高纯度、高质量的宏基因组DNA是后续宏基因组学生物信息分析的基础,宏基因组DNA提取方法根据样品的差异进行不同选择,提取手法主要分为直接提取法(原位裂解法)及间接提取法(异位裂解法),提取试剂常为细菌基因组提取试剂盒及氯仿抽提。原位裂解法主要通过去污剂(SDS等)、机械破碎(玻璃珠搅拌等)、酶处理(蛋白酶K等)释放DNA,加以重提纯化。刘艳超等[6]提取自来水中细菌宏基因组DNA时,直接将醋酸纤维滤膜上收集的自来水菌体加入裂解液进行裂解并提取DNA。在泥土及活性污泥微生物研究的DNA提取过程中,Xia等[7]在细胞裂解前直接利用缓冲液对环境样品进行洗涤处理再将微生物细胞与杂质分离,有助于去除腐殖酸、重金属离子等溶解抑制剂。Li等[8]发现先利用液氮冰冻研磨土壤等环境宏基因样品,再提取样品宏基因组DNA的效果最好。单独使用试剂盒提取虽操作简便,但提取效果最差,产物杂质多且干扰测序,在提取前用TENC缓冲液预处理,可以有效去除腐殖酸和滤杂质,有利于DNA提取[9]。除环境微生物样品外,肠道微生物DNA提取也通常使用间接提取法。Barry等[10]提取猫肠道微生物DNA时,使用灭菌玻璃珠搅拌分解粪便杂质后利用试剂盒提取。异位裂解提取法需先将微生物细胞提取出来后用温和的处理方法(脉冲凝胶电泳DNA回收法等)提取DNA,但由于操作繁琐,效率较低,使用相对较少[11]。
1.2 载体构建及克隆
宏基因组文库的构建形式沿用了分子克隆的基本原理及技术方法,在DNA提取之后与载体连接并转入宿主中。载体的选择主要针对感兴趣的功能基因或目标产物基因的特点分为大片段连接载体、表达载体、穿梭载体等几大类。
微生物产生的活性物质代谢途径常由多个基因簇联合调控,长片段的插入有助于获得完整的代谢途径调控基因簇[12]。目前常用的大片段插入载体有人工染色体和黏粒。细菌人工染色体插入片段长(350 kb),但克隆效率低;黏粒如pWE15可插入片度中等(40 kb)但克隆效率高。Entcheva等[13-14]利用细菌人工染色体载体和黏粒载体构建了插入片段大于50 kb和平均30 kb的宏基因组文库,成功合成抗菌素紫色杆菌素等。重组克隆子后,利用表达载体构建宏基因组文库既有利于提高宏基因表达,又方便活性检测。Henne等[15]就通过直接利用表达载体pBluescript SK+克隆获得了表达脂酶、酯酶及4-羟基丁酸脱氢酶的重组克隆子。相对于长片段载体,表达载体插入片段仅约10 kb,只适合于单一基因及小操纵子产物。穿梭载体能有效的扩大宿主范围,有助于提高外源基因的表达。外源基因的表达受宿主细胞的遗传类型(顺式元件、反式因子、tRNA丰度、密码子偏好等)、细胞基质、细胞的生理状态及初级代谢产物等的影响,Brady等[16]采用黏粒 pOS700Ⅰ穿梭载体,将其用大肠杆菌(Escherichiacoli)构建的宏基因组文库中感兴趣的克隆子外源片段转移到了链霉菌(Streptomyces)中,有效提高了转载基因的表达量。宿主的选择主要需考虑重组载体的稳定性及转化效率,大肠杆菌作为目前遗传背景研究最为透彻,应用最广泛的菌种。是宏基因组来源基因最常用的表达宿主,但大肠杆菌并不是一个高效表达外源基因的宿主细菌,对筛选新基因造成障碍。穿梭宏基因组文库,如利用穿梭载体将大肠杆菌等文库转入链霉菌或假单胞菌属(Pseudomonas)中,将会大大提高筛选效率[17]。
1.3 宏基因组文库筛选
宏基因组文库的筛选方式多样,主要有功能驱动筛选、化合物机构水平筛选、序列驱动筛选及底物诱导基因表达筛选等,每种筛选方法各有优缺点,可结合使用。
功能驱动筛选法是生物活性水平层面的筛选,通过检测并挑选活性的重组克隆子进行筛选,如通过可见性状筛选到了七叶苷水解酶,甘油脱水酶等[18],是新发现分子化合物的常用方法。功能性筛选依赖于功能基因或编码功能蛋白质的表达,常有外源宿主中基因不表达或表达产物活性低,造成了上千个克隆中仅有1%具有活性[15]。并且检测的手段很局限,工作量大且效率很低。
化合物结构水平筛选一定程度上避免了功能驱动筛选依赖生物活性的弊端,通过不同物质在色谱中的峰值差异,比较转入和未转入外源基因的宿主细胞或提取物色谱图,筛选出新结构化合物的克隆子。该筛选方式不一定要具有生物活性,可直接筛选出新结构化合物。Wang等[19]以链霉菌为宿主构建宏基因组文库,利用化合物结构水平筛选法从1020个克隆子中筛选出2个产生新化合物的克隆子并获得了全新小分子物质。
序列驱动筛选是DNA序列水平层面的筛选,根据同类酶基因可能具有的序列相似性为基础,利用相关功能基因的保守序列或相似序列设计杂交探针或PCR引物扩增及挑选阳性克隆[20]。Knietsch等[21]根据已知脱水酶基因保守序列设计PCR引物用于宏基因组DNA扩增,最后筛选出活性较强的产1,3-丙二醇脱水酶克隆子。序列驱动筛选不必依赖宿主表达克隆基因,筛选效率高。但在设计引物及探针时需了解目的基因片段相关信息,不适用于新物质挖掘。
底物诱导表达筛选基于相关基因表达需某些化合物(底物等)的诱导这一特性,通过建立特定的宏基因组文库,利用流式细胞仪筛选出表达受底物诱导的克隆子[22]。
2 宏基因组学在鱼类肠道微生物的应用
2.1 鱼类肠道微生物组成
动物复杂而具有动态的肠道生态系统对营养的吸收具有重要的作用,肠道微生物不仅与宿主生长发育和代谢等功能密切相关[23],还与很多疾病的发生有关(肝硬化、阿兹海默氏症等)[24-25]。1990年Moriarty等开始研究水生动物肠道微生物,水生动物的肠道微生物已被证实有利于宿主的营养、生理(产生维生素、氨基酸、消化酶和某些代谢产物)与免疫过程[26-28],但由于很多微生物无法通过实验室培养得到,传统的以培养为主的微生物研究方式受到了很多限制[29]。将宏基因组学研究手段运用于水生生物,对其胃肠道内含物及黏膜等的微生物进行测序,分析其微生物丰度、种类、结构组成和功能等方面的信息,不仅对鱼类生长发育调控、疾病预防等方面具有重要的意义,还规避了传统培养手段的不足[30]。
鱼类肠道中的优势微生物通常为细菌[31],主要由好氧菌和兼性厌氧菌组成,酵母菌也常在海水或淡水鱼类肠道微生物中发现,通常有三门核心微生物:厚壁菌门、变形菌门、拟杆菌门[32-33]。与恒温动物相比,鱼类肠道微生物丰度和复杂程度较低。外在或内在的因素变化(肠道的结构、水温、饮食因素、饲养条件等)都会影响鱼类肠道微生物的多样性和结构组成[34-35]。淡水鱼类与海水鱼类肠道微生物结构组成具有明显差异[36],在淡水鱼类物种中,气单胞菌属(Aeromonads)、假单胞菌属和A型拟杆菌(Bacteroides)是主要优势菌群,而海洋鱼肠道优势菌群组成则更丰富。如弧菌属(Vibrio)、无色菌属(Achromobacter)、交替单胞属(Alteromonas)、黄杆菌属(Flavobacterium)等[37],这或许与海洋鱼类更复杂的生境和多样化的饮食有关。Verner-Jeffreys等[38]利用宏基因组16S rRNA技术对不同生长时期的大西洋比目鱼(Hippoglossushippoglossus)肠道微生物进行研究发现,在未进食的幼鱼肠道中就发现弧菌属细菌,进食后亮弧菌(V.splendidu)、溶藻弧菌(V.alginolyticus)为优势菌群。但气单胞菌属和弧菌属用常规分离培养手段极难获得,Pond等[39]对虹鳟(Oncorhynchusmykiss)肠道微生物16S rRNA测序并分析肠道微生物组成,对比了传统微生物培养技术,结果显示,利用宏基因组学手段从虹鳟肠道中找到嗜麦芽寡养单胞菌(Stenotrophomonasmaltophilia)、真养罗尔斯通菌(Ralstoniaeutrophia)等传统手段无法获取的肠道微生物。单独使用常规微生物分离培养技术造成微生物信息不全面,分析结果往往产生偏离。而通过宏基因组手段常可发现无法培养的微生物,Einar等[40]通过对大西洋鲑(Salmosalar)肠道分离出的752个微生物16S rRNA测序,还发现了枯草芽孢杆菌(Bacillussubtilis)、氧化微杆菌(Microbacteriumoxydans)等大量被认为无法培养的微生物。鱼类肠道微生物组成受多因素影响,食性偏好及生境影响最为明显,为研究鱼类肠道微生物与宿主适应性之间的联系,Sullam等[41]对不同河流捕捉的的两种不同食性孔雀鱼(Trinidadianguppies)与人工培育孔雀鱼肠道微生物16S rRNA测序,比较其肠道微生物组成结构,研究发现,不但野生孔雀鱼与饲养孔雀鱼肠道微生物组成具有显著差异,同种不同河流生存的野生鱼肠道微生物组成也不同。Marianne河孔雀鱼梭杆菌目为优势微生物,而Guannpo港的孔雀鱼优势微生物为支原体目。实验室养殖孔雀鱼肠道微生物多样性低于野生鱼,个体差异小于野生孔雀鱼。Hennersdorf等[42]也利用宏基因组学技术研究印度尼西亚养殖褐点石斑鱼(Epinephelusfuscoguttatus)和野生石斑鱼肠道微生物,对比饲养与野生鱼肠道微生物组成发现,人工饲养的石斑鱼肠道微生物组成结构比野生石斑鱼更加平衡,肠道微生物多样性及丰度均较野生鱼低,而野生鱼肠道微生物组成个体间差异大于人工饲养鱼。可能由于饲养条件形成的规律食物和生境使宿主肠道微生物处于稳态,形成了固定的优势肠道微生物。鱼肠道微生物组成还与环境微生物存在正相关关系,通过对不同生长时期的罗非鱼(Oreochromis)幼鱼肠道微生物16S rRNA测序研究发现,随着幼鱼的生长发育,其肠道微生物种类与环境微生物种类部分重叠,表明水中的微生物会转移至肠道中,可能对宿主环境适应等具有重要的作用[43]。
2.2 鱼类肠道微生物与免疫
鱼类肠道微生物在抑制病原微生物方面具有重要作用,肠道内的优势菌群可以保护宿主免受环境复杂微生物的感染与侵害,某些肠道微生物代谢物质还能促进肠道上皮细胞增殖,促进免疫系统的应答反应[44]。Stagaman等[45]对68个正常野生型斑马鱼(Daniorerio)及61个缺乏B或T细胞受体功能的斑马鱼肠道微生物16S rRNA V4区测序比较,结果表明,适应性免疫对肠道微生物的组成具有过滤作用,肠道微生物的组成或许可以反映出宿主的免疫状况。Li等[46]对圆口铜鱼(Coreiusguichenoti)11个肠道内含物样品16S rRNA测序,对比健康鱼与病变鱼肠道微生物组成,研究发现,病变鱼肠内细菌多样性低于健康鱼,而病变鱼中变形菌门丰度显著高于健康鱼,其中气单胞菌属可能是造成感染疾病的关键。环境压力,如污染、低氧、温度骤变会使宿主免疫系统受损,导致病原体的入侵,使得肠道微生物组成出现改变。Jankauskien⊇等[47]发现,在海洋鱼类和淡水鱼类肠道中共有的乳酸菌通常不是肠道优势菌群,但生境出现变化后(如当其处于特定池塘培养系统中培养时)乳酸菌会成为肠道优势菌群并保持稳定,用以增进对新生境的适应性,增强免疫能力。通过改变肠道微生物结构来增强免疫能力的适应性手段不只在鱼类中发现,通常一些化学元素(抗生素、污染物、农药、杀虫剂)进入动物消化道中,均会对其肠道微生物组成产生重要影响[48]。在对雉科鸟类肠道内含物进行宏基因测序,并在京都基因与基因组百科全书代谢通路注释肠道微生物后表明,雉科鸟类肠道微生物组成中变形菌门明显高于已知的其他鸟类,在微生物代谢通路中化学物质降解占据较大比重。也许与雉科鸟类不善于飞翔导致其在地面土壤生境进食时摄入大量土壤有害物质有关[49]。除致病微生物(黄杆菌属、气单胞菌属、假单胞菌属)会影响鱼类肠道微生物组成以外,寄生虫也对鱼类肠道及黏膜微生物造成影响。Llewellyn等[50]对被鲑鱼虱(Lepeophtheirussalmonis)寄生后的大西洋鲑皮肤微生物进行16S rRNA V4区测序分析,结果显示,被寄生后的大西洋鲑体表微生物丰富度明显降低。有趣的是,被致病微生物感染的个体通常都伴随着鱼虱的寄生。可能是由于被感染后的个体免疫能力下降而易被寄生虫入侵。
2.3 鱼类肠道微生物与食性
通过分析鱼类肠道微生物的结构组成能反映出其生境状况、饮食等信息,而利用宏基因组学技术,探寻鱼类食性与肠道微生物间的关系,是宏基因组学研究的重要部分,也是目前关注度较高,研究开展较多的微生物宏基因组研究领域[51-52]。
不同食性鱼类肠道微生物中,为消化吸收不同类型的营养物质,常有标志性微生物存在[48]。Li等[53]利用宏基因组16S rRNA测序技术研究了8种自然环境下不同食性(肉食性为主、植食性为主和杂食性为主)鱼类与肠道微生物的关系。结果表明,不同食性的鱼类肠道微生物多样性和优势微生物群结构组成都具有明显差异,为通过鱼类肠道微生物组成分析鱼类主要食性提供了重要指标。通常认为,宿主食物偏好与其肠道微生物组成存在重要联系[54]。Ingerslev等[55]利用宏基因组测序技术研究虹鳟肠道微生物发现,虹鳟肠道微生物的组成由生境及所摄入的食物共同塑造,并且首次摄食可能起到主要的定殖作用。不同食性鱼类肠道微生物组成具有明显差异,并会随宿主摄食和生长状况改变出现变化[48],有些鱼类如孔雀鱼,在生长不同阶段会具有不同的食性偏好,这时其肠道微生物组成也会做出相应调整,协助宿主更好的吸收营养。不同食性鱼类肠道微生物组成由于功能不同(纤维素降解、蛋白质降解、几丁质降解等)具有差异。现有鱼类食性主要分为植食性鱼类、肉食性鱼类、滤食性鱼类、杂食性鱼类4大类。植食性鱼类以植物为主要营养来源,食物富含纤维素和多糖。纤维素的降解依赖于多种纤维素酶,但鱼类自身无法产生纤维素酶,对纤维素的利用只有依赖肠道微生物分解吸收[56],植食性鱼类肠道中特有微生物多为与纤维素降解有关的微生物。Ni等[57]对草鱼(Ctenopharyngodonidellus)肠道微生物16S rRNS测序研究发现,以植物为主要食物的草鱼肠道微生物中,鲸杆菌属(Cetobacterium)、气单胞菌属为优势菌群。Wu等[58]对草鱼肠道微生物宏基因组测序后结果表明,除气单胞菌属外,柠檬酸杆菌属(Citrobacter)、明串珠菌属(Leuconostoc)也在草鱼肠道微生物组成中占据较大比例。与投喂植物性食物饲养的虹鳟肠道微生物具有类似的组成结构[26]。气单胞菌属是淡水植食性鱼类肠道微生物优势菌群,与鲸杆菌属和明串珠菌属均为主要的纤维素降解菌[59]。鲸杆菌属还能发酵碳水化合物,产生维生素B12供宿主吸收利用。肠道微生物中,芽孢杆菌属也是主要生产纤维素酶的微生物之一。Ghosh等[60]对南亚野鲮(Labeorohita)肠道微生物16S rRNA测序后,在其肠道微生物中找到大量环状芽孢杆菌(B.circulans)和短小芽孢杆菌(B.pumilus);Saha等[33]发现在食植莫桑比克罗非鱼(O.mossambica)和草鱼肠道微生物中,环状芽孢杆菌和巨大芽孢杆菌(B.megaterium)为优势菌群;Peixoto等[61]对在亚马逊盆地食草野鱼肠道微生物16S rRNA基因测序也发现大量枯草芽孢杆菌。在多种不同的植食性鱼类肠道微生物中均发现芽孢杆菌属微生物的存在,并均为优势肠道菌群,这可能与植食鱼类需要其帮助消化吸收植物营养物质有关。肉食性鱼类肠道微生物多样性相比植食性和滤食性鱼类要略低,也许与该鱼类较为单一的食性偏好有关,肉食性鱼类肠道微生物组成中也有与纤维素代谢有关的微生物(鲸杆菌属、柠檬酸杆菌属)但量较少。Liu等[62]利用宏基因组16S rRNA技术研究了8种不同食性的野生鱼类,将两种肉食性鱼类翘嘴鳜(Sinipercachuatsi)和翘嘴鲌(Culteralburnus)肠道微生物与植食性鱼类肠道微生物对比研究,结果表明,两种肉食性鱼类肠道微生物以盐单胞菌属(Halomonas)、梭杆菌属(Fusobacterium)等以产生蛋白酶为主要功能的微生物为优势肠道菌群,酶代谢活性显示,肉食性鱼类中胰蛋白酶活性较高而纤维素酶代谢活性低,植食性鱼类中结果相反。草鱼肠道中产生纤维素酶的优势菌类芽孢杆菌属(Paenibacillus)只在植食性鱼类中发现,在肉食性鱼类中并不存在。Ghanbari等[63]通过宏基因组学手段研究滤食性鱼类并进行功能预测和对比其他食性鱼类发现,滤食性鱼类及杂食性鱼类肠道微生物种类及数量最高,结构组成最丰富,功能预测结果主要集中于碳水化合物代谢方面,肠道微生物组成除变形菌门、梭杆菌门、厚壁菌门这几种“核心菌群”外,还存在肉食性、植食性鱼类肠道中少见的放线菌、蓝藻细菌等多种微生物类型。这可能与滤食性杂食性鱼类不但摄食常见植物性虫幼体,还可以滤取个体微小的浮游动植物、有机碎屑、细菌凝聚体等有关,极大扩大了摄食种类,需要复杂的肠道微生物来进行分解吸收[64]。
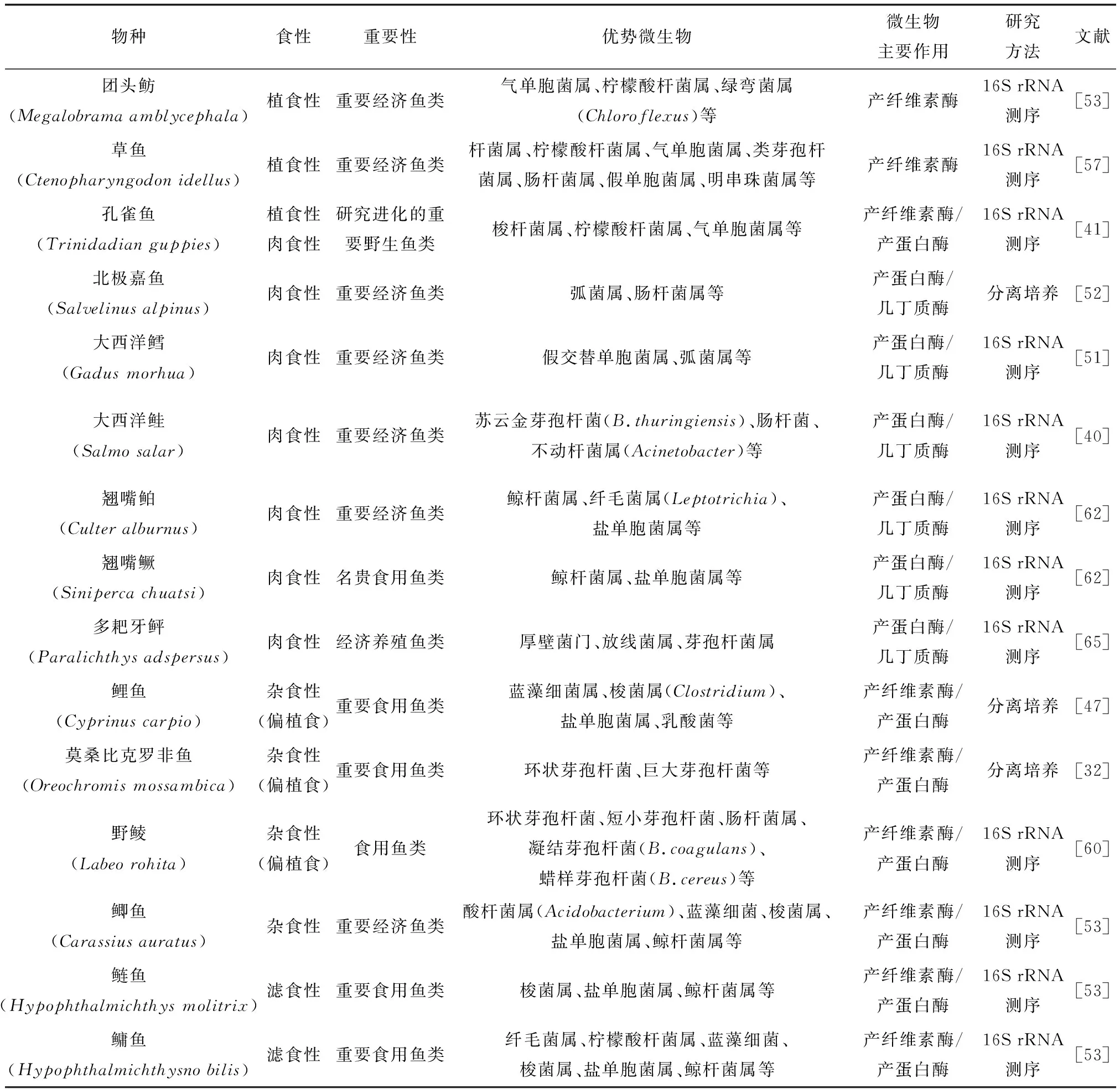
表1 不同食性代表性鱼类肠道优势微生物
3 结语与展望
宏基因组学研究手段绕过了传统微生物培养的弊端,以大数据测序文库覆盖样品中所含有的尽可能多的微生物基因信息,能够全面的分析样本内微生物的种类、丰度以及微生物的功能和代谢通路等信息,现大量运用于动物胃肠道微生物多样性分析,研究宿主、微生物与生境因素之间的相互关系。但是依然存在一些不足,就环境样品微生物研究方面而言,基因组学的手段无法说明微生物基因的表达量情况,当生境或其他因素变化时,同一微生物个体的基因表达也会由于适应环境出现变化,需结合宏转录组学,从转录组水平分析样本微生物的组成及活性基因表达情况,揭示特定生境因素,特定时间下微生物基因表达和调控机制,并且更易发现新的基因,这些基因在某些特定环境条件下可能发挥很大作用[66]。肠道微生物对宿主免疫功能具有重要作用,但由于目前对肠道微生物功能及作用研究有待深入,对鱼类肠道微生物与宿主间的互作机制还需加深了解。就研究鱼类肠道微生物与食性相关性方面。虽目前许多重要经济养殖鱼类(草鱼、鲤鱼、鲑鱼等)都已经进行肠道微生物相关方面的研究,但仅停留在客观描述层面,大多只对肠道微生物的种类及主要结构组成进行了简要概括,未深入探讨肠道微生物结构组成与食性间存在的关系,也少见将肠道微生物功能进行细化并与食性联合分析。在4种食性鱼类物种中,植食性鱼类肠道微生物及食性相关研究开展相对较多,肉食性、杂食性及滤食性鱼类物种肠道微生物研究较少。但鱼类少有单一性、专有性食性,而多为植食性或肉食性为主的杂食性。虽然16S rRNA扩增后测序对肠道样品要求(含量等)略低,但与全宏基因组直接高通量测序相比还存在一些问题,如扩增过程中对不同菌群具有偏好性,无法全面的反映微生物的真实信息。相信随着宏基因组测序成本的下降,分析手段常规化、简便化,越来越多的鱼类肠道微生物相关研究也会更加全面和深入。

