m6A修饰及其对病毒复制过程调控研究进展
薛鹏,蒋涛,沈兴家,2
m6A修饰及其对病毒复制过程调控研究进展
薛鹏1,蒋涛1,沈兴家1,2
1. 江苏科技大学生物技术学院,江苏省蚕桑生物学与生物技术重点实验室,镇江 212018 2. 中国农业科学院蚕业研究所,农业部蚕桑遗传改良重点实验室,镇江 212018
6-甲基腺嘌呤(6-methyladenosine, m6A)修饰是一种在真核生物中普遍存在的RNA修饰方式,在mRNA转运、稳定、翻译等过程中具有重要作用。m6A修饰在病毒复制周期中扮演不同的角色,病毒的复制和宿主对病毒的免疫反应都受到m6A甲基化的影响。本文总结了近年来关于m6A修饰方面的分子作用机制及其对病毒复制以及宿主免疫反应的影响相关研究,以期为病毒生命周期中的表观遗传调控研究提供一定的参考。
6-甲基腺嘌呤;RNA修饰;病毒;复制;免疫
RNA不仅能将遗传信息翻译成蛋白质,而且能调控多种生物学进程,因而其在生物系统中具有重要的作用。作为重要的遗传介质,mRNA的化学修饰是一种重要的RNA转录后调控机制,在众多已知的RNA修饰方式中,m6A甲基化修饰是真核生物mRNA中最普遍的一种修饰方式,于1974年首次被发现,并引起了人们的极大兴趣[1,2]。m6A修饰在从酵母、植物、果蝇到哺乳动物等真核生物到RNA病毒都具有较高的保守性[3]。在体外对甲基转移酶突变和底物偏好性的研究发现,m6A修饰主要集中在终止密码子附近及3′端非翻译区,其共有基序为[G/A/U][G>A]m6AC[U>A>C],而在拟南芥的起始密码子附近也发现了m6A甲基化修饰[4,5]。mRNA上m6A普遍存在而含量极少并且是非随机分布的,这意味着m6A修饰可能在转录后调控中具有重要的作用,而已有的研究表明,m6A修饰可以影响mRNA代谢过程,包括mRNA的加工、出核运输、翻译和稳定性,以及在RNA转录后水平调控基因的表达,进而影响各种生理过程,如生长、发育、生殖、细胞多能性、减数分裂、昼夜节律和疾病发生等[6,7]。
病毒被称为细胞的“寄生虫”,致病病毒对人类的生命健康构成了巨大威胁。病毒的生命周期是依赖宿主细胞的相关机制和途径进行的,从表观遗传学角度研究病毒活动过程对促进抗病毒机制的研究具有重要意义。人们最先在腺病毒(Ad)和甲型流感病毒(IAV)的mRNA上检测到m6A[8,9]。随后,在单纯疱疹病毒1型(HSV-1)、劳斯氏肉瘤病毒(RSV)、猴空泡病毒40 (SV40)、B77肉瘤病毒、禽流感病毒和猫白血病病毒等病毒RNA中也检测到了m6A[10]。研究发现m6A甲基化表观遗传修饰在病毒的侵染复制过程中具有重要作用。本文对m6A修饰的相关概念、m6A在病毒复制中的作用及对免疫反应的影响等方面进行了阐述,以期更深入地了解m6A修饰在病毒感染中的作用,为抗病毒药物的研发提供参考。
1 m6A修饰的分子机制
m6A是腺苷酸(A)在甲基转移酶复合物的催化下,第6位的N发生甲基化的修饰方式。RNA的m6A修饰是一个可逆的动态过程,此过程由甲基转移酶复合物、去甲基化酶和读取蛋白共同完成(图1)。
1.1 m6A甲基转移酶复合物
甲基转移酶复合物(Writers)由多种甲基转移酶(如METTL3和METTL14)和相关蛋白质亚基(如WTAP)组成。甲基转移酶样3 (METTL3)有催化活性,但METTL3需要和甲基转移酶样14 (METTL14)按1∶1的比例结合,形成异源二聚体,并在肾母细胞瘤1相关蛋白(WTAP)的引导下到达RNA上需要被修饰的位点,由METTL3将S-腺苷甲硫氨酸(SAM)上的甲基在转移到腺苷酸(A)上第6位的N上[11]。近年来又陆续发现并鉴定了KIAA1429[12]、RBM15、HAKAI[13]和METTL16[14]等m6A甲基转移酶复合物的新组分。
1.2 m6A去甲基化酶
m6A去甲基化酶(Erasers)负责去除RNA上的甲基化基团。迄今为止,在哺乳动物中只发现了两种m6A去甲基酶:肥胖相关蛋白(FTO)和ALKB同源蛋白5(ALKBH5),它们都属于Fe2+/α-酮戊二酸依赖的非血红素双加氧酶AlkB蛋白家族[15,16]。FTO首先氧化m6A生成氧化中间体6-羟甲基腺苷(hm6A),再进一步氧化生成6-甲酰腺苷(f6A),最终在其他酶的作用下,还原成腺苷。相较于FTO,ALKBH5直接将m6A还原成腺苷,不产生中间产物[17]。近期有研究表明,FTO更多的是在另一种甲基化修饰——6,2¢-O二甲基腺苷(m6Am)的去甲基化过程中行使功能[18]。
1.3 m6A读取蛋白
m6A的功能是通过RNA结合蛋白介导的,此类蛋白被称为m6A“阅读器”(Readers),Readers可以选择性地结合到含有m6A的RNA上。m6A读取蛋白通过YT521-B同源域(YTH)直接与m6A结合,从而识别发生m6A修饰的RNA[19]。人类中已发现5种含YTH域蛋白,包括两种亚型——YTHDFs和YTHDCs,其中,YTHDFs包括YTHDF1、YTHDF2和YTHDF3[20],YTHDCs包括YTHDC1和YTHDC2[21,22]。YTHDF2是第一个被鉴定的具有生物学功能的YTH家族蛋白[23,24],它选择性结合m6A修饰的mRNA,然后通过招募CCR4-NOT去腺苷酸酶复合物的CNOT亚基,促进mRNA的去腺苷酸化和降解[25]。YTHDF2主要存在于细胞质中,但在热休克压力下,YTHDF2会转移到细胞核中以保护5′非翻译区的m6A免受FTO去甲基化作用的影响,并促进mRNA的非帽依赖性翻译[26]。YTHDC1主要存在于细胞核中,能够通过与多种剪接因子的互作,调控mRNA的剪接[27,28]。YTHDC2在提高其靶mRNA的翻译效率和降低mRNA丰度方面具有双重作用[29]。
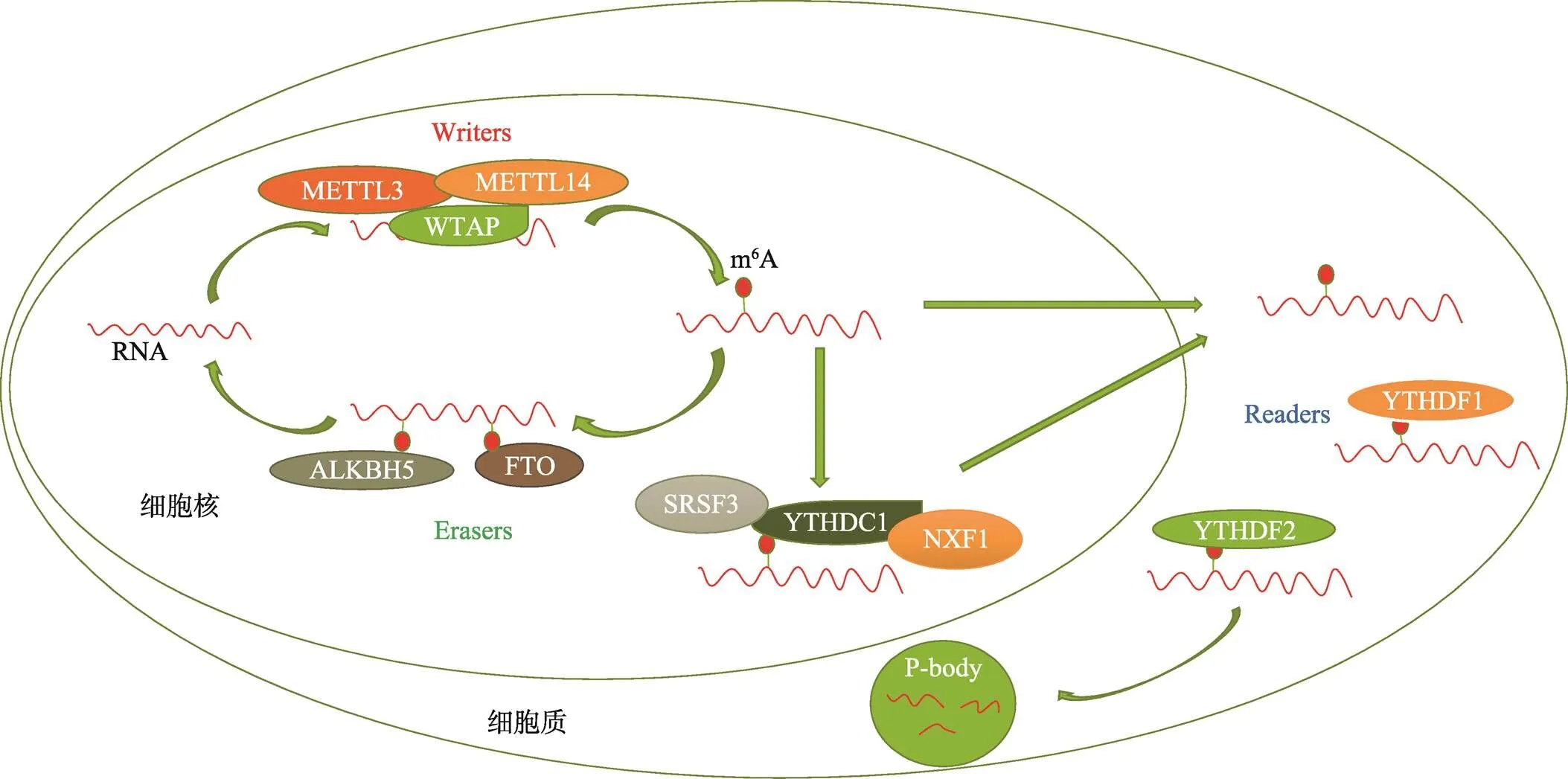
图1 m6A修饰的分子机制
METTL3、METTL14和WTAP组成甲基转移酶复合物(Writers)。ALKBH5和FTO是常见的去甲基化酶(Erasers)。YTHDF1、YTHDF2和YTHDC1是常见的读取蛋白(Readers)。SRSF3是剪接因子。NXF1是核RNA输出因子。P-body是RNA降解的场所。核内RNA在甲基转移酶复合物的作用下,形成m6A;而核内RNA上的m6A也可在去甲基化酶的作用下发生去甲基化。随后,在核内RNA的进一步加工过程中,核内的读取蛋白会与m6A位点结合;而当成熟的RNA出核后,细胞质内的读取蛋白会与RNA上的m6A位点结合。不同的读取蛋白结合到m6A位点后执行不同的功能,如YTHDC1能够识别RNA上的m6A位点,同时与SRSF3及NXF1相互作用,从而促进RNA的核输出;YTHDF1能够促进mRNA的翻译;YTHDF2识别RNA上的m6A位点后,能够引导RNA至P-body,从而促进RNA的降解。
2 m6A修饰与病毒复制
目前有关m6A修饰对病毒复制的调控及其机制的研究相对较少,m6A修饰作为一种表观遗传修饰,对不同病毒的复制具有完全相反的调控作用(表1)。
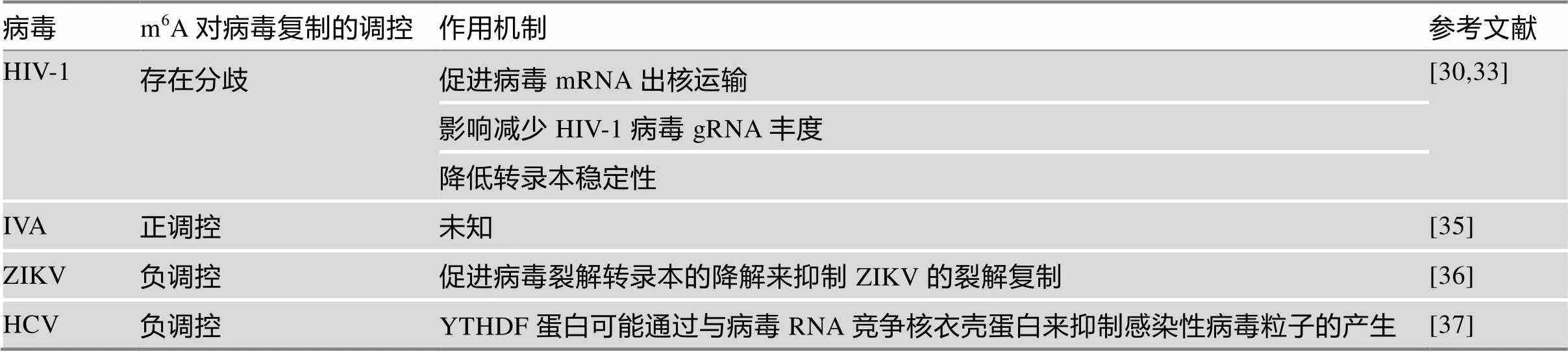
表1 m6A对病毒复制的调控及其机制
续表

病毒m6A对病毒复制的调控作用机制参考文献 HBV未知HBV转录本pgRNA 5¢端茎环上的m6A修饰促进pgRNA的逆转录[38] HBV转录本pgRNA 3¢端茎环上的m6A修饰降低HBV RNA的稳定性 KSHV存在分歧调节ORF50 pre-RNA的剪接[39,40] 影响病毒转录本的稳定性 SV40正调控促进晚期转录本的翻译[42] AMV负调控引发病毒mRNA的沉默和衰减来抑制病毒的复制[43]
2.1 m6A修饰对RNA病毒复制的调控
2.1.1 m6A修饰对人类免疫缺陷病毒Ⅰ型病毒(HIV-1)复制的调控
研究发现,HIV-1的感染会引起宿主和病毒mRNA的m6A丰度升高。Lichinchi等[30]通过shRNA介导METTL3、METTL14和ALKBH5的沉默,然后对HIV-1包膜糖蛋白GP120的RNA水平进行定量分析,并对感染后72 h的病毒衣壳蛋白p24进行免疫印迹,发现METTL3和METTL14的沉默,使GP120和p24表达水平降低,ALKBH5沉默使GP120和p24表达水平显著增加,表明m6A的修饰丰度与GP120和p24表达水平呈正相关。此外,HIV-1 Rev响应元件(RRE) RNA茎环Ⅱ区域的保守腺苷(A7883)发生m6A修饰,增加了HIV-1 Rev蛋白与RRE的结合,促进了RNA的出核运输,从而增强了HIV-1的复制。Kennedy等[31]发现YTHDF蛋白尤其是YTHDF2蛋白的过表达显著增强了293T细胞感染HIV-1病毒24和48 h后Nef、Tat和Rev的mRNA以及病毒基因组RNA (gRNA)的表达。Tirumuru等[32]观察到HeLa细胞中m6A识别蛋白YTHDF1-3的过表达使HIV-1感染被抑制,而在YTHDF1-3沉默后,增加了HIV-1的感染,尤其是YTHDF2,其过表达对HIV-1感染的抑制效果与叠氮胸苷(AZT)对HIV-1的抑制效果相近,推测YTHDF1-3可能通过抑制HIV-1的逆转录活性来抑制HIV-1的感染。Lu等[33]对m6A识别蛋白YTHDF1-3抑制HIV-1的逆转录进而抑制HIV-1的感染的机制进行了深入研究,发现YTHDF1-3降低了病毒gRNA的水平并抑制了早期和晚期的逆转录产物的产生,推测YTHDF1-3可能是通过影响病毒gRNA和逆转录产物的稳定性实现对HIV-1感染的调控。
2.1.2 m6A修饰对甲型流感病毒(IAV)复制的调控
IAV含有一个分段的、负性的单链RNA基因组,在其mRNA上发现了大约24个m6A修饰位点,其中8个位点集中在血凝素(HA) mRNA片段上[34]。Courtney等[35]发现,人肺上皮细胞系A549中关键的m6A甲基转移酶METTL3的突变能够抑制IAV复制,相反地,过表达m6A读取蛋白YTHDF2能够促进IAV复制和感染性病毒颗粒的产生。通过检测HA的mRNA和vRNA上的m6A位点并将其mRNA和vRNA上的甲基化位点突变,作者发现HA基因表达水平和IAV致病性降低。尽管实验结果表明m6A修饰能够增强IAV的复制,但具体的作用机制还有待进一步研究。
2.1.3 m6A修饰对寨卡病毒(ZIKV)复制的调控
ZIKV的RNA中含有丰富的m6A修饰位点,其复制受宿主甲基转移酶METTL3和METTL14以及去甲基化酶ALKBH5和FTO的调控,当METTL3和METTL14的表达量降低时,ZIKV的复制增加,而将ALKBH5和FTO沉默,会减少ZIKV的复制[36]。研究人员在分析读取蛋白YTHDF1-3对ZIKV复制的影响时发现,相较于YTHDF1和YTHDF3,YTHDF2的沉默引起ZIKV复制增加的程度最大;同样,YTHDF2的过表达降低ZIKV RNA的表达水平的效果最显著,进一步研究发现YTHDF2可能通过促进病毒裂解转录本的降解来抑制ZIKV的裂解复制,这表明m6A修饰在ZIKV病毒的复制过程中起负调控作用(图2)。

图2 m6A修饰在ZIKV裂解增殖中的作用
A:正常情况下,发生m6A修饰的裂解转录本会被YTHDF2识别并降解,产生的病毒粒子较少;B:在YTHDF2沉默的情况下,发生m6A修饰的裂解转录本的降解减少,产生的病毒粒子增多。gRNA是ZIKV的基因组RNA。
2.1.4 m6A修饰对丙型肝炎病毒(HCV)复制的调控
Gokhale等[37]在研究m6A修饰对HCV复制的影响时发现,METTL3和METTL14的表达量的变化对HCV RNA的复制和翻译没有影响,但能调控感染性HCV病毒粒子的产生及释放,并且这种调控作用是与m6A修饰丰度是负相关的。此外,在HCV感染过程中,YTHDF蛋白重新定位到脂滴——病毒装配位点,调节病毒粒子的产生,而YTHDF的沉默增加了感染性病毒颗粒的产生。HCV病毒E1基因上m6A修饰位点的突变,导致病毒RNA与核衣壳蛋白(或称核心蛋白)结合增加,而与YTHDF蛋白的结合减少,进而导致感染性病毒颗粒的增加,因此,Gokhale等[37]推测YTHDF蛋白可能通过与病毒RNA竞争核衣壳蛋白来抑制感染性病毒粒子的产生。
2.2 m6A修饰对DNA病毒复制的调控
2.2.1 m6A修饰对乙型肝炎病毒(HBV)复制的调控
与HCV不同,HBV是一种DNA病毒,通过一种称为前基因组RNA (pgRNA)的中间RNA完成其生命周期。Imam等[38]研究发现,pgRNA 5′端ε茎环和HBV转录本3′端都存在m6A修饰。METTL3和METTL14的沉默导致HBc和HBs两种病毒蛋白的表达增加,而ALKBH5和FTO的沉默降低了HBV病毒蛋白的表达。同样地,YTHDF2或YTHDF3的沉默显著延长了HBV转录本的半衰期,并增加了HBV蛋白HBs和HBc的表达,表明YTHDF蛋白也负向调控HBV蛋白的表达。在HBV转录本中,pgRNA 5′端茎环上的m6A修饰对pgRNA的逆转录起着正向调控的作用,而HBV 转录本3′端茎环上的m6A修饰对HBV RNA的稳定性进行负向调控,这表明m6A修饰位点的差异性可引起后续调控功能的差异。
2.2.2 m6A修饰对卡波氏肉瘤相关疱疹病毒(KSHV)复制的调控
KSHV在宿主细胞中建立潜伏感染仅需表达几个潜伏基因。当潜伏感染的细胞被重新激活时,进行裂解复制,表达大多数病毒基因并产生病毒粒子[39]。m6A修饰的阻断抑制了编码关键KSHV裂解开关蛋白复制转录激活因子(RTA)的pre-mRNA的剪接,阻断了病毒的裂解复制。RTA诱导m6A并增强其自身的pre-mRNA剪接。Ye等[39]的实验结果不仅证明了m6A在调控RTA pre-mRNA剪接中的重要作用,而且表明KSHV已经进化出一种通过操纵宿主m6A修饰,来使其在促进裂解复制方面发挥优势的机制。Tan等[40]过表达YTHDF3,发现病毒转录本的水平下降但相关病毒蛋白的水平没有显著变化。KiSLK细胞中YTHDF2沉默导致病毒粒子的产生增加到了原来的4倍,同时病毒mRNA ORF50、ORF57、ORFK8和ORF65的表达量上升了2~6倍进而导致蛋白质水平的升高,而YTHDF2过表达能够扭转这些影响,这表明YTHDF2可能通过促进病毒裂解转录本的降解来抑制KSHV的裂解复制,而这可能是细胞抑制病毒复制的一种防御机制。Hesser等[41]报道,在感染致癌的人类DNA病毒卡波西肉瘤相关疱疹病毒(KSHV)的细胞中,m6A水平显著升高,但发现m6A修饰机制的沉默对不同细胞中病毒基因的表达有不同的影响。在iSLK.219和iSLK.BAC16细胞中,METTL3和YTHDF2的沉默使病毒裂解转激活因子ORF50发生转录后积累,进而显著减少了病毒粒子的产生,这表明m6A起着促进病毒产生的作用。相比之下,在KSHV感染的B细胞中,METTL3或YTHDF2缺失时,ORF50蛋白表达增加,进而增加了病毒粒子的产生。虽然结果存在差异,但却都表明m6A修饰通过调控ORF50来调控KSHV的裂解复制。
2.2.3 m6A修饰对猴空泡病毒40 (SV40)复制的调控
SV40是一种DNA病毒,属于多瘤病毒家族。Tsai等[42]研究发现,过表达m6A读取蛋白质YTHDF2能够诱发更快速的病毒复制,而YTHDF2基因的突变失活或METTL3的沉默则会抑制病毒的复制。利用同义突变,使SV40晚期mRNA上的大多数m6A修饰位点发生突变,可以观察到突变型SV40比野生型SV40复制速度减慢,相较于野生型病毒,突变型病毒的转录本水平并没有显著变化,而VP1蛋白水平显著下降,因而Tsai等[42]推测m6A修饰主要通过促进晚期转录本的翻译来增强病毒基因表达,进而促进SV40病毒的复制。
2.3 m6A修饰对植物病毒复制的调控
植物病毒对于植物的正常生长与发育构成重大威胁。m6A表观修饰不仅发生在影响动物的病毒中,在植物病毒中也有报道。Martinez-Perez等[43]在拟南芥多功能紫花苜蓿花叶病毒(AMV)与衣壳蛋白(CP)相互作用的酵母双杂交筛选中,鉴定出了去甲基化酶AlkB家族成员atALKBH9B。在雀麦花叶病毒科的两个成员,AMV和CMV(黄瓜花叶病毒)的基因组中也发现了m6A的存在,并发现atALKBH9B的突变导致AMV基因组中m6A水平上升了35%,而超甲基化降低了AMV的感染效率。进一步的研究发现atALKBH9B与小干扰RNA(siRNA)的组成部分SGS3和处理小体(P-body)中的一种降解酶DCP1完全重叠,这表明m6A修饰可能通过引发病毒mRNA的沉默和衰减来负向调节AMV的复制。
3 m6A修饰对免疫反应的影响
免疫系统是高等生物抵御病毒侵染的重要屏障。目前,对于m6A在免疫系统中的作用及其在宿主病原体相互作用中的作用知之甚少。先天免疫提供对病毒感染的第一反应,入侵的病原体核酸被细胞质甲酸诱导基因Ⅰ (RIG-Ⅰ)样受体和膜结合的Toll样受体(TLRs)识别并结合,从而激活先天免疫反应。先天免疫必须区分宿主和病原体的核酸,以便在不激活自身免疫反应的情况下产生保护性免疫反应。当修饰过的核苷酸存在于RNA分子中时,会降低先天免疫反应的幅度,如含有m6A修饰的病毒RNA与RIG-Ⅰ结合较差,因而无法触发强烈的先天免疫反应[44]。Karikó等[45]的研究表明,经过m6A修饰的RNA刺激DC细胞只能引起少量的细胞因子的释放,同样地,当甲基化的RNA刺激表达TLRs的细胞时,也只引起少量免疫激活标志物的释放,而无法触发强烈的先天免疫反应。Lichinchi等[30]用HIV-1 (LAI病毒株)感染CD4+T细胞,发现细胞中总RNA中m6A的水平上升了约30%,这表明HIV-1感染T细胞可促进病毒和宿主RNA的甲基化,从而抑制免疫监视或扰乱宿主遗传网络,从而成功复制[46]。此外,ZIKV在潜伏期时,通过提高自身m6A水平来逃避宿主细胞的免疫监视,在裂解复制期间,ZIKV可动态修改宿主免疫相关转录本,通过篡夺m6A修饰机制来抑制宿主的抗病毒反应[10]。在IAV感染过程中过表达YTHDF2可提高感染性病毒粒子的释放,而YTHDF2介导的mRNA降解可能降低宿主抗病毒基因转录本,从而增强病毒复制[35]。总之,病毒通过借助宿主的m6A修饰机制,来逃避免疫系统的监察或抑制免疫系统的功能等,使病毒自身能够存活。
4 结语与展望
随着对m6A修饰机制研究的逐渐深入,发现其对RNA水平的调控作用变得更加复杂和多样。m6A修饰的调控功能通过读取蛋白来执行,一方面,读取蛋白能够促进经m6A修饰的mRNA的翻译,另一方面,也会通过降低靶mRNA的稳定性而促进其降解,有关这两种调控功能的选择的机制还有待研究。关于m6A修饰在病毒复制过程中的调控机制的研究表明,m6A修饰对病毒复制的调控是通过影响病毒mRNA或基因组RNA的稳定性来实现的,众多研究都证实了读取蛋白YTHDF2对病毒复制的影响,实验结果也表明YTHDF2的过表达能抑制多种病毒的复制。另外,也有实验表明YTHDF2能够促进病毒的复制。这些研究结果提示可以将靶向调控m6A修饰的药物作为抗病毒药物的开发方向,如3-脱氮腺苷(3-deazaadenosine, DAA)已经被证明可以通过阻断的S-腺苷高半胱氨酸(SAC)的水解,使S-腺苷甲硫氨酸(SAM)降解,从而阻断mRNA底物中m6A的加入[10]。Kennedy等[31]用DAA处理HIV-1感染的CEM-SS细胞,发现HIV-1的复制受到抑制,证明DAA对HIV-1复制的有效抑制作用。同时,有报道称DAA可以抑制RSV、IAV等病毒的复制[47,48]。在研究m6A修饰对病毒复制的调控时,也应对m6A修饰的动态变化对宿主细胞的影响给予高度关注,这或许可以为新型抗病毒药物的开发提供参考。此外,病毒RNA的高甲基化使部分病毒成功躲过免疫系统的监察,那么生物体内是否存在某些机制能够监测到这些异常的甲基化,进而反馈给免疫系统进行处理,这有待进一步的研究。
[1] Desrosiers R, Friderici K, Rottman F. Identification of methylated nucleosides in messenger RNA from Novikoff hepatoma cells., 1974, 71(10): 3971–3975.
[2] Perry RP, Kelley DE. Existence of methylated messenger RNA in mouse L cells., 1974, 1(1): 37–42.
[3] Ye F, Dan D, Gideon R, Chuan H. Gene expression regulation mediated through reversible m6A RNA methylation., 2014, 15(5): 293–306.
[4] LI YL, YU J, Song SH. Recent progresses in RNA N6-methyladenosine research., 2013, 35(12): 1340–1351.李语丽, 于军, 宋述慧. RNA 中6-甲基腺嘌呤的研究进展. 遗传, 2013, 35(12): 1340–1351.
[5] Zhao BS, Roundtree IA, He C. Post-transcriptional gene regulation by mRNA modifications., 2016, 18(1): 31–42.
[6] Roignant JY, Soller M. M6A in mRNA: an ancient mechanism for Fine-Tuning gene expression., 2017, 33(6): 380–390.
[7] Wu R, Jiang D, Wang Y, Wang X. N6-methyladenosine (m6A) methylation in mRNA with a dynamic and reversible epigenetic modification., 2016, 58(7): 450–459.
[8] Krug RM, Morgan MA, Shatkin AJ. Influenza viral mRNA contains internal N6-methyladenosine and 5'-terminal 7-methylguanosine in cap structures., 1976, 20(1): 45–53.
[9] Moss B, Koczot F. Sequence of methylated nucleotides at the 5'-terminus of adenovirus-specific RNA., 1976, 17(2): 385–392.
[10] Tan B, Gao SJ. RNA epitranscriptomics: regulation of infection of RNA and DNA viruses by N6-methyladenosine (m6A)., 2018, 29(1): 125–131.
[11] Zhang X, Jia GF. RNA epigenetic modification: N6-methyladenosine., 2016, 38(4): 275–288.张笑, 贾桂芳. RNA表观遗传修饰: N6-甲基腺嘌呤. 遗传, 2016, 38(4): 275–288.
[12] Schwartz S, Mumbach MR, Jovanovic M, Wang T, Maciag K, Bushkin GG, Mertins P, Ter-ovanesyan D, Habib N, Cacchiarelli D, Sanjana NE, Freinkman E, Pacold ME, Satija R, Mikkelsen TS, Hacohen N, Zhang F, Carr SA, Lander ES, Regev A. Perturbation of m6A writers reveals two distinct classes of mRNA methylation at internal and 5' sites., 2014, 8(1): 284–296.
[13] Horiuchi K, Kawamura T, Iwanari H, Ohashi R, Naito M, Kodama T, Hamakubo T. Identification of Wilms' tumor 1-associating protein complex and its role in alternative splicing and the cell cycle.2013, 288(46): 33292–33302.
[14] Pendleton KE, Chen B, Liu K, Hunter OV, Xie Y, Tu BP, Conrad NK. The U6snRNA m6A methyltransferase METTL16 regulates SAM synthetase intron retention., 2017, 169(5): 824–835.e14.
[15] Jia G, Fu Y, Zhao X, Dai Q, Zheng G, Yang Y, Yi C, Lindahl T, Pan T, Yang YG, He C. N6-methyladenosine in nuclear RNA is a major substrate of the obesity-associated FTO., 2011, 7(12): 885–887.
[16] Zheng G, Dahl JA, Niu Y, Fedorcsak P, Huang CM, Li CJ, Vågbø CB, Shi Y, Wang WL, Song SH, Lu Z, Bosmans RP, Dai Q, Hao YJ, Yang X, Zhao WM, Tong WM, Wang XJ, Bogdan F, Furu K, Fu Y, Jia G, Zhao X, Liu J, Krokan HE, Klungland A, Yang YG, He C. ALKBH5 is a mammalian RNA demethylase that impacts RNA metabolism and mouse fertility., 2012, 49(1): 18–29.
[17] Aik WS, Scotti JS, Choi H, Gong L, Demetriades M, Schofield CJ, Mcdonough MA. Structure of human RNA N6-methyladenine demethylase ALKBH5 provides insights into its mechanisms of nucleic acid recognition and demethylation., 2014, 42(7): 4741–4754.
[18] Mauer J, Luo X, Blanjoie A, Jiao X, Grozhik AV, Patil DP, Linder B, Pickering BF, Vasseur JJ, Chen Q, Gross SS, Elemento O, Debart F, Kiledjian M, Jaffrey SR. Reversible methylation of m6Am in the 5' cap controls mRNA stability., 2016, 541(7637): 371–375.
[19] Patil DP, Pickering BF, Jaffrey SR. Reading m6A in the transcriptome: m6A-binding proteins., 2017, 28(2): 113–127.
[20] Xiao W, Lu Z, Gomez A, Hon GC, Yue Y, Han D, Ye F, Parisien M, Dai Q, Jia G. m6A-dependent regulation of messenger RNA stability., 2014, 505(7481): 117–120.
[21] Meyer KD, Jaffrey SR. Rethinking m(6)A Readers, Writers, and Erasers., 2017, 33(1): 319–342.
[22] Patil DP, Chen CK, Pickering BF, Chow A, Jackson C, Guttman M, Jaffrey SR. m6A RNA methylation promotes XIST-mediated transcriptional repression., 2016, 537(7620): 369–373.
[23] Wang X, Lu Z, Gomez A, Hon GC, Yue Y, Han D, Fu Y, Parisien M, Dai Q, Jia G, Ren B, Pan T, He C. N6-methyladenosine-dependent regulation of messenger RNA stability., 2013, 505(7481): 117–120.
[24] Wang Y, Li Y, Toth JI, Petroski MD, Zhang Z, Zhao JC. N6-methyladenosine dification destabilizes developmental regulators in embryonic stem cells., 2014, 16(2): 191–198.
[25] Du H, Zhao Y, He J, Zhang Y, Xi H, Liu M, Ma J, Wu L. YTHDF2 destabilizes m6A-containing RNA through direct recruitment of the CCR4–NOT deadenylase complex., 2016, 7: 12626.
[26] Zhou J, Wang J, Gao X, Zhang X, Jaffrey SR, Qian SB. Dynamic m(6)A mRNA methylation directs translational control of heat shock response., 2015, 526(7574): 591–594.
[27] Xiao W, Adhikari S, Dahal U, Chen YS, Hao YJ, Sun BF, Sun HY, Li A, Ping XL, Lai WY, Wang X, Ma HL, Huang CM, Yang Y, Huang N, Jiang GB, Wang HL, Zhou Q, Wang XJ, Zhao YL, Yang YG. Nuclear m(6)A reader YTHDC1 regulates mRNA splicing., 2016, 61(4): 507–519.
[28] Ying Y, Chen YS, Sun BF, Yang YG. RNA methylation: regulations and mechanisms., 2018, 40(11): 964–976.杨莹, 陈宇晟, 孙宝发, 杨运桂. RNA甲基化修饰调控和规律. 遗传, 2018, 40(11): 964–976.
[29] Duan HC, Wang Y, Jia G. Dynamic and reversible RNA N6-methyladenosine methylation., 2018, 10(1): e1507.
[30] Lichinchi G, Gao S, Saletore Y, Gonzalez GM, Bansal V, Wang Y, Mason CE, Rana TM. Dynamics of the human and viral m(6)A RNA methylomes during HIV-1 infection of T cells., 2016, 1(4): 16011.
[31] Kennedy E, Bogerd H, Kornepati AR, Kang D, Ghoshal D, Marshall JB, Poling BC, Tsai K, Gokhale N, Horner SM. Posttranscriptional m6A editing of HIV-1 mRNAs enhances viral gene expression., 2017, 22(6): 830.
[32] Tirumuru N, Zhao BS, Lu W, Lu Z, He C, Wu L. Correction: N(6)-methyladenosine of HIV-1 RNA regulates viral infection and HIV-1 Gag protein expression., 2016, 5:e15528.
[33] Lu W, Tirumuru N, St Gelais C, Koneru PC, Liu C, Kvaratskhelia M, He C, Wu L. N6-methyladenosine- binding proteins suppress HIV-1 infectivity and viral production., 2018, 293(34): 12992–13005.
[34] Narayan P, Ayers DF, Rottman FM, Maroney PA, Nilsen TW. Unequal distribution of N6-methyladenosine in influenza virus mRNAs., 1987, 7(4): 1572–1575.
[35] Courtney DG, Kennedy EM, Dumm RE, Bogerd HP, Tsai K, Heaton NS, Cullen BR. Epitranscriptomic enhancement of influenza A virus gene expression and replication., 2017, 22(3): 377–386.e5.
[36] Lichinchi G, Zhao BS, Wu Y, Lu Z, Qin Y, He C, Rana T. Dynamics of human and viral RNA methylation during Zika virus infection., 2016, 20(5): 666–673.
[37] Gokhale N, Mcintyre ARR, Mcfadden M, Roder A, Kennedy E, Gandara J, Hopcraft S, Quicke K, Vazquez C, Willer J. N6-methyladenosine in flaviviridae viral RNA genomes regulates infection., 2016, 20(5): 654–665.
[38] Imam H, Khan M, Gokhale NS, McIntyre ABR, Kim G-W, Jang JY, Kim S-J, Mason CE, Horner SM, Siddiqui A. N6-methyladenosine modification of hepatitis B virus RNA differentially regulates the viral life cycle., 2018, 115(35): 8829–8834.
[39] Ye F, Lei X, Gao SJ. Mechanisms of Kaposi's Sarcoma- Associated herpesvirus latency and reactivation., 2011, 2011(1687–8639).
[40] Tan B, Hui L, Zhang S, Silva S RD, Lin Z, Jia M, Cui X, Yuan H, Sorel O, Zhang SW. Viral and cellular N 6 -methyladenosine and N6,2′-O-dimethyladenosine epitranscriptomes in the KSHV life cycle., 2017, 3(1): 108–120.
[41] Hesser CR, Karijolich J, Dominissini D, He C, Glaunsinger BA. N6-methyladenosine modification and the YTHDF2 reader protein play cell type specific roles in lytic viral gene expression during Kaposi's sarcoma- associated herpesvirus infection., 2018, 14(4): e1006995.
[42] Tsai K, Courtney DG, Cullen BR. Addition of m6A to SV40 late mRNAs enhances viral structural gene expression and replication., 2018, 14(2): e1006919.
[43] Martínez-Pérez M, Aparicio F, López-Gresa MP, Bellés JM, Sánchez-Navarro JA, Pallás V. Arabidopsis m6A demethylase activity modulates viral infection of a plant virus and the m6A abundance in its genomic RNAs., 2017, 114(40): 10755–10760.
[44] Durbin AF, Wang C, Marcotrigiano J, Gehrke L. RNAs containing modified nucleotides fail to trigger RIG-I conformational changes for innate immune signaling., 2016, 7(5): e00833–16.
[45] Karikó K, Buckstein M, NiH, Weissman D. Suppression of RNA recognition by Toll-like receptors: the Impact of nucleoside modification and the evolutionary origin of RNA., 2005, 23(2): 165–175.
[46] Kennedy EM, Courtney DG, Tsai K, Cullen BR. Viral Epitranscriptomics., 2017, 91(9): JVI.02263–16.
[47] Bader JP, Brown NR, Chiang PK, Cantoni GL. 3-Deazaadenosine, an inhibitor of adenosylhomocysteine hydrolase, inhibits reproduction of Rous sarcoma virus and transformation of chick embryo cells., 1978, 89(2): 494–505.
[48] Chiang PK. Biological effects of inhibitors of S-adenosylhomocysteine hydrolase., 1998, 77(2): 115–134.
Advances in m6A modification and its regulation of viral replication
Peng Xue1, Tao Jiang1, Xingjia Shen1,2
6-methyladenosine (m6A) is a prevalent modification of RNA in eukaryotes and plays an important role in the process of mRNA translocation, stabilization and translation. m6A exerts different influences on the viral replication cycle, and both viral replication and host immune response to the virus are affected by m6A. In this review, we summarize recent studies on the mechanism of m6A modification and its effects on viral replication and host immune response, in order to provide a reference for epigenetic regulation in the viral life cycle.
6-methyladenosine; RNA modification; virus; replication; immunity
2019-02-20;
2019-04-08
国家自然科学基金项目(编号:31672490),江苏省自然科学基金项目(编号:BK20151322)和江苏省高校自然科学研究重大项目(编号:15KJA180001)资助[Supported by the National Natural Science Foundation of China (No. 31672490), the Natural Science Foundation of Jiangsu Province (No. BK20151322) and Major Project of Natural Science Foundation for Universities of Jiangsu Province (No. 15KJA180001]
薛鹏,硕士研究生,专业方向:基因表达调控。E-mail: xuepeng0603@163.com
沈兴家,研究员,博士生导师,研究方向:基因表达调控。E-mail: shenxjsri@163.com
10.16288/j.yczz.19-042
2019/4/16 17:17:28
URI: http://kns.cnki.net/kcms/detail/11.1913.R.20190416.1717.001.html
(责任编委: 岑山)

