小桐子低温胁迫下microRNA实时荧光定量PCR内参的筛选与比较
孔春艳 陈永坤 王莎莎 郝大海 杨宇 龚明
(云南师范大学生命科学学院 生物能源持续开发利用教育部工程研究中心 云南省生物质能与环境生物技术重点实验室,昆明 650500)
microRNA(miRNA)是一类大小为20-24 nt、具有调控功能的内源性非编码RNA。众多的miRNA参与了植物对低温的感受、响应、信号转导和适应过程[1-2]。在miRNA研究中,通常需要对miRNA在植物各组织和器官中的表达量进行分析,确定miRNA表达量对于探究miRNA的生物学功能尤其重要[3-4]。实时荧光定量PCR(quantitative real-time PCR,qRT-PCR)技术具有高灵敏性、特异性、快速简单、低样本浓度需求等优点,是研究基因表达的首选方法[1-2,5]。然而,qRT-PCR结果中基因表达量的精确和准确性分析依赖于所选择的内参基因[6]。在qRT-PCR中,最常用的植物内参基因包括肌动蛋白(ACT)、甘油醛3-磷酸脱氢酶(GAPDH)、18S核糖体RNA(18S)和F-BOX家族蛋白(FP),但在miRNA定量检测中并不选择这些内参基因[7-8]。研究表明当选用ACT作为miRNA研究的内参时,由于目的miRNA与ACT的大小有较大的差异,在校正miRNA数据时并不理想[9]。不经筛选直接使用常见内参基因会降低试验结果的准确性[10-12]。内参基因的稳定性差导致基因表达量检测的误差,如在研究杂交兰(Cymbidium hybrid)花器官不同部位中八氢番茄红素脱氢酶基因(ChPDS)的表达模式时,以杂交兰泛素蛋白基因(ChUBQ)、杂交兰延伸因子基因(ChEF-1α)、杂交兰微管蛋白基因(ChTUB)、杂交兰肌动蛋白基因(ChACT)及杂交兰泛素蛋白基因(ChUBQ)和杂交兰延伸因子基因(ChEF-1α)组合进行校正的杂交兰ChPDS表达模式均为花瓣>唇瓣>蕊柱,而以稳定性最差的杂交兰RNA聚合酶β亚基基因(ChrpoB)进行校正时,杂交兰ChPDS相对表达量为花瓣>蕊柱>唇瓣[13]。因此,针对实际情况筛选内参基因很有必要性[1-2,5]。
小桐子(Jatropha curcas)是一种有巨大综合开发潜力的多用途能源植物树种,原产热带及亚热带地区,抗寒性较弱,是一种喜温冷敏植物[14-16]。Maes等[17]进行的世界范围内的调查表明,小桐子生长适宜的年均温度为19.3-27.2℃,最低月均温度要高于10.5℃。而随着小桐子栽培的北移和向高海拔山区发展,低温冷害已成为限制小桐子产业的主要环境因素[15-17]。为研究miRNA在小桐子低温胁迫与适应过程中的调控作用,本课题组前期进行了小桐子小RNA-seq分析,初步获得了差异表达与稳定表达的miRNA信息[18],但要进行进一步qRTPCR验证,需要筛选合适的内参基因用于miRNA定量分析。但目前尚未见对小桐子miRNA qRT-PCR内参基因筛选的报道。
本研究基于小桐子小RNA-seq数据[18],筛选低温处理前后表达量相对稳定的10个miRNA以及在其他物种miRNA定量分析中常用的U6作为候选内参,采用qRT-PCR方法对候选内参基因在低温胁迫下的表达稳定性进行分析,以期筛选出可用于小桐子低温胁迫下miRNA差异表达分析的稳定内参基因,为小桐子miRNA表达的定量分析提供参考。
1 材料与方法
1.1 材料
1.1.1 实验材料 选取饱满的小桐子种子,参照李忠光等[19]方法,用1.5% CuSO4溶液消毒,在恒温培养箱中萌发5 d,将发芽的种子种在营养土中,在相对湿度75%、26/20℃、16/8 h光周期的培养箱中培养21 d,至第3片真叶展开[20]。
1.1.2 低温处理 在小桐子长至第3片真叶展开时,转入1℃光照培养箱中低温处理5 d,每天取样1次(LT1d-LT5d,取用第2片及以上展开真叶)。以未处理的小桐子作为对照1(C0d);另在正常培养条件下第26天取样作为对照2(C5d)。依据前期结果,在1℃处理5 d时,小桐子幼苗死亡率约70%[20-21]。
1.2 方法
1.2.1 样品miRNA提取及cDNA合成 参照Plant miRNA Kit(OMEGA公司)说明书提取miRNA,2%琼脂糖凝胶电泳检测miRNA的质量,NanoPhotometer-N60超微量分光光度仪(Implen,Germany)测定浓度,并通过OD260/OD280和OD260/OD230检测miRNA的纯度。采用miRcute miRNA First-Strand cDNA Synthesis Kit(Tiangen,北京)反转 录 cDNA。 反 应 体 系 为 Reaction Buffer 10 μL、Enzyme Mix 2 μL、miRNA 2 μL,加水至 20 μL。42℃孵育1 h,并在95℃加热3 min失活逆转录酶(RT enzyme mix),进行miRNA加A尾反应以及cDNA第一链合成。cDNA用NanoPhotometer-N60超微量分光光度仪(Implen,Germany)测定浓度,每份样本终浓度为100 ng/μL,取1 μL用于qRT-PCR。
1.2.2 qRT-PCR各供选基因引物设计 在11个候选内参基因中,U6snRNA序列来自于Jatropha curcas Database(JCDB)和NCBI;10个来自小桐子低温胁迫下小RNA-seq[18]中表达稳定的miRNA,按照其序列设计引物,反向引物为:5′-GCTGTCAACGATACGCTACGTAACG-3′。 利 用 DNAMAN 8(Lynnon,USA)进行引物互补检测,减少引物自身延伸或两两互补延伸形成二聚体的可能性(表1)。

表1 候选内参基因引物序列
1.2.3 qRT-PCR分析 根据TaKaRa公司的SYBR Premix Ex TaqTMⅡ(Tli RNase H PLUS)试剂盒说明书配置10 μL反应体系,cDNA模板1 μL(10 ng/μL)、正反向引物各 0.4 μL(10 μmol/L)、2×TransStar Tip Green qPCR SuperMix 5 μL 和 ddH2O 3.2 μL。所有操作均在冰上进行,3个重复,3次生物学重复。反应程序为 95℃ 30 s;95℃ 5 s,60℃ 30 s,45 个循环,并在65-97℃进行熔解曲线分析。利用LightCycler®96 System(Roche,Switzerland)进行qRT-PCR反应。
1.2.4 数据处理 记录qRT-PCR分析得出的基因转录水平Ct值,每个基因在不同处理样本中的Ct值测定3个重复。qRT-PCR熔解曲线利用LightCycler 96 SW 1.1(Roche,Switzerland)软件进行作图及分析;利用 geNorm(https://genorm.cmgg.be/)、NormFinder(https://www.moma.dk/normfinder-software/) 和BestKeeper(https://www.gene-quantification.de/bestkeeper.html)分析11个候选内参基因的表达稳定性;利用Microsoft Excel 2016进行数据作图。
2 结果
2.1 miRNA质量检测
通过2%琼脂糖凝胶电泳检测提取的miRNA样品(图1)。可见提取的miRNA质量较好;各样品中miRNA的OD260/OD280、OD260/OD230的值均大于1.8(表2),miRNA质量满足后续实验的需求。

图1 提取的miRNA2%的琼脂糖凝胶电泳图

表2 7个miRNA样品的纯度与浓度

图2 内参基因的qRT-PCR分析
2.2 引物扩增特异性分析
以miRNA cDNA第一链为模板进行qRT-PCR扩增,用2%琼脂糖凝胶电泳检测(图2)。11条内参基因条带单一,通过测序、比对等检测,确定扩增产物为引物特异性扩增产物,符合预期结果。

图3 十一个候选内参基因的qRT-PCR熔解曲线
经熔解曲线分析,11条候选内参基因的熔解曲线均呈现明显的单一峰(图3),不存在引物二聚体,并且每个样本的重复性较好,进一步说明引物的特异性,可进行后续实验分析。
2.3 候选内参基因的表达分析
11个候选内参基因在不同温度处理的小桐子叶片中,平均Ct值变化范围为10.53-26.03(图4),其中U6Ct值最小,miR2612Ct值最大(图5)。

图4 11个候选内参基因平均Ct值分布

图5 各候选基因在不同处理下叶片组织中Ct值分布
2.4 内参基因的表达稳定性分析
利用GeNorm软件对11个候选内参基因在不同处理下叶片组织中稳定性(M值)分析(图6),M 值 由 高 到 低 为miR5672、miR5040、miR5535、miR1446、miR3624、miR5791、miR2612、miR159、miR3630、U6、miR6448,所选候选内参基因的M值均小于1.5,表明候选基因表达都较稳定,M值最低的是miR6448;其次是U6。可见miR6448在低温胁迫下表达最稳定,其次是U6。
通过对不同处理下叶片组织中内参基因的NormFinder分析(表3),发现稳定值最小的是miR6448(0.088);其 次 是U6(0.134)。 这 与GeNorm分析得出的结果一致。
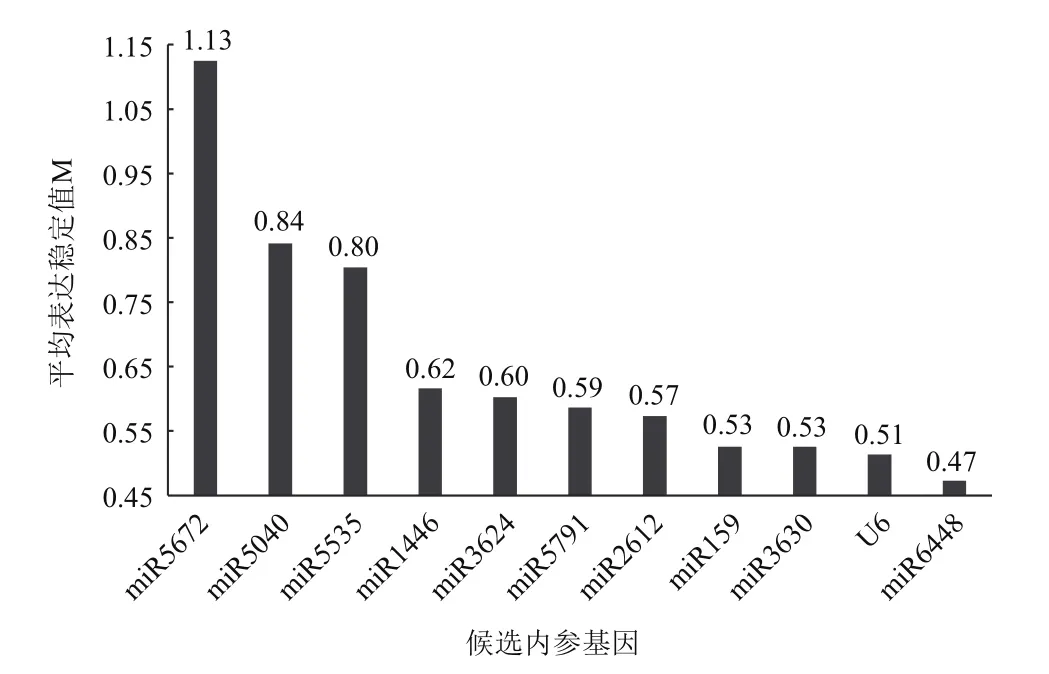
图6 不同处理下叶片组织中内参基因的GeNorm分析

表3 不同处理下叶片组织中内参基因的NormFinder分析
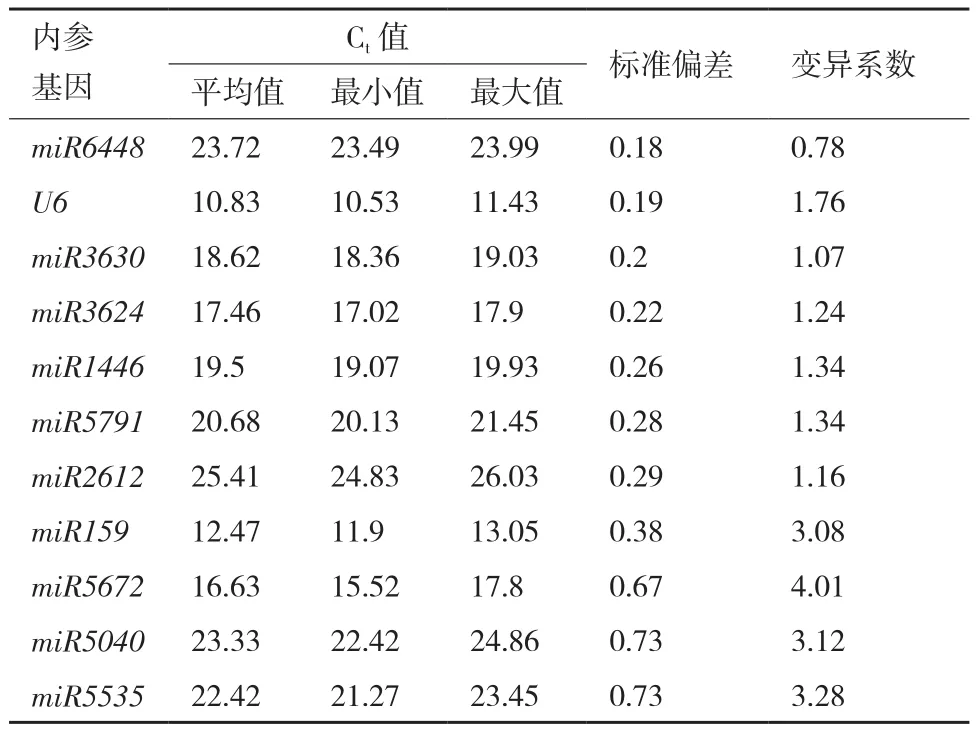
表4 不同处理下叶片组织中内参基因的BestKeeper分析
由BestKeeper分析标准偏差和变异系数,miRNA6448的标准偏差(0.18)和变异系数(0.78)都是最小的,表明miRNA6448在低温胁迫下的小桐子中表达最稳定。
3 讨论
进行qRT-PCR时,对内参基因的筛选非常有必要。内参基因在qRT-PCR中可以降低或校正基因定量过程中存在的误差,选择合适的内参基因对于基因定量表达的结果有重要意义[3,10]。而在选择内参基因时,除内参基因的稳定性,内参基因的表达丰度也会影响qRT-PCR结果[13,22-23]。内参基因与所检测基因之间的Ct值差异越小,扩增效率对计算结果的影响也会越小,计算的结果会更加准确[23]。如果目标基因和内参基因表达量(Ct值)过大,用内参基因来衡量目标基因的表达量就不完全可靠[10,23]。在本研究中,miR6448的 Ct值为 23左右,丰度适中,可以用于检测qRT-PCR试验中丰度较适中的转录本;U6的Ct值为10左右,可以用于miRNA qRT-PCR实验中丰度较高的转录本检测。
在进行miRNA研究时,提取的miRNA中往往弃除了mRNA等长链RNA,因此,相对链长较长的mRNA无法再用作miRNA定量研究的内参[24]。在qRT-PCR中,最常用的植物内参基因包括ACT、GAPDH、18S和F-BOX家族蛋白,通常在miRNA定量检测中并不选择这些内参基因[7-8]。常用于miRNA荧光定量的内参基因多为一些片段长度相对较短的基因,例如U6[3,25-26]。在大豆[27]、小麦[28]、龙眼[29]和百合[8]等植物 miRNA 的 qRT-PCR 分析中,miRNA的表达水平比最常用的ACT、GAPDH、F-BOX等蛋白质编码基因稳定得多。
在miRNA的qRT-PCR分析时,常常需要根据不同的植物材料和处理条件来选择合适的内参。茶树低温胁迫下,miRNA(PC-3p-222)在不同低温处理以及不同茶树组织中表达最为稳定,可作为茶树低温胁迫下miRNA的qRT-PCR的内参基因[30]。甘蔗芽在低温胁迫下,miR171/18S rRNA和miR171/miR5059表达最稳定[31]。盐胁迫下,U6适合作为大豆根组织miRNA研究的内参[32]。葡萄在盐胁迫和低温胁迫下,U6是表达最稳定的基因[1]。本研究发现,小桐子叶片组织对低温胁迫处理响应的miRNA中,miR6448和U6的表达表现稳定。可见miRNA内参基因除特异性外,像U6这样的基因也表现出一定的通用性。
4 结论
低温胁迫下表达最稳定的基因是miR6448和U6。miR6448适用于表达丰度适中的microRNA基因表达定量分析中;U6适用于丰度较高的microRNA的qRT-PCR分析。

