福建省常见尸食性蝇类的COⅠ及16S rDNA序列鉴定
卓荦,毛佳雄,陈建山,宋鹏,林澍,夏胜海,陈煌
(1.福建省公安厅刑事技术总队,福建 福州 350003;2.光泽县公安局,福建 光泽 354100)
死亡时间(postmortem interval,PMI)推断一直是法医学研究的重点和难点。近年来,建立在尸食性蝇类发育基础上的死亡时间推断是目前有效和精确的方法之一[1],利用该方法的关键在于蝇类的种属鉴定。传统的形态学种属鉴定需要专业且系统的昆虫分类学知识[2-3],绝大部分法医工作者很难进行准确鉴别。随着分子生物学技术的发展,DNA作为遗传信息的载体,具有种属特异性以及不受发育阶段影响的优势,应用尸食性昆虫种属特异性的遗传标记进行种属鉴定,将有效解决依赖形态学的鉴定难题。因此,分子鉴定作为形态学鉴定方法的补充手段受到了法医学者的广泛重视[4]。本研究拟通过基于线粒体细胞色素c氧化酶亚基Ⅰ(cytochrome c oxidase subunitⅠ,COⅠ)和16S核糖体脱氧核糖核酸(ribosomal deoxyribonucleic acid,rDNA)基因片段序列对福建省常见尸食性蝇类的种属进行分子鉴定,并探讨两种遗传标记对于蝇类种属鉴定的效力。
1 材料与方法
1.1 样本
收集福建省福州、宁德、莆田、泉州、厦门、漳州、南平、龙岩、三明9个地区自然环境下常见尸食性蝇类样本。选择室外自然环境下发生的非正常死亡案件现场,用捕虫网捕捉现场尸体表面停留的蝇类成虫,置于75%乙醇溶液中处死,固定48h后用蒸馏水漂洗,放入通风橱中通风干燥后保存。
1.2 方法
1.2.1 DNA提取
取样本胸肌组织置于1.5 mL微量离心管中研磨粉碎,加入20 mg/mL的蛋白酶K 10 μL,同时加入5% Chelex-100 150 μL,混匀后将其置于55℃水浴中消化过夜。消化完毕后,将离心管置于93℃下加热10 min,使混合液中的蛋白质变性,取出后立即冰浴10min,以9391×g离心8min,取上清液置于4℃冷藏保存备检。
1.2.2 PCR扩增
目前,针对尸食性蝇类线粒体COⅠ基因片段序列的引物设计扩增片段长度为272~1173bp[4]。过长的扩增序列对提取的DNA质量要求很高,测序结果需要拼接,容易产生错误,且成本较高,结合当前工作的实际情况,长片段序列不利于蝇类检验鉴定工作的推广实施。本研究引物设计参考蔡继峰[5]的研究报道,COⅠ扩增引物为:上游引物5′-CAGCTACTTTA TGAGCTTTAGG-3′,下游引物 5′-CATTTCAAGCTG TGTAAGCATC-3′,扩增片段长度278bp。16S rDNA扩增引物为:上游引物5′-CGCTGTTATCCCTAAGG TAA-3′,下游引物 5′-CTGGTATGAAAGGTTTGACG-3′,扩增片段长度289bp。
PCR 扩增体系为 30 μL,内含 10×PCR 缓冲液3 μL,dNTP混合物(2.5 mmol/L) 2 μL,上、下游引物(10μmol/L)各1μL,5U/μL的TaKaRa Ex Taq® DNA聚合酶0.2 μL,DNA模板2 μL,双蒸水补足。PCR循环参数:96℃预变性5 min;96℃变性20 s,52℃退火30 s,72℃延伸1 min,共10个循环;96℃变性20 s,52℃退火30 s,72℃延伸1 min,共35个循环;72℃延伸5min,于4℃保存。
1.2.3 电泳检测及测序分析
PCR扩增产物采用1%琼脂糖凝胶电泳(80 V,50 min)后,将凝胶板置于波长为254 nm的紫外灯下观察,利用凝胶成像系统拍照分析。扩增产物由北京六合华大基因科技有限公司测序。所得不同种属实验样本的正反向序列经拼接后,通过DNAMAN 6.0软件处理,截取278bp和289bp的相同长度核酸序列片段。所获得的样本COⅠ和16S rDNA的碱基序列片段,经BLAST(https://blast.ncbi.nlm.nih.gov/Blast.cgi)查询,与数据库中所有已知核酸序列进行序列同源性搜索和自动比对分析,比对完成后编辑样本序列并上传至GeneBank数据库注册。应用MEGA 10.0软件计算种内及种间遗传距离,选取模式为Kimura-2-parameter distance,并以非加权组平均法(unweighted pair-group method with arithmetic means,UPGMA)构建系统发育树,发育树节点自展值(bootstrap confidence level,BCL)选择Bootstrap方法进行检验,循环500次。
2 结 果
2.1 形态学鉴定结果
捕获的22个样本经形态学鉴定[6]共计3科5属6种:大头金蝇(Chrysomya megacephala)、绯颜裸金蝇(Achoetandrus rufifacies)、紫绿蝇(Lucilia porphyrina)、铜绿蝇(Lucilia cuprina)、棕尾别麻蝇(Sarcophaga peregrina)、家蝇(Musca domestica),具体种属分类见表1。
2.2 碱基位点分析
所采集样本16S rDNA基因片段序列的同源一致性达97.48%,其中:碱基A占39.50%~41.60%,平均值为40.98%;碱基C占12.20%~14.00%,平均值为13.30%;碱基G占6.30%~7.30%,平均值为6.84%;碱基T占38.10%~40.90%,平均值为38.86%。COⅠ基因片段序列的同源一致性达95.59%,其中:碱基A占28.50%~30.50%,平均值为29.11%;碱基C占15.30%~17.10%,平均值为15.77%;碱基G占14.70%~16.50%,平均值为15.83%;碱基T占37.70%~40.10%,平均值为39.30%。
2.3 序列种属同源性比对分析
所采集样本COⅠ和16S rDNA基因片段序列的同源性正确率分别达到90.90%和95.45%,其中COⅠ完全正确比对4个种属(20个样本),16S rDNA完全正确比对5个种属(21个样本)。种属正确比对样本的匹配一致性值(Max ident值)分别为:COⅠ在99.10%~99.71%,平均值为99.47%;16S rDNA在97.55%~100.00%,平均值为99.72%。紫绿蝇、铜绿蝇、绯颜裸金蝇、家蝇在两个基因片段序列的同源性比对正确率均达100.00%,种属比对情况见表2。
2.4 种内及种间遗传距离
所采集样本16S rDNA基因片段序列的种间遗传距离为1.8%~9.3%,种间遗传距离均数为1.8%~8.9%,种间最小遗传距离出现在铜绿蝇与紫绿蝇之间,种间最大遗传距离出现在棕尾别麻蝇和家蝇两个不同科之间。不同蝇种的种内遗传距离为0.0%~3.6%,种内遗传距离均数为0.0%~2.4%,其中种内遗传距离均数最大的为棕尾别麻蝇(2.4%),大头金蝇的种内遗传距离均数为0.6%,其余蝇种由于捕获数量不足,种内遗传距离范围未能客观反映。详见表3。

表1 所采集地区常见尸食性蝇类的生物学分类Tab.1 Biological classification of common necrophagous flies in the collected area

表2 COⅠ及16S rDNA基因片段序列在GeneBank数据库的比对情况Tab.2 Comparison of COⅠand 16S rDNA gene sequence fragments in GeneBank database

表3 各蝇种16S rDNA基因片段序列种间和种内的遗传距离Tab.3 The interspecific and intraspecific evolutionary divergence of 16S rDNA
所采集样本COⅠ基因片段序列的种间遗传距离为6.7%~14.0%,种间遗传距离均数为7.2%~13.6%,种间最小遗传距离出现在绯颜裸金蝇与大头金蝇之间,种间最大遗传距离出现在棕尾别麻蝇和家蝇以及棕尾别麻蝇和大头金蝇两个不同科之间。不同蝇种的种内遗传距离为0.0%~10.5%,种内遗传距离均数为0.0%~6.3%,其中种内遗传距离均数最大的为棕尾别麻蝇(6.3%),种内最大遗传距离出现在大头金蝇样本中,为10.5%,其余蝇种由于捕获数量不足,种内遗传距离范围未能客观反映。详见表4。
2.5 系统发育树的构建
应用MEGA 10.0软件对22个样本的基因片段序列进行分析,构建UPGMA系统发育树,按照不同科、属、种聚类,分子鉴定结果与形态学结果一致。由16S rDNA序列构建的系统发育树(图1)可见:各地区的大头金蝇聚集形成一个簇(BCL=95);铜绿蝇各样本形成一簇(BCL=100),并与紫绿蝇共同形成一簇,其共同的BCL值为92;绯颜裸金蝇、紫绿蝇、铜绿蝇以及大头金蝇4种蝇类以种属聚集成簇,其共同BCL值为81,构成了一个丽蝇科的簇;2只家蝇、3只棕尾别麻蝇分别聚集形成簇,其BCL值分别为100、94,与丽蝇科并列形成分支。由COⅠ序列构建的系统发育树(图2)可见:各地区的大头金蝇聚集成簇(BCL=95),并与绯颜裸金蝇形成一簇;不同地区的铜绿蝇聚集成簇(BCL=100),并与紫绿蝇共同形成一簇;不同地区的棕尾别麻蝇、家蝇也分别各自聚集成簇,BCL值均为100。上述不同地区不同种属的蝇类以种属聚集成簇,不同地区之间的样本聚集情况无明显差异。

表4 各蝇种COⅠ基因片段序列种间和种内的遗传距离Tab.4 The interspecific and intraspecific evolutionary divergence of COⅠ
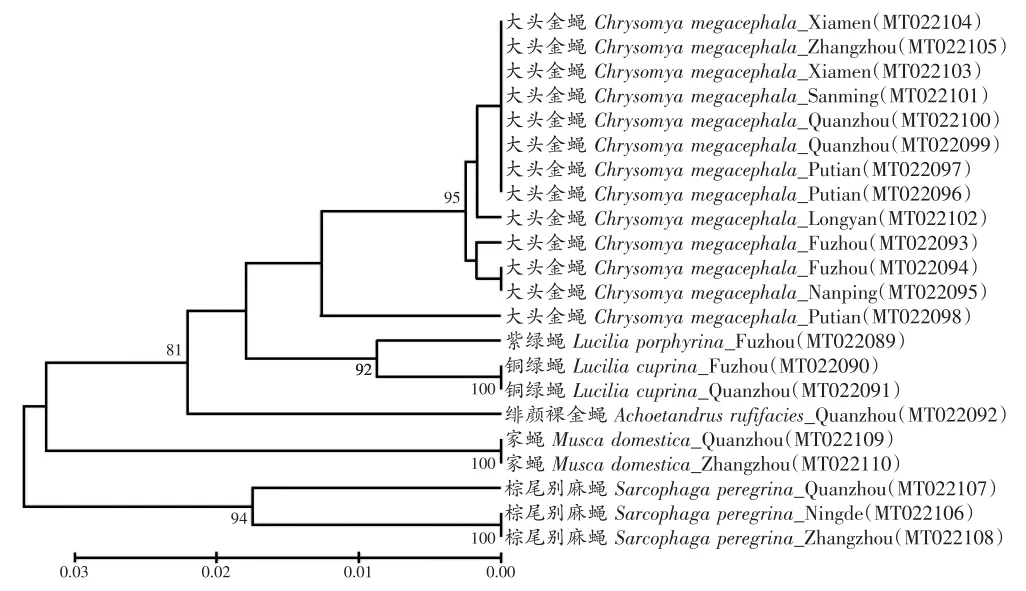
图1 各蝇种16S rDNA基因片段序列的系统发育树(UPGMA)Fig.1 Phylogenetic tree of 16S rDNA of flies by UPGMA
3 讨 论
分子鉴定方法作为形态学鉴定方法的补充手段,被越来越多的法医昆虫学者所接受[7-10]。其中,应用COⅠ及COⅡ序列对尸食性麻蝇进行分子鉴定被大多数学者所采用[11-13],但其缺点在于对样本质量要求高。近年来,寻找更易推广且种属鉴别力高的分子标记已成为昆虫分子鉴定研究的热点。线粒体16S rDNA是一种核糖体基因,在进化上较为保守,能够很好地反映各物种种属的进化,多应用于种属鉴定、进化分析等方面[14]。

图2 各蝇种COⅠ基因片段序列的系统发育树(UPGMA)Fig.2 Phylogenetic tree of COⅠof flies by UPGMA
本研究结果表明,福建省常见尸食性蝇类线粒体16S rDNA及COⅠ基因片段序列的同源一致性较高。各蝇种的种内遗传距离均数明显低于种间遗传距离均数,符合物种的遗传多态性特征,并且与前期其他地区的调查性研究结果[1,9,15-17]一致。根据 WELLS等[18]的研究,当遗传标记的种内遗传距离<1%、种间遗传距离>3%且与种间遗传距离范围无重叠,可应用于物种的分子鉴定。本研究中,不同蝇种16S rDNA和COⅠ基因片段序列的种内遗传距离均数均明显小于种间遗传距离均数,其中,最为常见的大头金蝇的16S rDNA种内遗传距离均数为0.6%,且样本最大种内遗传距离小于最小种间遗传距离,而16S rDNA基因片段序列种内最大遗传距离见于棕尾别麻蝇样本中,但仍然小于与其他蝇种的种间遗传距离。因此,16S rDNA对于福建省常见尸食性蝇类的鉴别具有良好的效力,可作为福建省常见尸食性蝇类种属鉴别的理想遗传标记。相较而言,COⅠ基因片段序列在棕尾别麻蝇样本的种内遗传距离均数为6.3%,与其他蝇种种间遗传距离范围没有交叉,表明COⅠ基因片段能够对不同种属的蝇种进行鉴别,但鉴别效力有待进一步验证。此外,本研究发现COⅠ的种内最大遗传距离为10.5%,与种间遗传距离范围有交叉重合。经分析,该结果为莆田地区捕获的样本,可能是由于不同地区大头金蝇的种群差异和地理隔离引起该样本的基因片段序列不同。
利用UPGMA对福建省常见尸食性蝇类样本构建16S rDNA和COⅠ基因片段序列的系统发育树,结果显示,不同地区相同种属的蝇种样本先聚集成簇,并与近缘种属形成姊妹簇或树形的分支部分。两个基因位点构建的系统发育树结果显示,聚类后BCL值多大于75%,说明聚类结果较可靠,且与形态学鉴定结果一致。所捕获的22个样本整体上被明确地分为丽蝇科、麻蝇科及蝇科三大分支,部分种属的样本形成了100%支持的独立簇分支,说明利用16S rDNA和COⅠ基因片段序列可以很好地对不同蝇种的样本进行区分。从系统发育树也可以看出,COⅠ基因片段序列中对于本研究出现的1只基因片段序列不同于其他样本的大头金蝇并不能够准确地鉴别,在发育树中该样本被错误地归到蝇科这一分支中,说明COⅠ基因片段序列对于存在序列差异性的样本的种属鉴别存在一定局限性。而利用16S rDNA基因片段序列却可以将该样本归入丽蝇科中,虽然并不能将其与绯颜裸金蝇完全区分,这主要是样本序列差异程度所决定的,在前期研究[16,19]中也有类似结论的报道,表明16S rDNA对大头金蝇与其他种属的鉴别有更高的价值。
本研究中COⅠ和16S rDNA基因片段序列在GeneBank数据库中的匹配一致性值(Max ident值)分别达到99.47%和99.72%。其中,16S rDNA对于紫绿蝇、铜绿蝇、绯颜裸金蝇、棕尾别麻蝇以及家蝇的同源性比对正确率均达到100%。不同蝇种的同源性比对中仍可见比对错误的样本出现,这种错误比对的现象在前期研究中亦有报道,有学者认为在近缘蝇种间存在杂交,可导致样本的基因序列出现错误比对或者与形态学鉴别不相符的现象[20-22]。因此,对于碱基差异较小的近缘种属的鉴别仍较困难,同时可能因为数据库中相关序列信息较少导致错误比对。随着各蝇种全基因序列的完善,种属的正确比对率将会进一步提高。
本研究通过对福建9个地区常见尸食性蝇类的调查研究并用16S rDNA及COⅠ基因片段序列进行种属鉴别,发现福建省的常见尸食性蝇类以丽蝇科为主,分布最广且最为常见的是金蝇属的大头金蝇。利用分子鉴定,线粒体COⅠ及16S rDNA两种基因片段序列均能够较为准确地对不同蝇种的种属进行鉴别;相较于COⅠ,16S rDNA对福建省常见的丽蝇科尸食性蝇类的种属鉴别价值更高,可以推广应用于命案勘验工作中精确鉴定尸食性昆虫种类,为死亡时间推断提供有力支撑。

