蛋白质-RNA相互作用鉴定技术研究进展
熊冰钰, 叶玙璠, 崔凌云, 郑天娇, 李茹玉, 徐永杰
信阳师范学院生命科学学院, 河南 信阳 464000
RNA是生命活动中一类极其重要的生物分子,种类繁多,现已发现包括mRNA、tRNA、snoRNA、miRNA、piRNA、lncRNA、circRNA等超过100种不同类型的RNA,在生命活动的各个方面均扮演着重要角色[1-2]。随着近年来对miRNA、lncRNA、circRNA等非编码RNA(ncRNA) 研究的深入,发现RNA的生物学功能具有惊人的多样性,而RNA功能的发挥与RNA结合蛋白(RNA binding protein, RBP)密切相关。RBP是一类可以结合特定RNA分子的蛋白质,在真核生物中种类较多,约占基因编码蛋白的2%~8%,迄今已发现近2 000种不同的RBP[3]。RBPs对RNA的识别主要通过特定RNA结合结构域(包括RRM、KH、dsRBD、ZnF、S1、TRAP、Pumilio和SAM等)来完成,但每种结构域识别的RNA序列是特异的,如KH结构域识别多聚胞嘧啶、RRM结构域识别富含AU序列的元件等[4-6]。RBP与其靶RNA相互作用,影响细胞中每个RNA分子的产生、定位和功能,对于维持从生物发生到降解的所有阶段的RNA代谢稳态至关重要,在决定细胞行为方面起着关键作用。揭示RNA与RNA蛋白质相互作用的分子机制,可以从本质上认识相关的细胞生理过程,因此,利用实验手段确定RBP-RNA结合以及结合的位点无疑是理解该RBP和RNA功能的关键,对于阐明重要生命过程、疾病发生的复杂分子机制具有重要意义。但由于RBP作用方式的复杂性、功能的多样性、作用空间位置的不确定性等原因,对其开展精确RNA-RBP互作研究一直是一个重大挑战。当前,在RNA领域,特别是ncRNA相关研究的迅速发展,加快了RBP-RNA相互作用技术的更新,开发出了一系列以RNA靶标的捕获杂交分析(capture hybridization analysis of RNA targets,CHART)、RNA免疫共沉淀(RNA immunoprecipitation, RIP)、紫外交联免疫沉淀(UV-crosslinking and immunoprecipitation,CLIP)、RNA Pull-down等为代表的体内外RNA-RBP互作鉴定技术。本文针对相关最新RNA-RBP互作技术进行了综述,比较分析了它们的技术原理、优缺点及其适用范围,并提出了未来需要解决的技术挑战,以期为新型技术的发现提供新的思路。
1 活体内研究RBP-RNA互作的技术
1.1 RNA纯化的染色质分离(ChIRP)技术
RNA纯化的染色质分离是一种通过纯化RNA分子从而获得其结合的染色质片段,进而鉴定与之相结合的基因组DNA和RBP的实验方法[7-8]。该方法在体内通过戊二醛将RNA与染色质交联,使RNA与染色质互作被固定下来,随后利用超声将染色质打断,再使用设计带有生物素或链霉亲和素标记的寡核苷酸探针与靶标RNA分子杂交,之后利用亲和素磁珠把靶标RNA拉下,同时与其共同作用的DNA染色体片段也会附在磁珠上,后续可以通过qRT-PCR、测序、免疫印迹法和质谱分析等鉴定与靶标RNA 互作的DNA和RBP。因此,能够在全基因组范围内鉴定RNA与染色质的互作,从而高通量检测与RNA结合的DNA或RBP,可能揭示许多生物学新途径,被不同领域的研究者们广泛采用。例如,Chu等[9]利用ChIRP结合质谱(MS)技术研究了X染色体失活过程中关键因子lncRNAXist的作用机制,发现来自染色质修饰、核基质和RNA重塑通路的81个蛋白质可与之互作,并伴随着从多能性到分化的转变。具体的相互作用蛋白主要包括RNA结合蛋白hnRNPK以及果蝇分裂末端同源蛋白Spen,hnRNPK参与Xist介导的基因沉默和组蛋白修饰,但不参与Xist定位;Spen同Xist的A-repeat结构域相互作用,是基因沉默所必需的,进而揭示XistlncRNA以模块化和受发育调控的方式与蛋白质结合,以协调染色质传播和沉默。随后Quinn等[10]对ChIRP技术进行了改造,演化成dChIRP(domain-specific chromatin isolation by RNA purification)新技术,该技术可以在活细胞中从单个RNA结构域的水平上剖析成对的RNA-RBP、RNA-RNA以及RNA-染色质之间的相互作用。其与ChIRP的区别主要在于反义寡核苷酸探针的设计上,dChIRP技术利用靶标RNA非重叠序列和非冗余序列设计生物素或链霉亲和素标记的反义寡核苷酸探针池,将RNA基因组定位信号提高了20倍以上,能够以较高的精度和灵敏度揭示RNA的结构和功能,大大提高了结果的准确性和敏感性。
1.2 RNA靶标的捕获杂交分析(CHART)技术
RNA靶标的捕获杂交分析是一种以分子杂交为基础,从可逆交联染色质提取物中富集内源性RNA及其靶标的方法,可以特异性鉴定与RNA互作的RBP或DNA[11-12]。与CHIRP技术类似,CHART也需要交联后用反义探针捕获,再用亲和素磁珠回收,最后将沉淀的RBP、RNA、DNA分别收集,进行分析。这两者主要的不同之处在于RNA靶标探针的设计,CHART通过RNase H试验寻找合适的靶标位点,设计生物素标记的短反义寡脱氧核糖核苷酸探针,从交联染色质提取物中特异性富集内源性RNA及它们的靶标[13]。Simon等[14]使用CHART以类似于蛋白质的染色质免疫沉淀的方式来研究果蝇剂量补偿相关的lncRNAroX2及与其相关的DNA和RBP靶标,结果表明roX2可结合在特定的基因组位点上,并与影响剂量补偿的男性特异性致死复合物蛋白质相结合,揭示了roX2的基因组靶标并在染色质水平发挥作用。
1.3 三分子荧光互补技术(TriFC)技术
双分子荧光互补技术(bimolecular fluorescent complimentary,BiFC)是基于两个不发光的荧光蛋白互补片段在借助其融合蛋白的驱动下重新形成活性荧光蛋白,直接作为报告基因的可视化关键性技术,是探究各种模式生物体内蛋白质与蛋白质间相互作用的重要手段[15]。而三分子荧光互补技术(trimolecular fluorescence complementation,TriFC)是基于BiFC技术基础上发展起来的一种新型技术,用于检测体内RNA与蛋白质之间的相互作用[16-17]。在TriFC技术中,通常选用传统的双分子荧光互补(BiFC)系统与两种不发生相互作用的RNA结合蛋白质(RBPs)或RNA识别结构域,再将RBP识别的潜在RNA序列融合进第三个表达载体,如果RBP分别与各自识别RNA序列相结合,那么这种相互作用将引导两个不相互作用的RBP相互靠近,从而实现荧光蛋白两片段的相互融合产生荧光(图1)。Zhu等[18]构建了基于红色mCherry的三分子荧光互补(TriFC)系统,用于监测mRNA-蛋白质相互作用的活细胞成像,研究了生理条件下细胞和病毒基因表达的转录后过程,证实活细胞中甲型流感病毒mRNAs与细胞TAP核输出途径中的ALY和UAP56蛋白互作,并通过免疫共沉淀试验进一步验证这些相互作用,这为深入揭示甲型流感病毒mRNAs的核输出机制奠定了基础。此外,Han等[19]开发了基于远红外的mneptune-TriFC系统用于观察活细胞和小鼠中mRNA-蛋白质相互作用的成像,发现PTB与HIV-1 mRNA的3′末端重复序列互作,表明PTB可能从潜伏期参与HIV激活中的HIV mRNA加工。因此,TriFC作为一个在活体实时检测RBP与RNA之间的相互作用的新工具,在动植物RNA-RBP互作研究中都得到较广泛应用[19-21]。

图1 TriFC技术原理示意图Fig.1 Schematic diagram of TriFC technology
1.4 RNA免疫共沉淀(RIP)技术
RNA免疫共沉淀是基于目的RBP特异性抗体富集RBP和靶RNA复合物来鉴定体内RNA-RBP互作的最常见方法[22-23],被广泛用于RBP的生物学功能研究中。例如,Pandey等[24]通过RIP技术发现lncRNA Kcnq1ot1同时可与H3K9甲基转移酶G9a和PRC2复合物互作,在细胞分裂过程中通过建立谱系特异性的转录沉默模式发挥作用;Rinn等[25]利用hnRNPK-RIP方法揭示lincRNA-p21与hnRNPK互作进而调节体细胞重编程。RIP主要包括细胞/组织的裂解和感兴趣的RBP抗体免疫沉淀天然RNA-RBP复合物,随后纯化RNA,再通过qPCR、基因芯片或高通量测序定量定性分析RNA,结合RNA的量化是用RIP富集的特定靶RNA的量与免疫沉淀之前来自裂解液的该RNA总量之比的百分数来表示[26]。由于RIP中RBP-RNA相互作用不是通过共价交联稳定的,因此,需要调整洗涤条件以确保RBP-RNA直接相互作用保留下来,而非特异性结合的RNA被除去。此外,动力学上不稳定结合的RBP可能在诸如超声波等剧烈的裂解条件下与其靶标RNA解离并与其他RNA重新结合,而温和的裂解条件似乎不会导致裂解后的相互作用,裂解后重新结合可能取决于RNA-蛋白质相互作用的动力学性质、裂解和免疫沉淀条件以及RBP及其RNA靶标的相对丰度[23,27]。早期RIP实验由于缺乏RNase消化步骤,不能鉴定转录本中的RBP特异结合位点,只能在整个转录水平分析RBP的结合情况。最近,Nicholson等[28]开发了一种称为DO-RIP(digestion-optimized RIP-seq)的RIP-seq方法,以解析RBP-RNA相互作用的特异结合位点,并以RNA结合蛋白HuR为例,分离出受HuR保护的20~70个核苷酸片段,确立了高分辨率的结合位点图谱。此外,通过使用包括总RNA和带有非特异性抗体的阴性对照免疫沉淀(IP)的对照样品,对整个转录组中具有高覆盖率的HuR结合位点进行定量分析,并进一步证实这种结合位点的定量富集与这些位点的体外相对结合强度成正比。
1.5 紫外交联免疫沉淀(CLIP)技术
紫外交联免疫沉淀主要利用了蛋白质和RNA在256nm紫外光照射下产生共价交联的特性,以保持生理条件下体内蛋白质与RNA复合物相互作用的真实状态,是研究RBP-RNA互作的重要技术之一[29-31]。该方法首先在紫外照射下,诱导活体细胞内RBPs和RNAs共价交联形成RBP-RNA复合物, 再用核酸酶消化部分RNA,然后用RBP特异性抗体将RBP-RNA复合物通过免疫沉淀进行富集,最后分离RNA和RBP,并对其通过qRT-PCR、WB、高通量测序和质谱鉴定等方法进行检测,从而高效探究RNA与RBP的调控作用。与RIP技术相比,CLIP技术增加了紫外交联和RNA酶消化步骤,保证了RBP-RNA的结合更加紧密,甚至能捕捉体内的瞬时相互作用,也避免了细胞裂解和纯化过程中RNA的丢失以及形成非特异的RBP-RNA复合物,同时RBP与RNA结合位点的检测也更加方便灵敏[23,29]。由于该方法基于紫外交联,而紫外线在穿透性、偏好性和交联效率等方面的局限,以及交联的RNA与非交联的RNA的分离,这便对原材料的量要求较大,很难在少数细胞情况下鉴定,因此,一般只运用于组织水平。尽管CLIP技术存在一些局限,但在近十来年里依然被广泛应用,并取得了丰富的成果,此外, CLIP技术与高通量测序的结合,可以在全基因组范围内捕获生物体内RBP-RNA的互作,更好地获得有价值的序列信息。而本身在交联效率、特异性和灵敏度上也在不断地改进,在CLIP的基础上又衍生出PRA-CLIP、CRAC、iCLIP、eCLIP、SpyCLIP等各具特色的新技术。
1.5.1 光活化核苷增强的CLIP(photoactivatable-ribonucleoside-enhanced CLIP,PRA-CLIP) 通常情况下蛋白质与RNA发生紫外交联的效率很低,只有1%~5%,严重制约了CLIP技术的应用。为了提高紫外交联效率,Hafner等[32]开发了光活化核苷增强的CLIP(PRA-CLIP),可以将交联效率提高100~1 000倍。该方法主要利用4SU(4-thiourdine,4-硫-脲嘧啶)、6SG(6-thioguanosine,6-硫-鸟嘌呤)等一些具有光学活性的核糖核酸衍生物,通过与活体细胞共培养将其插入到活体细胞的新生RNA转录本中,而365 nm紫外线可以加强核苷酸衍生物和蛋白质之间的交联,使得交联效率远高于传统的254 nm紫外光诱发的天然核酸与蛋白质的交联。此外,4SU或6SG的硫原子交联氨基酸后造成其碱基形成氢键的方式发生改变,在随后的反转录过程中4SU的插入导致T到C的转变,而6SG促使G到A的转变,这些突变位点提供了精确的交联位点信息,有利于更加准确的检测其特异性结合位点,也能消除背景噪音,极大地提高了分辨率。但是长时间的使用6SG或4SU可能导致细胞中毒,因此,需要注意检测细胞对这些核糖核苷酸的反应。PAR-CLIP技术不仅在人类细胞系中成功地应用于50多个RBPs的研究,而且在包括果蝇和小鼠等其他物种的细胞系以及线虫和酿酒酵母等整个生物体中都得到了广泛的应用[33-34]。例如,Ascano等[35]利用PAR-CLIP技术揭示了脆性X综合征(FXS)和自闭症谱系障碍(ASD)相关的RNA结合蛋白FMRP在体内能结合多种mRNA进而起到调控蛋白质表达的作用,并且这些调控在FXS和ASD中是相似的,为神经疾病寻求新的治疗靶点提供了基础。
1.5.2 cDNAs交联和分析(crosslinking and analysis of cDNAs,CRAC) 传统的CLIP技术是在非变性条件下纯化蛋白质,当目标RBP包含于多元RNP 复合物中时,易受复合物中其他蛋白质的干扰,加之有些抗体特异性不高和RNA污染等因素,往往导致CLIP结果特异性较差。为了提高CLIP的特异性, Tollervey等[36]基于CLIP开发了cDNAs交联和分析(CRAC)技术。该技术的关键是在目标RBP的C末端或N末端融合HTP(His6-TEV-Protein A)或PTH (Protein A-TEV-His6)标签,随后的CLIP纯化分两步进行,首先利用免疫球蛋白IgG结合标签中的蛋白A,然后经TEV蛋白酶切下目标RBP,再利用Ni 基质结合纯化组氨酸标签,而第二步纯化是在6 mol·L-1盐酸胍变性条件下,可有效地解除和目标RBP结合的其他蛋白质。经过两步亲和纯化,尤其第二步在变性条件下进行,能显著地提高结果的特异性,从而有效的鉴定RNA与RBP的结合位点。由于CRAC方法具有很高的特异性,已经成功应用于定位多个核糖体加工因子在核糖体RNA 上的结合位点,以及RNA相互作用、RNA解旋酶结合位点的鉴定中[37-38]。
1.5.3 单核苷酸分离CLIP(individual nucleotide resolution CLIP, iCLIP) 传统的CLIP方法在逆转录过程中,交联位点氨基酸残留会引起逆转录的突变、缺失甚至是终止,导致缺少5′接头的截短cDNA,而3′接头和5′接头是后续PCR扩增所必须的,致使截断短的cDNA在CLIP中没有扩增,为了解决这一问题,König等[39]在CLIP基础上开发了单核苷酸分离CLIP(iCLIP)技术。iCLIP技术可巧妙利用交联位置能够抑制逆转录的进行这一特点,通过RT引物将3′接头与cDNA相连,再在环化酶的作用下将cDNA环化,随后再通过RT引物中的BamHⅠ酶切位点使环状cDNA线性化,实现对正常和截短的cDNAs的扩增和测序,从而可以在单核苷酸水平上精确的确定蛋白质-RNA交联位点(图2)。iCLIP技术已经成功地应用于RBPs在选择性剪接、可变聚腺苷酸化、RNA甲基化、mRNA稳定性等方面的功能研究[40-43]。通过该技术确定了不同RBPs的高分辨率RNA剪接图谱,从而能够评估RBP结合在替代外显子周围的位置如何决定它们的剪接功能,更重要的是,除了识别RBP结合位点外,iCLIP还可以对RBP-RNA相互作用的动态变化进行定量分析。例如,通过定量iCLIP数据证实RNA结合蛋白hnRNPC与剪接因子U2AF65在许多剪接位点上存在竞争关系,hnRNPC的缺失导致U2AF65获得了数百个Alu元件的识别权,致使先前被抑制的Alu外显子错误的形成,这严重破坏了转录本功能,而hnRNPC在正常生理条件下能够阻止这种错误识别,进而在全基因组范围内维持转录组的稳定发挥了关键作用[44]。
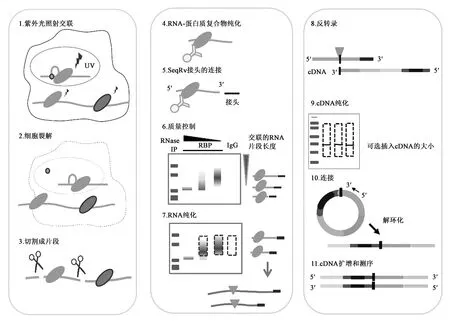
图2 iCLIP技术流程图Fig.2 Flowchart of iCLIP technology
1.5.4 优化的CLIP(enhanced CLIP,eCLIP) 尽管iCLIP很大程度地提高了分辨率,消除了PCR扩增的偏好性,提高了其扩增效率,但由于iCLIP反应步骤较多,环化效率不高,熟练操作较困难,且需消耗大量的原材料,这些都限制了iCLIP的广泛应用。Van Nostrand等[45]在iCLIP技术基础上对该技术进行进一步改进,提出了一种全新的CLIP方法,称为优化的CLIP(eCLIP)。该方法分两步去添加接头,首先将一个3′RNA接头连接到交联的RNA片段上,逆转录后再将一个3′ssDNA接头连接到逆转录终止端cDNA处,舍去了环化这一步,降低了连接失败导致的RNA片段损失,而且大大缩短了实验的手动操作时间(图3)。而eCLIIP技术结合高通量测序,极大的提高了建库的成功率,改进之后的方法相比普通的CLIP-seq而言,RBPs靶标识别率提高了2~3倍,该方法为RBPs在全基因组范围内的maps提供了一个更加强大、标准化的框架。利用此方法,科学家们揭示了众多蛋白调控RNA的新功能,如eIF4A3在转录后基因调控中的作用[46], MOV10-mRNA 3′UTR和MOV10L1-piRNA在哺乳动物睾丸发育中的调控机制[47]。

图3 eCLIP技术流程图Fig.3 Flowchart of eCLIP technology
1.5.5 基于SpyTag的CLIP(SpyTag-based CLIP,SpyCLIP) 基于SpyTag的CLIP(SpyCLIP)技术是中国科学院生物化学与细胞生物学研究所吴立刚团队最近将SpyTag-SpyCatcher这一共价交联系统创新性地应用到CLIP中开发出的一种新的CLIP技术[48]。该方法采用在RBP融合SpyTag和SpyCatcher之间形成共价键,可以承受用于去除非特异性相互作用的最苛刻的洗涤条件,极大地提高了所获蛋白质-RNA复合物的纯度;同时对CLIP建库的流程进行创新设计,绕过了放射性标记和PAGE膜纯化步骤,整个过程可以在磁珠上进行,易于自动化,避免了传统CLIP方法中多步限制规模化操作和大量损耗起始材料的效率限制性步骤;此外,还引入通用的Input文库用以消除背景,进一步提高了CLIP方法的特异性。这些核心改进不仅避免了现有CLIP方法对抗体的依赖,提高了数据的信噪比和可重复性,并大幅降低了操作的难度和时长,使得SpyCLIP方法能够适应高通量研究的需求。利用SpyCLIP这一新工具,研究人员在哺乳动物细胞中研究了内含子结合蛋白PTBP1和RBFOX2的RNA结合图谱,发现SpyCLIP可以真实、高效地捕获它们的剪接调控位点;此外,还研究了miRNA效应器蛋白AGO2的结合图谱,比较和揭示了3′UTR和CDS区miRNA靶位点的机制和特征。SpyCLIP对多种RBP均表现出杰出的灵敏度和特异性,为规模化、系统性研究蛋白质-RNA相互作用提供了一种简洁易用且具备高精度的新工具。
2 体外研究RBP-RNA互作的技术
2.1 RNA Pull-down技术
RBP-RNA互作的体内研究技术种类较多,而体外方法研究很少,当前使用最广泛的是RNA Pull-down技术。RBP-RNA互作的体内研究大多数是以RBP为中心,而RNA Pull-down则是以RNA为中心的方法鉴定与RNA相互作用的所有蛋白,已成为研究RNA(尤其是lncRNA、circRNA、miRNA等ncRNA)功能的主要技术手段之一[49-50]。该方法首先利用体外转录产生RNA或感兴趣的RNA的一部分,再利用生物素对RNA进行末端标记;然后与蛋白提取物孵育以允许RNA-蛋白质相互作用,再用链霉素亲和磁珠将特定的复合物从混合物中拉出来,并进行沉降;最后用质谱或蛋白质印迹鉴定这些蛋白质(图4)。Barnes等[49]用该方法确认了X染色体失活相关的lincRNAXist与EZH2等多梭蛋白复合体互作,有助于其在X染色体相互作用中的功能;Bai等[51]利用AR(雌激素)mRNA中富含AU元件的3′UTR在脂肪细胞裂解液中进行RNA Pull-down分析,鉴定到了重要的RNA结合蛋白HuR,揭示了HuR在脂肪细胞从转录后水平调节AR表达发挥关键作用。

图4 RNA Pull-down技术示意图Fig.4 Schematic of RNA Pull-down technology
2.2 RNA电泳迁移分析(RNA electrophoretic mobility shift assay,REMSA)技术
长期以来,电泳迁移分析(electrophoretic mobility shift assay,EMSA)一直是体外检测和鉴定DNA结合蛋白质及其靶序列的重要工具[52-53]。其原理是DNA与蛋白质互作时会形成DNA-蛋白质复合物,该复合物在凝胶中比未结合蛋白质的DNA迁移速度慢,从而形成一条相对滞后的条带,在典型的EMSA试验中,靶DNA序列可以用荧光探针、生物素标记或放射性同位素核标记进行检测。RNA电泳迁移分析(REMSA)是在EMSA基础上发展起来的,用于研究RBP与其相关的RNA结合序列相互作用的技术,可检测RBP和其相关的RNA结合序列的互作,并对RNA进行定性和定量分析[54]。与EMSA相比,REMSA主要是将DNA探针替换为RNA探针,其余方法步骤类似,但由于RNA的特殊性,RNA探针制作较麻烦且价格昂贵,而且探针为单链RNA容易降解,REMSA实验操作起来具有一定的技术难度。目前,REMSA已得到较为广泛的应用,例如,Yang等[55]利用REMSA方法证实Circ-HuR可与CNBP互作从而阻止其结合在HuR启动子上,进而抑制HuR的表达和胃癌进展;Gulappa等[56]利用REMSA发现LRBP可与黄体生成激素受体(LHR)mRNA结合,进而通过与真核生物起始因子5A(EIF5A)互作在转录后调节卵巢中LHR的表达来发挥作用。
3 展望
RBP与RNA互作在生命活动中扮演重要角色,随着生命科学的快速发展,更多、更深入的RNA及RBP相关分子机制的揭示离不开RBP-RNA相互作用鉴定技术的支撑,这必然促进RBP-RNA互作实验鉴定技术的发展。近些年,出现了以CLIP、CHART、TriFC、RIP、RNA Pull-down等为代表的一系列RBP-RNA互作分析方法,并广泛地应用到RNA和RBPP发挥功能的分子机制研究中,但这些技术或多或少都存在一些不足,例如,CHART技术染色质交联效率低并且耗时较长;TriPC易出现假阳性且对温度的要求比较严格;RIP技术特异性不高且需要抗体;CLIP实验易受到紫外交联的低效、非特异RNA 污染、复杂的实验步骤和RNA不稳定性等因素影响;RNA Pull-down技术难度大且成本较高。因此,RBP-RNA互作还有待进一步突破和完善,特别是快速、准确、经济地鉴定RBP-RNA之间的相互作用的实验技术。

