胶质母细胞瘤预后相关基因的数据挖掘分析
马胜男, 张晓康, 仪杨, 张昭, 姚婷婷, 赵清辉, 谢飞
北京工业大学生命科学与生物医学工程学院, 北京 100124
胶质母细胞瘤,又称多形性胶质母细胞瘤(GBM),最早是由Percival Bailey和Harvey Cushing于1926年提出,他们认为该肿瘤起源于原始的胶质细胞前体,由于坏死、出血和囊肿的存在而呈现出高度可变的外观[1]。在世界卫生组织(World Health Organization,WHO)分级中,多形性胶质母细胞瘤属于IV级恶性胶质瘤,是仅次于脑膜瘤的第二常见的中枢神经系统癌症,同时也是侵袭性最高的颅内恶性肿瘤。目前GBM的基本治疗方法为化疗药物替莫唑胺(TMZ)与放射治疗相结合,虽然采用了多种给药方式以达到维持TMZ浓度、降低TMZ神经毒性的目的[2],但治疗效果并不理想,患者复发率很高、预后差。此外,由于GBM中存在免疫活性抑制,除传统方式外还可采用嵌合抗原受体T细胞等免疫疗法进行治疗,但免疫疗法的联用方式仍在测试当中[3]。总之,在无任何治疗情况下GBM患者中位生存时间仅为3个月[4],而治疗后中位生存期一般也仅为1~2年[5-6]。
GBM的发生和发展与肿瘤基因表达密切相关[7-8]。根据组织病理学分类,GBM最早被分为四种亚型[9],2016年WHO在组织病理学基础上结合分子分型方法又将GBM更正为三种亚型[10],这说明基于基因表达谱数据的分子分型方法被越来越多地临床诊断标准所吸收和接受。本研究利用GBM患者的基因表达谱数据进行深度挖掘,从中筛选出与GBM患者预后密切相关的表达差异基因,从而为开发新的抗GBM药物提供潜在作用靶点,同时也为GBM患者预后评估提供新的依据。
1 材料与方法
1.1 基础数据获取
GBM患者的基因表达谱来自NCBI的微阵列/基因图谱公共数据库,我们从数据库中的GSE108474、GSE50161、GSE29796 3个基因表达谱芯片中获取了胶质母细胞瘤和正常组织中的基因表达谱。这3个基因表达谱芯片以GPL570平台为基础,其中GSE108474中包含了221个胶质母细胞瘤组织和28个正常组织,GSE50161中包含了34个胶质母细胞瘤组织和13个正常组织,而GSE29796中包含了14个胶质母细胞瘤组织和20个正常组织,同时数据库中还可获取包括病理学分型等患者的临床资料信息。
1.2 差异表达基因的鉴定
使用NCBI中线上工具GEO2R提取数据,用ggplot2 及R语言以|logFC|>2与adj.P<0.05为标准筛选差异表达基因(DEGs)并绘制火山图,设置log FC<0为下调基因,log FC>0为上调基因。利用Venn软件对筛选出的数据进行在线分析,找出3个数据集中共同存在的基因。
1.3 基因功能富集分析
利用Cluster Profiler及R 语言以FDR<0.05为标准进行功能富集分析,通过生物过程(BP)、分子功能(MF)、细胞成分(CC)来识别基因功能,同时进行KEGG-pathway富集分析。
1.4 蛋白互作网络的构建
利用STRING数据库检索蛋白互作网络(PPI)进行评估,研究差异基因间的相关性。Cytoscape重构后利用其中的MCODE插件找到定位紧密相连的核心区域,只选择节点数>10的独立网络进行分析。
1.5 基因表达验证及生存分析
利用GEPIA网站对来自TCGA数据库中的样本RNA测序表达数据进行分析。为进一步验证基因表达与预后的关系,制作Kaplan-Meier图说明DEGs基因表达水平与患者总体生存的相关性。
1.6 数据分析软件
基础数据获取自NCBI(https://www.ncbi.nlm.nih.gov/geoprofiles/?term=);差异表达基因鉴定采用ggplot2 版本3.3.0及R 3.6.2版本;Venn图绘制网址:http://bioinformatics.psb.ugent.be/webtools/Venn/;基因功能富集采用ClusterProfiler,版本3.14.3及R 3.6.2版本;蛋白互作网络网址:https://string-db.org;Cytoscape 版本3.7.2;基因表达验证及生存分析网址:http://gepia.cancer-pku.cn。
2 结果与分析
2.1 数据库中胶质母细胞瘤差异表达基因的筛选
本研究共包含了269个GBM组织和61个正常组织。运用在线分析工具在GSE108474、GSE50161、GSE29796 3个基因芯片中分别提取出1 367、1 479和2 633个差异表达基因(DEGs)(图1)。对3个基因芯片交叉数据集识别结果显示,GBM组织中共检测到344个交叉基因,包含259个上调基因(图2A),85个下调基因(图2B)。
2.2 胶质母细胞瘤差异表达基因功能富集分析
2.2.1 基因富集分析 利用Cluster Profiler对344个差异表达基因进行GO分析(图3,FDR<0.05)。结果表明,差异基因主要中在以下几个方面:①生物进程(biological processes, BP):包含146个亚类,差异基因主要表现在对细胞周期、核分裂、有丝分裂、细胞外基质组织形成等生物过程的影响;②细胞组成(cellular component,CC):包含45个亚类,差异基因主要表现在对浓缩染色体、细胞外基质、蛋白激酶复合物等物质组成的影响;③分子功能(molecular function,MF):包含13个亚类,差异基因主要表现在对细胞粘附分子、钙黏蛋白、纤连蛋白结合、细胞周期蛋白、组蛋白激酶、氧化还原酶活性,细胞外基质蛋白结构成分等相关功能蛋白的影响。

图1 胶质母细胞瘤差异表达基因筛选Fig.1 Screening of differentially expressed genes in glioblastoma

图2 胶质母细胞瘤芯片Venn图统计Fig.2 Venn diagram statistics of glioblastoma chip
2.2.2 KEGG通路分析 对344个差异基因进行KEGG pathway分析筛选共得出16条与差异表达基因密切相关的KEGG信号通路(图4,FDR<0.05),主要体现在细胞周期,p53、PI3K-AKT、AGE-RAGE、Hippo信号通路以及ECM受体相互作用方面。
2.3 胶质母细胞瘤差异表达基因蛋白互作网络建立(PPI)及分析
为更好地理解基因间的相互作用,采用在线工具对差异表达基因进行分析并获得蛋白相互作用(PPI)网络,同时实现互作关系的可视化(图5A)。结果表明,344个差异表达基因中有67个未被纳入到PPI网络(图5B),剩余的277个差异表达基因中有37个基因可形成核心模块,共包含618条边(图5C)。
2.4 核心基因的表达与生存相关分析
2.4.1 核心基因在正常人与胶质母细胞瘤患者中的表达 为了探索PPI网络中37个核心基因在胶质母细胞瘤患者与正常人之间表达水平的差异,采用在线工具对核心基因进行数据分析,结果表明这37个核心基因在GBM组中较正常组表达水平均显著升高(图6,P<0.05)。
2.4.2 核心基因表达与患者生存关系 为探索单个差异表达基因在总体生存中的潜在作用,利用在线工具对37个核心基因在TCGA数据库中的预后信息进行识别,并生成生存曲线,发现RRM2、PTTG1、E2F7基因与整体生存率密切相关,这3个基因的表达水平与患者中位生存期呈现显著性负相关(图7)。
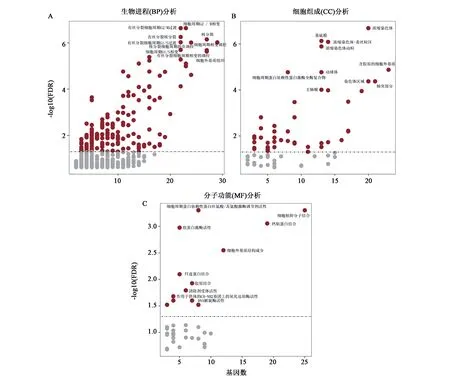
图3 差异表达基因GO分析Fig.3 The enriched GO terms for the differentially expressed genes
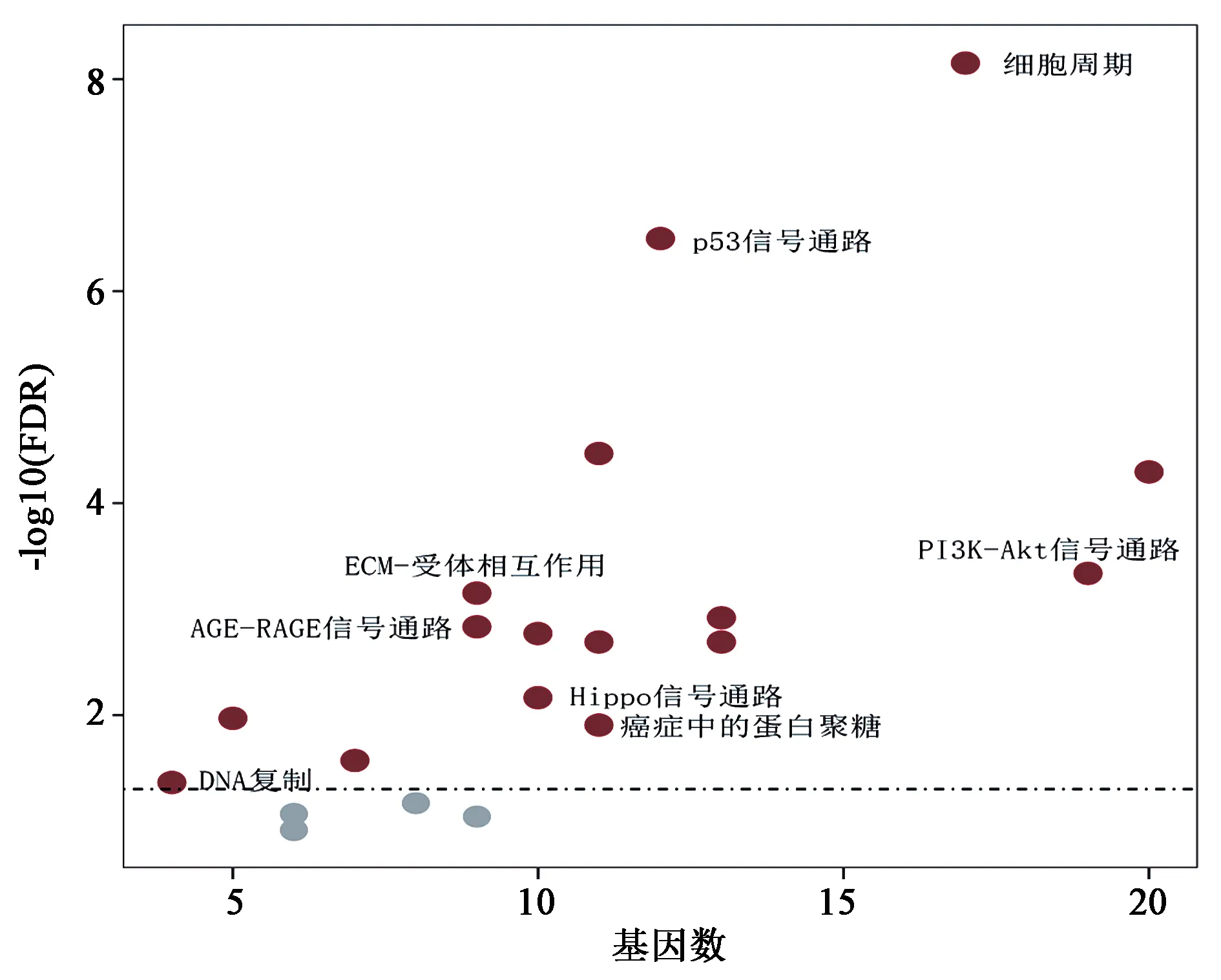
图4 差异表达基因KEGG pathway分析Fig.4 The enriched KEGG pathway for the differentially >expressed genes
3 讨论
本研究旨在通过对癌症公共数据库进行数据挖掘,寻找与胶质母细胞瘤预后密切相关的基因标志物,这些标志物不仅可以作为新的抗GBM药物研发的潜在靶点,同时也有助于对GBM患者的预后进行评估。研究中首先对GSE108474、GSE50161、GSE29796 3个数据集的269个GBM组织和61个正常组织分析发现344个交叉基因,其中259个基因表达上调,85个基因表达下调。基因功能富集分析发现差异表达基因主要集中在与细胞周期及与癌症进展关系密切的P13K-AKT等信号通路,进而构建基因网络对差异基因筛选得出37个核心基因,再通过在线分析工具进行基因表达量验证和存活率分析,发现3个基因(PTTG1、RRM2、E2F7)与GBM患者中位生存期呈现显著性负相关。数据分析方法及简要流程如图8所示。

图5 蛋白互作网络分析Fig.5 Analysis of protein interaction network

注:图中红色代表胶质母细胞瘤组织(n=163),灰色代表正常组织(n=207)。图6 核心基因在胶质母细胞瘤及正常组织中的表达量Fig.6 Expression of core genes in glioblastoma and normal tissues

图7 基因表达与患者生存率关系Fig.7 Relationship between gene expression and patient survival

图8 预后相关基因分析流程图Fig.8 Prognostic related gene analysis flow chart
在GO分析中,PTTG1主要富集在以下几方面:生物过程(BP)中对有丝分裂、细胞周期G2/M过渡、 有丝分裂核分裂的影响;细胞组分(CC)中对浓缩染色体组成的影响;分子功能(MF)中对钙粘蛋白结合的影响。RRM2主要富集在以下几方面:生物过程(BP)中对有丝分裂细胞周期G1/S过渡的影响、细胞组分(CC)中对细胞外基质成分、核基质的影响;分子功能(MF)中对细胞外基质结构成分形成、钙粘蛋白结合、纤连蛋白结合的影响。E2F7主要富集在以下几方面:生物过程(BP)中对有丝分裂细胞周期G1/S过渡的影响、细胞组分(CC)中对浓缩染色体的影响;分子功能(MF)中对钙粘蛋白结合的影响。从以上结果可以看出,PTTG1、RRM2、E2F7与有丝分裂过程中细胞周期过渡、钙粘蛋白质结合有显著相关性。
经调研发现,本研究所发现的3个与GBM预后显著相关的基因在肿瘤发生发展中起到关键作用。其中PTTG1即垂体瘤转化基因1,在染色体稳定性和DNA修复中起着核心作用。它也是一种致癌转录因子,过表达的PTTG1可能促进肿瘤转移。PTTG1基因的过表达导致染色体不稳定,增加了肿瘤形成和转移的可能性[11]。肾上腺皮质癌中PTTG1高表达患者的中位生存期为1.8年,而PTTG1低表达患者的中位生存期为9.0年。PTTG1的过表达与肾上腺皮质癌的不良生存相关[12]。非小细胞肺癌中PTTG1促进细胞迁移和侵袭,降低患者生存率[13]。RRM2即核糖核苷酸还原酶M2属于核糖核苷二磷酸还原酶小链家族。RRM2通过相应的核糖核苷酸催化脱氧核糖核苷酸生物合成,提供DNA合成必需的前体,并以细胞周期依赖性方式调节。RRM2可诱发NF-κB依赖的MMP-9激活,增强细胞侵袭性[14]。RRM2作为胰腺癌对吉西他滨反应的预测标志物,过表达可增强胰腺癌细胞侵袭性[15-16]。在头颈部和肝癌中作为预测治疗靶点调节Bcl-2,降低患者生存率[17]。RRM2与传统上被认为是肿瘤抑制因子的BRCA1为转录共激活因子,二者作用可避免癌基因诱发的复制应激、DNA损伤和细胞凋亡,提高GBM细胞存活率。值得注意的是,BRCA1-RRM2的相互作用不存在于研究的非GBM癌细胞系中[18]。转录因子E2F7具有两个DNA结合结构域,可独立于DP辅因子与DNA结合,参与血管生成、DNA损伤反应过程。E2F7异位表达导致E2F靶基因的抑制和G1期细胞的积累。此外,在S期E2F7增加与E2F调节启动子的缔合[19]。2015年,Chu等[20]研究表明, E2F7高表达导致他莫昔芬耐药,增加了患者复发的风险。2018年,Liu等[21]发现乳腺癌患者E2F7 mRNA水平升高与患者预后密切相关。肝癌组织中E2F7 mRNA表达水平明显高于癌旁组织,可通过增加Wnt信号通路活性降低患者生存率[22]。目前与GBM预后相关基因的研究主要集中在EGFR[9]、TP53[23,24]、MGMT[24,25]、IDH1[9,24,26]、IDH2[24,26]、1p19q[24]等基因上,尽管已有研究表明PTTG1、RRM2和E2F7基因与癌症发生发展密切相关,但这些基因的表达与GBM预后的关系还不明确,本研究为GBM患者的预后评估提供了新的基因标志物。
综上所述,我们通过对癌症公共数据库进行深入的数据挖掘,最终筛选出与GBM患者预后密切相关的3个基因:PTTG1、RRM2、E2F7,这3个基因在GBM的发生和发展中可能起到关键作用。本研究不仅可以为GBM瘤患者预后评估提供参考,同时也为寻找新的抗GBM药物作用靶点提供了重要的线索。

