基于COⅠ基因的10种水螨DNA条形码分析
张 旭,王柳玉,万胜豪,汤建强,王 琪,丁新雅,吴萍萍①
(淮北师范大学 生命科学学院,安徽 淮北235000)
0 引言
水螨(water mite),即为水体中生活的螨类,在分类学上,其隶属于节肢动物门Arthropoda、蛛形纲Arachnida、蜱螨亚纲Acari、真螨总目Acariformes、绒螨目Trombidiformes、前气门亚目Prostigmata、寄殖螨股Parasitengonina、水螨亚股Hydrachnidiae[1]. 水螨在水体中生态分布极为广泛,是淡水无脊椎动物的重要组成部分,也是研究底栖水生节肢动物生态学的功能类群[2]. 水螨的物种丰富度和种群数量与水体质量有着密切关系,因此水螨中许多种类可作为水体污染程度的生态指标[3-4]. 我国对于水螨的分类研究进展较为缓慢,严重限制利用水螨进行淡水环境监测等方向的应用研究. 其主要原因是水螨个体形态微小(0.2~0.8 mm),种类繁多,形态结构变化复杂,一些关键的鉴别特征需要通过解剖后才能观察到,加上目前从事水螨分类的研究人员逐渐减少,使得通过常规的形态学鉴定水螨物种非常困难. DNA条形码技术的出现为解决水螨物种的鉴定问题提供新思路.
DNA条形码技术是利用线粒体细胞色素氧化酶Ⅰ(cytochrome oxidase subunit I,COⅠ)基因中一段长度约658 bp的序列片段实现对物种的快速鉴定. 目前,COⅠ被认为是动物界中最适合的DNA条形码基因,已在鸟类、两栖类、昆虫等众多动物类群中得到广泛的应用[5-7],然而目前水螨的DNA条形码研究仅限于个别种类的修订和新种的描述,还未能在多数类群中开展[8-9]. 本研究以黄山及杭州等地区的水螨为研究材料,探讨利用DNA条形码技术在水螨物种鉴定中的可行性,为以后水螨的应用研究提供理论依据和技术支持.
1 材料与方法
1.1 样本来源
本研究共采集6种水螨9个样本,样本经常规采集后浸泡在无水乙醇中,-20 ℃保存. 根据水螨的分类标准进行形态学鉴定. 为充分验证DNA条形码在水螨物种中的可行性,本研究还从GenBank中下载我国已经发表的4种水螨11条序列(样本信息详见表1).
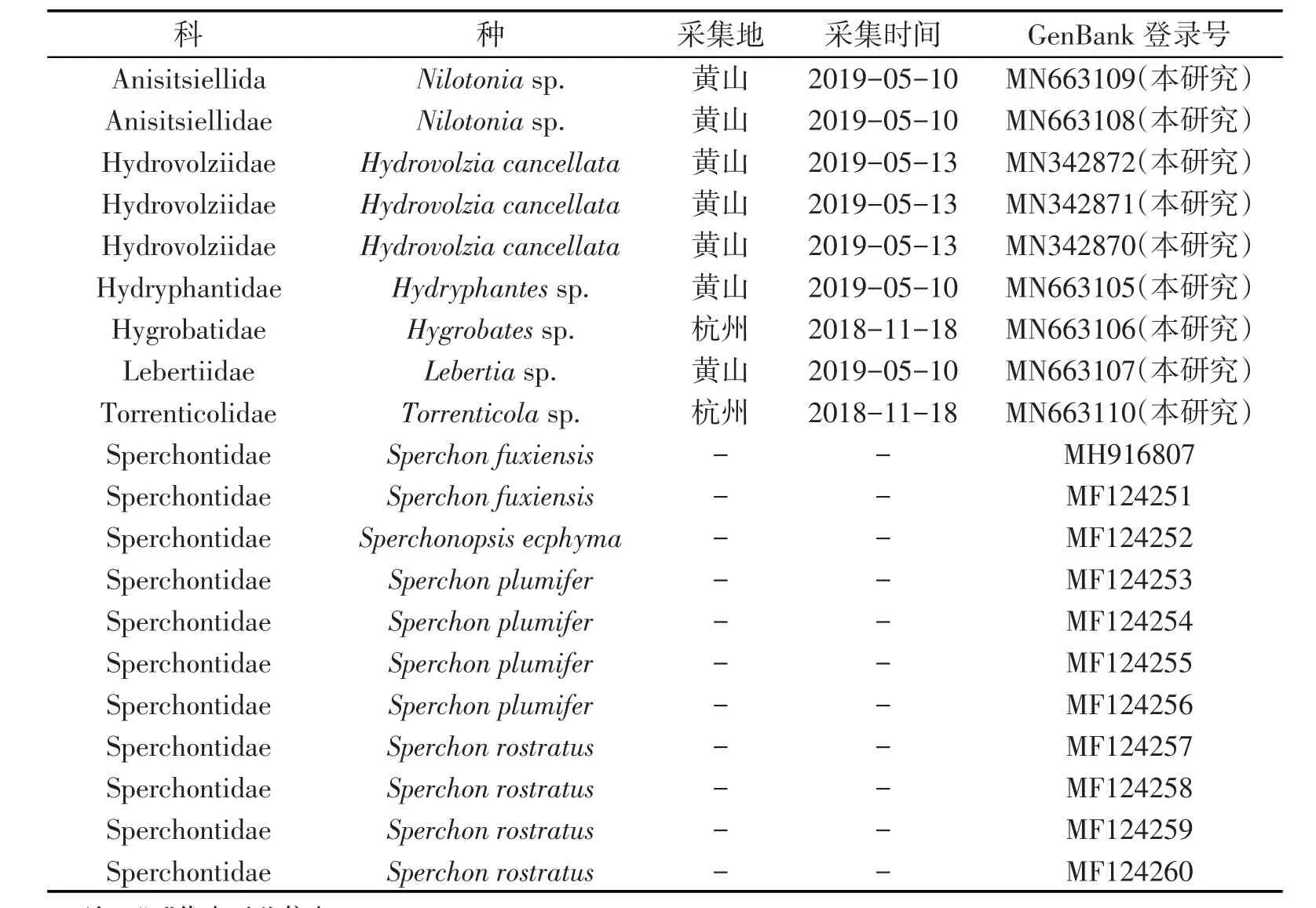
表1 样本信息
1.2 实验方法
将水螨样本用无菌水清洗后,用微量基因组提取试剂盒(TIANGEN DP316或QIAGEN 69504)提取总DNA. COⅠ基因扩增引物参考条形码的通用引物并进行修改COⅠF(5′-GGTCAACAAATCATAAA⁃GATATTGG-3′)和COⅠR(TTCAGGGTGACCAAAAAATCA -3′). PCR扩增程序为:94 ℃5 min,35次循环(94 ℃30 s,51 ℃30 s,72 ℃45 s),72 ℃10 min. 使用琼脂糖凝胶进行电泳检测,切下目标条带,使用胶回收试剂盒进行纯化(QIAGEN 28704). 将载体pEASY-T1 Simple Cloning Vector(TRANS CT111)与纯化后的PCR产物连接,进行蓝白斑筛选. 在每个样本中至少选择2个阳性克隆,送至上海华大基因公司进行测序.
1.3 数据分析
用EditSeq 5.0 软件打开测序结果,删除两端载体序列,在GenBank 和BOLD Systems 进行比对,通过分子鉴定,确保所获序列是目的序列. 用MEGA 7.0 软件进行序列比对,保留658 bp的高同源性序列,计算碱基组成,保守位点,变异位点,简约信息位点;基于K2P模型计算遗传距离并导出相应数据,使用Ex⁃cel软件绘制遗传频率分布直方图;使用DAMBE 6.6.47软件对序列的饱和度进行分析,以此检验序列是否适用于后续的分析;以绒螨科Trombiidae(GenBan登录号:KP979229)为外群釆用邻接法(neighbor-join⁃ing,NJ)构建系统发育树,分支的置信度用自展法进行1000次重复抽样检测.
2 结果与分析
2.1 GenBank与BOLD Systems的分子鉴定结果
本实验有9个样本成功得到COⅠ序列. 9条序列在GenBank和BOLD数据库中分子鉴定结果相似,仅在比对相似度上稍有差异(见表2). 所有序列均未能通过分子数据鉴定到具体的种类,序列的比对相似度在80.90%~89.38%(GenBank)和80.19%~89.26%(BOLD),其中有66.67%的样本(6个样本)的分子鉴定结果在属级阶上与形态学鉴定结果一致,有33.33%的样本(2 个Nilotonia sp. 的样本和1 个Hydry⁃phantessp. 的样本). 在科级阶元上也与形态学鉴定的结果不一致,仅能通过分子数据鉴定到目级阶元.有77.78%的样本(7个样本)通过GenBank和BOLD数据库比对的物种相同,而有22.23%的样本(1个Tor⁃renticolasp. 和1个Hygrobatessp.)在2个数据库中比对的结果为不同的物种.
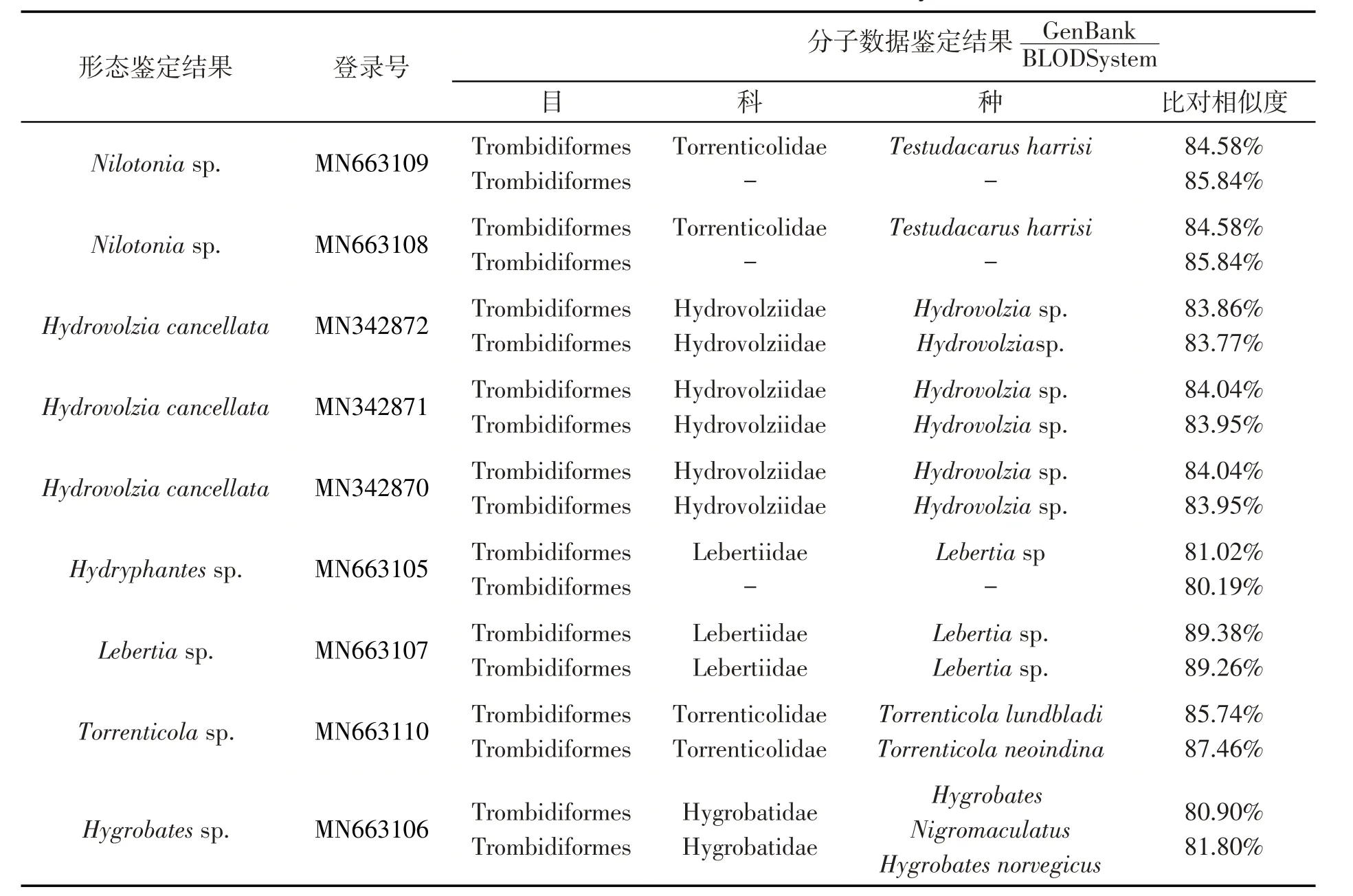
表2 基于COⅠ基因片段的9个水螨样本在GenBank 和BLOD Systems的分子鉴定结果
2.2 碱基序列分析
利用MEGA7.0 软件对20 条序列进行碱基序列分析. 结果表明T、C、A、G 的平均含量为35.8%、18.3%、30.6%、15.3%. A+T含量为66.4%,有明显的碱基偏好性. 保守位点、变异位点、简约信息位点分别为335个、323个、275个,在658个位点中分别占50.91%、49.09%、41.79%.
2.3 碱基替换饱和度分析
使用MEGA7.0软件进行碱基替换分析,核苷酸替换多为一致替换(527个),颠换数和转换数的平均比值相同,在第1、2、3位点的比值分别为0.8、1.8、1.0. 碱基置换主要发生在密码子第1个位点上,转换40个,其频率为总数的60.5%;颠换51个,其频率为总数的76.1%. 对COⅠ基因序列进行碱基替换饱和性分析时,使用DAMBE软件,以碱基替换频率为纵坐标,以TN93模型校正距离为横坐标,进行散点分析. 从饱和度分析散点图中可以看出(见图1),碱基替换颠换比率与遗传距离呈线性关系,没有出现饱和态势,能够构建系统发育树.
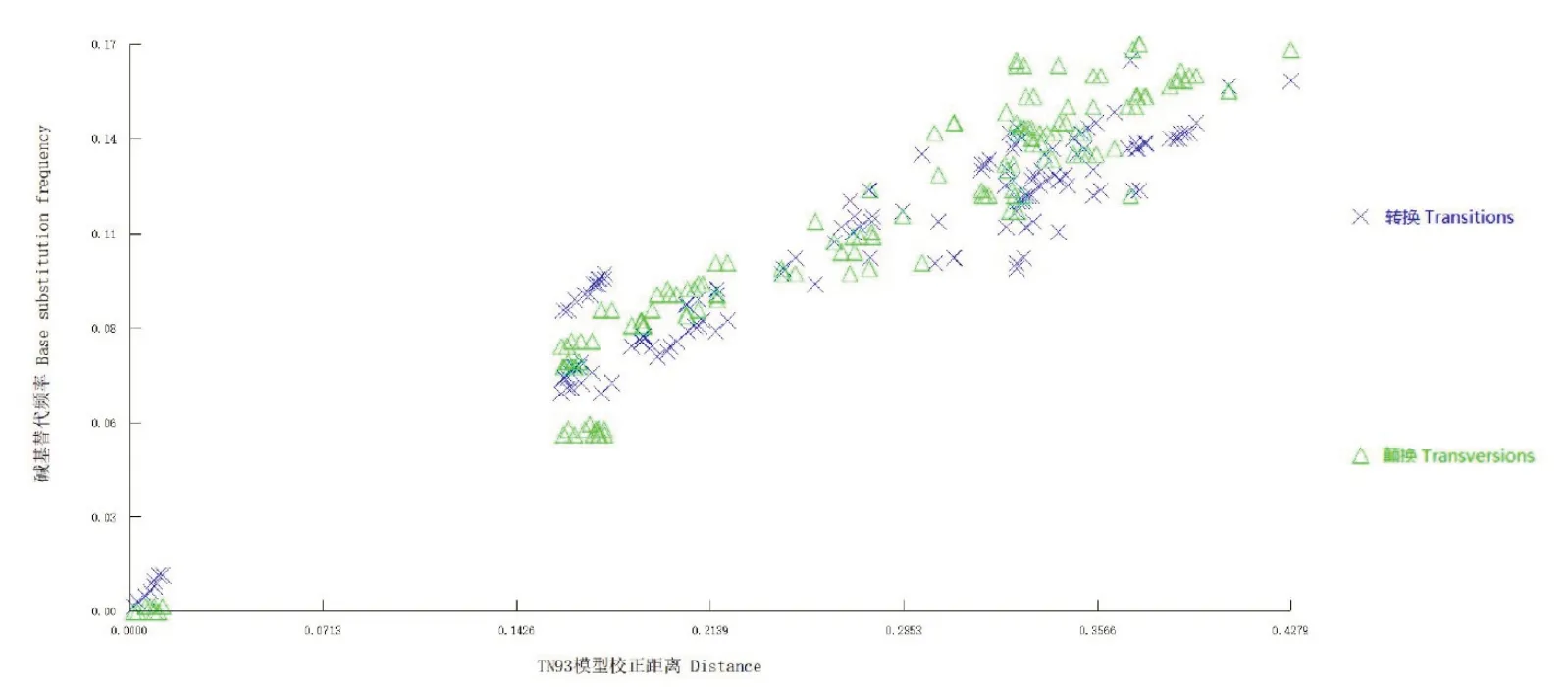
图1 饱和度分析散点图
2.4 遗传距离分析
基于MEGA7.0 软件(K2P 模型),计算遗传距离. 结果显示(见表3),种内遗传距离最大值变动范围0~0.012,其中Sperchon plumifer的种内遗传距离最大. 种间遗传距离变动范围0.158~0.365,平均种间遗传距离为0.276,Sperchon rostratus和Sperchon fuxiensis的种间遗传距离最小为0.158. 种间遗传距离最大值出现在Sperchon plumifer与Sperchonopsis ecphyma之间,为0.365. 最大种内遗传距离(0.012)小于最小种间遗传距离(0.158),存在明显的条形码间隔(见图2).
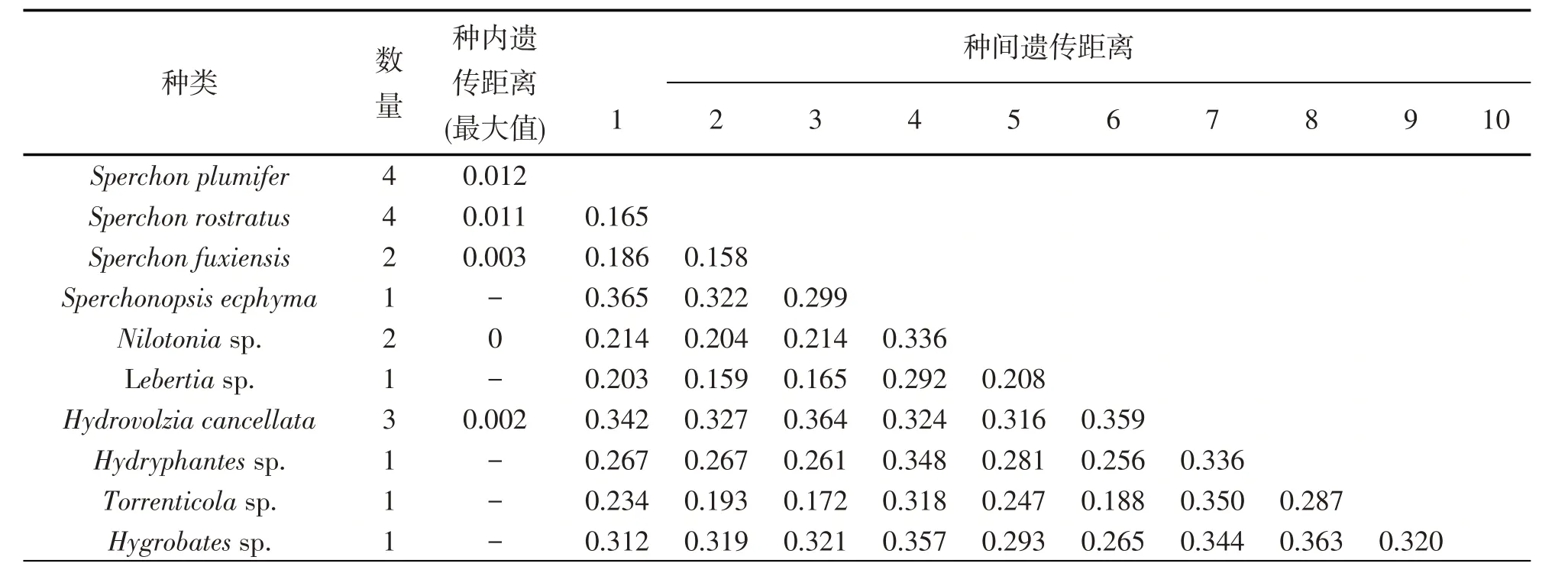
表3 基于COⅠ基因片段的10种水螨的种内与种间遗传距离

图2 遗传距离分布直方图
2.5 系统发育树的构建与分析
利用MEGA7.0软件以绒螨科Trombiidae为外群[10],构建NJ系统发育树,聚类结果如图3所示. 同一物种的所有序列均以100%的高支持率聚为一支. 例如:Sperchon plumifer的4 条序列;Sperchon rostratus的4条序列;Sperchon fuxiensis的2条序列;Nilotoniasp.的2条序列;Hydrovolzia cancellata的3条序列.
3 讨论
水螨是物种多样性十分丰富的类群(>6000种)[11],但目前在BOLD和GenBank数据库中注册的序列数量相对较少,其中GenBank数据库5451条,BOLD数据库9883条(截止到2019年10月). 根据本研究的序列比对结果,水螨的分子鉴定存在2 个方面的问题. 1)BOLD 和GenBank 中包含的物种信息比较简单,多数种类仅包含属名、科名甚至目名. 这就导致使用DNA条形码对物种进行分子鉴定时,很难比对到具体的物种名. 例如本研究中所有的样本均未能比对到具体的种名,仅有部分样本(66.67%)比对结果在属级阶元上与形态学鉴定结果一致. 2)BOLD 和GenBank 2 个数据库对水螨序列的比对相似度都偏低(BOLD 80.19%~89.26%,GenBank 80.90%~89.38%). 武宇鹏等的研究结果显示,果园螟蛾在BOLD数据库中分子鉴定的相似度在98.51%以上[7],而王嘉鹤等的研究结果显示,石斑鱼在GenBank 和BOLD 数据库中分子鉴定的相似度高达97.21%~100%[12]. 这2点均说明水螨的DNA条形码研究严重滞后,目前还未能满足使用数据库对物种进行分子鉴定的要求. 水螨的DNA条形码研究与其它动物类群相比还有很大的差距,补充水螨DNA条形码的数据量和完善数据库中的物种信息是目前水螨DNA条形码亟待开展的工作.
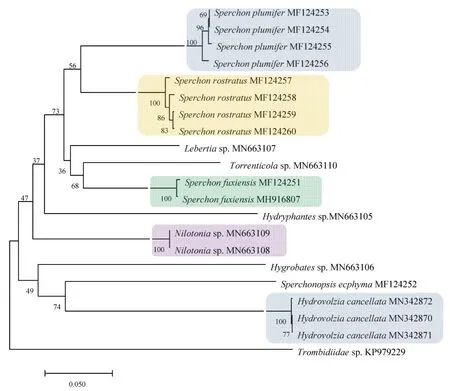
图3 基于COⅠ基因片段10种水螨的NJ系统发育树
Hebert曾提出,利用DNA条形码技术进行物种分类鉴定有2个前提条件,一:种内遗传差异小于种间遗传差异[13]. 二:物种在系统发育树中形成独立的分支[14]. 本研究的结果显示,同一水螨物种不同样本的序列差异小,种内最大遗传距离(0.012)小于种间最小遗传距离(0.158),种内、种间遗传差异未出现交叉,有显著的条形码间隔. 另外,本研究所构建的系统发育树也表明,同一水螨物种的所有序列都以100%的高支持率聚为一支. 由此可见,基于COⅠ基因完全可以满足DNA条形码理论的两个假设,证实COⅠ作为水螨类群DNA条形码基因是可行的.
值得探讨的是,Bernasconi曾在蝇类的条形码研究中证实COⅠ基因仅适合于低级阶元而不适合高级阶元的分析[15]. 本研究基于COⅠ基因所构建的系统发育树中,虽然同一水螨物种可以聚为单系,但是在属级及科级阶元上所显示的系统发育关系比较混乱. 例如Sperchon的3个物种尽管分为一簇,但它们与Lebertiasp.和Torrenttcolasp.这些不同科的物种聚为一个大支,出现同源交叉现象. 这也说明水螨类群中,COⅠ基因同样仅适用于种级阶元,而不能很好地用于高级阶元的系统发育分析.
相对于其它类群来说,水螨的DNA 条形码进展缓慢,虽然本研究在一定程度上证实COⅠ基因可以作为水螨的DNA条形码,但本次研究所涉及到的物种数较少,是否能够在所有的物种中都能得到相同的结论还需要后续的深入研究. 另外,本研究的结果也显示,COⅠ基因作为水螨类群的DNA条形码在高级阶元的分析上具有局限性,可以在后续的水螨DNA 条形码研究中开发其他基因,例如:线粒体的16S rRNA、COXⅢ以及核基因中的28S rRNA等[16-18].

