抑真菌伯克霍尔德氏菌Z1的分离鉴定及其抑菌物质分析
唐 滢,雷 平*,张翠央,毕世宇,郭照辉,2,刘清术,2
(1.湖南省微生物研究院,中国湖南 长沙 410009;2.湖南省农用微生物应用工程技术研究中心,中国湖南 长沙 410009)
植物病原真菌引起的土传病害严重影响农产品质量和数量,若防治不当将造成重大经济损失[1]。目前,化学农药仍是作物病害管理的主力军。然而,化学杀菌剂的长期使用会污染环境,引起病原菌耐药,甚至会导致作物根际有益菌群的丢失。而且,病原真菌能以孢子形式在土壤中长期存在,一次喷洒难以彻底清除。生防菌剂能在土壤中长期存活,可通过分泌抑菌物质等机制抑制病原菌生长,提高作物抗病能力,是绿色、环保的病害防治方法,能促进农业可持续发展,改善农产品质量。
植物根际中存在着一类能够促进植物根系生长和矿物质吸收、增强植物抗病性的细菌,即植物促生根际菌(plant growth promoting rhizobacteria,PGPR)[2]。芽孢杆菌属(Bacillus)、链霉菌属(Streptomyces)的许多PGPR已经被开发为商业生防菌剂,如枯草芽孢杆菌(B.subtilis QST 713,Serenade®MAX)、利迪链霉菌(S.lydicus WYEC 108,Actinovate®AG)等[3]。通常,健康作物的根际是分离PGPR的重要来源。近年来研究表明,作物经人工驯化种植后会丢失共生有益菌群[4]。例如:野生西红柿连续种植7年后,会丢失一部分共生菌且对镰刀菌和链格孢菌的易感性增强,而施用野生西红柿根际土壤后,其抗病能力得到恢复[5]。棉花、玉米等其他作物中也存在类似现象[6~7]。因此,在没有作物管理的自然环境中生长的野生植物是分离新PGPR的重要来源,但目前较少被关注。
伯克霍尔德氏菌属(Burkholderia)隶属于变形菌门、β-变形菌纲,具有丰富的代谢多样性及较强的适应能力,在环境中分布广泛,于1992年从假单胞菌属独立出来,成为新属。该属第一个成员为石竹伯克氏菌,于1942年从康乃馨中分离得到,原名石竹单胞菌,后改为石竹假单胞菌。伯克霍尔德氏菌属中的许多菌株通常在基因水平上有差异而表型没有差异,因此该属的新种鉴定工作非常复杂,难度大。研究人员将这些在基因序列上有差异而表型相似的菌株称为基因组变异型(genomovar)[8]。因此,伯克霍尔德氏菌属是一个异质性复系群,其中的许多菌株在分类学上颇具争议。随着序列分析技术的发展,研究人员对该属的多个种进行了重新分类。具有固氮、降解能力且营自由生活的一部分菌株(如B.xenovorans LB400)在2014年被移至Paraburkholderia属;生活在真菌体内且基因组非常小的菌株(如B.rhizoxinica)于2018年被移至Mycetohabitans属。截至2020年7月,该属已报道的菌株有143株,其中32株为正式有效发表的模式菌株[9]。许多伯克霍尔德氏菌都具有抑制植物病原菌生长、促进植物生长、修复土壤等功能,在农业生产和环境保护方面起着重要作用。
本研究从野生植株根际土壤取样,采用针对伯克霍尔德氏菌属的选择性培养基进行分离[10],经多次纯化后,获得伯克霍尔德氏菌Z1,随后探究了它对多种植物病原真菌的抑菌活性,初步分析了其分类地位及生理生化特征,并对其产生的抑菌物质进行了深入探讨。这些工作为靶向性筛选新PGPR开辟了新道路,而且所得菌株Z1具有的良好抑真菌活性也为将来开发新的生防菌剂奠定了基础。
1 材料与方法
1.1 材料
于2019年自湖南省长沙县青山铺镇山丘采集野生竹子根际土样。供试植物病原菌包括:水稻纹枯病菌立枯丝核菌(Rhizoctonia solani Kuhn)、香梨腐烂病原菌迂回壳囊孢菌(Cytospora ambiens)、西瓜枯萎病原菌腐皮镰刀菌(Fusariums solani)、辣椒炭疽病菌(Colletotrichum capsici)和油菜核盘菌(Sclerotinia sclerotiorum),其中,立枯丝核菌为湖南省植物保护研究所惠赠,其余均为本实验室保藏。菌株Z1已送至广东省微生物菌种保藏中心保藏,编号为GDMCC 61058。
菌株分离纯化所用培养基为针对伯克霍尔德氏菌属的选择性培养基,其配方由Haeckl等[10]在对多株伯克氏菌基因组序列进行分析后设计而成。病原真菌的培养采用PDA(potato dextrose agar)培养基;液体发酵培养采用NBY(nutrient broth yeast extract)培养基[11];平板培养采用CYMG(casitone yeast extract magnesium chloride glycerol)培养基,其配方如下:酪蛋白胨8 g,酵母提取物4 g,二水氯化镁4.06 g,50%甘油10 mL,琼脂15 g,去离子水1 L。血平板及革兰氏染色所用试剂盒均购自广东环凯微生物科技有限公司。
1.2 根际细菌的分离纯化
将5 g含有根系的根际土壤放入装有45 mL无菌Ringer洗液(7.5 g/L NaCl,0.35 g/L KCl,0.21 g/L CaCl2)的摇瓶中[12],于30℃、180 r/min振荡30 min。静置后,上清用Ringer洗液依次稀释至10-3、10-4、10-5,取200 μL涂布平板,每个梯度3个重复[13]。待出现菌落后,用接种针挑取边缘光滑的单菌落进行平板划线分离。
1.3 菌株Z1抑制植物病原真菌活性的测定
采用对峙培养法[14]检测筛选出来的细菌Z1的抑真菌活性。将在PDA平板上培养2~3 d的病原真菌用打孔器在菌丝生长外缘打若干孔,用接种针将菌丝块取出,放置在一个新的PDA平板中央,在距离中央3 cm处用接种环点接筛选出来的细菌,28℃共培养3~5 d,观察并测量抑菌圈大小。
1.4 菌株Z1的形态特征观察、全基因组测序及序列分析
将菌株分别接种于CYMG及血平板上,培养2~4 d后,肉眼观察菌落形态及周围是否出现溶血圈。取新鲜稀释菌液制备临时装片,借助蔡司相差显微镜(Zeiss,德国)观察菌株细胞形态。细菌染色采用革兰氏染色法。
取单菌落接种于NBY培养基中,培养2 d后收集菌体,无菌蒸馏水洗涤1次后再次悬浮,取1 μL作为16S rRNA基因的PCR扩增模板。PCR扩增体系(30 μL):PrimeSTAR®Max(TaKaRa公司,日本)15 μL、引物 27F/1492R(10 μmol/L)各 1 μL、模板 1 μL、ddH2O 12 μL。扩增条件:98 ℃ 5 min;94℃15 s,55℃15 s,72℃35 s,35个循环;72℃10 min。采用DNA胶回收试剂盒(上海捷瑞生物工程有限公司)回收目标PCR产物,并将其送至湖南擎科生物技术有限公司测序。从Z1的全基因组序列中调取recA基因序列。将所得recA及16S rRNA基因序列分别与EzBioCloud、NCBI数据库中相应序列进行比对[15],经Clustal Omega多序列比对分析[16]后,利用软件Mega 7.0构建系统进化树[17]。Z1的16S rRNA基因及recA基因的序列均已提交至GenBank,其序列登录号分别为MT804577和 MT798856。
用NBY培养基培养Z1,离心收集菌体,用TEN缓冲液(0.01 mol/L Tris-Cl,pH 8.0;0.001 mol/L EDTA,pH 8.0;0.1 mol/L NaCl)洗涤一遍后,离心并弃去上清,将菌体寄至武汉贝纳科技服务有限公司,采用Nanopore PromethION及Illumina Nova-Seq 6000进行全基因组测序。从JGI(Joint Genome Institute,https://img.jgi.doe.gov/)和NCBI(https://www.ncbi.nlm.nih.gov/)下载同源菌株的全基因组序列,用在线工具Genome-to-Genome Distance Calculator(http://ggdc.dsmz.de/ggdc.php)计算Z1与近缘菌株的DNA-DNA杂交值(digital DNA-DNA hybridization,dDDH)[18]。采用在线平均核苷酸一致性(Average Nucleotide Identity,ANI)计算工具(https://www.ezbiocloud.net/tools/ani)算得 ANI 值[19]。采用GCK 3.0(Gene Construction Kit 3.0)进行在线限制性片段长度多态性(restriction fragment length polymorphism,RFLP)分析。采用antiSMASH bacterial version(https://antismash.secondarymetabolites.org)预测Z1基因组中的次级代谢产物生物合成基因簇[20]。Z1的基因组序列及相关信息已提交至NCBI,其BioProject、BioSample及基因组序列登录号分别为 PRJNA649092、SAMN15658474及 JACFYL-000000000。
1.5 菌株Z1的生理生化特征分析
用法国梅里埃公司(bioMérieux)的试纸条API 20NE、API ID 32GN、API 50CH 及 API ZYM 检测Z1的生理生化特征。其中,API 20NE用于检测硝酸盐还原、七叶苷柠檬酸铁与凝胶水解及菌株对部分碳源的利用情况;API ZYM试纸条用于酶活测定;API 50CH试纸条用来检测菌株的发酵产酸情况;API ID 32GN用来检测菌株对32种碳水化合物的利用情况。各项操作均按照试纸条说明书进行。
1.6 代谢产物的提取及高分辨质谱分析
采用NBY培养基进行发酵,接种量为5%[11]。30℃培养72 h后,添加大孔树脂XAD-16(Amberlite®,美国),继续培养24 h后,离心收集菌体及XAD-16,弃上清,加入等体积甲醇浸提30 min,旋转蒸发仪浓缩后溶于1 mL甲醇中。
发酵液提取物经最高转速离心10 min后过滤,寄送至山东大学微生物技术国家重点实验室进行高分辨质谱(high resolution mass spectrum,HRMS)分析。所用仪器为Bruker Impact HD microTOF QⅢ质谱仪(Bruker Daltonics,德国),采用标准的电喷雾离子源。色谱柱为Thermo Scientific Acclaim C18 column(2.1 mm × 100 mm,2.2 μm)。流动相为含有0.1%甲酸的水及乙腈。
2 结果与分析
2.1 伯克氏菌Z1的分离及其抑真菌活性
从野生禾本科植物根际中分离到一株对多种植物病原菌均有较强抑制效果的细菌,命名为Z1。其对香梨腐烂病原菌迂回壳囊孢菌(C.ambiens)、西瓜枯萎病原菌腐皮镰刀菌(F.solani)、辣椒炭疽病菌(C.capsici)、油菜核盘菌(S.sclerotiorum)及水稻纹枯病菌立枯丝核菌(R.solani Kuhn)的抑菌圈直径分别 14 mm、4 mm、12 mm、12 mm 和 10 mm,显示出较广的抑菌谱(图1)。
2.2 菌株Z1的形态特征
菌株Z1在CYMG平板上可形成直径1~2 mm的菌落,圆形,黄白色,表面光滑,边缘整齐,不透明,不产生荧光,中间凸起不明显,培养48 h后可产生红色色素(图2A),但随着培养时间的延长该色素又会自行消失。这一现象在伯克霍尔德氏菌中很常见,具体机制至今不明。相差显微镜下菌体为杆状(图2B),革兰氏染色呈阴性。在血平板上培养48 h后,菌株Z1能产生明显的溶血圈(图2C),且培养时间越长,溶血圈越大。

图1 菌株Z1对5种植物病原真菌的抑菌活性(A)香梨腐烂病原菌迂回壳囊孢菌;(B)西瓜枯萎病原菌腐皮镰刀菌;(C)辣椒炭疽病菌;(D)油菜核盘菌;(E)水稻纹枯病菌立枯丝核菌。Fig.1 The inhibitory activity of strain Z1 against five phytopathogenic fungi(A)C.ambiens,the causative agent of fragrant pear canker;(B)F.solani,the causative agent of cucurbits fusarium wilt;(C)C.capsici;(D)S.sclerotiorum;(E)R.solani Kuhn,the causative agent of rice sheath blight.

图2 菌株Z1的菌落、菌体形态及溶血圈(A)CYMG平板上的菌落形态;(B)相差显微镜下的菌体特征(2 000×);(C)菌株培养48 h后,在血平板上出现明显的溶血圈。Fig.2 The colony,cell morphology and the hemolytic zone of Z1(A)Colony of Z1 on CYMG plate;(B)Z1 under phase contrast microscope(2 000×);(C)The hemolytic zone produced by Z1 on sheep blood agar after 48 h incubation.
2.3 菌株Z1的基因型特征
16S rDNA序列同源性是进行菌种分类的重要指标。大多数情况下,98.7%的16S rDNA序列同源性为判定新种的分界点[21]。但亦有超过该值的细菌被鉴定为新种,如2013年在International Journal of Systematic and Evolutionary Microbiology上发表的微杆菌属新种Microbacterium saccharophilum,其与同属菌株的最高同源性达99.87%[22]。以菌株Z1的菌液为模板,用引物27F/1492R成功扩增到了1.5 kb左右的条带(图3),测序后其大小为1 355 bp。在EzBioCloud数据库中比对该序列,得知其与Burkholderia contaminans LMG23-361的亲缘关系最近,同源性达99.56%。选取邻近17个菌株的同源序列构建进化树(图4),从图中可知,Z1与B.contaminans LMG23361的进化距离很近。
当16S rDNA序列同源性高于97%时,需要测定同源菌株间的dDDH值,该值大于70%为同种,低于70%为不同种[21]。由表1可知,Z1与其近缘菌株B.contaminans LMG23361、B.contaminans MS14、B.pyrrocinia Lyc2、B.pyrrocinia DSM10685及B.cepacia ATCC25416的dDDH值在44.50%~52.70%,低于阈值70%,说明菌株Z1与这5株近缘菌株的差异达到种间水平,可作为判定新种的依据。

图3 菌株Z1的16S rDNA扩增电泳检测图M:分子量标准DL5000;Z1:菌株Z1的16S rDNA扩增产物。Fig.3 Electrophoresis analysis of PCR products of the gene encoding 16S rRNA of strain Z1M:DL5 000 marker;Z1:The 16S rDNA PCR products of Z1.
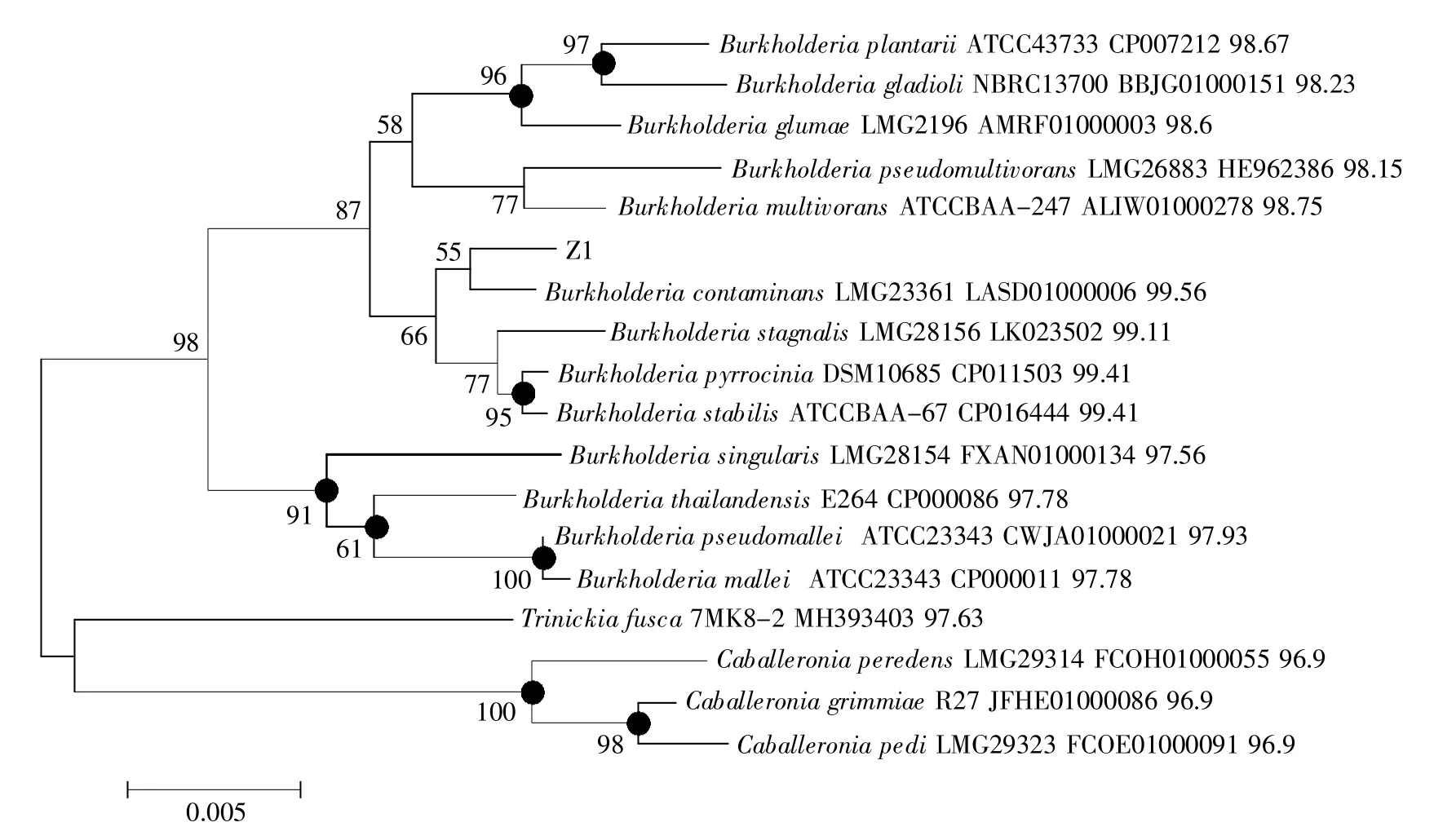
图4 菌株Z1的16S rDNA系统进化树(邻接法)图中显示了菌株Z1及17个来自EzBioCloud数据库的同源菌株的分类地位。替换模型为Kimura 2-parameter;分支处的数值为大于50%的步长值(重复1 000次)。黑点代表该分支与用最大可能性法构建时结果一致。菌种拉丁名后依次为菌株编号、序列登录号及与Z1的同源性。Fig.4 Neighbor-joining phylogenetic tree based on 16S rRNA gene sequence of strain Z1The relationships between strain Z1 and members of the genus Burkholderia are shown.Substitution model is Kimura 2-parameter.Bootstrap values(expressed as percentages of 1 000 replications)above 50%are shown at the branch points.Black dots denote branches recovered using maximum likelihood method.The strain number,sequence accession number and similarity are presented in order after the strain name.
ANI值指两个微生物基因组同源片段之间的平均碱基相似度,在近缘物种之间有较高的区分度。一项基于9万个基因组的分析表明,不同物种间的基因组ANI小于95%,同一物种的基因组ANI大于95%[23],因此可以95%的ANI为物种划分与物种聚类的标准。由表1可知,Z1与上述5个近缘物种的ANI值在91.54%~93.52%,均低于95%,虽达到判定新种的标准,但数值非常接近95%,故仍需更多数据才能确定其是否为新种。
由于伯克霍尔德氏菌的16S rDNA序列过于保守,为获得更准确的分类信息,我们对Z1与其同源菌株的recA基因序列同源性进行了分析。基因recA的序列已被证实能在伯克霍尔德氏菌属内区分70多个种,具有很好的鉴别功能[24]。在NCBI数据库中,与Z1的recA基因同源性最高的菌株为 B.pyrrocinia DSM10685,同源性 97.75%(表1)。从基于recA基因序列构建的进化树上亦可看出,Z1与B.pyrrocinia DSM10685的亲缘关系较近,但是二者位于不同分支,分支处的步长值(bootstrap value)为 82,可信度高(图 5)。
洋葱伯克氏菌群(Burkholderia cepacia complex,Bcc)包含多个表型相似、基因型不同的菌株,目前已鉴定出9个基因组变异型(genomovarⅠ~Ⅸ),每个基因组变异型又包含多个表型相似、基因型不同的菌株。其中,基因组变异型Ⅲ(B.cenocepacia)和基因组变异型Ⅱ (B.multivorans)为引起囊性纤维化的主要病原菌[25];基因组变异型Ⅸ的种名为B.pyrrocinia。Bcc中不同种间的recA基因同源性在94%~95%,同种相似度在98%~99%[25]。从表1可知,Z1的recA基因与B.pyrrocinia DSM-10685的同源性为97.75%,低于界定同种的阈值,但是比较接近。采用内切酶HaeⅢ对这两段序列进行RFLP分析,Z1的recA基因完全酶切消化后得到的DNA片段长度分别为144 bp、546 bp、583 bp、772 bp、843 bp、864 bp、876 bp、940 bp 和1 009 bp,而B.pyrrocinia DSM10685所得片段长度为 144 bp、538 bp、546 bp、583 bp、772 bp、864 bp、876 bp、940 bp和1 009 bp,两者仅有一个酶切位点的差异,因此我们认为Z1应为基因组变异型Ⅸ的一个新成员。
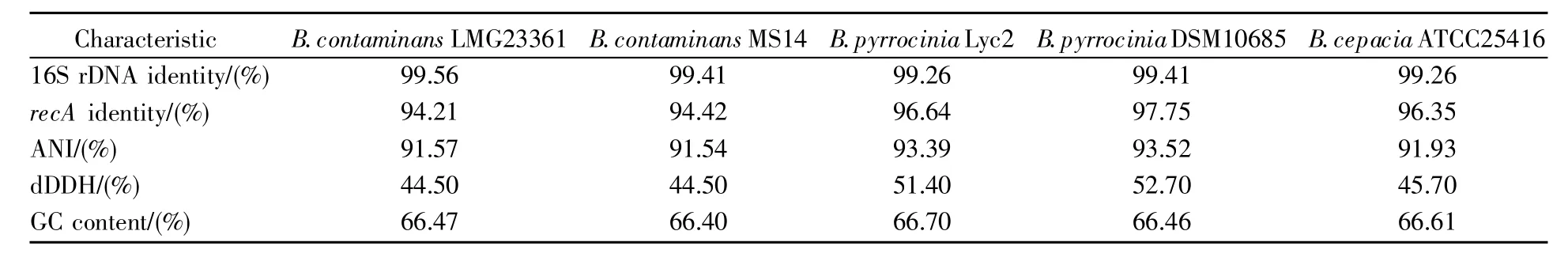
表1 菌株Z1与同属近缘菌株间的16S rDNA同源性、recA基因同源性、ANI值、dDDH值及GC含量比较Table 1 Comparison of the 16S rDNA and recA gene identity,ANI,dDDH,and GC content between Z1 and the closely related species of the genus Burkholderia

图5 基于recA基因序列分析构建的进化树Fig.5 Phylogenetic tree based on gene recA sequence analysis
2.4 菌株Z1的生理生化特征
为全面了解菌株Z1对碳源的利用情况及其酶活等各项生理生化特征,我们用梅里埃试纸条对Z1进行了测定。经试纸条测定,菌株Z1能将硝酸盐还原成亚硝酸盐,能水解七叶苷柠檬酸铁、凝胶及4-硝基苯-β-D-吡喃半乳糖苷,不能产生色氨酸酶、脲酶、精氨酸双水解酶、缬氨酸芳胺酶、胱氨酸芳胺酶、胰蛋白酶、胰凝乳蛋白酶、α-半乳糖苷酶、β-半乳糖苷酶、β-糖醛酸苷酶、α-葡萄糖苷酶、β-葡萄糖苷酶、N-乙酰-葡萄糖胺酶、α-甘露糖苷酶、β-岩藻糖苷酶,能产生碱性磷酸酶、酯酶(C4)、类脂酯酶(C8)、类脂酶(C14)、亮氨酸芳胺酶、酸性磷酸酶、苯酚-AS-BI-磷酸水解酶,能发酵阿拉伯糖、木糖、D-侧金盏花醇、半乳糖、葡萄糖、乳糖、纤维二糖、麦芽糖、岩藻糖产酸,能同化葡萄糖、葡萄糖酸钾、N-乙酰葡萄糖胺、阿拉伯糖、甘露糖、甘露醇、乙酰葡萄糖胺、麦芽糖、羊蜡酸、己二酸、苹果酸、柠檬酸钠、苯乙酸、D-核糖、肌醇、蔗糖、辛二酸盐、L-丙氨酸、5-酮基-葡萄糖酸盐、L-丝氨酸、D-蜜二糖、L-岩藻糖、D-山梨醇、组氨酸、2-酮基-葡萄糖酸盐、L-脯氨酸。经与文献中同源菌株相应数据[26~28]比较,发现菌株Z1的大部分生理生化特征与吡咯伯克霍尔德氏菌一致,仅个别指标存在差异(表2),这支持了其为基因组变异型Ⅸ中一员的结论。
2.5 基因组分析
伯克霍尔德氏菌生活范围广,适应能力强,代谢种类多样,因而其基因组也较大,平均约7.5 Mb,大小位列细菌的前5%。伯克霍尔德氏菌的大多数种都含有两条染色体,0~6个质粒。生活在真菌体内的B.rhizoxinica(现已更名为M.rhizoxinica)例外,只含有1条染色体,其基因组大小为3.75 Mb。不同于链霉菌等其他细菌,大多数伯克霍尔德氏菌的必需基因与非必需基因分别位于两条不同的染色体上[29]。
经全基因组测序及拼接获得菌株Z1的基因组序列,其含有两条染色体(3.7 Mb和3.5 Mb)及1个大质粒(1.2 Mb),总长8.4 M,GC含量66.28%。经在线次级代谢产物生物合成基因簇预测软件antiSMASH分析,我们发现其基因组中含有萜类(terpene)、细菌素(bacteriocin)、非核糖体肽(nonribosomal peptide,NRP)等15个次级代谢产物的生物合成基因簇,其中一个长约94 kb的基因簇与occidiofungin的生物合成基因簇相似度达88%。
2.6 菌株Z1的抑真菌物质分析
伯克霍尔德氏菌产生的抑真菌物质种类丰富,包括 burkholdines[30]、occidiofungins[31]及 pyrrolnitrin[32]等。其中,burkholdines和occidiofungins属于环脂肽,能够裂解血细胞,且两者结构非常相似,在骨架上只有一个氨基酸不同(图6)。Occidiofungins由1个β-羟基天冬酰胺(β-hydroxy asparagine,BHN)、1 个 β-羟基酪氨酸(β-hydroxy tyrosine,BHY)、两个丝氨酸、1个甘氨酸、1个天冬酰胺、1个2,4-二氨基丁酸(2,4-diamino butyric acid,DABA)及1个来源于脂肪酸途径的非蛋白氨基酸脱水缩合而成。而burkholdines的骨架中没有DABA,取而代之的是天冬酰胺。两者均含有1个由11个碳原子组成的直链烷烃及1个木糖。
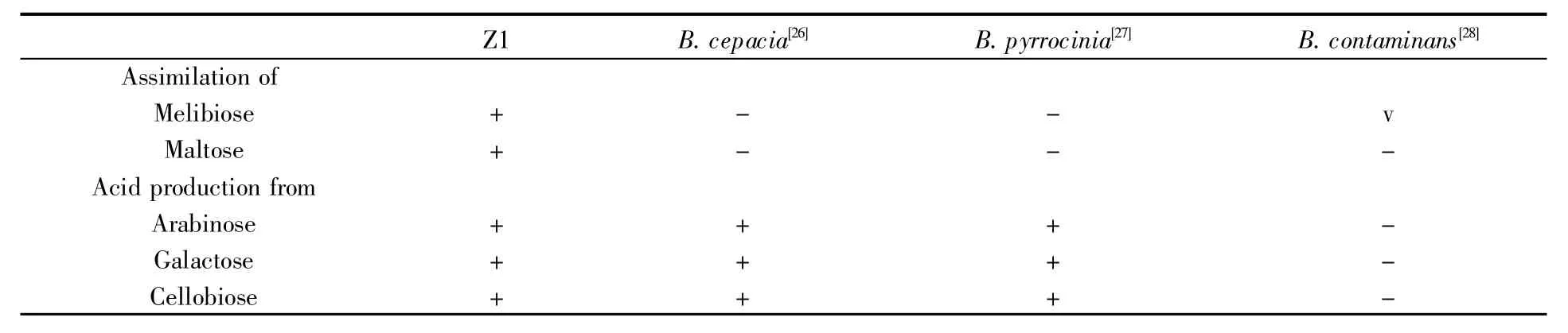
表2 菌株Z1与同属近缘菌株的表型差异特征Table 2 Differential phenotype characteristics of strain Z1 and strains of closely related species of the genus Burkholderia
菌株Z1能抑制多种植物病原真菌及白色念珠菌,其基因组中含有与抑真菌环脂肽occidiofungin生物合成基因簇高度同源的基因簇。为进一步探讨Z1抑真菌活性物质的成分,我们对该菌的发酵液进行了高分辨质谱分析。从质谱结果中可找到质荷比为1 216.588 3[M+H]+(calcd for C52H86N11O22,1 216.594 3,4.9×10-6)的峰,该值与occidiofungin B的相对分子质量非常接近;除此之外,亦能找到质荷比为1 214.571 4[M+H]+(calcd for C52H84N11O22,1 214.578 7,6.0×10-6)及 1 230.564 1[M+H]+(calcd for C52H84N11O23,1 230.573 6,7.7×10-6)的峰,这与 burkholdine-1213和 burkholdine-1229的相对分子质量也非常接近。对于1 215.556 5[M+H]+、1 083.515 3[M+H]+、1 231.550 1[M+H]+和1 099.507 4[M+H]+,目前尚未在已报道文献中找到与其相对分子质量一致的化合物,但是它们的二级质谱碎片离子与文献报道的burkholdine-1097的许多碎片离子[33]相同,且两两相差132 Da,与丢失木糖的片段吻合,木糖的相对分子质量为150 Da(图7),因此我们推测其可能为burkholdine的新衍生物。

图6 Occidiofungin B、burkholdine-1213及burkholdine-1229的结构、分子式和相对分子质量红色部分为两种环脂肽在骨架上的不同之处。Fig.6 The structure,chemical formula and monoisotopic mass of occidiofungin B,burkholdine-1213 and burkholdine-1229The red color indicates the difference between the backbones of occidiofungins and burkholdines.
据报道,某些能产生occidiofungins或burkholdines的伯克氏菌株可在血平板上产生溶血圈[34]。菌株Z1在血平板上培养48 h后,菌落周围亦出现了非常明显的溶血圈(图2C)。结合高分辨质谱分析结果,我们推测菌株Z1产生的抑真菌物质很可能为 occidiofungin B、burkholdine-1213、burkholdine-1229及burkholdine的新衍生物。
3 讨论
伯克霍尔德氏菌属的细菌能以自由生活或寄生的方式广泛存在于陆地、水域等多种环境中。许多伯克霍尔德氏菌均具有生物防治的功能,例如:B.rinojensis能抑杀节肢动物,可开发为杀虫剂[29];B.contaminans等菌株能合成多种抗真菌物质。有些伯克霍尔德氏菌能促进植物的生长,在农业生产中可将其用作生长调节剂或复合肥料,如具有固氮功能的B.ambifaria和B.caribensis。另外,有的伯克霍尔德氏菌还可以分解土壤中残留的农药、净化污水[29]。20世纪90年代,美国环保局批准用于多种植物病原真菌防治的菌剂Deny®、Blue Circle®及Intercept®等的主要成分均为伯克霍尔德氏菌[35],在我国亦有类似产品,如“亚宝”。此外,也有一些伯克霍尔德氏菌株是动植物病原菌。
本研究采用选择性培养基从野生竹子根际靶向性筛得伯克霍尔德氏菌Z1,其具有广谱抑真菌活性(图1)。与其16S rRNA基因及recA基因序列同源性最高的菌株分别为B.contaminans LMG-23361 和 B.pyrrocinia DSM10685(表 1,图 4~5)。两者都是Bcc成员,其基因组中均含有大量抑菌活性天然产物的生物合成基因簇,但不含有主要致病因子[36]。这将大幅降低大规模应用时的安全风险。虽然Z1与多株近缘菌株间的dDDH值及ANI值均低于新种鉴定阈值,达到界定新种的标准(表1),但有差异的生理生化指标不多(表2)。鉴于伯克霍尔德氏菌属新种鉴定的复杂性,本论文不敢贸然将该菌认定为新种。该菌与近缘菌株间确实存在差异。Bcc菌群目前已有9个基因组变异型,每个基因组变异型又包含多个菌株,它们的形态特征非常相似,但基因组水平上的差异达到新种鉴定标准[8]。综合本文对Z1及其近缘菌株的基因组序列分析及生理生化特征的测定结果,我们认为菌株Z1极可能是Bcc基因组变异型Ⅸ(B.pyrrocinia)中的一员。

图7 菌株Z1代谢产物的高分辨质谱分析质荷比分别为 1 230.564 1[M+H]+(A)、1 231.550 1[M+H]+(B)、1 099.507 4[M+H]+(C)、1 214.571 4[M+H]+(D)、1 215.556 5[M+H]+(E)及1 083.515 3[M+H]+(F)的衍生物的母离子的二级质谱图。两个或多个衍生物共有的碎片离子用红色标出,衍生物A与B、D与E之间相差1 Da的碎片离子用蓝色表示。质荷比为547.332 9[M+H]+、718.393 7[M+H]+和832.437 7[M+H]+的碎片离子与文献[33]报道一致,其中832.437 7[M+H]+为burkholdines特有的碎片离子。Fig.7 The HRMS analysis of the metabolites of Z1Shown here are the MS2 spectrum for the masses with a m/z of 1 230.564 1[M+H]+(A),1 231.550 1[M+H]+(B),1 099.507 4[M+H]+(C),1 214.571 4[M+H]+(D),1 215.556 5[M+H]+(E)and 1 083.515 3[M+H]+(F)respectively.The numbers in red color indicate the MS2 fragments shared by two or more different derivatives,while the blue numbers highlight fragments with an increase of 1 Da compared with A and B,D and E,separately.The mass fragments with m/z of 547.332 9[M+H]+,718.393 7[M+H]+and 832.437 7[M+H]+are consistent with the ones in former publications,while the 832.437 7[M+H]+is unique for burkholdines.
结合抑菌活性、高分辨质谱、血平板试验及基因组序列分析结果,我们认为菌株Z1产生的抑真菌活性物质应为环脂肽occidiofungins和burkholdines。尽管Z1基因组中仅存在与occidiofungin生物合成基因簇高度相似的基因簇(相似度88%),但这两种环脂肽结构类似,考虑到腺苷酰化结构域(adenylylation domain)宽松的底物特异性,这在生物合成途径上是完全可行的。目前已报道的能产生occidiofungins的伯克霍尔德氏菌有B.pyrrocinia Lyc2[37]、B.contaminans MS14[31];能产生burkholdines的菌有B.ambifaria 2.2N[33]。虽然两种环脂肽结构非常相似,但既能合成occidiofungins又能合成burkholdines的菌株在国内外尚没有文献报道,且Z1还能合成burkholdine的新衍生物(图7),这为研制具有自主知识产权的新生防菌剂奠定了基础。