陆地棉盐胁迫应答基因GhPEAMT1的克隆及功能分析
王娜,赵资博,高琼,何守朴,马晨辉,彭振,,杜雄明,
陆地棉盐胁迫应答基因的克隆及功能分析
王娜1,赵资博2,高琼1,何守朴1,马晨辉1,彭振1,2,杜雄明1,2
1中国农业科学院棉花研究所/棉花生物学国家重点实验室,河南安阳 455000;2郑州大学农学院/棉花生物学国家重点实验室郑州基地,郑州 450001
【】磷酸乙醇胺N-甲基转移酶(phosphoethanolamine N-methyltransferse,PEAMT)是植物磷酸胆碱合成的关键酶,而磷酸胆碱是可以增强植物抗性的甘氨酸甜菜碱合成底物胆碱的前体。通过对陆地棉磷酸乙醇胺N-甲基转移酶基因()的克隆、表达模式分析,以及功能验证,探究在陆地棉响应盐胁迫中的生物学功能,为棉花耐盐品种的选育提供基因资源。根据转录组测序数据分析,最终确定为耐盐候选基因;通过聚合酶链式反应(PCR)扩增目的基因;通过生物信息学分析基因结构特征、预测蛋白质相对分子质量以及进化关系;利用荧光定量PCR(qRT-PCR)分析耐盐棉花品种早熟长绒7号和感盐棉花品种南丹巴地大花在NaCl胁迫后不同时间点不同组织的表达特征;构建亚细胞定位载体,进行烟草的瞬时转化,确定蛋白质在细胞中的位置;构建基因超表达载体,通过花序浸染法转化拟南芥,分析转基因拟南芥种子在盐胁迫下的萌发率和转基因拟南芥主根长度;利用病毒诱导基因沉默技术(virus induced gene silencing,VIGS)对早熟长绒7号棉花品种进行目的基因沉默,提取棉花叶片的RNA进行实时荧光定量用以检测基因沉默效率,随后用40 g·L-1的NaCl溶液的处理对照棉花植株(CK)、注射TRV2:00的棉花植株、和沉默成功的棉花植株,测定棉花叶片过氧化氢酶(catalase,CAT)、谷胱甘肽过氧化物酶(glutathione peroxidase,GPX)的活性。克隆获得的2个同源基因和,2个基因的CDS全长分别为1 488和1 485 bp;构建进化树发现,与海岛棉同源基因的亲缘关系最近,而与其他物种相比,与可可同源性最高;盐胁迫条件下,和在耐盐品种早熟长绒7号叶片和根中的表达量显著高于感盐品种南丹巴地大花。亚细胞定位显示该基因位于细胞质中;超表达转基因拟南芥后,在100和150 mmol·L-1NaCl的MS培养基上,转基因拟南芥的种子萌发率显著高于野生型拟南芥。在50和100 mmol·L-1NaCl的MS培养基上,转基因拟南芥的主根长极显著高于野生型拟南芥的主根长;用40 g·L-1的NaCl溶液处理24 h后,和沉默成功的棉花植株叶片比CK和注射TRV2:00的棉花植株的棉花叶片萎蔫严重,盐胁迫后,与注射空载棉花植株相比,注射TRV2::GhPEAMT1A棉花植株过氧化氢酶(CAT)活性降低、谷胱甘肽过氧化物酶(GPX)活性降低。可以增强拟南芥对盐胁迫的耐性,能够响应盐胁迫并起正向调控作用。
陆地棉;盐胁迫;;基因克隆;基因沉默
0 引言
【研究意义】棉花作为世界上最重要的天然纤维作物和最重要的经济作物,既是纺织工业的主要原料,也是国防、医药、化工等工业部门的原料[1-2]。土壤盐渍化是全球面临的重大问题,由于人类的活动,农作物的生长受到了土壤盐渍条件的限制,严重降低了农作物的产量,非生物胁迫每年造成数亿美元的损失,严重地威胁着农业的可持续性发展[3-5]。棉花是中度耐盐作物,它的盐容忍阈值为7.7 ds·S-1[6-7]。虽然棉花是耐盐作物,但是盐碱地高浓度的盐分依然制约着棉花的产量[8]。因此,解析植物盐胁迫的响应机制可有助于提高作物的耐盐性[7]。【前人研究进展】棉花渗透调节盐胁迫条件下,外界渗透势降低容易造成根系细胞脱水,失去膨压,进而叶片萎蔫影响棉花的生理功能。因此,棉花的耐盐机理之一,是在盐胁迫条件下能对渗透胁迫进行调节[3]。棉花渗透调节有2种方式:一种方式是吸收大量的无机盐离子积累到液泡中,提高浓度,降低水势,使其能在较高的渗透条件下吸水,保持膨压,正常生长,但这种耐胁迫能力有限,毕竟Na+在胞质积累过度会造成离子毒害;另一种方式主要依靠棉花体内合成一些小分子有机物质如脯氨酸、甜菜碱、甘油、甘露醇、海藻糖等来提高细胞的渗透压,降低水势。防止细胞脱水和保持细胞各种酶在高盐环境下的功能[9]。目前,已知的有机渗透调节物质中,甜菜碱是最重要的一种。甜菜碱是一种水溶性生物碱,是甘氨酸甜菜碱(glycinebetaine,GB)的简称,广泛存在于植物中,它的积累可以增强植物的抗性[10-11]。此外甜菜碱合成后几乎不再被进一步代谢,属于永久性或半永久性渗透调节剂,因此,甜菜碱被认为是最有希望的渗透保护剂,在植物抗盐、耐旱研究中已越来越受到重视。陈少良等[12]研究发现,生长于中国西北盐碱干旱地区的胡杨,在100 mmol·L-1NaCl处理下叶中甜菜碱浓度增加243倍,根中增加9倍。在高等植物中,合成甘氨酸甜菜碱(GB)的底物是胆碱(choline,Cho),磷酸胆碱(phosphocholine,P-Cho)在磷酸胆碱磷酸酶的水解作用下可生成胆碱(Cho)和磷脂酸(phos- phatidic acid,PA)[13]。磷酸胆碱(P-Cho)是由磷酸乙醇胺N-甲基转移酶(phosphoethanolamine N-methyltransferse,PEAMT)通过连续3次甲基化生成,PEAMT的连续3次的催化被认为是磷酸胆碱(P-Cho)合成中的限速步骤[14]。研究发现在高等植物中胆碱(Cho)的合成速度受到严格控制,而磷酸乙醇胺N-甲基转移酶在甜菜碱的底物胆碱的合成中起到重要作用[15-16],因此,PEAMT在植物的耐盐胁迫调控中具有重要的作用。【本研究切入点】近年来,研究表明磷酸乙醇胺N-甲基转移酶(PEAMT)促进GlyBet的合成,有助于提高植物抗盐的能力。然而目前棉花中PEAMT基因的研究较少。基于PENG等[17]发表的转录组和蛋白组数据,发现无论在mRNA水平还是蛋白质水平均表达量较高且在耐盐性不同材料差异显著。因此,研究的功能,进一步解析它在棉花中的耐盐机制。【拟解决的关键问题】本研究通过克隆磷酸乙醇胺N-甲基转移酶基因(和)cDNA全长,并对核酸序列和蛋白质序列进行生物信息学分析,以及亚细胞定位载体、基因超表达载体和基因沉默载体的构建,进而对其生物学功能进行验证,确定的功能,为解析在棉花盐胁迫应答中的调控机制提供科学依据,同时也可为棉花耐盐品种的选育提供基因资源。
1 材料与方法
1.1 试验材料
试验材料为陆地棉品种早熟长绒7号(耐盐)和南丹巴地大花(感盐)。这些材料的种子均由中国农业科学院棉花研究所国家棉花种质资源中期库提供,并经过多代自交,且性状稳定。拟南芥哥伦比亚野生型(Col-0)和本氏烟草由中国农业科学院棉花研究所种质资源课题组提供。
1.2 总RNA的提取和cDNA的制备
选取陆地棉耐盐品种早熟长绒7号和感盐品种南丹巴地大花。通过霍格兰营养液培养,在苗期三叶一心期用200 mmol·L-1NaCl溶液处理,分别在处理前(0 h)和处理后0.5、4、12、24和72 h取棉花的叶片和根,3个生长一致的棉花植株混合取样,-80℃保存备用。采用天根生化科技(北京)有限公司的多酚多糖植物总RNA提取试剂盒(NO.DP441),提取经过处理后的根和叶中的RNA。用TaKaRa公司的PrimeScriptTMRT Reagent Kit with gDNA Eraser(Perfect real time)试剂盒(NO.RR047A),将提取的RNA反转录为cDNA,用于后续试验。
1.3 GhPEAMT1的克隆
根据(Gh_A03G080)和(Gh_D02G1225)的基因序列,利用primer premier 5软件设计特异引物(表1)。利用TOYOBO的KOD-Plus-Neo高保真酶进行PCR扩增,得到基因片段。纯化后,连接到Blunt-zero载体上,并转化Trans1-T1大肠杆菌感受态中。筛选阳性单克隆,送深圳华大基因科技有限公司测序,获得正确的和片段。吸取测序正确的菌液20 μl加入含有20 mL LB(Kan+)液体培养基中,37℃过夜培养,用北京全式金生物技术有限公司的质粒小提试剂盒提取质粒。
1.4 生物信息学分析
用Protparam(https://web.expasy.org/protparam/)分析GhPEAMT1氨基酸的理化性质;用CDD(https:// www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)在线软件预测蛋白质的保守结构域;用Signal(http://www.cbs. dtu.dk/services/SignalP/)进行跨膜结构的分析;用CELLO V.2.5(http://cello.life.nctu.edu.tw/)在线软件对蛋白质进行亚细胞定位预测;用DNAMAN软件进行基因序列比对;运用MEGA7软件对陆地棉基因及其他物种同源基因构建进化树。
1.5 GhPEAMT1的表达模式分析
为了研究不同组织中的表达模式,使用Primer Premier 5设计特异引物(表1)。使用LightCycler480Ⅱ(Roche,德国)对1.2方法获得的NaCl胁迫的不同时期cDNA样本进行qRT-PCR试验。按照北京全式金生物技术有限公司的荧光定量试剂盒(TranStart Top Green qPCR SuperMix,AQ131)说明书确定qRT-PCR的反应体系和反应程序。内参基因为(AY305733),每个反应设置3个重复。采用2-ΔΔct计算基因的相对表达量。
1.6 GhPEAMT1亚细胞定位载体的构建
分别以CPEAMT1A-F、CPEAMT1A-R和CPEAMT1D-F、CPEAMT1D-R为引物(表1)和1.3中提取的质粒为模板进行PCR扩增。用限制性内切酶Ⅰ37℃酶切,按照NEB的小牛肠碱性磷酸酶(CIP,#M0290S)的步骤对酶切产物进行碱性磷酸化,回收,16℃过夜连接到表达载体P438-GFP中。将构建好的表达载体通过冻融法转入农杆菌GV3101中,得到亚细胞定位载体P438-GFP-GhPEAMT1A和P438-GFP- GhPEAMT1D。
1.7 GhPEAMT1过表达载体的构建及拟南芥的遗传转化
扩增和的CDS片段,用限制性内切酶Ⅰ和HⅠ对目的片段和表达载体PBI121进行双酶切,然后连接,重组得到过表达载体PBI121-GhPEAMT1A和PBI121-GhPEAMT1D。使用冻融法将其转入农杆菌GV3101,筛选阳性单克隆菌。通过花序浸染法浸染拟南芥,收获的种子即是T0代。把消毒后的种子,平铺在含有卡那霉素抗性的MS培养基上,在培养箱中培养约15 d后,获得拟南芥阳性苗。把阳性苗种植到营养土中,待拟南芥长到7—8片叶片,取叶片进行PCR检测,确定目的基因是否转入拟南芥中,此时为T1代拟南芥。继续种植,最后得到纯合的T3代。
1.8 转基因拟南芥种子盐胁迫处理
将拟南芥种子在0.1%的HgCl2溶液中振荡浸泡4 min,灭菌水清洗6次后,分别把转基因拟南芥种子和野生型拟南芥种子点种在含不同浓度NaCl(0、50、100、150和200 mmol·L-1)的MS培养基中,4℃,春化48 h后,放入23℃的光照培养箱中,16 h光照/8 h黑暗萌发,生长10 d后观察统计拟南芥种子的萌发率,然后选取生长一致的野生型拟南芥和转基因拟南芥转移到不同浓度NaCl的MS培养基中,垂直置于恒温培养箱中培养,一周后统计根长,试验重复3次。
1.9 病毒诱导GhPEAMT1沉默载体的构建
基因沉默载体的构建选用的载体是TRV2,选取特异的目的片段,片段大小为300 bp,酶切位点为Ⅰ和HⅠ。用Primer Premier5软件设计引物为VPEAM1TA-F、VPEAMT1A-R和VPEAMT1D-F、VPEAMT1D-R(表1)。以1.4提取的质粒为模板进行PCR扩增,用限制性内切酶Ⅰ和HⅠ对目的片段进行酶切,然后连接到TRV2沉默载体上,测序正确导入农杆菌LBA4404中,挑取单克隆,验证条带大小,得到基因沉默载体TRV2::GhPEAMT1A和TRV2::GhPEAMT1D。
1.10 GhPEAMT1沉默棉花植株的盐胁迫处理
把TRV2::GhPEAMT1A、TRV2::GhPEAMT1D、TRV2:00、CLA1和辅助菌液在含有卡那霉素和利福平的LB液体培养基中置于28℃的摇床里过夜培养(约16 h),菌液离心收集菌体,用重悬液(MES、MgCl2、乙酰丁香酮)重悬,测OD值为1.0—1.2,把辅助载体重悬液和各个载体的重悬液按1﹕1混合,然后在25℃黑暗环境中静置2 h。选取在24℃,16 h光照/8 h黑暗环境下生长7 d的棉花幼苗完全展开的子叶进行注射,注射之后黑暗处理24 h,置于23℃的温室继续培养。等出现白化苗后,对试验组和对照组单株取样,进行沉默效率的检测,三叶期对试验组和对照组分别进行盐处理,盐浓度为40 g·L-1,每株浇灌100 mL盐水。试验重复3次。
2 结果
2.1 GhPEAMT1在转录组和蛋白质组中的表现
通过比较陆地棉耐盐品种早熟长绒7号和感盐品种南丹巴地大花苗期叶片盐胁迫诱导的转录组和蛋白组数据[17],图1-A表明,Gh_A03G0803()和Gh_D02G1225()2个基因在NaCl 胁迫后4和24 h具有显著的差异表达,尤其是在24 h时表达差异最大,其中,在早熟长绒7号中的表达差异倍数为5.22,在早熟长绒7号中的表达差异倍数为6.71。图1-B表明,2个基因在蛋白质水平受盐胁迫4和24 h时有显著的差异表达,其中GhPEAMT1D蛋白表达差异最大,耐盐陆地棉早熟长绒7号中的表达差异倍数为4.70,感盐陆地棉南丹巴地大花中的表达倍数为3.90。表明和可能是耐盐相关基因。

表1 研究中使用的引物

A:差异表达基因;B:差异丰度蛋白。E:早熟长绒7号;N:南丹巴地大花;0:CK;4:200 mmol·L-1 NaCl处理4 h;24:200 mmol·L-1 NaCl处理24 h。转录组数据无生物学重复,蛋白组数据有3个生物学重复
2.2 GhPEAMT1的克隆与序列分析
用200 mmol·L-1NaCl溶液处理后12 h的早熟长绒7号和南丹巴地大花的cDNA为模板,通过基因克隆、测序得到正确的和的CDS序列,经分析,这两个基因的CDS区核酸序列分别在2个品种中均表现无差异。的CDS全长为1 488 bp(图2-A),编码495个氨基酸,蛋白质分子式为C2528H3924N670O749S19,相对分子质量为56.296 kD,理论等电点为6.27,具有亲水性,不具有跨膜结构。的CDS全长为1 485 bp(图2-B),编码494个氨基酸,蛋白质的分子式为C2520H3906N662O746S19,相对分子质量为56.021 kD,理论等电点为6.02,具有亲水性,不具有跨膜结构。通过保守结构域分析,都具有2个高度保守甲基转移酶结构域(PF08241和PF13489)(图2-C)。

A:GhPEAMT1A克隆电泳图;B:GhPEAMT1D克隆电泳图;C:GhPEAMT1蛋白保守结构域分析;M1:Trans2K®Plus II Marker;1和3:早熟长绒7号;2和4:南丹巴地大花
将陆地棉GhPEAMT1A的氨基酸序列与亚洲棉(,XM_017755899)、海岛棉(,XM_016811336、XM_ 016835662)、雷蒙德氏棉(,XM_012623073)、拟南芥(,NP_188427.2、NP_973993.1)、水稻(,XM_015785333XP_015622327)、玉米(,NP_001307897.2)、可可(,XM_ 007036740)和杨树(,XM_ 024582451)进行序列比对,发现GhPEAMT1的氨基酸序列与以上物种存在高度保守区域(图3-A),进化树分析表明与海岛棉同源基因的亲缘关系最近,同源性较高,而棉种与其他物种相比,与可可同源性最高(图3-B)。
2.3 GhPEAMT1的表达特征分析
通过对经NaCl溶液不同处理时间棉花中表达的分析(图4),结果表明,不同的棉花品种在不同组织和时期和的表达量呈不同程度上调,但是表达特征有所不同。在12和24 h时,耐盐棉花品种早熟长绒7号中的和表达量显著高于南丹巴地大花,且24 h表达量达到最高;相对于叶片中的表达趋势,在根中有所区别。2个基因在早熟长绒7号根中受NaCl胁迫12 h表达量达到最大值,整体表现为先升后降。感盐品种南丹巴地大花的表达量在4、12和24 h显著低于早熟长绒7号。因此,推测2个基因在耐盐品种中响应盐胁迫过程中起重要作用。

A:GhPEAMT1氨基酸的多序列比对。Gbar_A03G010420:海岛棉,XM_016811336;Gbar_D02G013090:海岛棉,XM_016835662;Thecc1EG012768t1:可可,XM_007036740;Gorai.005G132000:雷蒙德氏棉,XM_012623073;Gh_A03G0803:陆地棉,GhPEAMT1A;Gh_D02G1225:陆地棉,GhPEAMT1D;AT1G48600:拟南芥,NP_973993.1;AT3G18000:拟南芥,NP_188427.2;LOC_Os01g47540:水稻,XM_015785333;LOC_Os01g50030:水稻,XP_015622327;Ga03G1288:亚洲棉,XM_017755899;Potri.012G047400:杨树,XM_024582451;GRMZM2G122296:玉米,NP_001307897.2;黑色线段为保守结构域的位置;B:GhPEAMT1的系统进化分析;黑色方框表示目的基因
A: Multiple sequence alignment of. Gbar_A03G010420:, XM_016811336; Gbar_D02G013090:, XM_016835662; Thecc1EG012768t1:, XM_007036740; Gorai.005G132000:, XM_012623073; Gh_A03G0803:, GhPEAMT1A; Gh_D02G1225:, GhPEAMT1D; AT1G48600:, NP_973993.1; AT3G18000:, NP_188427.2; LOC_Os01g47540:, XM_015785333; LOC_Os01g50030:, XP_015622327; Ga03G1288:, XM_017755899; Potri.012G047400:, XM_024582451; GRMZM2G122296:, NP_001307897.2; The black line indicates the position of conserved domain; B:Phylogenetic analysis of; The black box indicates the target gene
图3 GhPEAMT1氨基酸序列比对和进化树分析
Fig. 3 Amino acid sequence alignment and phylogenetic tree analysis for gene GhPEAMT1

A:GhPEAMT1A在早熟长绒7号和南丹巴地大花叶片中的表达;B:GhPEAMT1A在早熟长绒7号和南丹巴地大花根中的表达;C:GhPEAMT1D在早熟长绒7号和南丹巴地大花叶片中的表达;D:GhPEAMT1D在早熟长绒7号和南丹巴地大花根中的表达。E:早熟长绒7号;N:南丹巴地大花
2.4 陆地棉GhPEAMT1的亚细胞定位分析
运用CELLOV.2.5软件预测GhPEAMT1位于细胞质中,通过激光共聚焦扫描显微镜观察,发现空载体在细胞膜、细胞质和细胞核中均能检测到绿色荧光蛋白,而目的基因融合蛋白在细胞质中检测到绿色荧光蛋白(图5),说明GhPEAMT1A和GhPEAMT1D位于细胞质中。
2.5 转基因拟南芥的获得与鉴定
通过花序浸染法得到转基因拟南芥种子,用MS培养基(Kan+)筛选得到阳性植株,使用基因特异引物对筛选出的阳性植株进行PCR鉴定,去除假阳性植株,为了得到纯合的单拷贝阳性植株,继续筛选阳性植株与非阳性植株比值为3﹕1的株系,直至得到纯合株系。获得9株转阳性植株(图6-A),获得5株转阳性植株(图6-B),通过RT-PCR分析,和在转基因植株中均能正常表达,在野生型拟南芥中未检测到转录信号(图6-C)。
2.6 转基因拟南芥的耐盐性分析
为了进一步研究的耐盐性,通过对WT、转拟南芥和转拟南芥在不同浓度NaCl(0、50、100、150和200 mmol·L-1)处理下进行萌发率和根长的数据统计(图7)。0 mmol·L-1NaCl浓度时野生型与转基因拟南芥的萌发率和主根长无显著差异;50 mmol·L-1NaCl浓度时野生型拟南芥萌发率无显著差异,主根长有显著差异,但能正常生长;100 mmol·L-1NaCl浓度时野生型拟南芥萌发率显著低于转基因拟南芥,野生型拟南芥的主根显著长于野生型拟南芥;150 mmol·L-1NaCl浓度时野生型拟南芥的萌发率显著低于转基因拟南芥的萌发率,野生型拟南芥叶片发黄逐渐死去,转基因拟南芥叶片发黄且主根长度较短。
2.7 GhPEAMT1沉默后的功能分析
对陆地棉早熟长绒7号中的和进行沉默。棉花中与叶绿素合成相关基因发生沉默,棉花的叶绿素合成受到干扰,后期新出现的真叶表现为白化[18]。侵染棉花植株后观察表型,当出现白化表明基因沉默成功(图8-A)。然后单株取棉花的叶片,通过实时荧光定量(qRT-PCR)验证对照(CK)、TRV2:00、和沉默后棉花植株的表达量,CK棉花植株和TRV2:00棉花植株表达量无显著变化,目的基因沉默后棉花植株的表达量显著降低,结果表明,成功沉默(图8-B)。用40 g·L-1NaCl溶液对基因沉默棉花植株、TRV2:00棉花植株和CK棉花植株进行盐胁迫处理。观察盐处理后的表型发现盐胁迫48 h后,棉花1沉默的植株比对照植株萎蔫严重(图8-C和图8-D)。

图5 GhPEAMT1A和GhPEAMT1D亚细胞定位

A—D:0、50、100和150 mmol·L-1 NaCl处理下拟南芥萌发率情况;E—H:0、50、100和150 mmol·L-1 NaCl处理下拟南芥根伸长情况;I:不同NaCl浓度下拟南芥的萌发率;J:不同NaCl浓度下拟南芥的主根长度。*:差异显著(P<0.05),**:差异极显著(P<0.01)。下同
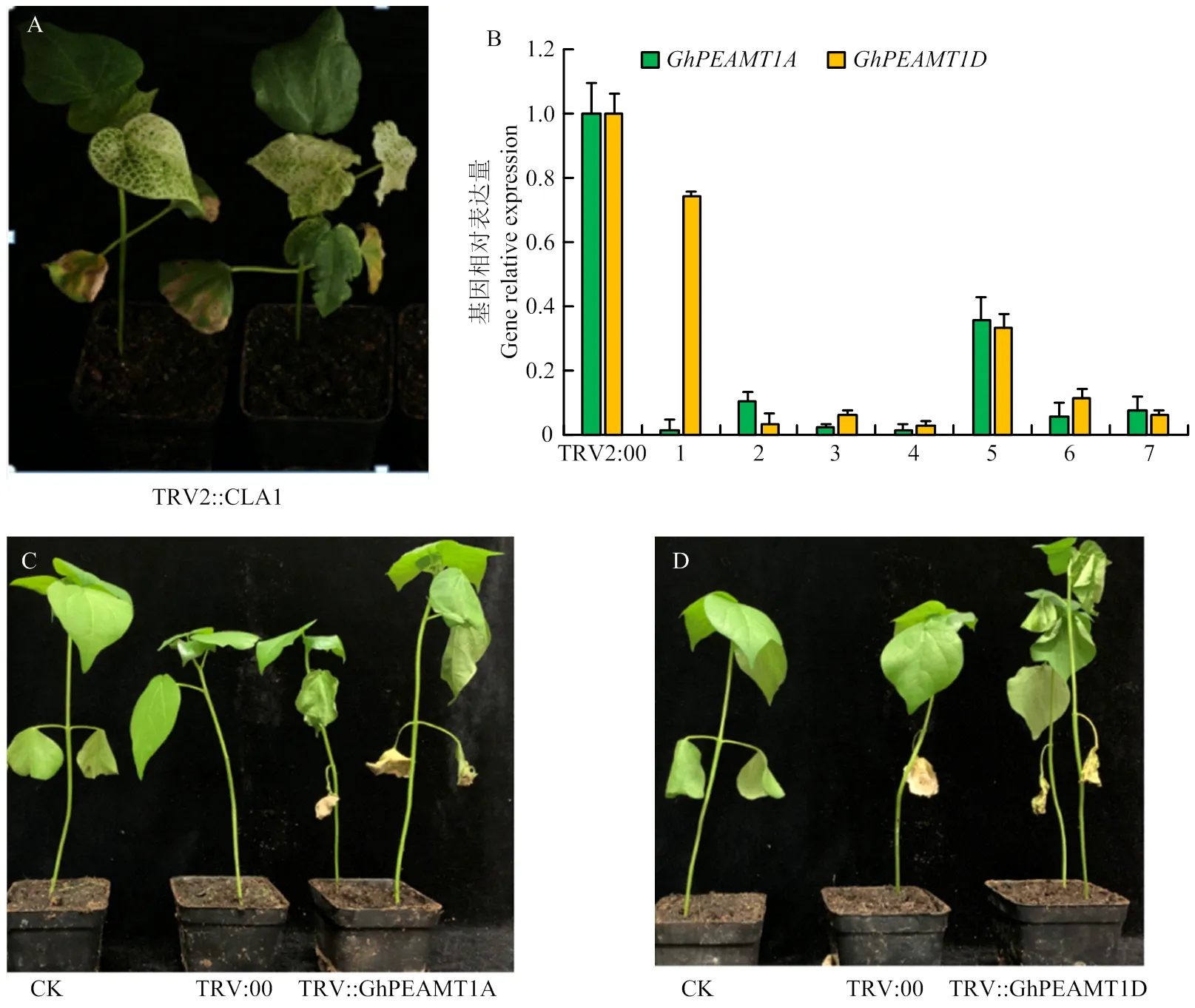
A:沉默标记基因TRV2:CLA植株;B:病毒诱导基因沉默后,GhPEAMT1A/D沉默效率分析,1—7:注射TRV2::GhPEAMT1A、TRV2::GhPEAMT1D菌株的株系;C—D:盐胁迫48 h后,CK、TRV2:00、TRV2::GhPEAMT1A、TRV2::GhPEAMT1D棉花幼株表型
盐胁迫后,注射TRV2::GhPEAMT1A棉花植株与注射空载棉花植株相比过氧化氢酶(CAT)活性降低、谷胱甘肽过氧化物酶(GPX)活性降低(图9-A和图9-B)。沉默TRV2::GhPEAMT1D棉花植株TRV2:00相比CAT、GPX活性都略微升高,但没有显著差异(图9-A和图9-B),由此推测沉默后,CAT、GPX的活性降低,直接影响活性氧清除系统的不同成员如CAT、GPX、POD的活性(随着胁迫时间延长,最终结果都是降低),继而表现出对盐敏感。而沉默后对活性氧清除系统没有影响,推测沉默的植株受盐胁迫后可能影响其他代谢途径。
3 讨论
当植物受到盐胁迫时,可发生多种胁迫应答反应,例如,可以积累一些可溶性物质来提高细胞渗透压来减轻盐胁迫对植物造成的伤害[19]。植物PEAMT基因通过提高Cho的含量促进渗调物质甘氨酸甜菜碱(GB)的生成,从而增强了植物对盐胁迫或干旱胁迫的耐受能力[20]。研究表明,在转基因植物中GB含量低在很大程度上是由于Cho合成能力不足[21]。其中磷酸乙醇胺N-甲基转移酶(PEAMT)是磷酸胆碱合成的关键酶[22],因此,研究植物中的PEAMT的基因在应对盐胁迫中所起的作用显得非常重要。近年来,随着分子生物学的发展,通过基因工程技术培育和筛选耐盐性的作物得到了大力的发展。截至目前,研究人员已克隆、鉴定出一批与耐盐相关的基因,并研究了抗逆境功能,通过遗传转化还获得了一些转基因株系。比如,用NaCl处理菠菜叶片后,PEAMT的酶活都高于未处理的[23];Peel等[11]克隆了玉米中的PEAMT基因在盐碱胁迫下的玉米叶片,PEAMT酶的活性增加了400%;Li等[24]在辽宁蓬碱中克隆了PEAMT基因,在盐胁迫后,基因的表达量升高;对大麦的根中进行了PEAMT基因转录调控的分析,在24 h和3 d上调表达[25];在无隐芒子中克隆出,干旱胁迫诱导8 d时,在叶和根中表达量分别是未干旱对照的43.35倍和13.25倍,表明可能是无芒隐子草抗旱性相关的基因[26]。过量表达PEAMT基因的转基因植株通过增加胆碱和GB的合成来提高自身对盐胁迫的耐受能力[14,27],表明PEAMT基因参与植物对非生物逆境的响应。Mou等[27]研究表明,拟南芥突变体的Cho生物合成量减少约64%,从而导致突变体植株对盐渍高度敏感。

A:盐胁迫48 h后,TRV2:00、TRV2::GHPEAMT1A棉花幼苗CAT的活性;B:盐胁迫48 h后,TRV2:00、TRV2::GHPEAMT1D棉花幼苗GPX的活性
目前,PEAMT基因在棉花中研究比较少。本研究在转录组学和蛋白质组学数据分析的基础上筛选到的该耐盐相关的候选基因。前人的研究表明不同植物PEAMT基因的mRNA转录水平受到盐胁迫诱导上调表达[28-29]。WU等[30]通过对玉米进行实时定量PCR分析,表明受到盐胁迫的诱导和高温抑制;MOU等[27]对盐角草启动子区分析含有脱落酸(ABA)诱导元件、热激应答元件和低温诱导元件等顺式调控元件,表明在转录水平上可能受赤霉素、低温、光、ABA与热激的调控,这和盐草角适应其恶劣多变的环境相关。本研究对盐耐性不同的棉花品种进行表达特征分析表明在受到盐胁迫后根和叶在12和24 h表达量最高,且在耐盐棉花品种中的表达量高于感盐品种,因此,推测陆地棉在植物耐盐中有着重要作用。继而对目的基因的功能进行验证,通过转基因过表达拟南芥与野生型相比,在100 mmol·L-1NaCl胁迫下萌发率和主根长显著较高;通过VIGS技术沉默后陆地棉的抗氧化能力相比对照植株下降,由此推测在棉花在盐胁迫响应中能提高抗氧化能力,继而表现对盐胁迫的耐受性。
土壤盐渍化已经严重影响到世界范围内不少作物的产量等性状。而当前利用基因工程手段来提高作物的耐盐性是解决这一世界难题的有效策略之一。也有大量研究表明向作物转入与耐盐相关的基因可以在一定程度提高作物的耐盐性。本研究通过转基因拟南芥,提高了耐盐性;通过沉默该基因导致棉花沉默株系对盐敏感,都证明该基因是耐盐相关基因,这为进一步棉花基因工程育种,培育棉花抗逆新品系提供理论和试验依据。
4 结论
从陆地棉中克隆了2个同源基因和,其CDS长度分别为1 488和1 485 bp,编码495和494个氨基酸,推导的多肽均包含2个高度保守甲基转移酶结构域。不具有跨膜结构,在进化中功能高度保守。陆地棉和的表达在盐胁迫后被诱导,推测在响应盐胁迫过程中起正向调控作用。
[1] 李忠旺, 陈玉梁, 罗俊杰, 石有太, 冯克云, 陈子萱. 棉花抗旱品种筛选鉴定及抗旱性综合评价方法. 干旱地区农业研究, 2017, 35(1): 240-247.
LI Z W, CHEN Y L, LUO J J, SHI Y T, FENG K Y, CHEN Z X. Screening and evaluation for drought resistance of cotton varieties. Agricultural Research in the Arid Areas, 2017, 35(1): 240-247. (in Chinese)
[2] 孟超敏, 蔡彩平, 郭旺珍. 棉花抗逆育种研究进展. 南京农业大学学报, 2012, 5(5): 29-38.
MENG C M, CAI C P, GUO W Z. Advances in cotton stress resistance breeding. Journal of Nanjing Agricultural University, 2012, 5(5): 29-38. (in Chinese)
[3] MUNNS R, TESTER M. Mechanisms of salinity tolerance. Annual Review of Plant Biology, 2008, 59(1): 651-681.
[4] ZHANG X, ZHEN J, LI Z, KANG D, YANG Y, JIN K, HUA J. Expression profile of early responsive genes under salt stress in upland cotton (L.). Plant Molecular Biology Reporter, 2011, 29(3): 626-637.
[5] MAHAJAN S, TUTEJA N. Cold, salinity and drought stresses: An overview.Archives of Biochemistry & Biophysics, 2005, 444(2): 139-158.
[6] MAAS E V. Crop salt tolerance-current assessment. Journal of the Irrigation and Drainage Division, 1977, 103(2): 115-134.
[7] MAGWANGA R O, KIRUNGU J N, LU P, CAI X Y, XU Y C, WANG X X, ZHOU Z L, HOU Y Q, AGONG S G, WANG K B, LIU F. Knockdown of ghAlba_4 and ghAlba_5 proteins in cotton inhibits root growth and increases sensitivity to drought and salt stresses. Frontiers in plant science, 2019, 3389(10): 1292-1241.
[8] DEINLEIN U, STEPHAN A B, HORIE T, LUO W, SCHROEDER J I. Plant salt-tolerance mechanisms. Trends in Plant Science, 2014, 19(6): 371-379.
[9] 黄滋康, 季道藩, 潘家驹. 中国棉花遗传育种学. 济南: 山东科学技术出版社, 2003.
HUANG Z K, JI D F, PAN J J.. Jinan: Shandong Science and Technology Press, 2003. (in Chinese)
[10] ASHRAF M, FOOLAD M R. Roles of glycine betaine and proline in improving plant abiotic stress resistance. Environmental & Experimental Botany, 2007, 59(2): 206-216.
[11] PEEL G J, MICKELBART M V, RHODES D. Choline metabolism in glycinebetaine accumulating and non-accumulating near-isogenic lines ofand.Phytochemistry, 2010, 71(4): 404-414.
[12] 陈少良, 李金玉, 毕望富, 王沙生. 盐胁迫条件下杨树盐分与甜菜碱及糖类物质变化. 植物学通报, 2001, 18: 587-596.
CHEN S L, LI J Y, BI F W, WANG S S. Genotypic variation in accumulation of salt ions, betaine and sugars in poplar under conditions of salt stressChinese Bulletin of Botany, 2001, 18: 587-596. (in Chinese)
[13] MCNEIL S D, NUCCIO M L, ZIEMAK M J, ET A L. Enhanced synthesis of choline and glycine betaine in transgenic tobacco plants that overexpress phosphoethanolamine N-methyltransferase. Proceedings of the National Academy of Sciences of the United States of America, 2001, 98(17): 10001.
[14] RICARDA J, OLIVER B, JOHN S, JOSETTE M. Biochemical characterization of two wheat phosphoethanolamine N-methyltransferase isoforms with different sensitivities to inhibition by phosphatidic acid.The Journal of biological chemistry, 2009, 284(46): 31962-31971.
[15] WERETILNYK E A, SMITH D D, WILCH G A, SUMMERS P S. Enzymes of choline synthesis in spinach (response of phospho-base n-methyltransferase activities to light and salinity).Plant Physiology, 1995, 109(3): 1085-1091.
[16] HANSON A D, RHODES D. 14C tracer evidence for synthesis of choline and betaine via phosphoryl base intermediates in salinized sugarbeet leaves. Plant Physiology, 1983, 71(3): 692-700.
[17] PENG Z, HE S, GONG W, XU F, PAN Z, JIA Y, GENG X, DU X. Integration of proteomic and transcriptomic profiles reveals multiple levels of genetic regulation of salt tolerance in cotton. Bmc Plant Biology, 2018, 18(1): 128.
[18] ZHANG J, WANG F, ZHANG C, ZHANG J, YU C, LIU G, ZHAO Y, HAO F, ZHANG J. A novel VIGS method by agroinoculation of cotton seeds and application for elucidating functions of GhBI-1 in salt-stress response. Plant Cell Reports, 2018, 37(18): 1091-1100.
[19] KLÁRA KOSOVÁ, ILJA PRÁŠIL, PAVEL VÍTÁMVÁS. Protein contribution to plant salinity response and tolerance acquisition.International journal of molecular sciences, 2013, 14(4): 6757-6789.
[20] GOU W, ZHENG P, CHEN F, ZHANG L, CUI Z, CAO M, ZHANG L, HU J. Accumulation of choline and glycinebetaine and drought stress tolerance induced in maize () by three plant growth promoting rhizobacteria (pgpr) strains. Pakistan Journal of Botany, 2015, 47(2): 581-586.
[21] NUCCIO M L, RUSSELL B L, NOLTE K D, RATHINASABAPATHI B, GAGE D A, HANSON A D. The endogenous choline supply limits glycine betaine synthesis in transgenic tobacco expressing choline monooxygenase.The Plant journal for cell and molecular biology, 1998, 16(4): 487-496.
[22] 王名雪, 陆平, 许菲, 潘琪芳, 唐克轩, 赵静雅. 枸杞胆碱合成关键酶基因PEAMT的克隆及生物信息学分析. 上海交通大学学报(农业科学版), 2013, 31(1): 1-7.
Wang M X, LU P, XU F, Pan Q F, Tang K X, Zhao J Y. Cloning and bioinformatics analysis of choline biosynthetic gene PEAMT in. Journal of Shanghai Jiaotong University, 2013, 31(1): 1-7. (in Chinese)
[23] SUMMERS P S, WERETILNYK E A. Choline synthesis in spinach in relation to salt stress. Plant physiology, 1993, 103(4): 1269-1276.
[24] LI Q L, XIE J H, MA X Q, LI D. Molecular cloning of Phosphoethanolamine N-methyltransferase (PEAMT) gene and its promoter from the halophyte Suaeda liaotungensis and their response to salt stress. Acta Physiologiae Plantarum, 2016, 38(2): 39.
[25] GHORBANI A, FEIZPOUR A, HASHEMZAHI M, GHOLAMI L, HOSSEINI M, SOUKHTANLOO M, VAFAEE B F, KHODAEI E, MOHAMMADIAN R N, BOSKABADY M. The effect of adipose derived stromal cells on oxidative stress level, lung emphysema and white blood cells of guinea pigs model of chronic obstructive pulmonary disease. Daru Journal of Pharmaceutical Sciences, 2014, 22(1): 26.
[26] 段珍, 张吉宇, 狄红艳, 霍雅馨. 无芒隐子草基因克隆及表达特性分析. 西北植物学报, 2014, 34(12): 2367-2373.
Duan Z, Zhang J y, Di H Y, Huo Y x. Clone and expression characterization ofgene inActa Botanica Boreali-Occidentalia Sinica, 2014, 34(12): 2367-2373. (in Chinese)
[27] MOU Z, WANG X, FU Z, ET A L. Silencing of phosphoethanolamine N-methyltransferase results in temperature-sensitive male sterility and salt hypersensitivity in. The Plant Cell, 2002, 14(9): 2031-2043.
[28] SAHU B B, SHAW B P. Isolation, identification and expression analysis of salt-induced genes in, a natural halophyte, using PCR-based suppression subtractive hybridization. BMC Plant Biology, 2009, 9(1): 69.
[29] KING J, WEI Y D, BHUIYAN N H, SELVARAJ G, LIU W P, LIU G S. Transcriptional regulation of genes involved in the pathways of biosynthesis and supply of methyl units in response to powdery mildew attack and abiotic stresses in wheat.Plant molecular biology, 2007, 64(3): 305-318.
[30] WU S, YU Z, WANG F, LI W, YE C, LI J, TANG J, DING J, ZHAO J, WANG B. Cloning, characterization, and transformation of the phosphoethanolamine N-methyltransferase gene () in maize (L.). Molecular Biotechnology, 2007, 36(2): 102-112.
Cloning and Functional Analysis of Salt Stress Response Gene
WANG Na1, ZHAO ZiBo2, GAO Qiong1, HE ShouPu1, MA ChenHui1, PENG Zhen1,2, DU XiongMing1,2
1Institute of Cotton Research, Chinese Academy of Agricultural Sciences/State Key Laboratory of Cotton Biology, Anyang 455000, Henan;2School of Agricultural Sciences, Zhengzhou University/Zhengzhou Research Base, State Key Laboratory of Cotton Biology, Zhengzhou 450001
【】Phosphoethanolamine-N-methyltransferase (PEAMT) is the key enzyme in the synthesis of plant phosphocholine, which is the precursor of choline, a glycine betaine synthesis substrate that can enhance plant resistance. The biological function ofgene in response to salt stress in upland cotton was studied by cloning, expression pattern analysis and functional verification,so as to provide gene resources for breeding salt tolerant cotton varieties.【】According to the screening of transcriptome sequencing data,gene was selected as a salt-tolerant candidate gene. the target gene was amplified by polymerase chain reaction (PCR). gene structure characteristicspredict protein relative molecular mass and evolutionary relationship were analyzed by bioinformatics; Salt-tolerant cotton variety Earlistable 7 and salt-sensitive cotton genotype Nandanbadidahua were treated with 200 mmol·L-1NaCl solution, and cotton leaves and roots were taken for fluorescence quantitative PCR (qRT-PCR) to analyze the expression characteristics in tissues. Subcellular location vector was constructedfor transient transformation of tobacco to determine the location of protein in the cell. The gene overexpression vector was constructed, andwas transformed by inflorescence infection method, and the germination rate and taproot lengthof transgenicunder salt stress were analyzed. Virus induced gene silencing (VIGS) technology was used on Earlistable 7to silence the target gene and the efficiency of gene silencing was verified by real-time fluorescence quantitative analysis of the cotton leaves. Then the enzyme activities of catalase (CAT) and glutathione peroxidase (GPX) in the leaves of cotton were measured. 【】Two homologous genes,and, were cloned and the CDS lengths were 1 488 bp and 1 485 bp, respectively. The phylogenetic tree showed thathad the closest genetic relationship with the Sea island cotton, andhad the higher homology withcompared with other species. Under salt stress conditions, the expression levels ofgene andgene in leaves and roots of salt-tolerant Earlistable 7 were significantly higher than that of salt-sensitive Nandanbadidahua. Subcellular localization shows that the gene is located in the cytoplasm.Overexpression of this gene forfound that the germination rate of transgenicwas significantly higher than that of wild-type on MS medium of 100 and 150 mmol·L-1NaCl. The taprootroot length of transgenicwas significantly longer than that of wild-type on the MS medium of 50 mmol·L-1and 100 mmol·L-1NaCl.After treatment with 40 g·L-1NaCl solution for 24 h, the cottonleaves with successfully silencedandgene were more wilting than those of CK and TRV2:00 injections. After salt stress, the TRV2::GhPEAMT1A cotton plants were reduced in catalase (CAT) and glutathione peroxidase (GPX) content in comparison with the unloaded injected cotton plants. 【】overexpression and VIGS experiments show that thegene can respond to salt stress and play a positive regulatory role.
Upland cotton; salt stress;gene; gene cloning; gene silencing

10.3864/j.issn.0578-1752.2021.02.002
2020-06-13;
2020-08-27
国家重点研发计划(2017YFD0101600)、中央级公益性科研院所基本科研业务费专项(161012018004)
王娜,E-mail:15517960172@163.com。通信作者杜雄明,E-mail:dxm630723@163.com。通信作者彭振,E-mail:pengzhen01@caas.cn
(责任编辑 李莉)

