基于扩散磁共振的脑组织微结构成像研究综述
徐永红 丁 玲
(燕山大学电气工程学院,河北 秦皇岛 066004)
引言
扩散磁共振成像(diffusion magnetic resonance imaging, dMRI)技术由常规磁共振成像发展而来,在过去的20年中,已经成为神经成像研究中必不可少的工具[1]。在微观环境下,dMRI对水分子的扩散运动十分敏感,因此可以作为组织微结构的探针,在微米尺度上测量组织结构,或以非侵入方式研究组织微结构与脑功能连接,得到关于细胞完整性和组织微结构的信息[2]。作为扩散磁共振成像的实现方式之一,扩散张量成像(diffusion tensor imaging, DTI)是用来评估生物组织结构完整性最简单、最常用的模型[3]。DTI模型用平均扩散系数(mean diffusivity,MD)和各向异性分数(fractional anisotropy,FA)等指标反映白质组织特性,但缺乏特异性描述,也无法描述多个纤维交叉、弯曲等复杂环境下的结构特征。目前已提出了很多方案避免上述DTI模型的限制,包括直接对DTI进行扩展[4-6]、扩散方向变换[7]、扩散谱成像(diffusion spectrum imaging,DSI)[8]、基于Q空间球面的成像方法[9-10]、球面反卷积(spherical deconvolution,SD)[11-13]等,这些技术可获取体素中纤维族的方向,却无法提供组织微结构相关信息。
传统组织学是通过解剖来获取亚微米级的组织细胞结构信息,扩散磁共振图像则是显示毫米级的平均信息,是体素内组织结构的统计描述。dMRI作为组织微结构的探针包括两步:第一步是利用给定组织结构来预测信号的正向问题,第二步是解决用给定测量信号计算微结构特征的逆问题[14]。解决逆问题的难点在于样本的磁共振信号已经被平均化,有关特定细胞属性的信息大量丢失。因此,微结构成像的首要任务是:认识到哪些微观结构的特性在这种平均情况下仍然存在,结合组织结构建立数学模型,将模型拟合到MR信号的测量值上,以估计组织微结构特征。目前,组织学上的许多工作都是在相对广泛的组织范围内探索更多的统计变化,例如:白质通路中轴突的密度和直径分布决定了它的信息承载能力;细胞的不同密度、形状和构型可以区分不同类型的脑肿瘤;大量蛋白质沉积是阿尔茨海默症的标志,而上述应用中微结构成像技术则显现其特有优势[15]。
目前,微结构成像是扩散磁共振成像技术发展与临床研究的热门领域,虚拟组织学在临床应用上显现出巨大潜力。图1所示为1985—2019年扩散磁共振微结构成像领域文献数量随时间增长的曲线,当前文献增长正处于指数增长阶段。

图1 1985-2019年微结构成像文献数量的增长Fig.1 The relationship between number of documents and the time from 1985 to 2019
笔者从微观结构与宏观信号关系出发,概述扩散磁共振成像原理;随后介绍微结构成像的经典模型和最新进展,主要集中在多隔室模型和深度学习算法的应用上;最后对微结构成像技术发展趋势进行展望。
1 扩散磁共振成像原理
由于扩散过程受环境几何结构影响,因此磁共振可用于无创地探测环境结构。在生物组织中,影响扩散的边界长度往往很小,导致常规磁共振成像技术无法解决。扩散磁共振成像技术在常规MRI基础上发展而来, 目前已成为研究组织微结构最有前景的方法之一。
1.1 水分子的扩散特性
自然界物质中的分子不停地进行布朗运动,即扩散。从本质上扩散可以看作是一个统计过程:在成像体素中宏观观察到大量粒子位移的系统平均值。单个粒子被描述为自旋位移概率密度函数P(rt|r0,t),表示在经过时间t后,在rt位置发现初始位置r0的自旋的概率。体素中包含大量自旋,用位移密度函数P(r,t)表示时间t内的粒子位移r=rt-r0的概率。
组织的微观结构决定粒子的移动,从而确定粒子位移的概率密度函数P(r,t)。相反,概率密度函数的特征也可以反映组织的微观结构。扩散磁共振成像的目的是检测不同组织中水分子扩散位移的概率密度函数,在较长的扩散时间下测量获取隔室尺寸;反之,则提供有关分子运动的信息,即扩散系数。
1.2 扩散与磁共振成像
原子核的进动磁化在磁体孔周围的线圈中产生感应电流,由此能够及时监控磁化强度m(r,t)的变化。Bloch-Torrey方程从微观上准确描述磁化强度的变化[16],在复杂生物组织中,忽略微观各向异性后可将Bloch-Torrey方程简化为
(1)
式中,m(r,t)是磁化强度,2是描述各向同性扩散的拉普拉斯算子,由此测量到的体素内宏观信号可表示为
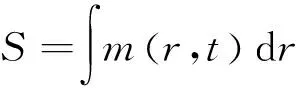
(2)
在不均匀磁场Bz(r,t)中扩散时,每个原子核相位φ沿运动轨迹R(t)对位置有关的拉莫尔频率γBz(r,t)积分得到
(3)
早在1950年,Hahn就提出梯度磁场中粒子随机运动出现相位滞后效应[17];之后,Stejskal和Tanner引入梯度脉冲自旋回波序列(pulsed gradient spin echo, PGSE),将MR信号与介质中自旋扩散关系简化[18]。具体是在常规磁共振成像的自旋回波序列180°脉冲的两侧,加入一对大小方向均相同的扩散敏感梯度脉冲(见图2),其中:第一个梯度脉冲引起所有质子自旋,从而引起相位变化;第二个梯度脉冲使相位重聚,但此时相位分散不能完全重聚,从而导致信号衰减。

图2 梯度脉冲自旋回波序列原理Fig.2 Principle of pulsed gradient spin echo
扩散在两个梯度脉冲之间自旋位移r=rt-r0产生净相位变化,有
Δφ=φΔ-φ0=γδG(rΔ-r0)=qr
(4)
为了方便描述体素内分子多方向空间扩散情况,将扩散梯度磁场的参数进行组合并称为q,有
q=γδG
(5)
式中,γ是磁旋比,G和δ分别为扩散梯度脉冲的强度和持续时间。
由于处理MR信号衰减E(q)比处理MR信号本身更容易,所以一般采用信号衰减。宏观MR信号衰减可通过样本中所有自旋信号求和获得,有
(6)
当N→ ∞,信号衰减总和可表示为整体自旋概率密度的积分,即
(7)
该方案最初由Stejskal提出,通过傅里叶变换,将扩散衰减信号E(q)与平均位移概率密度相联系。对式(7)进行傅里叶变换求逆,获得平均位移概率密度函数P,重建概率密度函数获取位移的“Q空间图像”。
此外,磁共振成像纵向弛豫时间T1和横向弛豫时间T2直接受局部生物物理结构和生物化学环境的影响,对脑组织微结构完整性的变化敏感。特别地,在给定的组织隔室中有更多的水时,该隔室体现为较长的T1时间,在T1加权图像上呈现较低的信号。
1.3 生物组织中的扩散

E(q)=e-q2D(Δ-(δ/3))=e-bD
(8)
式中,扩散系数D是自旋在扩散介质的属性。
上述方程将脉冲持续时间的影响合并到信号中,由此定义自由扩散梯度因子b值,即
b=q2(Δ-(δ/3))=(γδG)2(Δ-(δ/3))
(9)
式中,b值表示扩散加权强度,是概括所施加扩散梯度和扩散时间的组合效应参数。
目前,已有各种方法模拟生物系统中的扩散,每种方法都有不同的假设和复杂度。最简单的模型是假设自由扩散,以单一扩散系数为特征。由于测得的扩散系数D与扩散时间及组织样本相关,故引入表观扩散系数(apparent diffusion coefficient, ADC)。特别是急性缺血性卒中成像,在缺血开始后的几分钟内ADC明显减少,其他成像方法则在几个小时后才能观察到。迄今为止,这仍是大脑 dMRI最常见的临床应用之一。
扩散张量模型是ADC模型的简单扩展,对扩散的各向异性建模,并从中提取描述组织微观结构的指标。常用指标FA被广泛用于神经疾病和精神疾病研究中,已经成为临床研究中最常用的指标之一。但FA难以解释各向异性发生的变化,因为不同的原因(如髓鞘质减少、轴突变性等)都可能导致FA发生相同的改变。此外,大脑中存在多种纤维结构(如交叉纤维、扇形纤维等),图3为纤维结构示意,DTI则无法描述这些复杂纤维中的扩散。

图3 纤维取向示意。(a)交叉;(b)扇形;(c)平行;(d)弯曲Fig.3 Schematic illustrating the fiber orientations. (a) Crossing; (b) Fanning; (c) Parallel; (d) Bending
2 微结构成像与多隔室模型
2.1 微结构成像
微结构成像是为了克服传统扩散磁共振成像的一些缺点而发展的一种新技术。该技术的目的在于获取关键微结构参数的可靠指标,如轴突直径、纤维密度和方向等。微结构成像的一般策略是建立数学模型,模型将组织细胞结构的特定属性、随时间变化的水分子扩散模式与扩散MR信号相联系,将模型拟合到MR信号的测量值上,以解决估计组织微结构属性这一逆问题[18]。成像过程中,在扫描仪上设置不同的参数(扩散时间、b值等)来获取不同的扩散MR图像,不同扫描参数在图像的每个体素上提供一组测量值,之后根据测量值来拟合模型并估计参数。
微结构成像包含两种关键模型——信号模型和隔室模型。DTI[3]、扩散谱成像[8]、Q空间成像[9-10]、扩散峰度成像[19]、双扩散编码的扩展方法[20-21]等信号模型,将每个体素视为单一均匀隔室,因此仅能提供一个复合视图;隔室模型则将体素中的信号视为多个隔室共同作用的结果,假设每个隔室对应于不同的细胞成分,且表现出不同的扩散模式。图4是利用多隔室模型微结构的成像过程,其中将脑组织结构的微观特征与MR信号相联系,MR信号为一组不同梯度强度、梯度方向、扩散时间的扩散信号;白质微观结构的建模为简单、理想化的几何模型,将不同的组织模型和分布情况组合为多隔室模型,之后选择优化算法,将多隔室模型拟合到扩散数据中,估计纤维方向分布和组织特征参数等。
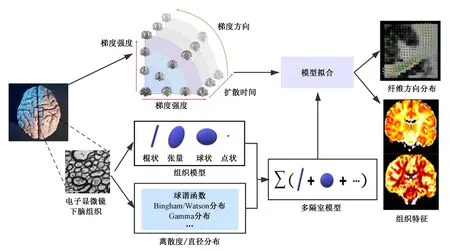
图4 多隔室模型成像Fig.4 Multi-compartment model image
2.2 多隔室模型
微结构成像依赖于将组织结构的微观特征与磁共振信号相关联所建立的多隔室模型,并向探测更加精细组织微结构信息的方向快速发展。接下来从多隔室模型估计纤维方向与纤维结构两方面展开介绍。
2.2.1估计纤维方向
2003年,Behrens等[22]提出了DTI模型的简单替代方案“球棍模型”(ball-and-stick model)。假设白质MR信号由两种不同扩散模式的水组成:一种被困于轴突内部,仅可沿纤维方向移动,用棍状模型表示;另一种在轴突周围自由扩散,表示为各向同性扩散的张量模型。与DTI模型不同,球棍模型设定所有纤维垂直方向的扩系数相同,拟合模型可估计纤维密度和纤维方向。
随后Assaf等[23]提出阻碍与限制复合扩散模型(composite hindered and restricted model of diffusion,CHARMED), 类似地将轴突内外分为两个隔室来表示扩散过程:限制扩散和受阻扩散。轴突建模为多个竖直平行、不可渗透圆柱体的集合,半径服从Gamma分布。轴突内使用限制在圆柱内的扩散模型,轴突外部受阻部分使用扩散张量建模。
传统的球棍模型和CHARMED模型存在着一定的局限性,模型假设每个体素内的所有纤维具有单一方向。因此,在原有模型基础上,提出更多的扩展方案来应对不同的纤维群。例如,假设在轴突内部用多个棒状隔室表示纤维交叉结构[24-25];对于一般的纤维结构,把信号看作是单个纤维信号与纤维方向分布的卷积,然后用单个纤维信号的模型,对信号进行反卷积,以便于估计纤维的方向分布[11-12,26]。其他技术使用简单的参数模型来表示纤维的方向分布,如Watson和Bingham分布。
2.2.2估计纤维结构
还有一部分技术着重估计描述纤维束组成的参数,如轴突密度和直径分布。最早在1997年Stanisz等[23]提出了一种神经组织三室模型:水分子扩散在细长椭圆形的轴突内、球形胶质细胞内以及细胞外空间,但需要提供大量扩散梯度及多个b值的低噪声测量数据。
Assaf等[27]对上述模型做出简化,并对CHARMED模型进行扩展,引入AxCaliber技术。其中,假设隔室之间不可渗透,并且没有胶质细胞隔室,特别是将轴突直径建模为两参数的伽马分布。实验表明,在切除的神经组织样本上,AxCaliber技术估计的轴突直径分布与组织学上的测量结果一致。但是AxCaliber技术需要假定已知的纤维方向,且在大脑纤维方向变化区域无法绘制纤维特性图,采集过程中需要高梯度磁场,扫描时间过长都会导致无法在临床上应用。Alexander等[28]提出ActiveAx技术,将优化的高角度分辨率扩散成像(high angular resolution diffusion imaging, HARDI)与简化的模型相结合,从而解决这些限制。该模型旨在最大程度地减少复杂性,使用四室组织模型和优化的获取协议,在受试者可接受的扫描时间内实现轴突直径和密度的定向估计。
之后,Zhang等[29]提出神经突起方向离散度与密度成像模型(neurite orientation dispersion and density imaging,NODDI),包含细胞内、细胞外和脑脊液隔室的三室模型。NODDI采用更经济的协议,由两个b值分别为700和2 500 s/mm2的HARDI外壳组成,采集时间缩短至10 min,可以用于临床研究。NODDI模型可由扩散信号估计出4个微观结构参数:轴突内部体积分数(intra-neurite volume fraction,Vic)可以反映神经密度,方向离散度(orientation dispersion index,OD)可以量化轴突方向变化,程度参数表示方向离散的程度,以及脑脊液体积分数(volume fraction of the isotropic compartment, Viso)。
2.3 模型优化
虽然已陆续提出多种隔室模型用于估计纤维方向和纤维结构,但在模型设计、模型拟合的算法以及采集方案上仍有一些优化策略来提高模型的稳定性。
在传统NODDI模型中,Watson分布只能模拟水分子各向同性的扩散分布,而不能模拟纤维呈扇形或交叉的各向异性。为了克服这一局限性,Tariq等[30]提出用Bingham分布替换Watson分布,从而可以对扇形纤维建模,但这不适用于交叉纤维。以往也有采用Bingham分布的混合形式[31-32],在确定每个体素使用Bingham分布的数量以及拟合方法上存在困难,致使在实际情况下难以利用。2017年,Zucchelli等[33]结合FORECAST框架与NODDI技术的优势提出NODDI-SH模型,该模型能够获得准确的微结构信息,包括从交叉到扇形各种纤维分布的体素中细胞内、细胞外和脑脊液体积分数等。与传统NODDI方法相比,NODDI-SH可同时在每个体素中重建纤维方向分布分数,便于纤维示踪成像与脑连接的研究。
除了对模型本身进行改进外,还有部分研究针对模型中算法优化。传统方法是采用非线性程序拟合上述模型,但存在计算复杂度高、拟合时间久、在临床上受试者人数较多难以应用等缺点。Daducci等[34]在2015年提出通过凸优化加速微观结构成像(accelerated microstructure imaging via convex optimization,AMICO)的框架,利用凸优化的通用性,将已有模型重新描述为等价但简单的线性系统,将拟合速度提高了一个数量级,并在ActiveAx和NODDI模型上证明了该框架的有效性, 表1对AMICO框架应用到ActiveAx和NODDI算法与模型原始算法拟合全部体素所需的时间进行比较;ActiveAx原有算法将模型拟合到所有体素(即44个底物×100个噪声试验×6组信噪比)大约花费11天,AMICO框架则可以大大减少计算时间,相同实验数据仅用不到20 s即可完成拟合。原始NODDI算法将模型拟合到所有体素(即80 个底物×6组信噪比×250个梯度方向)所需的时间约为40 h。在相同的数据上,AMICO方法仅用了6 min。2017年,Harms等[35]对最常用的微结构隔室模型NODDI和CHARMED的几种优化算法和初始化策略的性能进行研究,发现无梯度Powell共轭方向算法在运行时间、拟合准确度和精度方面优于其他常用算法。实验在图形处理单元(graphics processing unit, GPU)上实现,大大缩短了运行时间,可以在几秒到几分钟内实现全脑数据集的运算。Farooq等[36]提出一种新的多功能优化技术——交叉微结构成像(microstructure imaging of crossing,MIX),使用已有拟合方法分开考虑,将随机搜索算法和梯度方法组合,能够在多个纤维方向估计详细的轴突特征,实现交叉白质纤维的微结构成像。

表1 实验数据在原始算法和AMICO框架下拟合所需时间[34]Tab.1 Time required for experimental data to fit under the original algorithm and AMICO framework[34]
磁共振信号通过随时间变化磁场梯度进行扩散编码。目前广泛应用的技术使用标准单扩散编码(single diffusion encoding,SDE)序列[1],SDE通常以脉冲梯度自旋回波序列实现。除此之外,还有常用的双扩散编码(double diffusion encoding,DDE)序列[37]、振荡梯度自旋回波(oscillating gradient spin echo,OGSE)序列[38],为目前估计的组织特征提供了更高的灵敏度,以及对新特征的额外敏感性。从微观孔径的分布中,DDE和Q空间轨迹成像可以辨别各向异性,SDE无法区分;DDE还可以增强交换的敏感性,并改善对血容量的估计; 在轴突方向分散或不确定的情况下,OGSE在简单的隔室模型中增强了对轴突直径的敏感性。
2.4 微结构成像数据集与工具箱
脑组织微结构成像是扩散磁共振成像技术发展与临床研究的热点方向,已有多种公开数据集与工具箱供研究使用。
在现有研究中,常用人类连接组项目(Human Connectome Project,HCP)提供的MGH健康成人扩散磁共振数据集[39]作为实验数据,或在框架中提供支持HCP数据的接口。最近人类连接组项目发布WU-Minn HCP完整数据集,包含1 206名健康年轻成人的3 T MR成像数据[40],其中有部分受试者的高角度分辨率扩散成像。UK Biobank作为大规模前瞻性流行病学研究,最近发布10 000个参与者多种模式脑成像的原始数据和处理后数据供研究使用[41]。多维扩散MRI(multi-dimensional diffusion MRI, MUDI)挑战提供了5位健康成人志愿者的全脑MR数据集,数据已经过降噪、运动校正、涡流校正等预处理。
目前已有一些方法和软件从dMRI估计微观结构特征,下面重点介绍几种微结构成像的通用工具箱。Dmipy是基于Python语言开发的微结构成像工具箱[42],它将微结构成像方法模块化,动态地生成现有模型并设计、拟合和重建任何多隔室模型参数。cuDIMOT工具箱利用GPU并行计算体系和CUDA,设计和拟合非线性模型[43]。MIX优化工具箱通用于多隔室模型,特别是在多个纤维方向下估计详细的轴突特征[36]。CAMINO是一个开源工具箱,实现对扩散MRI的重建与处理,还包含Monte-Carlo扩散模拟,在隔室模型中估计轴突密度和直径等[44]。
还有部分工具箱专门用于微结构多隔室模型:NODDI Matlab工具箱在并行多个CPU线程之间,对体素进行拟合[29];AMICO通过凸优化加速微观结构成像,适用于NODDI和ActiveAx技术[34];DSI Studio工具箱支持受限扩散成像方法和CHARMED模型[45]。在CAMINO、AMICO和NODDI中,纤维定向是通过扩散张量估计得到的,其余参数(如指定体积分数、半径指数、密度指数、方向分布等)由非线性回归确定。
2.5 临床应用
基于模型的dMRI对脑组织微观结构的变化提供特异性描述,已初步应用于临床神经系统疾病的研究与诊断中,近年来在脑组织发育、神经系统退行性疾病、神经损失与变性、脑肿瘤等方面极富发展潜力。
2.5.1脑组织发育
Karmacharya等[46]用DTI、NODDI和高斯混合模型评估新生儿白质的发育,并检测与冠心病相关的微结构异常,部分区域FA、Vic、OD等指标随年龄增长而变化,其中高斯混合模型对组织异常最敏感。Kelly通过NODDI和DTI对早产儿和足月儿的脑发育情况进行研究[47],发现早产儿会出现白质区域FA、Vic较低和OD较高的情况。
2.5.2神经系统退行性疾病
NODDI衍生参数为DTI观察白质变化和解释疾病机理提供补充信息。Wen等[48]使用DTI、NODDI和Q空间成像来研究早期阿尔兹海默症(Alzheimer disease,AD)中的白质变化,从轻度认知障碍个体的扣带回、丘脑辐射、大钳观察到较低的FA和较高的OD、MD及径向扩散系数。Parker等[49]在利用 NODDI 研究 38位年轻AD患者和22位正常对照者的灰质皮质时,发现病人组患者组所有区域的OD值均显著降低,某些区域的轴突密度明显降低。大脑皮质的NODDI指标提供了除皮质变薄之外有关AD病理学的信息。
2.5.3神经损失与变性
2019年,NODDI被应用于超急性期、急性期和亚急性期的卒中患者中,以显示其受损的脑区;NODDI与DTI和DKI相比,具有更高的敏感性[50]。Mastropietro等[51]应用 NODDI和DKI,研究慢性卒中的皮质脊髓束微结构变化,发现患病区域持续存在FA减少和OD增加,表明可能与神经变性而引起的纤维减少有关。Irie等[52]对正常颅压脑积水研究发现,皮质脊髓束的OD、Vic降低,FA与ADC值升高。
2.5.4脑肿瘤
NODDI能够反映由于胶质瘤等级不同和细胞增殖引起的结构变化,为临床诊断和治疗胶质瘤提供更多信息。Wen等[53]发现,NODDI能够显示出常规扫描和DTI上未观察到的病变。Ivan等[54]应用DTI、DKI、NODDI三种模型的相应指标,对胶质瘤恶性程度等级进行评估,发现平均峰度、Vic、OD等指标可作为胶质瘤等级标志物,不同等级之间存在显著差异。Li等[55]发现随肿瘤分级的增加,Vic、ODI相应升高,ADC降低;低等级胶质瘤患者与正常人的Vic、OD指标存在显著差异。
3 深度学习算法应用于微结构成像
并行成像等快速磁共振成像技术的出现,有效地加快了MRI扫描和图像重建;基于压缩感知理论的快速磁共振重建方法,利用磁共振图像在变换域中的稀疏性来减少采集样本数,从而缩短扫描时间。深度学习算法已成功应用于计算机视觉领域[56],用其从dMRI数据中解码微结构信息受到越来越多的关注,在磁共振成像重建时间及重建准确性上显现出特有的优势[57-58]。
传统微结构成像方法通过扩散加权成像数据拟合模型,最终获取微结构参数,往往需要多个步骤,每一步信息会因自由度的降低而部分丢失,但是对于如何选取要保留或丢弃的信息,传统方法无法提供后续步骤到早期步骤的反馈。深度学习算法则提供更为灵活的解决方案:图像本身可以用于学习过程中,不需要进行特征提取,将每一层的计算结果反向传播,以调整前面层的权重,根据最终目标对所有层共同优化。
NODDI与AMICO框架都需要在大量的扩散梯度下才能够准确估计微结构,在临床应用上受限。Golkov等[59]开发了一种基于深度学习的方法用于NODDI微观结构估计,该方法使用多层感知机(multilayer perceptron, MLP),在减少扩散梯度数量的情况下仍能够进行准确预测。深度网络完成训练后,数据仅需通过网络正向传递,因此计算速度明显优于NODDI和AMICO框架,但是无法获取二维空间邻域信息。Gibbons等[60]开发一种多维深度学习方法,在二维卷积神经网络训练后,能够从高度欠采样的Q空间数据中,同时生成NODDI指标和广义各向异性分数参数。卷积神经网络在更大范围上获取空间邻域信息,可提高参数估计性能。此方法适用于临床脑卒中成像,在临床允许的时间范围、较少采样数量下,仍能提供准确度较高的参数估计。Nath提出了一种新的数据驱动方法,探索白质纤维重建的未开发信息[61],利用残差块深度神经网络,对观测的dMRI信号与真实结构之间的非线性映射进行建模,在多个数据集上进行验证,并与其他微结构重建方法进行比较。
基于优化学习深度网络通过展开传统的迭代优化过程进行构建,已成功应用于求解正则化最小二乘问题[62-63],在速度和精度上都显现出优势。Ye等[64-65]受AMICO框架启发,提出一种深度网络的方法MEDN(microstructure estimation using a deep network,MEDN),基于优化学习的策略与NODDI模型估计微观结构。MEDN利用字典框架对扩散信号建模:首先利用展开和截断的迭代过程,解决稀疏的重建问题;之后将上一阶段的输出作为输入,计算最终的微结构参数;结果显示,MEDN的准确性高于Golkov等的MLP算法。随后,Ye等[66-67]提出基于改进的长短时记忆神经单元的深度网络,扩散信号在空间域和角域共同稀疏表示,网络将历史信息自适应于迭代更新中,以解决稀疏重建问题,再将稀疏表示映射到组织微结构测量,并在NODDI模型、球面平均技术模型上进行评估。
深度学习算法应用于微结构成像,存在着一定的局限性。首先,尽管深度学习算法能够对某些组织微结构参数进行准确估计,但如何做出估计却是一个黑匣子;其次,网络的最佳结构和超参数,以及训练模型所需的训练集数量因任务而异。在一项任务中,实现了高性能的超参数不一定会在其他任务中获得良好的性能。虽然深度学习算法已经成功地应用到一些任务中,但目前建立的网络只适用于解决单一数据类型下的一类微结构参数估计,所以仍不具有通用性。
4 结论与展望
在微结构成像技术中,多隔室模型近年来广受关注,许多学者对此进行了研究,衍生出多种不同模型及优化算法,用于组织微结构信息检测。发展至今,微结构成像已经成为扩散磁共振成像中一个重要方法,且被用于脑发育及老化、神经损伤、肿瘤性疾病等应用研究中。未来的研究可以着眼于以下几个方面:
1)扩散磁共振成像利用传统的PGSE序列估计组织特征,而非PGSE编码方案可能对组织特征有更高的敏感性,或可挖掘到微观结构新的特征信息[68]。例如,双扩散编码和Q空间轨迹编码从微观孔径的分布来辨别组织中的各向异性,振荡梯度自旋回波序列提高了简单隔室模型轴突直径的敏感性。这一方向是目前非常活跃的研究领域,并有希望提出能够被广泛应用的实际方法。
2)虽然研究者已经通过多隔室模型对微观结构参数进行估算,但还出现了一些异常结果,如模型估计出的轴突直径高于组织学测量的3倍以上[69-70]。考虑这些异常与模型本身设计有关,存在模型假设不合理的情况,如固定扩散系数、轴突建模为理想的直圆柱型等,还忽略了隔室交换、树突分支、轴突起伏和神经胶质细胞等的影响。目前,仍缺乏能够描述真实物理效应的生物物理模型,未来模型设计需要进一步考虑组织结构的实际形态与细节描述,以便从dMRI获取微结构特征的更多准确信息。
3) 微结构成像技术在大脑的应用正在不断增加,使用不同的模型来对信号加以解释,并且将这一思路延伸到大脑之外的应用。可根据大脑的研究模式对其他组织成像进行解释,如心脏[71]、胎盘[72]、肝脏[73]、肾脏[74]、肌肉组织[75]等,这在部分研究中已经显现出较好结果。利用微结构成像,以非侵入的方式,帮助临床医生在疾病诊断、疗效进展、辅助手术等方面提供更多有用信息。

