基于加权基因共表达网络分析筛选结肠腺癌预后关键基因
李卓阳, 张皓旻, 刘格良, 陈浩然, 智 鹏, 陈熙勐, 卢学春, 贺培凤
1.山西医科大学管理学院,山西 太原 030001;2.中国人民解放军总医院第二医学中心血液科 国家老年疾病临床医学研究中心
结肠癌是最常见的恶性肿瘤之一,是我国恶性肿瘤死亡的第4常见原因[1]。结肠腺癌(colon adenocarcinoma,COAD)是发生于腺上皮细胞的恶性肿瘤,是结肠癌最主要的病理类型之一,目前的治疗手段包括外科手术切除、化学疗法、免疫疗法等。由于复发等因素,术后患者的5年生存率仅为60%~70%[1-3],提高患者的生存优势仍是一个挑战。
全转录组测序(RNA-seq)可同时检测全部已知基因的表达模式,通过差异表达基因分析,可鉴定肿瘤与正常组织之间的差异表达基因[4]。近年来,研究者基于癌症基因图谱(The Cancer Genome Atlas,TCGA)、基因表达综合(Gene Expression Omnibus,GEO)等开源数据库开展的生物信息分析发现,COAD的发生与大量基因的显著变化有关。同时,基因间调控机制复杂,且其发展涉及多种信号通路异常[5-7]。目前,结肠癌已有预测预后基因的筛选研究,但未见基于基因表达与临床表型关系筛选预后基因的相关研究。加权基因共表达网络分析(weighted gene co-expression network analysis,WGCNA)是一种根据表达谱数据探索特定基因模块与临床表型间相关关系的系统生物学方法,可用于鉴定候选生物标志物、预后基因及治疗靶标[8-9]。
本研究应用WGCNA方法,对来自TCGA和GEO数据库的COAD RNA-seq数据,构建COAD的差异基因共表达网络,筛选与COAD发生发展密切相关的基因模块,随后结合Kaplan-Meier方法鉴定COAD预后基因,为COAD预后相关基因的进一步基础及临床研究提供方向和指导。
1 材料与方法
1.1 数据获取从TCGA数据库[10]中获取COAD转录组数据和相应的临床信息。其中,转录组数据包括398例COAD样本和39例正常结肠组织样本;临床信息包括COAD患者的生存状态和生存时间。
从GEO数据库[11]中以“colon adenocarcinoma”检索COAD基因表达谱数据。筛选条件包括:研究物种为人或小鼠;全基因组表达芯片数据或转录组测序数据;生物学样本组织来源类型一致;有对照组;每组至少3个生物学重复;实验设计思路清晰以及数据质量良好。最终筛选得到GSE110224数据集[12]。该数据集包括17例原发性COAD样本和17例配对的正常结肠组织样本,基于GPL570平台分析原发性COAD的整体基因表达变化。
1.2 差异表达基因分析采用R语言软件包limma,分别对来自TCGA和GEO的转录组数据进行数据标准化和差异表达基因(differentially expressed genes,DEGs)分析,得到两组DEGs。差异表达基因的筛选标准,TCGA为|logFC|≥1、FDR<0.05;GEO为|logFC|≥1、FDR<0.05。当多个探针与一个相同的基因匹配时,则以平均值作为该基因的表达值。最后采用R语言软件包ggplot2对DEGs的表达模式进行可视化。
1.3 加权基因共表达网络分析采用R语言软件包WGCNA,分别对来自TCGA和GEO的转录组数据进行加权基因共表达网络分析。填补缺失值后,构建邻接矩阵并转换为拓扑重叠矩阵。采用动态剪切法构建基因聚类树状图,将表达相似的基因聚类为不同的基因共表达模块,规定每个模块最少基因数目为50。随后采用Pearson相关分析计算每个基因与各个模块、不同临床特征基因显著性的相关系数r值和P值,获取肿瘤相关性最高的模块及该模块中的基因。最后采用R语言软件包VennDiagram,将相关系数最高模块中的基因与TCGA、GEO的DEGs取交集,得到最终的DEGs,用以后续分析。
1.4 GO富集和KEGG通路富集分析采用R语言软件包clusterProfiler,对上述DEGs进行GO[13-14]富集分析和KEGG通路[15]富集分析。以FDR<0.05、P<0.05为阈值,筛选富集的GO条目和KEGG通路。
1.5 蛋白互作网络构建及COAD预后相关的核心基因筛选采用STRING v11.0开源数据库(https://string-db.org/cgi/input.pl)构建蛋白互作(protein-protein interaction,PPI)网络,以描述DEGs编码蛋白之间的相互作用关系。选择物种为“Homosapiens”,设置可靠性阈值>0.4,去除游离节点后下载PPI网络数据。将数据导入Cytoscape软件对PPI网络进行可视化,并使用Cytohubba插件中的MMC算法,筛选值最高的10个节点作为COAD预后相关的核心基因。
1.6 生存分析确定COAD预后相关的关键基因采用R软件语言包survival,基于TCGA数据库的COAD基因表达谱数据和临床信息(患者生存状态和生存时间),通过Kaplan-Meier生存分析法,分析与患者总生存期(overall survival, OS)显著相关的基因,以初步筛选出与患者预后不良有关的基因。随后采用在线分析工具GEPIA2(http://gepia2.cancer-pku.cn/),分析上述10个核心基因与患者无病生存期(disease-free survival,DFS)之间的关系。以P<0.05为阈值,筛选同时与OS和DFS有关的基因,作为COAD预后相关的关键基因。
1.7 HPA数据库验证关键基因的蛋白质表达人类蛋白质图谱(The Human Protein Atlas,HPA)(https://www.proteinatlas.org/)数据库是利用转录组学和蛋白质组学技术,从RNA和蛋白水平研究人类不同组织和器官中的蛋白表达情况。采用HPA数据库,分析关键基因所编码的蛋白在肿瘤组织和正常组织中的不同表达之处,进一步明确与COAD预后相关的关键基因。
2 结果
2.1 差异表达基因筛选从TCGA数据库下载COAD数据集,经分析得到差异表达基因3 544个,其中上调基因1 293个,下调基因2 251个。从GEO数据库下载GSE110224数据集,经分析得到差异表达基因515个,其中上调基因223个,下调基因292个(见图1)。
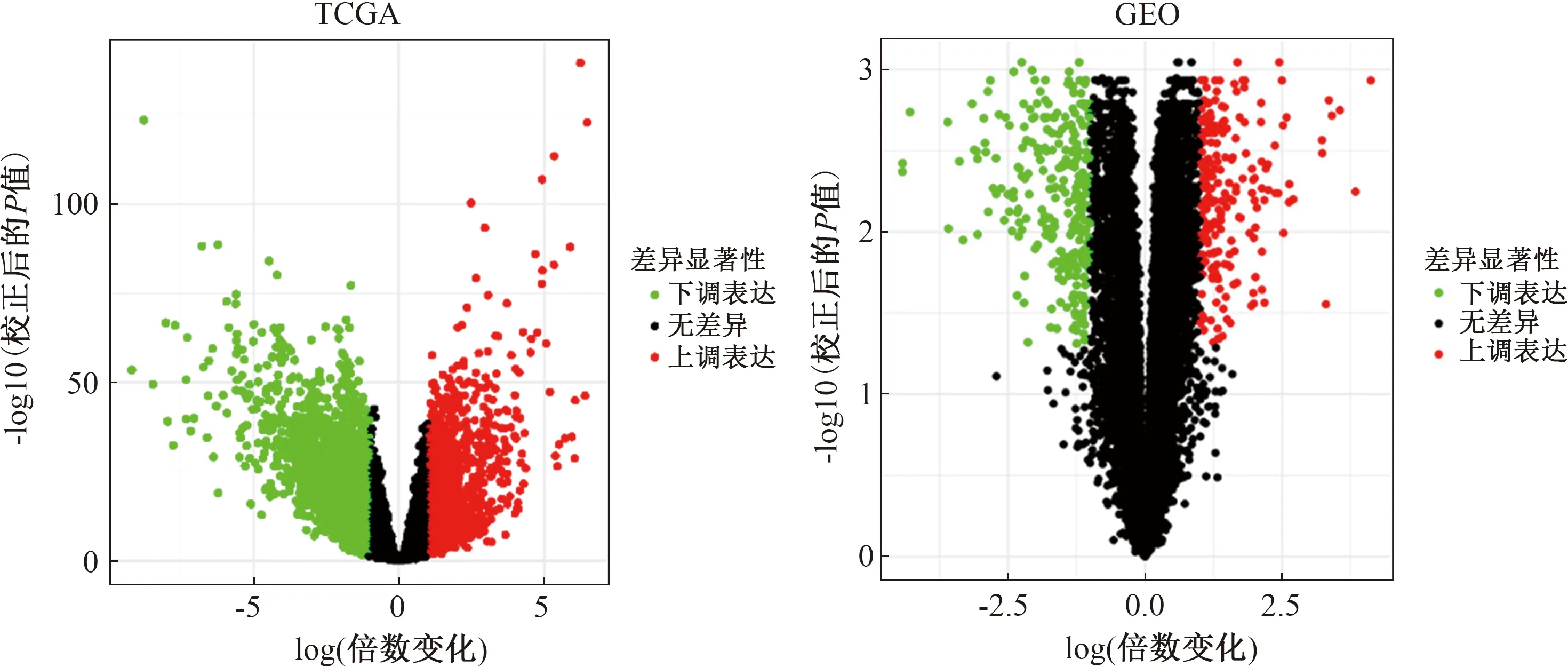
图1 TCGA和GEO中COAD的DEGs火山图
2.2 加权基因共表达网络分析基于加权基因共表达网络分析,来自TCGA和GEO的所有基因分别被分为20个和18个模块(见图2~3)。如图所示,与肿瘤正相关性最强的模块分别是TCGA黄色(r=0.54,P=1e-34)和GEO棕色(r=0.65,P=3e-05),与肿瘤负相关性最强的模块分别是TCGA棕色(r=-0.87,P=1e-135)和GEO青色(r=-0.63,P=7e-05)。分别获取模块TCGA黄色和GEO棕色、TCGA棕色和GEO青色的共同基因,将两组基因合并后再与TCGA和GEO的DEGs取交集,得到最终的153个DEGs,进行后续分析(见图4)。
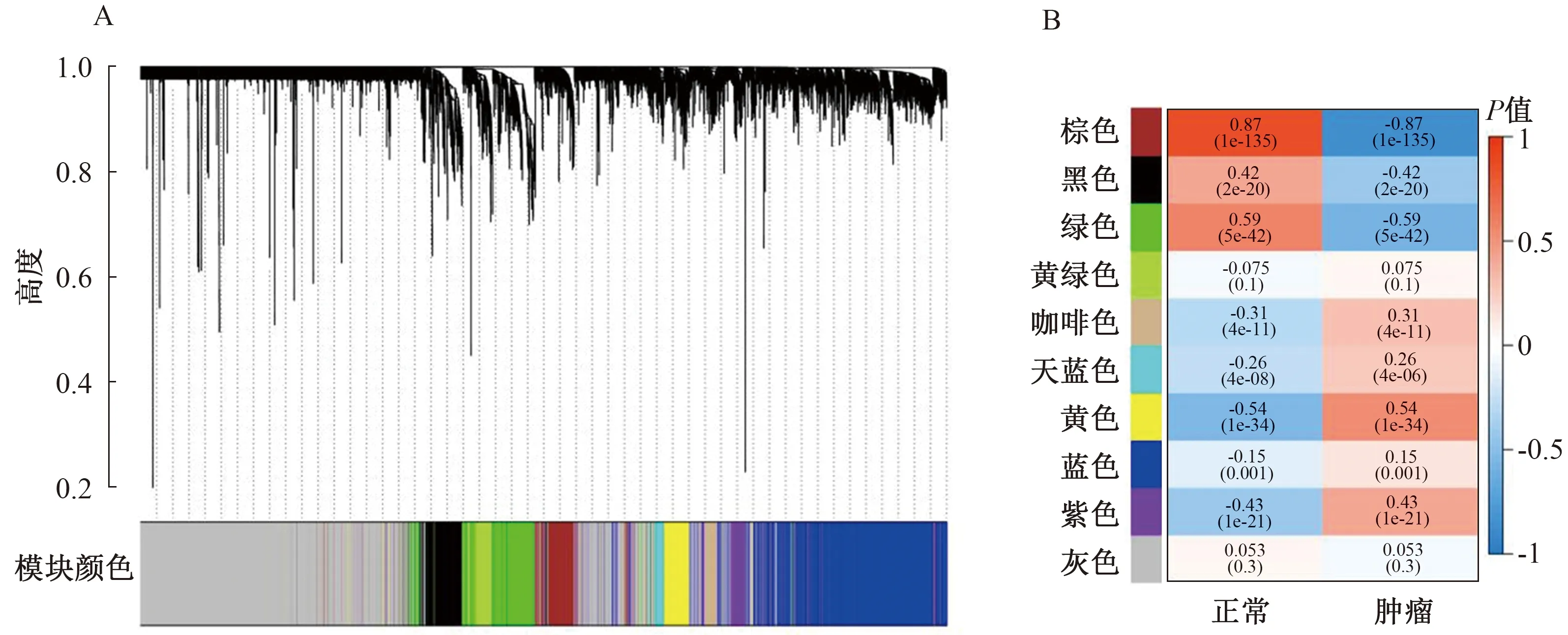
注:A:基因聚类树状图;B:基因模块与肿瘤之间相关性的热图。
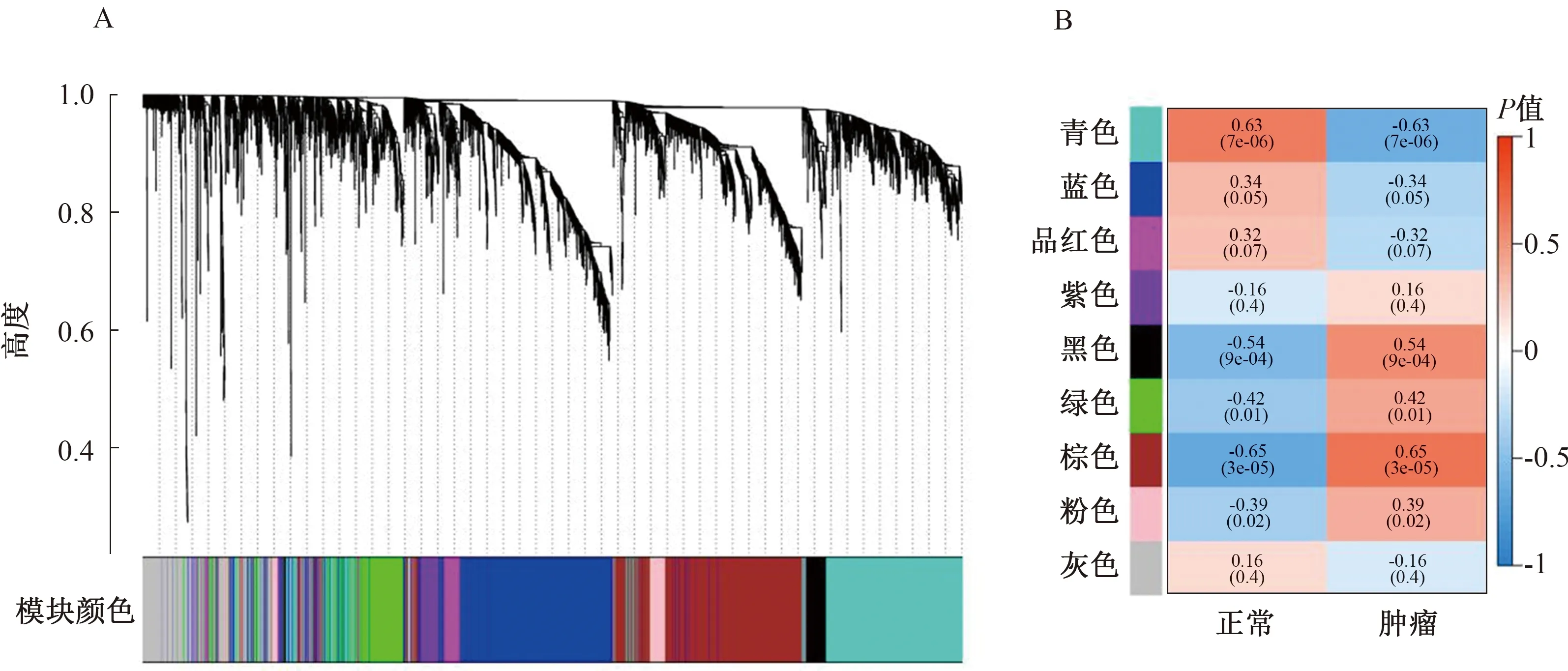
注:A:基因聚类树状图;B:特征基因模块与COAD关系图。
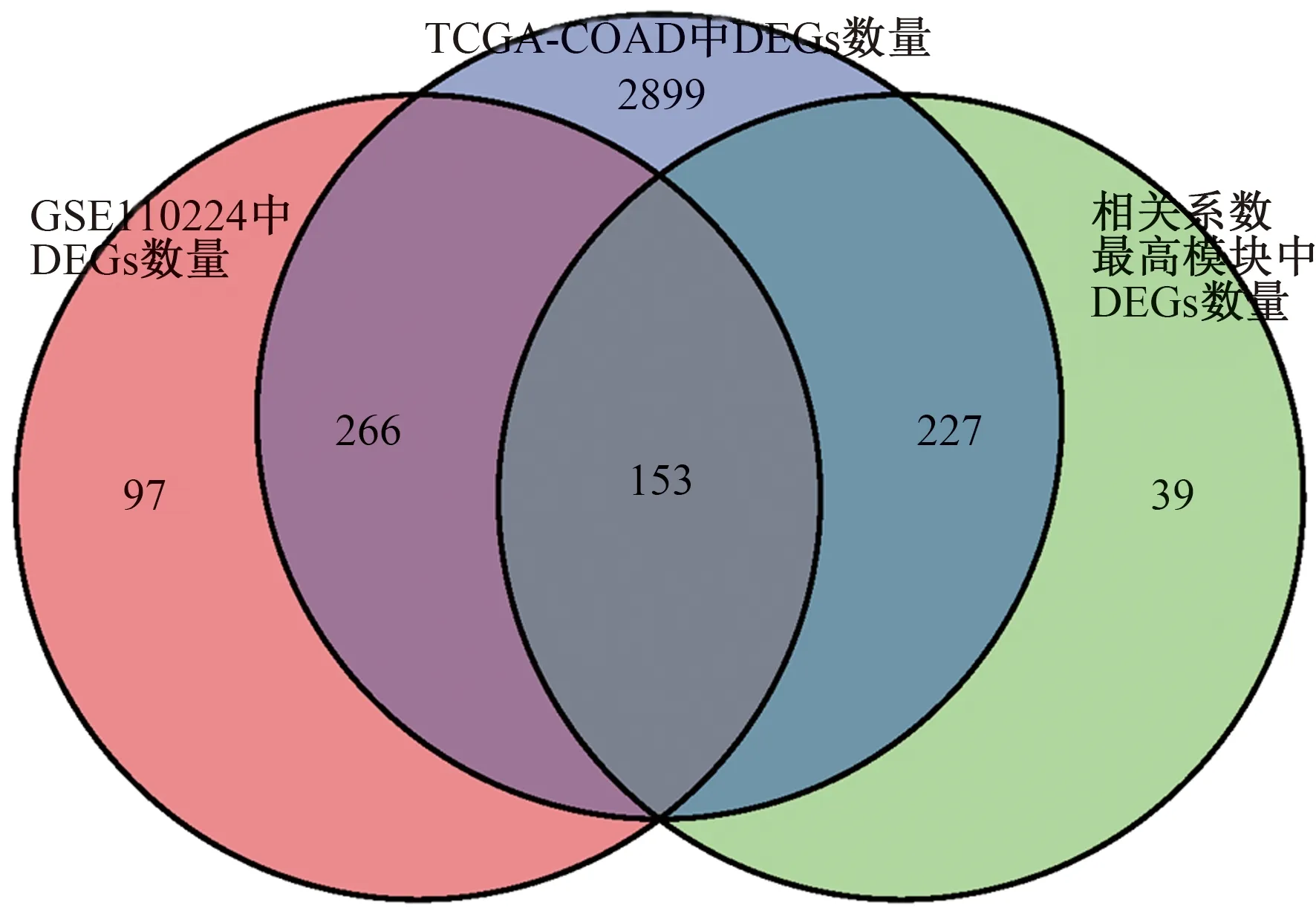
图4 DEGs与WGCNA模块中基因的韦恩图
2.3 GO富集和KEGG通路富集分析GO富集从三个方面注释了基因的生物学特性:生物学进程(biological process,BP)、细胞成分(molecular function,CC)和分子功能(molecular function,MF)(见图5)。其中,BP集中于离子运输、激素代谢等;CC主要与细胞的顶端部分、顶质膜、刷状缘、微绒毛等细胞结构有关;MF则主要与离子和分子跨膜转运蛋白,以及碳酸盐脱水酶活性、类固醇脱水酶活性等有关。
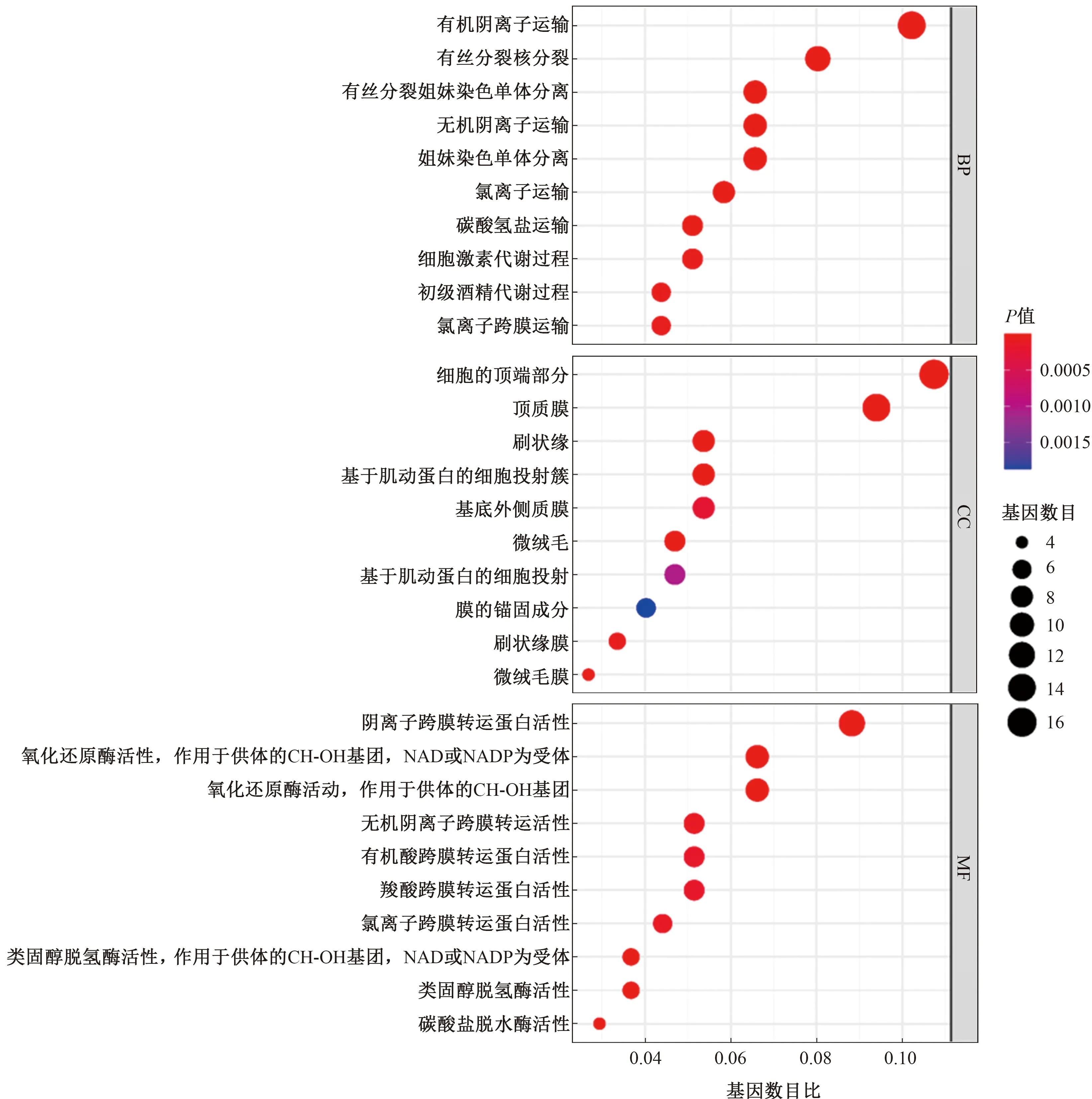
注:横坐标代表富集在各GO条目的基因数目比率,纵坐标代表GO条目名称。
筛选得到KEGG通路富集有21条(见图6),发现这些基因主要参与胆汁分泌、类固醇激素合成、戊糖和葡萄糖醛酸酯相互转化,以及氮、视黄醇、络氨酸、丙酮酸等代谢过程。
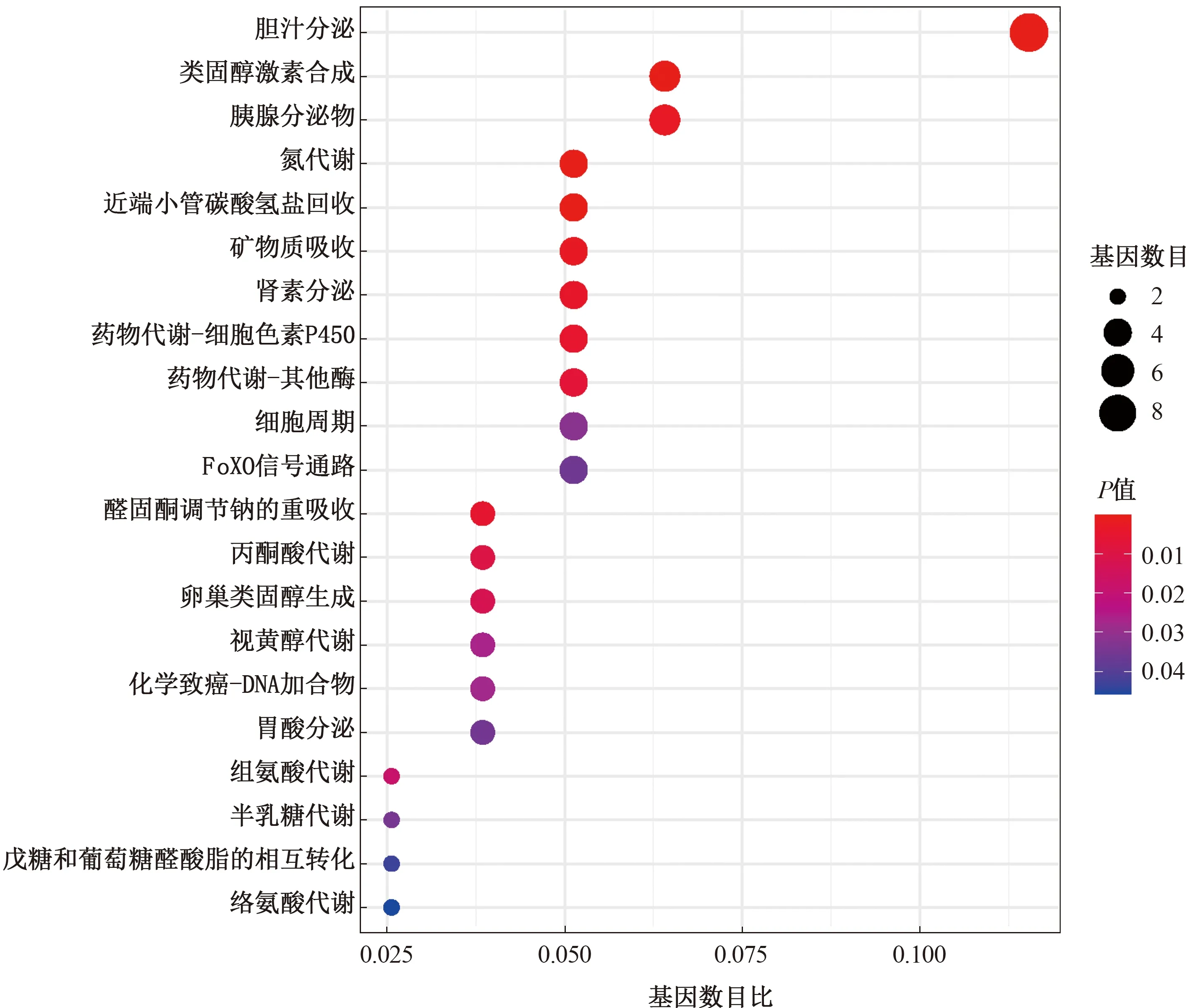
注:横坐标代表富集在各通路的基因数目比率,纵坐标代表通路名称。
2.4 PPI网络构建和COAD预后相关核心基因筛选PPI网络中共出现153个节点和385条连线(见图7A)。根据Closeness算法,凝聚素Ⅰ复合物亚基G(non-SMC condensin I complex subunit G,NCAPG)、细胞分裂周期6(cell division cycle 6 homolog,CDC6)、核受体亚家族1,组H,成员4(nuclear receptor subfamily 1, group H, member 4,NR1H4)、甲状腺素受体结合因子13(thyroid hormone receptor interactor 13,TRIP13)、氯离子通道辅助蛋白1(chloride channel, calcium activated, family member 1,CLCA1)、肠促胰高素样肽1类似物胰高血糖素(glucagon,GCG)、核苷酸还原酶M2肽(ribonucleotide reductase M2 polypeptide,RRM2)、蔗糖酶异麦芽糖酶复合物(sucrase isomaltase,SI)、周期蛋白B1(Cyclin B1,CCNB1)、叉头框M1(forkhead box M1,FOXM1)等10个基因为其中的核心基因(见图7B)。

注:A:153个DEGs的PPI网络;B:153个DEGs的核心基因。
2.5 关键基因的确定与验证10个核心基因中,OS分析结果显示,CLCA1低表达患者的OS明显比高表达组短(P<0.001,见图8);DFS分析结果显示,TRIP3低表达、CLCA1低表达与患者的DFS显著相关(P<0.05,见图9)。选择CLCA1基因作为关键基因。根据HPA数据库,与正常结肠组织相比,肿瘤组织中CLCA1基因的蛋白质水平显著降低(见图10)。
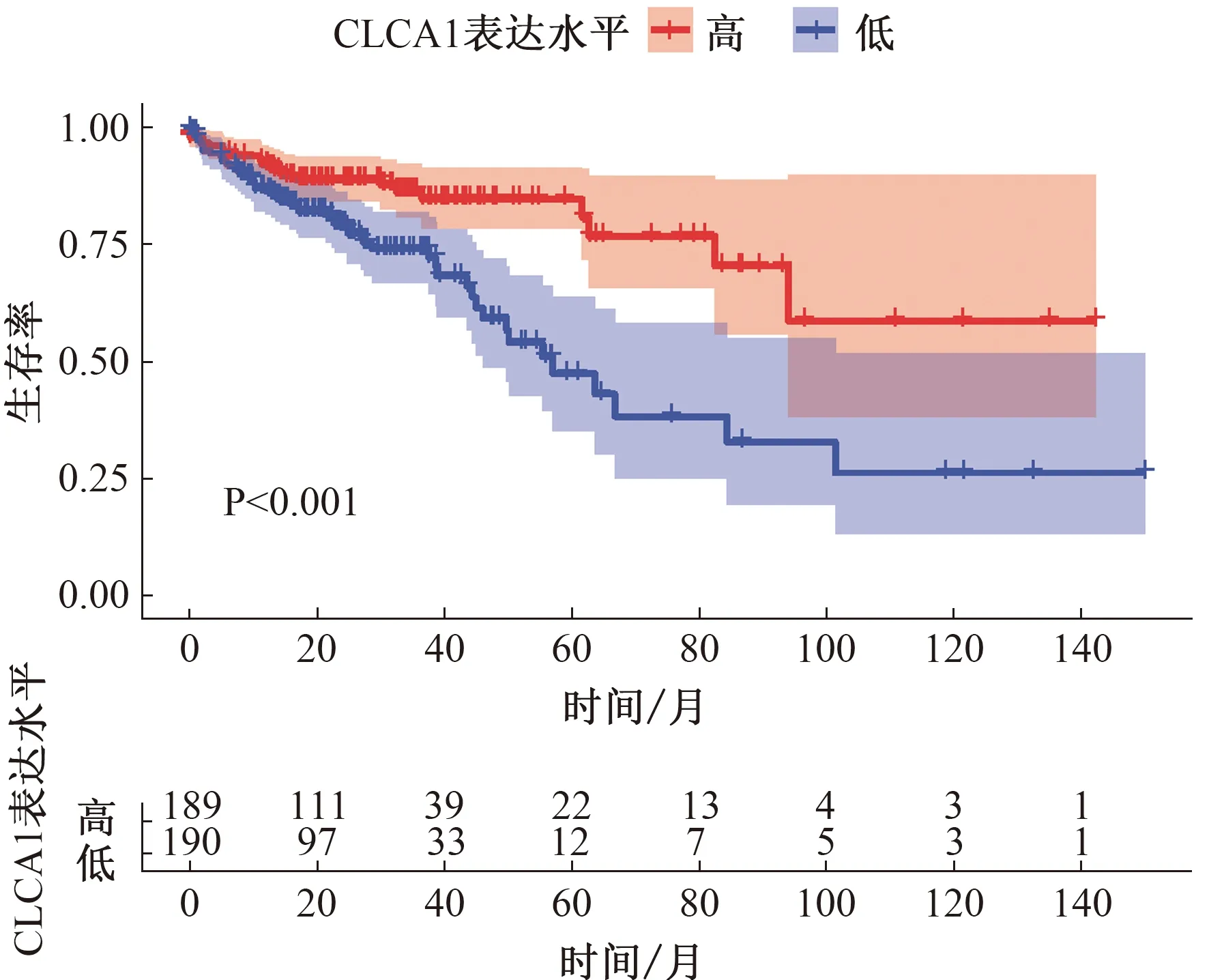
图8 OS分析结果
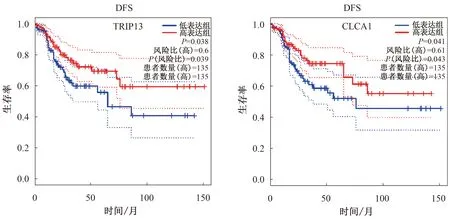
图9 10个核心基因的DFS分析结果
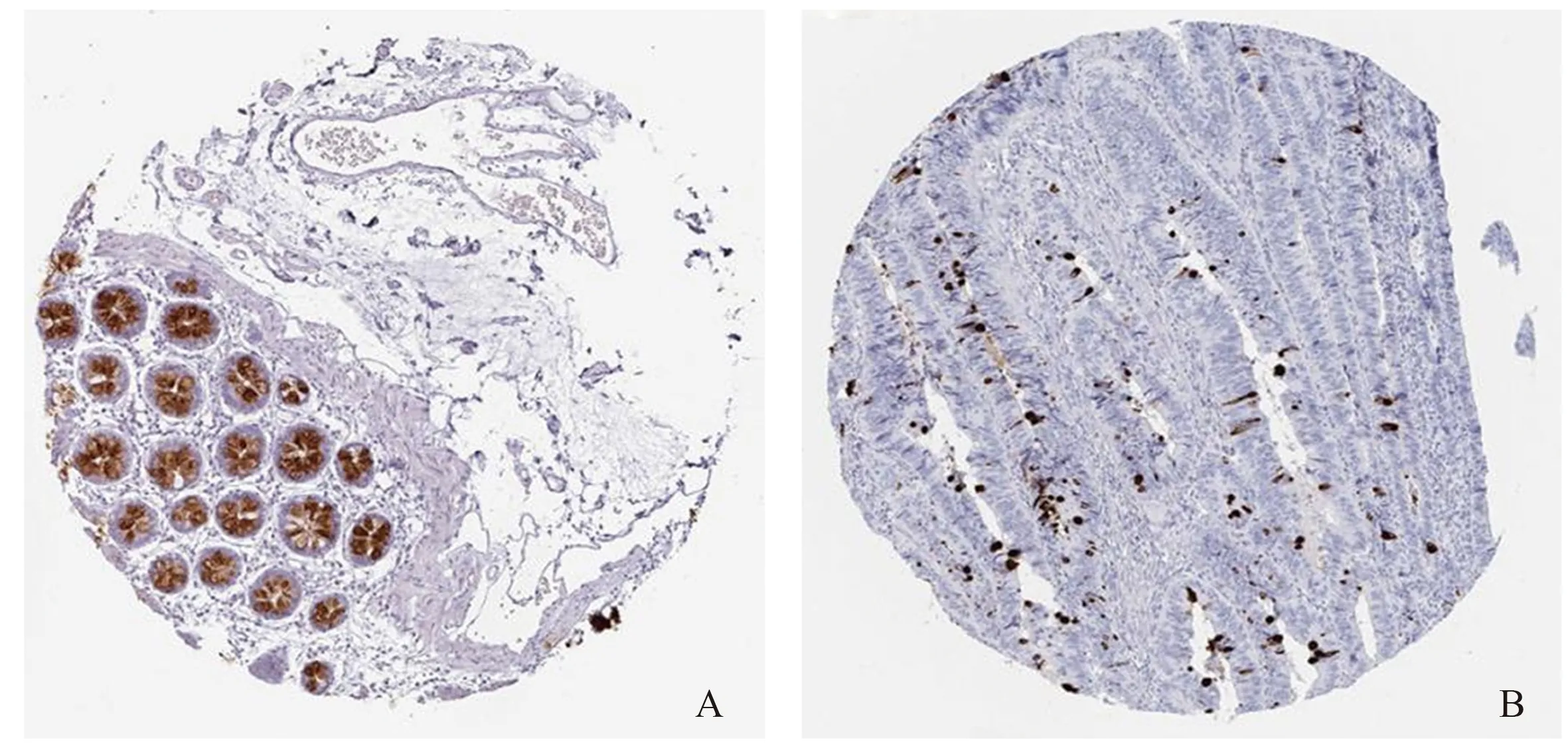
注:A;正常结肠组织,B;结肠腺癌组织。
3 讨论
本研究通过对TCGA和GEO数据库中的COAD转录组数据进行差异表达基因分析和加权基因共表达网络分析,发现COAD主要与患者体内的153个基因异常表达有关。富集分析发现这些基因大多与离子运输、激素代谢等生命活动有关。最后通过蛋白互作网络分析和生存分析,发现CLCA1基因与COAD患者的不良预后显著相关。
细胞内离子通道在所有细胞中无处不在,研究表明包括K+、Cl-、Ca2+和Na+在内的离子通道在胃肠道癌症中均有表达和失调,这可能是导致正常细胞向癌细胞转化的重要原因[16]。本研究中DEGs主要与无机和有机离子通道的激活和抑制等生物学进程有关,提示离子通道的异常表达或功能障碍对COAD患者体内癌细胞转化、侵袭和转移等过程具有重要作用。Warburg等发现癌细胞会比正常细胞消耗更多的葡萄糖[17]。葡萄糖进入细胞后参与细胞质内的糖酵解活动,其终产物丙酮酸经过酶促反应转化生产乳酸;乳酸可通过多种机制促进肿瘤的血管形成、细胞迁移和逃避免疫监视[18]。本研究中部分DEGs参与丙酮酸代谢活动,这提示癌细胞可能是通过糖酵解途径改变机体内能量代谢方式,从而促进COAD的发生和发展。此外,本研究中部分DEGs富集于胆汁分泌信号通路,提示COAD患者的病情进展与胆汁分泌具有密切联系。已有研究证实,相对于健康人群,结直肠癌患者的肠道微生物组群落发生变化[19-20]。Ridlon等[21]发现,饮食中饱和脂肪会诱导胆汁分泌增多,进入肠道后产生脱氧胆酸和石胆酸等二级胆汁酸,激活细胞信号级联反应,从而促进结直肠癌细胞的增殖和迁徙。
钙激活的氯离子通道蛋白参与细胞内信号传导并激活特定的细胞应答,如与癌症相关的增殖、凋亡、迁移和血管生成[22],并被认为是新兴的药物靶点[23-24]。CLCA1基因是钙激活的CLCA家族成员,胃肠道中CLCA1主要在小肠、结肠和阑尾表达,为胃肠道提供防护作用。目前,针对CLCA1在结直肠癌中的机制已有研究。CLCA1可以通过提高黏附分子E-cadherin和肠碱性磷酸酶的表达从而促进肠上皮分化[25],而敲除CLCA1(Caco-2细胞系)则可以抑制细胞分化并促进细胞增殖[26]。同时,有研究发现CLCA1表达水平的升高可抑制Wnt信号通路和上皮-间充质转化(epithelial-mesenchymal transition,EMT)过程,表明该基因具有肿瘤抑制作用[25,27-28]。此外,c-myc这一原癌基因的产物参与细胞增殖和凋亡的调节,有研究发现CLCA1的转录则与c-myc的转录具有一定相关性[29],但其中具体机制仍有待明确。
寻找新的治疗靶点一直是结直肠癌研究的关注要点。目前已有包括西妥昔单抗、帕尼单抗在内的以EGFR为靶点的药物被批准应用于临床中的靶向治疗,但患者仍表现出耐药性[30]。离子通道的功能表达及其受类固醇激素和生长因子的调控是卵巢癌发生发展的重要组成部分,也可能与患者产生耐药性有关[31]。Musrap等[32]发现,CLCA1是聚集形成的卵巢癌细胞中上调较多的蛋白;而使用氯离子通道阻滞剂或敲除CLCA1则会降低癌细胞形成聚集体的能力,表明CLCA1可能是恶性肿瘤新的治疗靶点[33]。CLCA1的低表达与结直肠癌的低生存率和高疾病复发率有关[22],将其作为靶标进行靶向药物研发,有望为COAD的治疗提供新方法。
本文的不足之处在于,虽基于WGCNA方法对COAD潜在的预后基因展开了一系列的生物信息学分析和预测,但该分析结果仍需进一步的基础或临床实验加以验证。
综上,进一步明确CLCA1对于COAD发生和发展的具体机制,可能有利于提高临床预后的判断力和个性化治疗方案的优化。建议将CLCA1作为靶点进行针对性的临床研究和靶向药物研发,使得更多COAD患者获益。

