基于流式细胞术和基因组Surve y的花榈木基因组大小及特征分析
夏诗琪,欧阳天林,温 强,周成钏,楼浙辉,宋 颖,3,刘丽婷★
(1.江西省林业科学院,江西 南昌 330013;2.江西省林业科技实验中心,江西 赣州 341600;3.华南农业大学·林学与风景园林学院,广东 广州 510642)
花榈木(Ormosia henryi)为豆科(Leguminosae)红豆属(Ormosia)常绿乔木,在中国属珍贵树种,多生于亚热带地区的低海拔常绿阔叶林中。花榈木树形优美,树姿挺拔,是优良的园林绿化或防火树种。花榈木适合材用,其木质细腻,纹理精美。在药用价值方面,其全株均可入药,具有治疗抑郁、镇定安神等多种功效[1-2]。近年来,由于人类对花榈木资源的不合理开发利用,花榈木的野生资源已处于濒危状态,同时因其种子种皮致密坚硬,不易萌芽,自然繁殖能力较弱,当前花榈木的野外种群数量锐减[3],现已被列为国家二级重点保护野生植物。
现有文献表明,国内外对花榈木的研究多集中在种苗扩繁、药效分析[4-5]、遗传分析、资源分布及评价[6-8]等领域,在遗传特性方面的研究还较少。为进一步对花榈木群体遗传学等领域开展研究,发掘、保护和利用其遗传资源,有必要开展花榈木基因组的研究。
基因组大小(DNA C值)是一个评价生物单倍体细胞核DNA含量的关键指标[9],基因组特征研究是植物基因资源开发和分子机制研究的前提[10-11]。目前测定基因组大小的方法主要有流式细胞术和基于K-mer分析的基因组Survey测序。流式细胞术常用于评估基因组大小及倍性水平,基因组Survey测序具有速度快且数据量大等优点[12-13]。近年来,基于流式细胞术及Genome Survey技术的豆科植物基因组研究已逐渐开展,但多集中在草本植物,如大豆(Glycine max)、绿豆(Vigna radiata)、苜蓿(Medicago sativa)、鹰嘴豆(Cicer arietinum)等均已完成了全基因测序[14],对豆科中木本植物的基因组研究还较少。本研究以花榈木为材料,采取流式细胞术和基于K-mer分析的基因组Survey测序两种方法,估测花榈木基因组大小,并获得基本特征信息,以期为花榈木的系统进化和基因资源开发等研究奠定基础。
1 材料与方法
1.1 材料
花榈木样品采自江西省林业科学院苗圃,取健康花榈木植株的新鲜叶片,经液氮速冻后置于-80℃超低温冰箱保存,备用。内参植物为豌豆(Pisum sativum)。
1.2 方法
1.2.1 细胞核悬浮液制备
细胞核悬浮液的制备是流式细胞术的基础。取0.8 mL mGb解离液进行预冷,加入1 g待测花榈木样品并迅速垂直切碎其组织,使之与解离液充分接触。静置10 min后用400目滤网过滤至离心管中,经离心后弃上清液,收集细胞核沉淀再加入200μL解离液即得到细胞核悬浮液。选用非特异染料碘化丙啶(propidium iodide,PI)对细胞核DNA进行荧光标记,在暗处染色20 min后备用。
1.2.2 流式细胞仪检测
单独测定内参植物和待测植物的荧光峰强度,并以此为依据,调整二者的混合比例,使共进样品时两种植物细胞核浓度一致,然后进行检测。利用BD FACScalibur流式细胞仪对染色后的细胞核悬浮液样品上机检测,电压为290 V,采用488 nm蓝光激发,检测PI的荧光强度,每次检测收集参数设定为10 000。变异系数(coeffcient of variation,CV)需要控制在5%以内[15]。使用Modifit 3.0分析软件作图分析。
1.2.3 基因组大小计算
PI染色时,其嵌入量与DNA含量呈正比,因对照样品的基因组大小已知,根据待测植物与内参植物的荧光比值即可测定待测植物的DNA含量。计算公式为:待测植物DNA含量=内参植物DNA含量×待测植物的荧光强度/内参植物的荧光强度。观察花榈木和豌豆PI-DNA复合体的荧光峰值,得出二者DNA含量的比值,再与豌豆的C值相乘,即可计算出花榈木的C值。
1.2.4 基因组DNA提取与建库测序
采用改良CTAB法进行花榈木基因组DNA提取,DNA质量检测合格后,经Covaris超声波破碎仪随机打断,构建插入片段为400 bp的DNA文库,在Illumina Hiseq X-ten上进行双末端(Paired-End)测序,经SOAP nuke v1.6.5软件质控过滤后得到高质量数据,利用SOAP de novo进行数据组装,采用模拟数据拟合的方法评估基因组杂合率,用CASAVA软件对原始图像数据进行识别。
1.2.5 基因组大小预测和杂合度估计
将测序所得的高质量数据用于K-mer分析,K-mer是从测序数据中提取出的长度为K的寡聚核苷酸序列[16]。本研究取K=41进行分析,对序列进行频率作图,得到K-mer分布曲线,根据公式计算基因组大小(基因组大小=K-mer总数/K-mer期望深度)。基因组重复序列比例根据K-mer曲线分布图的拖尾现象评估,基因组的杂合率通过杂合峰值/纯合峰值来确定。
1.2.6 样品污染评估
样品污染问题在基因组研究中有着决定性的地位[17],在开展基因组调查前,需查明所提取的样品DNA是否存在污染物种。本研究中,对过滤后的高质量数据随机抽取10 000条reads(read1和read2各5 000条)数据,运用Blast软件与NCBI核苷酸数据库(NT库)进行比对,计算与NT库比对上的reads占总reads数目的比例,查看比对上的物种是否为样本的近缘物种,以此判断样品有无污染。在NT库比对结果中,若为同源比对,则认为样品材料未被污染。
1.2.7 GC含量分布分析
物种GC(碱基对)含量是评估调研图分析准确性和后续基因组精细组装难度的重要指标之一[18]。利用contigs覆盖深度分布与GC含量分布构建GCdepth关联图进行分析。
2 结果与分析
2.1 流式细胞术测定花榈木基因组大小
使用流式细胞术测定物种基因组大小时,常使用内参法。本研究以已知基因组大小的豌豆(基因组大小为4.45 Gb)为内参植物,进行细胞流式实验,估算花榈木的基因组大小。同时对豌豆和花榈木的PI发射荧光强度进行测定分析,得到图1。由图1可知,豌豆和花榈木所呈现的峰在形状上均尖而细,碎片背景也非常少,二者测定峰的位置没有重叠干扰,保证了用豌豆做内参的准确性。经2次重复测定,得到花榈木与内参植物豌豆荧光强度的比值,由此测算得到花榈木的基因组大小为2.99 Gb和3.01 Gb(表1)。
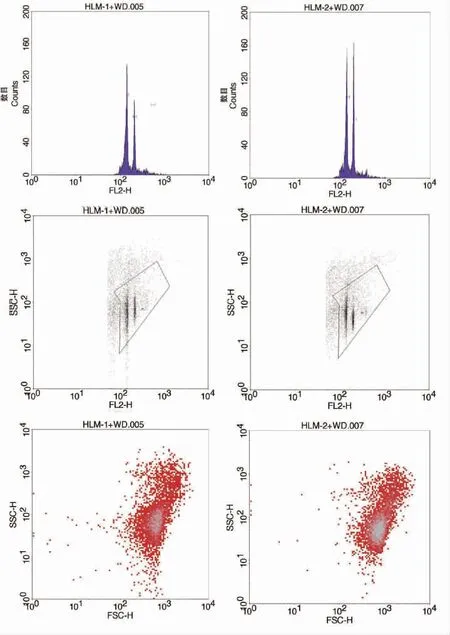
图1 花榈木流式细胞术检测Fig.1 Flow cytometry detection of Ormosia henryi
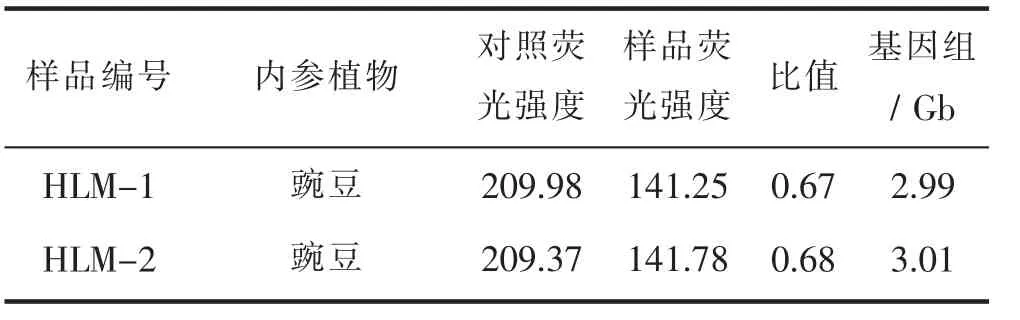
表1 花榈木基因组测定Tab.1 O.henryi genomic assay
2.2 Genome Survey测序数据统计及质量评估
基于Illumina Hiseq平台进行双PE150测序,获得花榈木reads数量190 771 231对,共57.23 Gb原始数据(表2)。以Q20与Q30为指标衡量测序质量,其中,Q20比率为96.47%、Q30比率为90.68%,测序错误率正常(<0.05%)。

表2 花榈木测序数据统计Tab.2 Sequencing data statistics of O.henryi
2.3 K-mer分析以及基因组大小估计
利用K-mer的分析方法来预测花榈木基因组的大小、杂合率和重复序列等基因组特征。当取K=41时,得到其K-mer的频率分布情况(图2),K-mer曲线在depth=28附近出现主峰,经计算后得到花榈木基因组大小为3.05 Gb,修正后为3.01 Gb。由图2可知,K-mer分布曲线出现较为明显的拖尾现象。根据Kmer的深度分布,估计重复序列比率为82.23%,以杂合峰值与纯合峰值的比值计算得到花榈木基因组杂合率为1.04%。利用SOAP de novo软件预测得到Kmer总数为85 418 954 938(表3)。
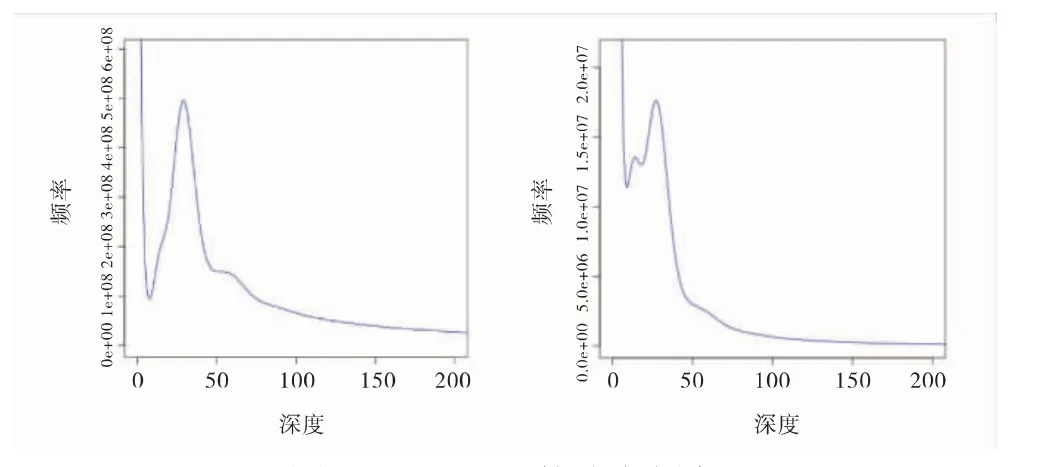
图2 K-me r17的分布频率Fig.2 Distribution frequency of K-mer17

表3 基因组特征统计Tab.3 Feature statistics of genome sequences
2.4 基因组数据初步组装
利用SOAP de novo软件对花榈木有效基因组数据进行初步组装和拼接,本研究K-mer值取41得到最佳拼接效果(表4),共获得4 066 523条contigs,基因组总长度为1 343 713 339 bp,最长的contig长度为54 197 bp,N50长度为656 bp,N90长度为127 bp。在此基础上进一步组装得到3 866 097条scaffolds,拼接总长度为1 364 689 951 bp,最长序列为59 305 bp,N50为762 bp,N90为130 bp。
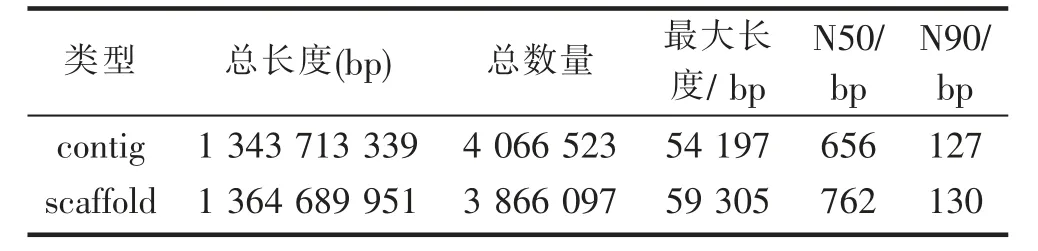
表4 基因组组装统计Tab.4 Statistics of outcome of genome assembly
2.5 样品污染评估——核苷酸比对结果
随机选取10 000条单端reads,与NT库BLAST比对,核苷酸比对结果显示(表5),比对率最高的6个物种均为豆科不同属物种,大豆、黄羽扇豆(Lupinus luteus)、鹰嘴豆、百脉根(Lotus corniculatus)、水黄皮(Millettia pinnata)、蚕豆(Vicia faba),表明此研究中的样品不存在污染,可正常用于后续分析。

表5 原始数据文库与NT库比对Tab.5 Blast of raw date with NT database
2.6 GC含量分布分析
针对组装的contigs统计GC含量,并进行了GC含量与测序深度的关联分析(图3),结果表明,GC含量主要集中在40%,没有明显的GC偏向性,GCdepth散点未出现明显的分层现象,说明未出现外源污染情况,不影响后续的拼接和注释。经计算分析,得到花榈木基因组GC含量为37.17%。
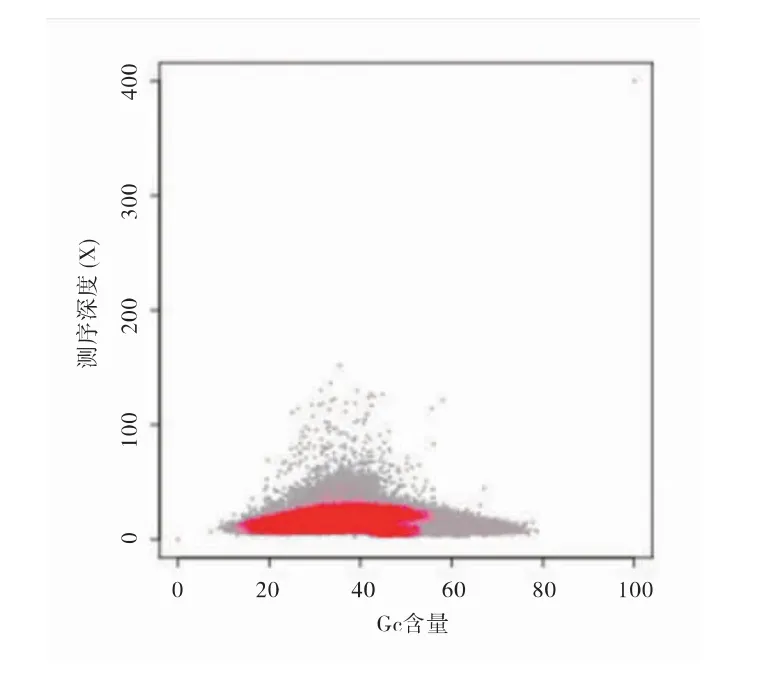
图3 GC-depth分布Fig.3 The distribution of GC-depth
3 结论与讨论
基因组大小是物种最基础的基因多样性特征参数,指一个物种单倍体基因组的DNA含量[19]。每个物种都有其独特的C值,通过测定物种基因组大小,可以对物种全基因组测序、物种鉴定、系统分类及进化、遗传资源挖掘与保护等方面的研究提供参考和理论依据[20-21]。目前基因组大小的测定方法主要有基因组调查测序法、流式细胞术、实时荧光定量PCR法、孚耳根微显影技术、Feulgen染色图像密度分析等[22-23]。其中基于K-mer分析的Genome Survey是一种更高效、准确的方法,能够在开展全基因组测序工作之前,对目标物种基因组特征进行估测,进而可为后续全基因组测序策略选择提供数据参考[23-24]。同时,流式细胞术由于具有操作快捷简便、分辨率和准确率高等优点,也被广泛运用于物种染色体倍性及核型分析、基因组大小测定、种质鉴定等研究[15-16]。
在使用流式细胞术对基因组大小进行测定时,其准确性直接受样品处理、对照标准选择、流式测定条件、荧光染料种类、浓度及染色时间等因素影响[25]。本研究中,使用PI进行DNA特异性染色,PI的吸光波长为480~580 nm,荧光波长为623 nm,采用波长为488 nm的蓝光激发,其与PI的最大激发波长相一致,由此可以保证流式细胞术测定基因组大小的准确度。本研究经流式细胞术实验后得到的直方图分辨率高、峰图平滑且完整,表明以PI作为荧光染料测定花榈木基因组大小可行,结果具有参考价值。Genome Survey分析中,BLAST结果亦未发现其他科树种或动物类、微生物类的高比率情况,表明此研究中的样品不存在污染,可正常用于后续分析。
随着高通量测序技术的迅速发展,目前已有多种豆科草本植物的全基因组被解析,如大豆、豌豆、紫花苜蓿(Medicago sativa)、蚕豆、绿豆和蒺藜(Tribulus terrestris)、苜蓿等作物,在遗传学和基因组学方面已建立模式系统,为豆科生物学研究提供了一定的基础理论指导。花榈木为豆科红豆属树种,目前对豆科木本植物参考基因组的研究鲜有报道。本研究通过流式细胞术对花榈木基因组大小进行测定,所得的基因组大小为2.99 Gb和3.01 Gb。在此基础上,利用了基于Illumina Hiseq测序平台的K-mer分析技术进一步测定及验证,得到花榈木的基因组大小为3.01 Gb,与前者所得基本一致。两种方法结合使用对花榈木基因组进行分析与评估,获得基因组的大小、重复序列、GC含量等信息,提高了研究的可靠性,使所得结果更全面、准确。
通常情况下,当GC含量处于25%~65%时,分析结果可信度较高[26]。本研究中,花榈木的基因组GC含量为37.17%,无明显偏性,说明测序中不含有污染,结果可靠。重复序列比例为82.23%,杂合率高达1.04%,表明该基因组为高重复高杂合基因组。为了进一步获得高质量的全基因组图谱,后续研究策略可考虑结合三代测序PacBio和Illumina测序平台,辅以高通量染色体构象捕获(Hi-C)技术及相应的拼接组装软件进行基因组的组装,以完成花榈木全基因组的测序研究。

