结肠腺癌能量代谢相关mRNA-LncRNA预后预测模型的构建与验证*
黄浩东,刘 颖,刘小株,龚 军,段敏捷,王惠来,向天雨△
(1.重庆医科大学医学数据研究院,重庆 400016;2.重庆市急救医疗中心药剂科,重庆 400014;3.重庆医科大学附属大学城医院信息中心,重庆 401331)
结直肠癌(colorectal cancer,CRC)是常见的恶性肿瘤之一。其发病率居全球癌症的第3位,病死率居全球癌症的第2位[2]。在我国,CRC的发生率和病死率也逐年上升[3]。结肠癌最常见的病理组织学分型为结肠腺癌(COAD)。信使RNA(messenger RNA,mRNA)是指导功能性蛋白质合成的直接模板[5]。长链非编码RNA(long non-coding RNA,LncRNA) 是一种长度大于200 bp且不具备明显蛋白编码能力的RNA转录物[6]。本研究旨在研究COAD中代谢相关RNA对预后的影响,寻找关键分子和调控通路,以更好地指导COAD的预防、诊断和治疗。
本研究通过癌症基因组图谱(TCGA)获取了COAD患者的转录本数据和临床病理数据。采用相关性分析与Cox回归相结合的方法构建COAD患者能量代谢相关mRNA-LncRNA预后预测模型,并通过受试者工作特征(AUC)和Kaplan-Meier曲线等分析方法,在高风险组和低风险组中进行预后预测模型的评估。以便为基于mRNA和LncRNA的COAD分子机制研究提供新视角。
1 材料与方法
1.1 数据收集
从TCGA(https://portal.gdc.cancer.gov;level3)下载TCGA-COAD项目COAD组织与正常组织的RNA-seq数据集和相关临床资料。 从GENCODE (https://www.gencodegenes.org/human/)数据库下载基因的注释信息(包括19 955个mRNA和16 888个LncRNA注释信息)。将下载的转录本数据注释分为mRNA与LncRNA两个部分。
1.2 能量代谢相关mRNA与LncRNA的鉴定
利用edgeR包对差异表达基因进行筛选COAD和正常对照组间的差异表达的mRNA(DEGs)与差异表达的(DELs),筛选条件为错误发现率(FDR)<0.05,且基因表达差异倍数|logFC|>1,结果以火山图的形式表示。从MSigDB (version 7.0;http://software.broadinstitute.org/gsea/msigdb/)数据库收集能量代谢相关基因组,将DEGs与能量代谢相关基因组相交得到能量代谢相关DEGs。对DELs与能量代谢相关DEGs 的进行共表达分析,选择相关系数(r)>0.6且P<0.05的共表达对来得到能量代谢相关的LncRNA。
1.3 功能富集分析
使用 DAVID(https://david.ncifcrf.gov/)数据库[9]将共表达分析得到的能量代谢相关LncRNA调控的mRNA进行GO功能富集,以明确这些基因所涉及的生物学过程、分子功能及细胞组成。同时进行KEGG通路分析,以明确涉及的特定能量代谢过程,结果以气泡图形式进行展示。
1.4 预后模型的构建
将共表达分析得到的能量代谢相关mRNA与LncRNA表达量与患者临床信息合并,并进行单因素Cox回归和多因素Cox回归,以P<0.05为差异具有统计学意义。最后风险得分(risk score)= (基因1的风险系数×基因1的表达量)+(基因2的风险系数×基因2的表达量)+(基因3的风险系数×基因3的表达量)+…+(基因n的风险系数×基因n的表达量)。
1.5 预后模型的验证
以中位风险评分作为分界点,将COAD患者分为高风险组和低风险组。利用Kaplan-Meier 曲线和受试者工作特征 (ROC) 曲线评估能量代谢相关特征的预测能力,并与其他临床特征的预测能力进行对比。为了验证风险评分是否能独立于其他临床病理特征,分别采用:(1)非参数检验检测患者不同临床病理特征分组中风险评分是否有差异;(2)单因素Cox回归和多因素Cox回归验证风险评分是否为患者不良预后的独立危险因素。
1.6 统计学处理
采用SPSS25.0和R语言(4.3.0版)进行统计分析和画图,差异表达分析采用edgeR包实现;代谢相关基因与LncRNA 采用Pearson相关分析进行共表达分析,通过cor.test函数实现;cox回归分析采用survival包实现;通过Enhanced Volcano包绘制火山图;通过survminer包绘制Kaplan-Meier 曲线,log-rank法检验P值;通过survival ROC包绘制ROC曲线;通过ggplot2包绘制气泡图;通过forestplot包绘制Cox回归森林图;非参数检验采用Mann-WhitneyU检验。以P<0.05为差异具有统计学意义。
2 结 果
2.1 能量代谢相关mRNA与LncRNA的鉴别
共收集到TCGA-COAD项目中的471个COAD组织与41个正常组织,其中有完整临床病理特征信息的患者387例,基于GENCODE数据库共标记19 551个mRNA 与14 036个LncRNA。edge包筛选出4 851个DEGs与1 468个DELs(图1A)。通过搜索MsigDB数据库,共下载了1 384个能量代谢相关基因,其中碳水化合物代谢294个,脂质代谢742个,氨基酸及衍生物代谢375个。将上述能量代谢相关基因与DEGs相交,结果显示326个DEGs与能量代谢相关(图1B)。将326个能量代谢相关DEGs与1 468个DELs进行了326×1 468个Pearson相关分析,根据r>0.6且P<0.05,得到共表达碱基对2 079对,其中125个mRNA,上调46个,下调79个;451个LncRNA,上调261个,下调190个。r排名前10的碱基对见表1。

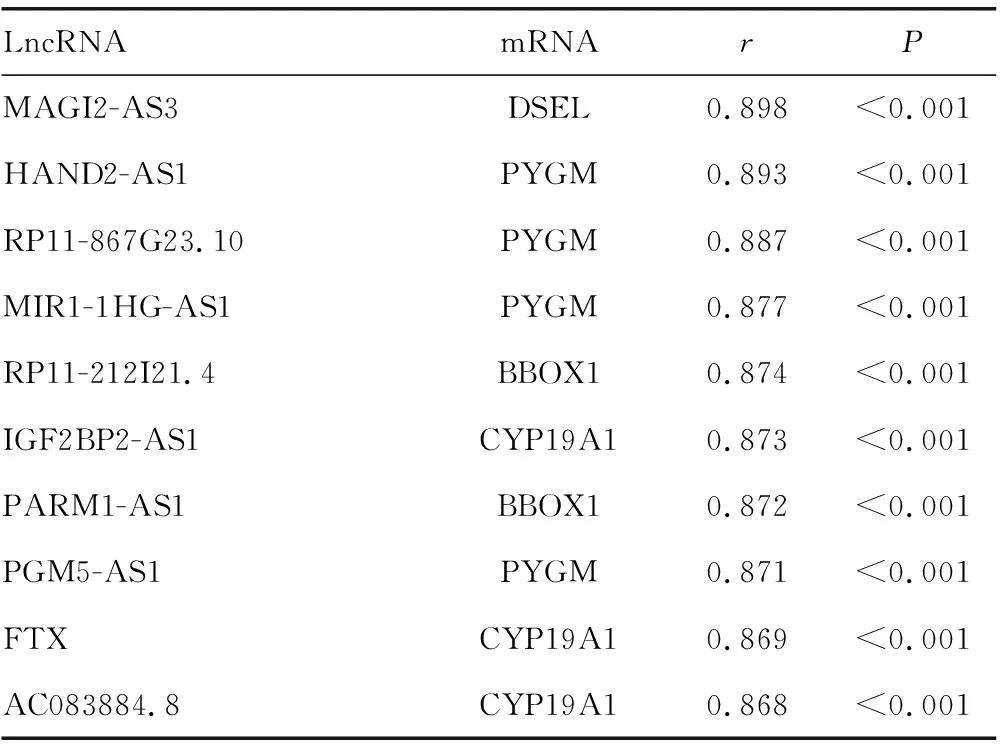
表1 r排名前10的碱基对
2.2 功能富集分析
将共表达分析得到mRNA进行KEGG与GO通路分析。KEGG通路结果显示共表达分析得到的LncRNA可能通过各种代谢途径参与了COAD的形成过程(图2A)。GO生物学过程富集结果显示与氧化还原过程、脂质代谢过程和病毒转录等相关(图2B)。GO细胞组成富集结果显示与细胞质、细胞外泌体和细胞膜等相关(图2C)。GO分子功能富集结果显示与蛋白质同源二聚活性、铁离子结合和磷酸吡哆醛结合等相关(图2D)。
2.3 预后预测模型的构建
将上述的125个能量代谢相关DEGs和451个能量代谢相关DELs与临床预后数据相结合,单因素Cox回归分析得到 15个mRNA与22个LncRNA与预后相关;将其纳入多因素Cox回归分析,结果显示LRP2、CTC-428G20.6、LINC02257、PRR7-AS1、RP11-29G8.3、RP11-677M14.3这6个碱基与COAD患者预后相关(表2),根据此结果,得到风险得分=LRP2×4.037 98+CTC-428G20.6×(-1.937 56)+LINC02257×1.046 69+PRR7-AS1×0.869 12+RP11-29G8.3×(-1.459 44)+RP11-677M14.3×0.841 68。


表2 多因素Cox回归结果

续表2 多因素Cox回归结果
2.4 预后模型的验证
2.4.1能量代谢相关DEGs与DELs的生存分析
将上述得到的风险得分以中位数分为高风险组(风险得分>中位数)与低风险组(风险得分 <中位数),Kaplan-Meier分析得到的生存曲线见图3A,高、低风险组的中位数生存期分别为5.066年和8.334年。图3B、图3C和图3D分别为1年、3年和5年生存时间对应的ROC曲线。可以看出风险得分在预测1年生存期时对应的AUC=0.767,与其他的临床病理特征相比最高;在预测3年生存期时,对应的AUC=0.694,低于病理分期Stage AUC=0.745与M分期=0.704;在预测5年生存期时,对应的AUC=0.691,低于病理分期Stage AUC=0.706。综合来说,风险得分在预测短期生存期时有一定的优势,在预测长期生存期时,与病理分期预测性能相似。

2.4.2风险得分与不同临床病理特征的分析
为比较风险得分是否与COAD患者的临床病理特征具有相关性,以风险得分公式得出的值,以不同临床病理特征为分组,进行Mann-WhitneyU检验,分析不同组间的风险得分值的差异,结果如表3所示。从表中得出风险得分与COAD患者的不同临床病理特征相关性不大。

表3 不同临床病理特征与风险得分的Mann-Whitney U检验
2.4.3风险得分与患者临床病理特征的生存分析
如图4所示,风险得分与患者的其他临床病理特征进行单因素Cox回归与多因素Cox回归检验,得出高风险得分是患者不良预后的独立危险因素。

3 讨 论
近年来,随着生物信息学的发展,越来越多的研究利用mRNA或LncRNA的表达量预测肝癌、乳腺癌、胰腺癌和结直肠癌等患者的预后[10-13]。能量代谢相关mRNA和LncRNA的结直肠腺癌预测模型尚未构建。能量代谢过程参与生命发生、发展的全过程,在COAD的进展中起重要作用,糖、脂质和氨基酸代谢过程产生三磷酸腺苷(adenosine triphosphate,ATP),而肿瘤细胞的恶性特征(快速增殖、侵袭和迁移)需依靠大量ATP维持[4,14]。并且一些研究表明,LncRNA可以通过调节能量代谢相关基因影响癌症进展。如:WANG等[7]研究发现,LINRIS在总生存率较差的患者CRC组织中上调,敲除LncRNA LINRIS减弱了CRC细胞中Myc介导的糖酵解途径,从而抑制CRC细胞的生长。TANG等[8]研究发现,LncRNA GLCC1通过稳定c-Myc的泛素化,从而重新编程葡萄糖代谢促进CRC增殖。因此寻找出能量代谢相关生物标志物并建立预后预测模型有利于COAD患者的个性化治疗。
本研究通过对比COAD组织与正常组织,筛选出DEGs与DELs,然后通过DEGs与MsigDB数据库提供的糖、脂质、氨基酸三大类代谢基因组得到能量代谢相关DEGs,再将其与DELs进行共表达分析进一步筛选得到能量代谢相关DELs与DEGs。对这些能量代谢相关DEGs进行富集分析发现它们除了在hsa01100:Metabolic pathways通路聚集较多外,Count数较高的通路还有hsa00564:Glycerophospholipid metabolism、hsa03320:PPAR signaling pathway和hsa04975:Fat digestion and absorption等。而生物学过程富集于氧化还原过程与脂质代谢过程。然后,本研究进一步采用单因素Cox回归与多因素Cox回归得到具有预后预测意义的RNA,其中包含1个mRNA和5个LncRNA,分别为LRP2、CTC-428G20.6、LINC02257 、PRR7-AS1、RP11-29G8.3和RP11-677M14.3。其中LRP2是唯一1个mRNA,LRP2基因编码的Megalin是一种配体结合的跨膜蛋白,通过和不同的配体结合发挥神经及内分泌调节、抗凋亡等作用[15-17]。Megalin/LRP2已在多篇文章中报道,如JAKOVAC等[18]推测其可能有助于鳞状上皮中异常细胞的更好存活和肿瘤发生;ANDERSEN等[19]表明,黑色素瘤细胞中的Megalin对细胞的生存较重要,因为黑色素瘤细胞中的Megalin/LRP2表达的降低会导致其增殖和存活率降低。并且LRP2已被FEDIRKO等[20]证实参与CRC中维生素D的代谢过程,从而增加西欧人群CRC的患病风险。XIAO等[21]通过生物信息学方式发现LINC02257高表达会导致COAD预后不良,与本研究结果相符。其他4个LncRNA尚未见文献报道。最后,本研究利用上述筛选出的6个能量代谢相关的RNA构建了COAD预后预测模型。
为了验证这6个能量代谢相关RNA是否具有预后意义,本研究根据构建的预后模型建立了一个风险得分公式,按照其中位数分为了低风险组与高风险组。在Kaplan-Meier生存分析中,低风险组与高风险组的中位数生存期分别是5.066年和8.334年(log-rank检验显示P<0.05),低风险组COAD患者生存期明显高于高风险组生存期。而风险得分在1、3和5年的ROC曲线下面积分别是0.767、0.694和0.691。其中在1年的ROC曲线下面积相比于COAD患者其他临床病理特征的ROC曲线下面积最高,而在3年与5年的ROC曲线下面积略低于COAD患者病理分期,因此,风险得分在预测短期生存期时有一定的优势,在预测长期生存期时,与病理分期预测性能相似。此外本研究对风险得分与不同的临床病理特征进行非参数检验,结果显示,不同的临床病理特征与风险得分不相关。为了探讨高风险得分是否为COAD患者不良预后的独立危险因素,本研究纳入临床病理特征与风险得分进行Cox回归分析,结果显示高风险组生存时间明显低于低风险组(HR=3.78,95%CI:2.29~6.22,P<0.05),高风险得分为COAD患者不良预后的独立危险因素。本研究也有一些不足之处,例如研究筛选出的6个RNA,其中4个LncRNA(CTC-428G20.6 、PRR7-AS1、RP11-29G8.3和RP11-677M14.3)还没有相关报道,对其如何影响COAD的发展尚不清楚,因此需要进一步的前瞻性实验研究。
本研究利用6个RNA(LRP2、CTC-428G20.6、LINC02257 、PRR7-AS1、RP11-29G8.3和RP11-677M14.3 )建立的COAD患者的能量代谢相关预后预测模型具有较好的性能。对COAD的个性化治疗具有一定的积极意义,但仍然需要进一步的研究。

