靶向G-四链体的抗番茄丛矮病毒卟啉类衍生物的筛选
谢从宝, 祝强强, 张 涛, 宁丽红*,, 位灯国*,
(1.华中农业大学 农业微生物资源发掘与利用全国重点实验室,动物医学院,武汉 430070;2.湖北洪山实验室,武汉430070;3.华中农业大学,兽药残留国家基准实验室及农业部兽药残留检测重点实验室,武汉 430070;4.华中农业大学深圳营养与健康研究院,深圳 518000;5.中国农业科学院 深圳农业基因组研究所和岭南现代农业科学与技术广东省实验室深圳分中心,深圳 518000;6.华中农业大学 交叉科学研究院,武汉430070;7.华中农业大学 理学院,武汉 430070;8.华中农业大学 园艺植物生物学教育部重点实验室,园艺林学学院,武汉 430070)
植物病毒病,又称“植物癌症”,每年在全世界范围造成的经济损失高达 600 亿美元,其中仅粮食作物损失就高达 200 亿美元[1-2]。蔬菜作为我国仅次于粮食作物的第二大作物,近年来种植规模和面积不断增加,蔬菜病毒病的发病率也呈指数级增长,然而目前可用于防治植物病毒病的药剂较少,因此,寻找抗植物病毒药物的新靶点,开发高效、低毒的植物病毒生态农药成为近年来农药学研究的热点。
G-四链体(G4s)是一种特殊的核酸高级结构,4 个鸟嘌呤碱基通过Hoogsteen 氢键配对形成一个鸟嘌呤碱基平面 (G-quartet),多个碱基平面堆积成非常稳定的G-四链体结构[3]。形成G-四链体的序列广泛存在基因组中的某些重要生物学功能区域,其在病毒基因组中的形成或解链可调节基因的复制、转录和翻译等过程,进而影响病毒增殖[4]。前期工作表明,植物病毒基因组中存在大量的G-四链体可形成序列 (putative G-quadruplex sequences,PQS),开展G-四链体结构-功能研究,筛选靶向G-四链体配体,有望发现新的植物病毒抑制剂。
番茄丛矮病毒 (tomato bushy stunt virus,TBSV)属于番茄丛矮病毒科 (Tombusviridae) 的番茄丛矮病毒属 (Tombusvirus)[5]。感染TBSV 会导致寄主生长发育迟缓、矮化 (如番茄等),叶片出现斑点、变形或黄化 (如天竺葵等),果实变形或缺失[6-7]。该病毒于1935 年首次在爱尔兰的番茄上被发现,广泛分布在多个欧洲国家、北美及南美地区,是一种植物检疫有害生物。然而,近年来在我国也分离出TBSV,其给蔬菜、花卉及其他农作物的产量及质量带来严重损失。有关TBSV 的基因组复制、转录和翻译等分子机制的研究已较为成熟,TBSV 常被作为研究病毒与寄主互作的模式病毒[8-9]。揭示TBSV 基因组中G-四链体的结构及功能,有望为植物病毒基因中的G-四链体研究奠定基础。
TBSV 是一种正义ssRNA 病毒,全长4775 nt,基因组含有5 个ORF,编码5 个蛋白。ORF2 (gp2基因) 编码一个33 kD 的蛋白 (p33)[9]。ORF1 (gp1基因) 通过通读p33 终止密码子翻译生成92 kD 的蛋白 (p92),其具有复制酶活性。蛋白p33 和p92的正常表达是病毒复制的第1 步,p33 蛋白协同p92 蛋白结合单链核酸,二者与病毒RNA 及寄主相关因子共同形成复制酶-RNA 复合体,来调控病毒增殖[10]。ORF3 (gp3基因) 编码41 kD 的衣壳蛋白 (p41)。ORF4 (gp4基因) 和ORF5 (gp5基因) 分别编码移动蛋白 (p22) 和多功能蛋白 (p19),这两种蛋白质从重叠的基因序列中表达。p22 是一种RNA 结合蛋白,决定病毒运输,可与细胞膜和细胞壁结合。p19 是一种多功能蛋白,充当病毒基因沉默的抑制子。p19 蛋白与siRNA 结合,阻止siRNA 与沉默复合体整合。在感染时,亚基因组以独特的方式进行转录:侵染前期,sg mRNA2 先转录;侵染后期,sg mRNA1 转录[11-12]。TBSV 基因组与亚基因组的结构示意图见图1。
本研究拟基于基因组数据库中5 株TBSV 毒株进行生物信息学分析,鉴定TBSV 基因中PQS的保守性,然后综合保守性分析、序列特征和所处基因组位置等信息,选取两条位于gp1和gp2区域的PQS 进行进一步研究,通过生物物理试验证明该序列可折叠成稳定的G-四链体结构,而小分子卟啉可调节G-四链体的稳定性,并呈现抗病毒活性。卟啉衍生物是广泛存在于自然界中的天然产物,天然产物衍生物具有高效、低风险特性,克服了常规化学农药普遍存在的高毒、高残留和对其他非靶标生物的毒害。对其进行修饰改造,使其有望成为良好的生态友好型农药。
1 材料与方法
1.1 供试材料和仪器
本研究所用的寡核苷酸均购自TSINGKE (中国武汉),形成寡核苷酸的G-四链体在缓冲液(20 mmol/L K2HPO4/KH2PO4pH 7.0 和100 mmol/L KCl) 或10 mmol/L Tris-HCl (pH 7.0)、100 mmol/L KCl 和0.1 mmol/L EDTA (乙二胺四乙酸,pH 7.5)中退火,95 ℃孵育5 min 后逐渐冷却至室温,置于4 ℃备用。化合物N-甲基中卟啉IX (NMM)、端粒酶抑制剂Braco 19 和Pyridostatin (PDS),购自北京大学国家化合物库 (PKU-CNCL)。8%宁南霉素水剂 (ningnanmycin AS,CAS:156410-09-2),购于德强生物股份有限公司 (PD20097122)。利福平(Rif)、壮观霉素(Spec)、乙酰丁香酮和2-(N-吗啡啉) 乙基磺酸(MES),纯度 > 95%,均购自索莱宝(Solarbio);含有绿色荧光蛋白 (GFP) 的TBSV病毒克隆载体,由华中农业大学李峰课题组提供。
LB (Luria-Bertani) 液体培养基:将10 g 胰蛋白胨(tryptone)、5 g 酵母浸出物(yeast extract)、5 g NaCl,完全溶于适量 ddH2O 中,并定容至 1 L,121 ℃高压蒸气灭菌 15 min,室温保存。
Chirascan V100 圆二色光谱仪 (英国应用光物理公司);MOS-500 分光光度计(bio-logic,法国);RF-5301PC 荧光光谱仪 (日本岛津)。
1.2 生物信息学分析
综合利用QGRS Mapper、Quadparser、Quadfinder 和G4 Hunter 等G-四链体预测软件对病毒基因组序列中具有(G2+N1-7)3G2+特征的G-四链体可形成序列进行分析。从NCBI 的Genome 数据库(https://www.ncbi.nlm.nih.gov/)中下载5 条病毒毒株的全基因组序列。利用MEGA 6 软件的Alignment-ClustalW 功能,将不同病毒毒株的研究序列进行序列比对,并通过DNAMAN 6.0 进行作图。每条PQS 的保守性为具有相同PQS 序列的毒株数目除以参与比较的总毒株数目 (若存在多个位置相同的序列,则取不同位置的平均值)[13]。
1.3 工作曲线Job Plot
在G-四链体和化合物NMM 的浓度之和保持在3 μmol/L 不变的情况下,通过连续改变化合物与G-四链体之间的比例,评估TBSV G-四链体和NMM 之间的结合比例[14]。将含有适量NMM 的样品与相应量退火后的G-四链体混合,在30 ℃下孵育10 min 后测定荧光信号。在激发波长405 nm处,溶液的荧光强度随加入的NMM 摩尔分数的变化而变化。当比例等于化合物与核酸的计量比时,化合物的荧光响应最强。
1.4 CD 光谱分析
将TBSV-PQS2 (20 μmol/L) 与TBSV-PQS4(20 μmol/L) 分别溶于10 mmol/L Tris-HCl (pH 7.0)和 0.1 mmol/L EDTA (pH 7.5) 或10 mmol/L Tris-HCl (pH 7.0)、100 mmol/L KCl 和0.1 mmol/L EDTA (pH 7.5) 溶液中。在室温下使用1 mm 径长的石英比色皿进行CD 光谱分析。光谱采集范围为360~220 nm,扫描速度为50 nm/min。带宽为1.0 nm,响应时间为2.0 s。CD 熔融实验中波长固定为260 nm,温度从4 ℃以1 ℃/min 升至95 ℃,每摄氏度扫描一次,将温度与对应CD 光谱信号值进行拟合得到熔融曲线。每一组CD 光谱信号都会采集一组缓冲液的数据作为背景扣除[15]。
1.5 紫外光谱与荧光光谱
通过紫外吸收光谱确定化合物NMM 的紫外吸收波长,并以此波长为参照,综合荧光光谱结果确定NMM 的激发波长。仪器参数设置为:扫描波长为200~600 nm,速度为中速。光径为1 cm的石英比色皿,样品体积为100 μL。
荧光光谱数据用荧光光谱仪在室温下采集。10.0 μmol/L 退火后的RNA 样品与3.0 μmol/L 的NMM 在缓冲液(20 mmol/L K2HPO4/KH2PO4pH 7.0、100 mmol/L KCl 和0.1 mmol/L EDTA pH 7.5)中混合,30 ℃下孵育10 min 后采集光谱数据。
为了测定NMM 随RNA 样品的荧光变化,控制NMM 的浓度恒定为3 μmol/L,0~10 μmol/L 退火后的RNA 分别与NMM 在缓冲液20 mmol/L K2HPO4/KH2PO4pH 7.0, 0.1 mmol/L EDTA (pH 7.5) 中混匀,在30 ℃下孵育10 min 后进行荧光光谱测量。激发波长为405 nm,发射狭缝宽度和激发狭缝宽度均为5 nm,扫描范围为415~800 nm,所有的荧光测量试验均在室温下进行。利用TBSVPQS2 滴定NMM 的荧光变化,按 (1) 式计算KD值。
式中:Fmin表示游离配体NMM 在最大发射波长611nm 处的荧光发射强度;Fmax表示饱和时的最大荧光强度,F表示存在不同浓度RNA 时的荧光强度,M表示TBSV-PQS2 的浓度。
1.6 农杆菌侵染性克隆
将病毒基因组克隆到载体,再通过体外转录获得病毒RNA,是制备RNA 病毒侵染性克隆的常用方法。本研究用绿色荧光蛋白基因 (GFP) 代替TBSV 病毒的外壳蛋白基因 (CP) 构建的表达载体,通过检测荧光强度,筛选抑制病毒活性的小分子配体。农杆菌在含有10 mg/L Rif 和10 mg/L Spec 的LB 培养基中扩大培养48 h,培养条件为28 ℃,摇床参数220 r/min;1 L LB 培养基中含酵母抽提物5 g、氯化钠10 g 和色氨酸10 g,用氢氧化钠调整pH 值至7.0,121 ℃高压灭菌20 min。将培养菌液与LB 培养基按体积比1 : 100 的比例诱导。诱导液中加入40 μmol/L 的乙酰丁香酮、4.5 mmol/L 的MES 和适量的抗生素Rif 和Spec;侵染前,将转化后的农杆菌悬浮液置于悬浮缓冲液(含有150 μmol/L 乙酰丁香酮和10 mmol/L 氯化镁) 中,调节OD600值至0.01~0.1,室温静置2~4 h。注射方式为叶背注射,每株烟草注射3 片叶片,每片叶片主叶脉两侧各注射一个点,每点注射范围约1 cm²。注射24 h 后将烟草置于自动转盘上,每小时拍照记录1 次,连续观察7 d。
2 结果与讨论
2.1 TBSV- PQS 序列的生物信息学分析
NCBI 中有5 条完整的TBSV 的基因组序列。通过Quadpaser 程序对TBSV (NC_001554.1,见表S1) 的正链及负链进行PQS 搜索,发现了16 条G2-PQS,不存在G3-PQS。除了TBSVPQS14、TBSV-PQS15 和TBSV-PQS16 这3 条分布在TBSV 基因组的负链,其余均分布在TBSV基因组的正链 (表1,图2)。由表1 可以看出:12条G2-PQS 单独存在于基因中,4 条存在于G-四链体簇中;仅有一条TBSV-PQS13 存在于非编码区,其他序列在CDS 区域。通过MEGA 6 及DNAMAN 6.0 软件对5 株TBSV 毒株中每个PQS 的序列保守性进行分析,发现预测的G2-PQS 除3 条序列 (TBSV-PQS7、TBSV-PQS14 和TBSV-PQS15) 外,其他G2-PQS 序列的保守性均为100% (表1,图2),其中G2-PQS、G3-PQS 和G-四链体簇结构如图3 所示。

图2 G2-PQS 在TBSV 基因组的分布及保守性Fig.2 Distribution and conservation of PQS in TBSV genome
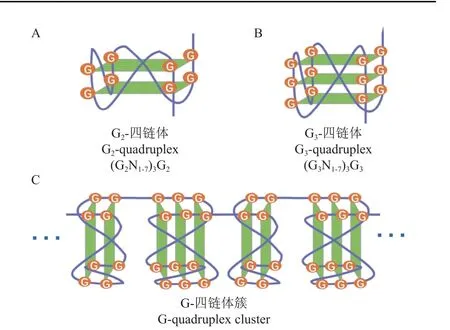
图3 G2-四链体(A)、G3-四链体(B)和G-四链体簇(C)的结构示意图Fig.3 Schematic diagrams of the G2-quadruplex (A),G3-quadruplex (B), and G-quadruplex clusters (C)

表1 TBSV 中PQS 的序列信息及所在基因组的位置Table 1 Sequence information and the location of the genome of PQS in TBSV
与其他正单链RNA 病毒类似,TBSV 的复制也涉及多步反应。复制相关蛋白p33 和p92 的正确翻译是病毒基因组复制的第1 步,也是最关键的一步。在TBSV 病毒基因组序列中,只有近5′端的ORF (如p33 和p92) 才可以被直接翻译,位于3′端的ORF (如p41、p22 和p19) 则不能被直接翻译表达。p92 蛋白的N 端包含着完整的p33 的序列,TBSV 的gp1基因 (编码p92 蛋白的基因) 是通过通读gp2基因 (编码p33 蛋白的基因)的终止密码子的方式而得来的。在宿主细胞中,通读产物p92 的含量仅为p33 的5%。TBSV 病毒复制需要这两个蛋白的共同参与,暗示着p92 中的p33 区域与独立的p33 在复制中的功能可能不同。病毒复制的第2 步需要形成复制酶-RNA 复合体,其中包含p33、p92、病毒RNA 模板以及其他宿主因子。所以,p33 与复制酶-RNA 复合体的组装及定位有关,p92 具有3 个RNA 结合域,可以与病毒基因组RNA 结合,与子代病毒RNA 合成直接相关。TBSV 亚基因组的转录与p41、p22和p19 的表达相关,多种RNA 顺式作用元件和病毒蛋白参与了这一过程。在转录过程中,病毒编码的p33 和p92 蛋白参与了整个转录过程,并对病毒RNA 和sg mRNAs 间的表达比例起着重要的调节作用[5,9]。因此,本研究重点关注gp1基因和gp2基因中的G-四链体结构。
根据loop 长度选择合适的G4 序列:G-四分体数目、loop 长度及loop 组成等因素对DNA 及RNA G-四链体的稳定性及拓扑结构均会产生一定的影响。本研究中,按照 (G2+N1-7)3G2+特征的序列来预测G-四链体,但过长的loop 并不利于G2-PQS 结构的形成和稳定[16-17]。Qin 等[18]发现,所有测试的loop 长度为1 nt 的DNA G2-PQS 均形成了平行构象,而loop 长度为3 nt 的序列均形成了反平行构象。其原因可能是loop 长度为1 nt 或3 nt的G-四链体具有很高的热稳定性,可以克服loop组成改变的影响[18]。在人类的基因组中,loop 长度为1~3 nt 是G2-PQS 最常见的序列,并且loop 长度为{1、1、1}的集合约占所有G2-PQS 的4.6%[19-20]。
因此,综合考虑序列保守性、PQS 所在基因的功能和PQS 序列特征3 个方面,本研究选择了表1中红色的序列作为进一步研究对象:TBSV-PQS2(5′-GGAUGGAGUGGAGG-3′) 和TBSV-PQS4 (5′-GGCGGUUGGAAGAUGG-3′)。
2.2 TBSV-PQS 的体外结构鉴定
采用CD 光谱分析TBSV-PQS2 和TBSVPQS4 序列能否折叠成G-四链体结构。结果发现RNA G-四链体几乎全部采取平行构象,CD 特征峰在240 nm 附近显示负峰,在265 nm 附近显示正峰。因此,在CD 光谱中,RNA G-四链体比DNA G-四链体呈现的拓扑多样性更少[21]。如图4A,TBSV-PQS2 在不含 K+条件下,G-四链体结构的特征峰很弱;而在缓冲溶液 (10 mmol/L Tris-HCL(pH 7.0)和 100 mmol/L KCl ) 中,TBSV-PQS2 的CD 光谱在240 nm 附近显示负峰,在265 nm 附近显示正峰,证明其形成了典型的平行RNA G-四链体结构。在近生理浓度钾离子条件下更容易形成G-四链体结构,说明G-四链体结构具有在体内形成的潜力。
与TBSV-PQS2 一样,在不含钾离子的溶液中,TBSV-PQS4 G-四链体CD 特征信号很低,而在高K+浓度溶液条件下,TBSV-PQS4 的CD 特征信号在265 nm 附近出现了一个正峰,在240 nm附近出现负峰,也是一个平行的RNA G-四链体体结构 (如图4B)。说明K+浓度同样影响TBSVPQS4 G-四链体的形成。

图4 TBSV-PQS2 与TBSV-PQS4 的圆二色光谱图Fig.4 Circular dichroism (CD) spectra of TBSV-PQS2 and TBSV-PQS4
2.3 与TBSV-PQS2 和TBSV-PQS4 互作的G-四链体配体筛选
为了筛选能够与TBSV-PQS 相互作用的小分子配体,选择几种已经被广泛研究可以与G-四链体结合且抑制病毒增殖的小分子配体,包括卟啉类化合物NMM[22]、吖啶类化合物Braco 19[23-24]和喹啉类化合物PDS[25-26](结构式见图式1)进行初步的配体筛选研究,发现典型的G-四链体配体NMM与TBSV-PQS2 和TBSV-PQS4 存在较强的相互作用 (如图式1 和图S1 所示)。

图式 1 3 种配体的分子结构式Scheme 1 Molecular structural formulas of three ligands
G-四链体配体对不同的G-四链体结构可能具有选择性,研究发现,NMM 可以稳定G-四链体结构,调控病毒的增殖过程。NMM 结合G-四链体后,为了有效地进行π-π 堆叠,可以调整其空间构象以匹配G-四链体结构,从而增强其荧光强度[27]。这种特殊性质使其成为适用于G-四链体的特异性荧光探针。
本研究首先通过紫外荧光法测定了TBSVPQS2 和TBSV-PQS4 与NMM 的相互作用。结果表明,加入TBSV-PQS 后NMM 的紫外吸收峰发生了红移 (图5A),这是由于卟啉化合物插入核酸后,卟啉大环的电子云密度受核酸碱基上电子云的影响而发生了改变,说明NMM 能够很好的结合TBSV-PQS。NMM 本身具有很弱的荧光[28],单独检测发现,其在最大发射波长611 nm 仅显示出微弱的荧光信号 (图5B);而当加入G-四链体后,荧光会增强,TBSV-PQS2 与NMM 有荧光响应,且最大荧光强度与不加入G-四链体时的比值为:FINMM+TBSV-PQS2/FINMM= 40 (cTBSV-PQS2/cNMM= 3)。在NMM 溶液中加入TBSV-PQS4 后,荧光强度也会增强,且最大荧光强度与不加入G-四链体时的比值为:FINMM+TBSV-PQS4/FINMM= 27 (cTBSV-PQS4/cNMM= 3),说明相比于TBSV-PQS2,TBSVPQS4 与NMM 的结合较弱。

图5 NMM 与TBSV-PQS 互作的紫外光谱(A)和荧光光谱(B)Fig.5 UV (A) and fluorescence spectra (B) of the interaction between NMM and TBSV-PQS
CD 研究发现,加入NMM 后,TBSV-PQS2和TBSV-PQS4 在265 nm 处的CD 信号明显增强,也证明了NMM 可以稳定TBSV-PQS 结构(cTBSV-PQS2/cNMM= 1.5)。相同浓度下,TBSVPQS2 与NMM 结合后,呈现的CD 峰强于TBSVPQS4 与NMM 结合后呈现的CD 峰 (图6A,6B),这与紫外荧光光谱的检测结果相一致。

图6 TBSV-PQS2 (A)和TBSV-PQS4 (B)与NMM 的CD 光谱图Fig.6 CD spectra of TBSV-PQS2 (A) and TBSV-PQS4 (B) with NMM
CD 熔融实验是一种考察G-四链体稳定性的方法,在不加入化合物NMM 的条件下,TBSVPQS2 的熔融温度显著高于TBSV-PQS4,说明TBSV-PQS2 的稳定性显著高于TBSV-PQS4 (图7A),TBSV-PQS2 在自然条件下稳定存在的可能较大;加入化合物NMM 后,TBSV-PQS2 的稳定性进一步提高 (Δtm= 9.2 ℃) (图7B),说明NMM 可以显著稳定TBSV-PQS2 的结构。因此选取TBSVPQS2 作为进一步研究的对象。

图7 TBSV-PQS2 与TBSV-PQS4 的热熔解曲线(A)和TBSV-PQS2 在存在/不存在NMM 时的热熔解曲线(B)Fig.7 Thermal melting curve of TBSV-PQS2 and TBSV-PQS4(A), and thermal melting curve of TBSV-PQS2 in the presence/absence of NMM(B)
荧光滴定分析用于测量NMM 与TBSV-PQS2的结合强度。如图8A 所示,TBSV-PQS2 诱导的荧光强度呈剂量依赖性增加。根据荧光滴定数据及Benesi-Hilde brand 方程拟合,确定NMM 与TBSV-PQS2 之间的解离常数(KD)为3.81 μmol/L(图8B)。

图8 NMM 与不同浓度TBSV-PQS2 的荧光滴定光谱(A)和使用线性拟合方程测定NMM 与TBSV-PQS2 的结合常数(B)Fig.8 Fluorescence titration spectra of NMM with different concentrations of TBSV-PQS2 (A) and determination of binding constants of NMM to TBSV-PQS2 using a linear fitted equation (B)
为了进一步确定TBSV-PQS2 与NMM 的结合比,以NMM 占样品总浓度的比例为横坐标,相对荧光强度(△FI = 实际荧光值 - 对应浓度下NMM 荧光值)为纵坐标绘制Job Plot 工作曲线,用Origin 8.0 软件拟合相对荧光值,发现拟合交点为0.56,表明NMM 与TBSV-PQS2 的结合比为1 : 1(图9),说明两者之间只存在一个结合位点。
2.4 卟啉衍生物NMM 对TBSV 病毒增殖的抑制作用
TBSV-PQS2 和NMM 的CD 熔融实验结果表明,NMM 可以稳定这个G-四链体结构,但TBSV-PQS4 与NMM 的结合不如TBSV-PQS2 与NMM 的结合稳定,因此,选择稳定性更好的TBSV-PQS2 进行TBSV-GFP 侵染性克隆,用于筛选抑制TBSV 病毒的小分子化合物。
以商品化植物病毒抑制剂宁南霉素作为阳性参照,选取不同浓度的G-四链体配体,如Braco19、PDS 和NMM 进行抗病毒活性试验。结果(图10)发现,与PDS、Braco19 相比,NMM在低浓度 (20 μmol/L) 时即可显著降低荧光蛋白的表达,抑制病毒增殖,即使5 μmol/L 的NMM 也呈现出了抑制效果 (图S2)。

图10 体内筛选抑制TBSV 病毒增殖的小分子化合物Fig.10 In vivo screening of small molecule compounds that inhibit the proliferation of TBSV virus
3 结论
本研究基于对5 株TBSV 毒株的生物信息学分析,在TBSV 毒株基因组中预测出16 条PQS本研究序列。综合考虑序列保守性、PQS 所在基因组的功能和PQS 序列特征3 个方面,选择TBSV-PQS2 和TBSV-PQS4 作为进一步研究对象。CD 光谱结果表明,PQS2 和PQS4 在近生理浓度钾离子条件下均能够形成平行G-四链体结构。通过紫外光谱和荧光光谱筛选出经典的G-四链体配体卟啉衍生物NMM 与TBSV-PQS2 和TBSV-PQS4 存在较强的相互作用。G-四链体的稳定性除了取决于序列本身G 碱基的含量、Loop 的长度之外,还与溶液环境和所结合的配体密切相关。植物病毒中PQS 的loop 一般比较长[29],这直接导致植物病毒中的G-四链体的稳定性可能相对较差。结合CD 熔融实验结果,进一步选择结构稳定性更强、更容易在生物体内存在的TBSVPQS2 作为研究对象,证明了NMM 可以稳定TBSV-PQS2 的结构,并通过Job Plot 曲线法验证了TBSV-PQS2 G-四链体与NMM 的结合比为1 : 1。为了探究G-四链体配体抗植物病毒的潜力,针对TBSV-PQS2 进行抗病毒活性进行研究,发现卟啉衍生物NMM 可以显著降低荧光蛋白的表达,显示出很好的抗TBSV 活性,可能延迟病毒的复制。G-四链体在TBSV 中的结构与功能的研究为理解病毒复制机制、设计防治TBSV 的农用药物或培育抗病品种提供理论基础,对设计高效、低风险的生态农用药物具有广泛的参考价值。

