高温胁迫下 2个棉花品种转录组可变剪切差异分析
丁宇 王马寅 唐敏强 李子阳 谢尚潜

摘要:棉花(Gossypium hirsutum L.)是一种重要的经济作物,是世界上第二大的天然纺织纤维来源和重要的食用油来源。棉花对生物和非生物胁迫高度敏感,尤其是高温胁迫极易影响花粉的活力和花药的开裂。为了解析棉花在高温胁迫下基因转录水平的相应变化机制,开展了热敏和耐热2个棉花品种的全长转录组响应高温胁迫处理变化的研究。通过转录本的可变剪切分析发现,2个棉花品种在高温胁迫下可变剪切的总数显著增加。热敏品种Che61-72中发现了2 900个差异表达基因,并且差异基因在加热前样本(R0)和加热12 h后的样本(R12)中分别识别到了 13 251 个和25 296个可变剪切事件,其中内含子保留事件增加得最多,有3 837个。耐热品种新陆早36号中发现了 2 437 个差异表达基因,在加热前样本(T0)和加热12 h后的样本(T12)中鉴定到了11 248个和13 769个可变剪切事件,外显子跳跃事件变化得最大,增加了4 144个。富集分析发现,2个品种的差异基因都显著富集到了光系统Ⅰ的光捕获、叶绿体类囊体膜和光合作用-天线蛋白通路中,并筛选出5个关键基因(CPB3、A0A1U8IZF2、A0A1U8KCA2、A0A1U8NDW4和A0A1U8NI70),均被注释为叶绿素a/b结合蛋白,它们参与了调控棉花光合作用动态平衡。本研究为棉花在高温胁迫的调节机制的深入研究提供了理论依据,为后续耐高温的种质改良及新品种培育提供了数据支持。
关键词:棉花;高温胁迫;全长转录组;纳米孔测序;可变剪切
中图分类号:S562.01文献标志码: A
文章编号:1002-1302(2023)05-0001-10
棉花(Gossypium hirsutum L.)是一种重要的经济作物,是世界上第二大的天然纺织纤维来源和重要的食用油来源[1]。近年来,由于温室效应,地表溫度升高对植物生长产生不同程度的影响[2]。棉花在高温胁迫下花粉活力会受严重影响并导致花药的开裂[3-4]。因此,从不同水平了解棉花抗高温胁迫分子机理,选育耐高温棉花材料是十分必要的。
随着测序技术的发展以及基因组研究的兴起,棉花抗高温胁迫已经开展了相关的研究。已有研究表明,棉花的酶活性、萌发、生长、根系发育、营养发育和开花最适温度在25~31 ℃[5],高温环境通过减弱棉花光合作用和新陈代谢,引起授粉能力减弱[6],影响棉花花朵的定型、生产率和所产生的纤维的质量[7]。Liang等通过纳米孔测序转录组分析了棉花的抗热胁迫机制,揭示了富集在核糖体、激素信号转导以及内质网蛋白质加工途径的差异表达基因、叶绿体a/b结合蛋白基因和lncRNAs对棉花耐热机制的调节作用[8]。Reddy等研究发现,在20~30 ℃的最适温度下,棉花生物产量增加,超过最适温度则导致产量严重下降[9]。高于最适温度会抑制棉花光合作用[7-10],同时伴随着碳水化合物的进一步消耗和呼吸作用的增强[11]。Barua等发现,热休克蛋白与棉花等作物的光合作用能力密切相关[12]。Min等发现,在高温敏感品系种,棉花花药在高温诱导下GhCKI高度表达,该基因可能在高温胁迫中有重要作用[13]。Wang等研究发现,miRNAs通过调节与生长稳态、活性氧、叶绿体功能、植物-病原菌相互作用和植物激素信号转导途径相关的基因来发挥作用[14]。Ma等揭示了lncRNA对高温下棉花雄性生殖的作用,并发现了GhHRK1基因在高温胁迫反应中的负调控作用[15]。这些研究结果为理解棉花抗高温胁迫的基因水平的调控机制提供了支持,但高温胁迫条件下关键调控基因转录本水平的调控解析仍有待进一步研究。
目前,三代测序技术已经被广泛运用于植物的全长转录本研究,主要包括鉴定复杂的可变剪切事件、全长转录变异体、融合转录本和选择性多聚腺苷事件。可变剪切事件的发生是调控基因表达的一种重要调控机制,也是促进转录组和蛋白质组多样性的动力[16]。三代测序相较于二代测序在序列长度、转录本识别方面的优势,更有助于识别复杂的剪接异构体,研究植物响应各种胁迫的基因调控机制[17]。前人利用三代测序技术已经在高粱(Sorghum bicolor)[18]、玉米(Zea mays)[19]、异源多倍体棉花(allopolyploid cotton)[20]和花竹(Phyllostachys edulis)[21]中分别鉴定到可观的可变剪切事件。多倍体棉花中与耐热机制相关的可变剪切事件仍有待进一步研究。
本研究基于纳米孔测序的全长转录组数据,分析了热敏(Che61-72)和耐热(新陆早36号)2个品种的棉花在热刺激处理前后的可变剪切差异,发现高温胁迫增加了可变剪切事件,并进一步分析了在高温胁迫中差异表达基因的功能,以及可变剪切事件的解析。本研究为棉花高温胁迫的调节机制的深入研究提供了理论依据,为后续耐高温的种质改良及新品种培育提供了数据支持。
1 材料与方法
1.1 数据收集与处理
收集来自NCBI的2个棉花品种(热敏品种Che61-72和耐热品种新陆早36号)的纳米孔测序数据,此数据由石河子大学在2021年3月4日上传发布,登录号为PRJNA706603。测序样品使用光照培养箱(温度为40 ℃)进行热应激处理,在处理0 h和12 h后测序。使用pipeline-nanopore-ref-isoforms程序(https://github.com/nanoporetech/ pipeline- nanopore-ref-isoforms)对纳米孔测序全长转录组数据进行处理,再用minimap2 v2.24软件将测序数据比对到棉花的参考基因组(https://www.ncbi.nlm.nih.gov/genome/?term=Gossypium+ hirsutum)[22],参数设置为-ax splice,-secondary=no-C5。
1.2 注释与可变事件的鉴定
将cDNA_Cupcake(https://github.com/Magdoll/cDNA_Cupcake)的collapse_isoforms_ by_sam.py脚本用于比对数据去除冗余的转录本和合并同种转录本,使用SQANTI3 v4.3软件[23]对处理后的转录本数据结果与基因组注释进行比较分析。可变剪切事件通过SUPPA v2.3软件[24]使用默认参数进行识别鉴定,SUPPA可以从注释生成转录事件和局部选择性剪切事件,把可变剪切事件分为以下7种类型:外显子跳跃(SE),外显子互斥(MX),5′端选择性剪切(A5),3′端选择性剪切(A3),内含子保留(RI),替代第1个外显子(AF),替代最后1个外显子(AL)。
1.3 基因表达水平的量化和差异表达分析
通过Salmon定量软件[25]将全长reads比对到参考基因组上,并对匹配质量大于5的reads进行量化。通过每百万计数(CPM)估计基因表达水平(CPM=映射到转录本的reads/样本中对齐的总reads×106)。使用DESeq2 R包[26]对2个棉花品种不同时间点的样品进行差异表达分析。在该分析中,由DESeq2确定的P<0.01、|foldchange|≥2被认为是差异表达基因(DEG)。
1.4 差异表达基因的功能富集分析
使用eggNOG v2.0.0软件[27]进行差异基因注释,从差异基因的GO数据库中获得注释背景信息。利用R的clusterProfiler R包[28]对差异基因进行GO富集分析,P值小于0.05的GO条目被认为是差异表达基因显著富集。进一步使用KOBAS v3.0[29]分析KEGG通路中DEGs的富集情况(α=0.05)。
2 结果与分析
2.1 数据统计分析
本研究从NCBI公共数据库中选取了棉花的耐热品种(新陆早36号)和热敏品种(Che61-72)的纳米孔测序的转录组样本进行分析,其中热敏品种Che61-72对照组(加热前样本,R0)和试验组(加热12 h后样本,R12)总的reads分别为19 301 677和21 456 617,总碱基数为15.6 G和19.4 G,GC含量为43.2%和42.5%。 序列的平均长度为849 bp和951 bp,N50长度为918 bp和1 064 bp。在耐热品种新陆早36号的对照组(加热前样本,T0)和试验组(加热12 h后样本,T12)中,分别获得了 19 219 360 和16 564 945条reads,总的碱基数为14.7 G和13.6 G,GC含量分别为43.3%和42.6%,序列N50长度为868 bp和934 bp,平均长度为801 bp和964 bp(表1)。
2.2 数据的处理与注释
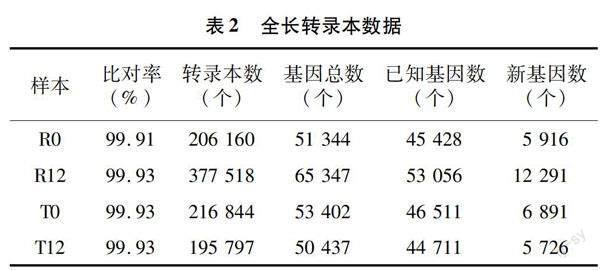
比对2个不同品种棉花获得转录本,使用cDNA_Cupcake对全长转录组序列进一步去除冗余转录本。4个样品的转录组序列比对到基因组上的
比对率分别为99.93%、99.93%、99.91%和99.93%(表2)。
使用SQANTI3v.4.3对转录组进行分析和注释,转录本总数最多的是热敏品种Che61-72的试验组(R12),得到了377 518个转录本,基因总数也最多,其中已知基因数为65 347个,新基因数量为 12 291 个。而转录本最少的是耐热品种新陆早36号的试验组(T12),有195 797个。基因总数为 50 437 个,其中已知基因数为44 711,新基因数有 5 726 个。对新的基因进一步进行注释,并用于下游分析。
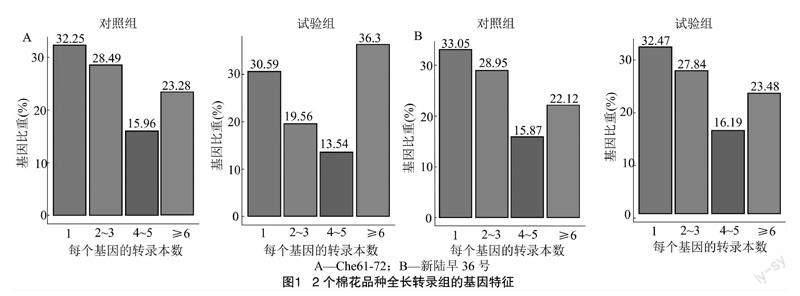
在热敏品种Che61-72的R0中每个基因只有一个转录本的现象占的比重最大,有32.25%,其次是具有2~3个转录本,占了28.49%。而在R12中具有6个以上转录本的基因的比重最高,有36.3%。在耐热品种新陆早36号的T0和T12中,每个基因具有1个转录本的现象比重最大(图1)。经过热应激处理后,2个品种中6个以上转录本的数量增加,可能是生物体内基因需要产生更多的可变剪切体来应对热应激反应。这些结果表明,使用纳米孔测序平台进行大规模转录本测序是重建转录本的有效方法,有利于复杂转录事件的研究。
2.3 热应激处理后可变剪切事件的广泛动态变化
在高等动植物基因组中,大部分基因在转录过程中存在可变剪切,从而引起同個基因的多个转录本。植物在胁迫条件下会产生更多的剪切变异来微调其基因的表达模式[30]。本研究使用了SUPPA软件去识别可变剪切事件,可变剪切事件分为7种类型:外显子跳跃(SE),外显子互斥(MX),5′端选择性剪切(A5),3′端选择性剪切(A3),内含子保留(RI),替代第1个外显子(AF),替代最后1个外显子(AL)(图2-A)。在热敏品种Che61-72的R0样品中,从35 162个基因中检测到了66 638个可变剪切事件,在R12样品中,从61 097个基因中检测到了137 248可变剪切事件(图2-B)。研究结果表明,经过40 ℃热应激的外部条件刺激12 h后,R12与R0相比表达的基因变多,可变剪切事件增加,特别是内含子保留(RI)增加了27 591,占了总的可变剪切数量的30.06%,其次是3′端选择性剪切(A3),占了28.46%(图2-C)。在耐热品种新陆早36号的T0样品中,从31 687个基因识别到了 59 639 个可变剪切事件,其中内含子保留(RI)事件最多。在T12样品中,从34 977个基因中识别到了69 483个可变剪切事件,而3′端选择性剪切事件最多,占了总事件的30.73%(图2-B和图2-C)。在40 ℃热应激刺激后, T12与T0相比表达的基因和可变剪切事件都有所增加,但耐热品种对热应激的反应没有热敏品种这般强烈,最多的3′端选择性剪切事件增加了4 023个。3′端选择性剪切(A3)是棉花中最普遍的形式[31]。已有研究表明,内含子保留(RI)可能是植物应对胁迫的重要分子机制[32]。棉花在热应激处理后,内含子保留(RI)事件发生了显著变化,这表明内含子保留(RI)可能在响应高温胁迫的转录后转录组调整中发挥作用。
2.4 响应热应激的差异表达基因与可变剪切的变化
为了探究热应激条件下基因表达的差异,分析了热敏品种Che61-72和耐热品种新陆早36号在温度为40 ℃条件刺激12 h后的基因表达情况。在Che61-72中发现了2 900个差异基因(DEGs),其中1 592个基因表达上调,1 308个基因表达下调。差异基因在R0样本中共识别到了13 251个可变剪切事件,在R12样本差异基因中,可变剪切事件大量增加,一共识别到了25 296个可变剪切事件,其中RI事件增加得最多,有3 837个(图3-A)。在新陆早36号中发现了2 437个差异表达基因,其中1 369个上调基因,1 068个下调基因。在T0样本中鉴定到了11 248个可变剪切事件,在T12样本中鉴定到了13 769个可变剪切事件,在热应激后,可变剪切事件增加了2 521个,其中外显子跳跃事件变化得最大,增加了4 144个。
从2个不同品种的棉花样本的可变剪切变化中研究发现,热敏品种Che61-72对热应激反应非常强烈,不同类别的可变剪切事件都呈上升趋势,而耐热品种新陆早36号的不同类别的可变剪切事件除了外显子跳跃显著增加和外显子互斥少量增加之外,其他5个类型都有减少的现象,这可能是与耐热品种中耐热机制对热应激的自身调控有关。
为了防止因为材料本身的差异导致的结果误差,本研究继续分析了热敏品种(R0)和耐热品种(T0)之间的基因表达的差异情况。一共发现了318个差异基因,其中206个基因表达上调,112个基因表达下调。其与热敏品种的热应激的表达差异基因中有37个共有基因(图3-C),与耐热品种的热应激的表达差异基因中有38个共有基因(图3-D)。在后续的功能注释分析时,剔除共有差异基因。
2.5 差异表达基因的GO与KEGG富集分析
进一步分析差异表达基因的功能,本研究对Che61-72和新陆早36号的差异基因进行了GO富集分析。在Che61-72的差异基因中,一共富集到了973条GO通路。在生物过程通路中,差异基因主要富集到了光合作用、光系统Ⅰ的光捕获、对光刺激的反应、蛋白质折叠等;在细胞成分通路中,主要富集在胞质、叶绿体类囊体膜和质体球等;在分子功能类别中,主要富集在DNA结合转录因子活性、折叠蛋白的结合和水通道活动等。在新陆早36号的差异基因被856条GO通路注释,在生物过程中,差异基因主要涉及到了核糖体的结构成分、核糖核酸绑定和叶绿体类囊体膜等条目;在细胞成分中,主要涉及了质体球、蛋白质泛素化和核斑点中;在分子功能类别中,主要富集在血红素结合、细胞质大核糖体亚基和细胞质小核糖体亚基等条目之中(图4)。
利用KOBAS v3.0分析了2个样本差异基因的KEGG富集通路。在Che61-72的差异基因中,一共被122条通路注释到,分别是光合作用-天线蛋白、昼夜节律-植物、代谢途径和次生代谢物的生物合成等通路;新陆早36号的差异基因一共富集到了113条通路之中,其中有光合作用-天线蛋白、核糖体、代谢途径和次生代谢物的生物合成等通路(图5)。通过富集分析发现,在GO条目中2个品种的差异基因都显著富集在光系统Ⅰ的光捕获、叶绿体类囊体膜中,在KEGG通路中最显著富集于光合作用-天线蛋白中,可见热应激对棉花的光合作用过程产生了影响。
2.6 响应热应激的信号通路和基因分析
在本研究中,2个品种的差异基因都显著富集到了光系统Ⅰ的光捕获、叶绿体类囊体膜和光合作用-天线蛋白中。在光系统I的光捕获之中,Che61-72 富集上了39个差异基因,其中2个基因上调,37个基因下调。使用string蛋白质数据库(https://cn.string-db.org/)对基因进行注释,发现有11个基因被注释成叶绿素a/b结合蛋白。新陆早36号富集到了35个差异基因,表达下调,有9个基因被注释成叶绿素a/b结合蛋白。在叶绿体类囊体膜之中,Che61-72富集上了52个差异基因,有6个基因表达上调,46个基因表达下调。在新陆早36号富集到了48个差异基因,其中11个基因表达上调,37个基因表达下调。富集到的基因分别被注释成叶绿素a/b结合蛋白、核糖核蛋白A、伴侣蛋白、光系统Ⅰ反应中心亚基和蛋白质曲率类囊体1A。在KEGG最显著富集的通路光合作用-天线蛋白中,Che61-72富集到了41个差异基因,2个基因表达上调,39个基因表达下调,新陆早36号在通路中富集到了37个差异基因,表达下调。此通路的基因都属于捕光叶绿素a/b结合蛋白(LHC)家族。
在热敏品种所富集上的基因中,在热應激前(R0)的转录本总数为952个,可变剪切的总数为735个,其中内含子保留的类型最多有231个,其次是3′端可变剪切有221个。在热应激12 h后(R12),转录本数量为565个,比R0减少了387个,可变剪切总数有571个,最多的类型是3′端可变剪切有167个,其次为内含子保留152个。与R0相比,内含子保留的数量减少的最多,减少了79个。在耐热品种所富集上的基因里,热应激前(T0)的转录本总数为776个,可变剪切总数为609个,3′端可变剪切和5′端可变剪切的数量最多,分别为171个和178个。热应激12 h后(T12)转录本数量和T0相比,减少了383个,可变剪切的数量减少了281个,其中3′端可变剪切减少的数量最多,减少了77个(表3)。
在自然条件下,强烈的阳光会削弱光合作用,导致光合活性受损。本研究中,在热应激12 h的条件下处于这3条通路上的基因几乎都呈现下调表达(图6),导致转录本数目和可变剪切数量减少,其中CPB3、A0A1U8IZF2、A0A1U8KCA2、A0A1U8NDW4和A0A1U8NI70同时参与了这3条通路的调控。
3 讨论与结论
纳米孔测序技术作为一种长读长测序技术,并且可以表征全长转录本以及量化转录表达,它能够在转录和转录后水平上剖析植物转录本的复杂性[33]。本研究分析了纳米孔测序平台的2个棉花品种的全长转录本数据。分析测序结果得到了超过63 Gbp的有效数据,并且读取的平均长度超过800个碱基。超过 99% 的reads成功比对到棉花基因组,这表明纳米孔测序reads的高质量。大规模、高质量的纳米孔数据保证了转录本鉴定和定量的准确性,这表明纳米孔测序是一种具有成本效益的植物全长转录本分析方法。
可变剪切会产生多个转录本,并且可能会从同一基因产生不止一种蛋白质,它会因对植物生长和发育产生负面影响的环境压力而改变。植物胁迫相关基因特别容易发生选择性剪切事件,这通常会调节活性和非活性异构体之间的比例以响应非生物胁迫,从而微调关键胁迫调节因子的表达[32]。本研究在热应激的胁迫条件下,从全转录水平展示了棉花的热敏品种Che61-72和耐热品种新陆早36号的可变剪切的变化。2个品种的棉花在高温胁迫下,可变剪切的總数大量增加,特别是热敏品种Che61-72,在被热刺激的12 h之后,可变剪切的数量增加了2倍多,并且内含子保留的事件最为显著。然后分析了2个棉花品种在热应激12 h前后基因表达模式的变化。在热敏品种Che61-72中发现了2 900个差异基因,并且差异基因在R0样本中识别到了13 251个可变剪切事件,在R12样本中识别到了25 296个可变剪切事件,其中内含子保留事件增加得最多。在新陆早36号中发现了2 437个差异表达基因,在T0样本中鉴定到了11 248个可变剪切事件,在T12样本中鉴定到了13 769个可变剪切事件,与热敏品种不同的是,耐热品种的外显子跳跃事件变化得最大。
高温会导致光合作用装置的直接损害,而光系统Ⅱ通常被认为是热诱导的光合作用失活的主要目标,光系统Ⅰ被认为可以确定生命系统中的全球焓量,因此温度升高的影响关于光系统Ⅰ可能对于调节变化环境中所有光合自养生物的光合反应至关重要[34]。叶绿体的宏观结构会因热刺激下被暴露而改变,类囊体膜的完整性受到损害,在35~45 ℃ 的温度范围内可以观察到膜堆积减少和类囊体膜的一般重组[35]。差异基因GO和KEGG富集分析发现,2个品种的差异基因都显著富集到了光系统Ⅰ的光捕获、叶绿体类囊体膜和光合作用-天线蛋白通路中,并且筛选出5个关键基因(CPB3、A0A1U8IZF2、A0A1U8KCA2、A0A1U8NDW4和A0A1U8NI70),均被注释为叶绿素a/b结合蛋白(LHC),属于光捕获叶绿素a/b结合(LHC)蛋白家族,是光捕获复合体的组成部分,作为一个光受体,它捕获并将激发能量传递给与其密切相关的光系统。Liang等在棉花纳米孔测序转录组分析中也发现叶绿体a/b结合蛋白基因(LHC)有利于耐热品种的光合稳态,参与棉花抗热调节机制[8]。在高温处理下的大麦(Hordeum vulgare)和葡萄(Vitis vinifera)中, 叶绿体a/b结合蛋白(LHC)的蛋白丰度和转录本均下降[36]。紫花苜蓿(Medicago sativa)叶片中的3种叶绿a/b结合蛋白在24 h的高温处理下,表达丰度提高,但在延长的高温胁迫过程中(72 h)蛋白丰度下降[37],表明叶绿体素结合蛋白的丰度变化可能与耐热性有关,其可能取决于高温胁迫的强度、持续时间和植物种类。此外,在低温处理的芒属植物中,叶绿素a/b结合蛋白基因上调表达,表明可能有助于保护光合作用[38]。本研究中,高温胁迫是导致棉花叶绿素a/b结合蛋白基因下调的根本原因,5个LHC基因与调控棉花光合作用动态平衡密切相关。
本研究分析了2个棉花品种在高温胁迫下转录组的可变剪切事件,发现在热处理后可变剪切事件的总数量增多,其中热敏品种增加了70 610个,耐热品种增加了9 844个。这表明转录后剪接在棉花应对高温胁迫的生物调节中起重要作用。富集分析发现,差异基因显著富集到了光系统Ⅰ的光捕获、叶绿体类囊体膜和光合作用-天线蛋白通路中,并筛选出5个关键基因,它们均为叶绿素a/b结合蛋白基因,参与高温胁迫下调控棉花光合作用动态平衡。本研究探索了棉花在高温胁迫条件下的调节机制,为后续耐高温的种质选育奠定基础、提供重要了的数据支持。
参考文献:
[1]Han Z,Wang C,Song X,et al. Characteristics,development and mapping of Gossypium hirsutum derived EST-SSRs in allotetraploid cotton [J]. Theoretical and Applied Genetics,2006,112(3):430-439.
[2]Grover A,Mittal D,Negi M,et al. Generating high temperature tolerant transgenic plants:achievements and challenges [J]. Plant Science,2013,205:38-47.
[3]Zahid K R,Ali F,Shah F,et al. Response and tolerance mechanism of cotton Gossypium hirsutum L. to elevated temperature stress:a review [J]. Frontiers in Plant Science,2016,7:937.
[4]Pettigrew W T. The effect of higher temperatures on cotton lint yield production and fiber quality [J]. Crop Science,2008,48(1):278-285.
[5]Burke J J,Wanjura D F. Plant responses to temperature extremes[M]//Physiology of cotton. Berlin:Springer,2010:123-128.
[6]Oosterhuis D M,Snider J L. High temperature stress on floral development and yield of cotton [M]//Oosterhuis D M. Stress physiology in cotton. Cordova,Tennessee:The Cotton Foundation,2011:1-24.
[7]Reddy K R,Kakanl V G,Zhao D,et al. Interactive effects of ultraviolet-B radiation and temperature on cotton physiology,growth,development and hyperspectral reflectance [J]. Photochemistry and Photobiology,2004,79(5):416-427.
[8]Liang Y,Gong Z,Wang J,et al. Nanopore-based comparative transcriptome analysis reveals the potential mechanism of high-temperature tolerance in cotton (Gossypium hirsutum L.) [J]. Plants,2021,10(11):2517.
[9]Reddy K,Hodges H,Reddy V. Temperature effects on cotton fruit retention [J]. Agronomy Journal,1992,84(1):26-30.
[10]Reddy V,Reddy K,and Baker D. Temperature effect on growth and development of cotton during the fruiting period [J]. Agronomy Journal,1991,83(1):211-217.
[11]Soliz L M A,Oosterhuis D M,Coker D L,et al. Physiological response of cotton to high night temperature [J]. Am J Plant Sci Biotechnol,2008,2:63-68.
[12]Barua D,Downs C A,Heckathorn S A. Variation in chloroplast small heat-shock protein function is a major determinant of variation in thermotolerance of photosynthetic electron transport among ecotypes of Chenopodium album[J]. Functional Plant Biology,2003,30(10):1071-1079.
[13]Min L,Zhu L,Tu L,et al. Cotton Gh CKI disrupts normal male reproduction by delaying tapetum programmed cell death via inactivating starch synthase [J]. The Plant Journal,2013,75(5):823-835.
[14]Wang Q,Liu N,Yang X,et al. Small RNA-mediated responses to low-and high-temperature stresses in cotton [J]. Scientific Reports,2016,6(1):1-14.
[15]Ma Y,Min L,Wang J,et al. A combination of genome-wide and transcriptome-wide association studies reveals genetic elements leading to male sterility during high temperature stress in cotton [J]. New Phytologist,2021,231(1):165-181.
[16]Barash Y,Calarco J A,Gao W,et al. Deciphering the splicing code [J]. Nature,2010,465(7294):53-59.
[17]Zhao L,Zhang H,Kohnen M V,et al. Analysis of transcriptome and epitranscriptome in plants using PacBio Iso-Seq and nanopore-based direct RNA sequencing [J]. Frontiers in Genetics,2019,10:253.
[18]Abdel-Ghany S E,Hamilton M,Jacobi J L,et al. A survey of the sorghum transcriptome using single-molecule long reads [J]. Nature Communications,2016,7(1):1-11.
[19]Wang B,Tseng E,Regulski M,et al. Unveiling the complexity of the maize transcriptome by single-molecule long-read sequencing [J]. Nature communications,2016,7(1):1-13.
[20]Wang Q,Rio D C. JUM is a computational method for comprehensive annotation-free analysis of alternative pre-mRNA splicing patterns [J]. Proceedings of the National Academy of Sciences,2018,115(35):EB181-EB190.
[21]Wang X,Xu Y,Zhang S,et al. Genomic analyses of primitive,wild and cultivated citrus provide insights into asexual reproduction [J]. Nature Genetics,2017,49(5):765-772.
[22]Li H. Minimap2:pairwise alignment for nucleotide sequences [J]. Bioinformatics,2018,34(18):3094-3100.
[23]Tardaguila M,de la Fuente L,Marti C,et al. SQANTI:extensive characterization of long-read transcript sequences for quality control in full-length transcriptome identification and quantification [J]. Genome Research,2018,28(3):396-411.
[24]Trincado J L,Entizne J C,Hysenaj G,et al. SUPPA2:fast,accurate,and uncertainty-aware differential splicing analysis across multiple conditions [J]. Genome Biology,2018,19(1):40.
[25]Patro R,Duggal G,Love M I,et al. Salmon provides fast and bias-aware quantification of transcript expression [J]. Nature Methods,2017,14(4):417-419.
[26]Love M I,Huber W,Anders S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2 [J]. Genome Biology,2014,15(12):550.
[27]Cantalapiedra C P,Hernández-Plaza A,Letunic L,et al. eggNOG-mapper v2:functional annotation,orthology assignments,and domain prediction at the metagenomic scale [J]. Molecular Biology and Evolution,2021,38(12):5825-5829.
[28]Yu G,Wang L G,Han Y,et al. clusterProfiler:an R package for comparing biological themes among gene clusters [J]. Omics,2012,16(5):284-287.
[29]Xie C,Mao X,Huang J,et al. KOBAS 2.0:a web server for annotation and identification of enriched pathways and diseases [J]. Nucleic Acids Res,2011,39(Web Server issue):W316-W322.
[30]Chaudhary S,Jabre I,Reddy A S N,et al. Perspective on alternative splicing and proteome complexity in plants [J]. Trends in Plant Science,2019,24(6):496-506.
[31]Kim S,Kim T H. Alternative splicing for improving abiotic stress tolerance and agronomic traits in crop plants [J]. Journal of Plant Biology,2020,63(6):409-420.
[32]Laloum T,Martín G,Duque P. Alternative splicing control of abiotic stress responses [J]. Trends in Plant Science,2018,23(2):140-150.
[33]Cui J W,Shen N,Lu Z G,et al. Analysis and comprehensive comparison of PacBio and nanopore-based RNA sequencing of the Arabidopsis transcriptome [J]. Plant Methods,2020,16(1):85.
[34]Ivanov A G,Velitchkova M Y,Allakhverdiev S I,et al. Heat stress-induced effects of photosystem Ⅰ:an overview of structural and functional responses [J]. Photosynthesis Research,2017,133(1/2/3):17-30.
[35]Allakhverdiev S I,Kreslavski V D,Klimov V V,et al. Heat stress:an overview of molecular responses in photosynthesis [J]. Photosynthesis Research,2008,98(1/2/3):541-550.
[36]Shakeel S N,Aman S,Haq N U,et al. Proteomic and transcriptomic analyses of Agave americana in response to heat stress [J]. Plant Molecular Biology Reporter,2013,31(4):840-851.
[37]Li W M,Wei Z W,Qiao Z H,et al. Proteomics analysis of alfalfa response to heat stress [J]. PLoS One,2013,8(12):e82725.
[38]Spence A K,Boddu J,Wang D F,et al. Transcriptional responses indicate maintenance of photosynthetic proteins as key to the exceptional chilling tolerance of C4 photosynthesis in Miscanthus×giganteus[J]. Journal of Experimental Botany,2014,65(13):3737-3747.
收稿日期:2022-05-17
基金項目:国家自然科学基金(编号:32060149、31760316);海南省自然科学基金(编号:320RC500、321RC469)。
作者简介:丁 宇(1996—),女,海南海口人,硕士研究生,主要从事转录组、基因组等生物信息学研究。E-mail:woshidingyuya@163.com。
通信作者:谢尚潜,博士,教授,主要从事生物信息学和基于三代测序的基因组学和转录组学研究。E-mail:sqianxie@foxmail.com。

