山桃杂交群体抗蚜优系的背景选择与标记优劣比较
刘苏宁,别航灵,王君秀,陈雪嘉,王新卫,2,王力荣,2,曹珂
山桃杂交群体抗蚜优系的背景选择与标记优劣比较

1中国农业科学院郑州果树研究所,郑州 450009;2中国农业科学院西部农业研究中心,新疆昌吉 831100
【目的】筛选‘橡皮油桃’(果实大、感蚜)ב帚形山桃’(果实小、抗蚜)F2代杂交群体中,‘橡皮油桃’遗传背景回复率高且表现抗蚜的优系,探讨桃背景选择技术体系的建立方法。【方法】首先,利用3种方法选择背景标记,分别为前期研究得到的高多态性SNP(Pre-work SNP)、全基因组范围内随机挑选的SNP(Random SNP)、以及对SNP进行功能注释后,选择突变位点影响起始密码子与终止密码子的功能性SNP(Functional SNP),3种方法最终筛选的SNP个数统一设定为775个。然后,利用这775个SNP分别计算3种不同背景标记在F2代杂交群体中121个单株的双亲回复率。通过比较3种背景标记中‘橡皮油桃’背景回复率较高的前10个单株在两两之间的重复情况,比较不同背景标记的重复性。在完成F2群体单株的抗蚜性、单果重与可溶性固形物含量评价后,分别选择单果重与可溶性固形物含量2种性状的极端表型单株各10株,通过比较两类表型单株‘橡皮油桃’回复率的差异显著性,衡量不同选择方法的优劣。最后,以抗蚜定位区间内的SNP为抗蚜前景标记,筛选F2群体内‘橡皮油桃’遗传背景回复率高且表现抗蚜的优系。【结果】Pre-work SNP、Random SNP、Functional SNP 3种背景标记的F2代单株‘橡皮油桃’背景回复率分别在36.34%—71.99%、31.75%—74.92%、4.51%—66.53%。3种背景标记筛选的‘橡皮油桃’回复率较高的前10株中,Pre-work SNP与Random SNP和Functional SNP的重复单株分别均有2个,Random SNP与Functional SNP的重复单株有6个,即Random SNP和Functional SNP两种选择方法的重复率更高。Pre-work SNP、Random SNP、Functional SNP 3种背景标记在以单果重为选择目标时,极端表型单株间‘橡皮油桃’背景回复率显著性分别为0.069、0.26和0.092,即Pre-work SNP筛选的后代‘橡皮油桃’背景回复率与单果重表型相关性最高,Functional SNP次之,Random SNP差异不显著。在以可溶性固形物含量为选择目标时,极端表型单株间‘橡皮油桃’背景回复率显著性分别为0.77、0.65和0.31,3种背景标记差异均不显著。基于Pre-work SNP和Functional SNP的计算结果,本研究筛选出2个‘橡皮油桃’回复率高的单株,分别为N20和N36,其中N20携带抗蚜标记,单株表现抗蚜,平均单果重为34.42 g,可溶性固形物含量为16.1%,为山桃F2群体的优异单株。【结论】在本研究群体内,Pre-work SNP相较于Functional SNP与Random SNP,在单果重这一表型上与‘橡皮油桃’回复率的相关性较强,印证该背景标记选择方式的优异性,以这种背景标记挑选出的单株N20在目标性状的优异表现也支持这一结果。本研究提供一种背景选择的思路以及判断不同背景标记在研究群体内优劣性的方法,可为有效提高抗性育种效率提供参考。
桃;抗蚜性;SNP;前景选择;背景选择
0 引言
【研究意义】桃蚜(Sulzer)繁殖能力极强,在短时间内即可形成庞大的种群数量,是桃树生产上的主要害虫[1]。近年来,杀虫剂的大量使用使桃蚜产生抗药性,导致防治难度增加[2-4],抗性资源的发掘利用是控制虫害更为经济有效且环保的方法[5]。但由于果树生育周期较长,通过常规育种获得优良品种至少需要10—15年,且常规育种存在目标性状选择效率低、背景回复速度慢等问题[6],分子标记技术的出现和应用为果树抗性育种提供了更为高效的方法[7]。分子标记辅助选择主要包括对目标性状的前景选择和对遗传材料的背景选择两部分[8],前景选择确保目的基因在后续杂交或者回交过程中能够逐代传递下去,而背景选择则保证子代在具备目标性状的前提下,更快地回复到优良亲本的遗传背景[9]。本研究利用3种不同的方法选择背景标记,通过比较不同背景标记与育种目标性状表型结果的一致性来衡量不同选择方式的优劣,为抗桃蚜新品种培育奠定基础。【前人研究进展】近年来,常规育种辅助分子标记以加速育种进程,在农作物与园艺作物的抗性育种或品种改良工作中广泛应用,Rai等[10]利用173个SSR(simple sequence repeats)标记,对前景选择得到的BC1F1单株进行背景选择以转移,培养软粒小麦品种。赵雅楠[11]在无核抗寒葡萄胚挽救育种工作中,选用无核标记GSLP-569对杂种株系进行早期的无核性状辅助选择,同时利用抗寒标记S241-717进行早期抗寒性状的辅助选择。而在桃背景选择标记以及抗蚜前景标记的开发上,GUAN等[12]借助二代测序技术和生物信息学工具,在360个普通桃品种中共鉴定了16 658 391个SNP(single nucleotide polymorphism),通过生物信息学分析得到775个分布于桃全基因组的、高质量的、多态性高的SNP,可以用于背景选择。王力荣等[13]以‘橡皮油桃’ב帚形山桃’杂交后代F2群体为材料,对抗蚜性状进行集群分离分析(BSA),进而采用35个InDel(insertion- deletion)进行加密,将抗性位点定位到950 kb的区间。【本研究切入点】本研究以抗蚜定位区间内的SNP位点作为前景标记,而在背景标记的筛选上,除前期研究的775个SNP位点外[12],另外在染色体上随机选择775个SNP位点,以及选择影响起始密码子和终止密码子功能的775个SNP位点,利用这3种方式选择背景标记,计算F2代单株背景回复率并比较不同背景标记与表型结果的一致性。【拟解决的关键问题】本研究以一个将桃野生近缘种的抗蚜性位点渐渗入普通桃的杂交群体为试材,比较桃背景标记的筛选方法,在筛选母本‘橡皮油桃’背景回复率高、携带抗蚜位点优系的同时,探索桃分子育种技术体系建立的方法。
1 材料与方法
1.1 试验材料
用于本研究的F2群体单株数为121株,其母本为‘橡皮油桃’(果实大、感桃蚜),父本为‘帚形山桃’(果实小、抗桃蚜)。群体定植在中国农业科学院郑州果树研究所新乡实验基地,栽培管理条件一致。
1.2 表型分析
本试验共评价3种表型,分别为抗蚜性、单果重与可溶性固形物含量。
4月下旬,在果园中出现蚜虫时,收集果园中的无翅蚜虫,接种到温室大棚育苗钵中的山桃杂交后代实生苗上,每株实生苗的新梢上接种蚜虫100头左右,1周后调查单株抗性,依据卷叶程度对每个单株单独进行抗性评价,抗性分级标准参考王力荣等[14]并略作改动。1级:未发现蚜虫;2级:为害较轻或少量卷叶;3级:为害较重,卷叶数量超过新梢叶量的50%。
在果实成熟期摘取10—20个桃果实,电子秤称总质量后计算平均单果重。依据桃种质资源描述规范[15]将单果重分成5级的方法,在本群体中,极小:<10.0 g;小:10.0 g—20.0 g;中:20.0 g—30.0 g;大:30.0 g—40.0 g;极大:≥40.0 g。平均单果重精确到0.1 g。
称量平均单果重后,取桃子果肉挤出桃汁,糖度计测定可溶性固形物(SSC)含量。同样地,依据桃种质资源描述规范[15],将可溶性固形物含量分为5级,极低:<8.0%;低:8.0%—10.0%;中:10.0%—12.0%;高:12.0%—14.0%;极高:≥14.0%。可溶性固形物含量精确到0.1%。
对群体单株完成表型评价后,依据群体内表型分布情况,分别选取各性状的极端差异表型单株做后续分析。
1.3 山桃杂交群体基因组重测序和SNP鉴定
取F2代单株幼嫩叶片置于液氮中研磨,提取叶片基因组总DNA,植物基因组DNA提取试剂盒购自北京艾德莱生物科技有限公司,基因组重测序由安诺优达基因科技(北京,中国)有限公司完成,对121个单株及亲本DNA样品构建测序文库,文库质检合格后,使用Illumina Hiseq 2500测序平台进行高通量测序,测序读长为150 bp。获得的原始数据(raw data)去除接头序列并进行数据质控后得到过滤后数据(clean data),有效测序数据利用BWA软件[16-17],比对到桃Lovell基因组[18](Lovell_Prunus_persica_ v2.0.a1)以产生SAM文件,SAM文件经SAMTOOLS[19]转换格式为BAM文件后继续进行质控,使用GATK[20]软件进行SNP的检测,从变异文件中提取SNP变异后,对SNP进行缺失率和等位基因频率(MAF)的过滤,得到最终的SNP变异信息。其中,SNP过滤条件为:QUAL<40,QD<2.0,MQ<40.0,FS>60.0,SOR>3.0,MQRankSum <-12.5,ReadPosRankSum <-8.0。
1.4 抗蚜前景标记的选择
笔者实验室前期对群体单株进行抗蚜性鉴定,依据抗性分级结果进行基因定位,鉴定的抗蚜区间为3号染色体上物理距离为950 kb的区间内,物理位置为Chr3:24570169—Chr3:25524683,在抗蚜区间内选择与连锁信号最强的标记距离最近的SNP,作为本研究的前景标记。
1.5 抗蚜背景标记的选择
采用3种不同的方法挑选抗蚜背景标记,首先,在前期研究中,借助二代测序技术和生物信息学工具,在360个普通桃品种中共鉴定了16 658 391个SNP,过滤MAF<0.05,测序深度小于10×,质量评分小于1 000,π<0.48,等位基因数大于2的SNP后,得到775个高质量、分布于桃全基因组的SNP位点[12]。本研究选择这些SNP位点的位置信息,提取杂交群体的相应位点基因型,作为第一种抗蚜背景标记,以下简称“Pre-work SNP”。此外,通过随机选择的方式,结合SNP鉴定的结果文件,依据各染色体的总变异位点数目,等比例从各染色体中随机选择位点,利用Linux系统下的shuf命令在各染色体随机选择相应数目的位点,如从1号染色体随机选择165个位点:shuf -n 165 Chr.01.txt >> shuf1.txt,从各染色体中随机选择的位点数目相加共计775个,以下简称“Random SNP”。最后,利用ANNOVAR[21]软件对SNP鉴定结果文件中所有的SNP进行功能注释,共有1 160 518个变异位点被注释,外显子区域的变异位点有132 624个,其中影响起始密码子与终止密码子功能的突变位点有1 129个,依据这些突变位点在各染色体上的位点数目,从中等比例随机挑选,从各染色体中随机选择的位点数目相加共计775个,以下简称“Functional SNP”。最后,利用VCFTOOLS[22]软件分别提取杂交群体中各单株在上述3种不同挑选方式所得到的775个位点的SNP信息,并筛选有效差异位点。
1.6 双亲背景回复率的计算
亲本1背景回复率(%)=[1-(有效差异位点数/有效位点总数)]×100。式中,有效位点总数是指在不同背景标记下双亲中存在多态性的位点总数;有效差异位点包含于有效位点,是指F2代单株与亲本1表现不一致的SNP位点个数。亲本2背景回复率计算方法同上。
2 结果
2.1 山桃F2群体表型结果
经对山桃F2群体表型评价,该群体内对桃蚜抗性1级单株36株,抗性2级单株49株,抗性3级单株28株;群体平均单果重变化范围为11.02—79.62 g,平均值为27.23 g,变异系数CV值为53.58%。可溶性固形物含量变化范围为8%—16.1%,平均值为11.45%,变异系数CV值为18.21%。
2.2 山桃F2群体重测序及SNP鉴定
平均每个个体的raw data为1.7 G,总测序数据量达到206 G,测序质量Q30>91%,各样本的序列GC含量分布正常,建库测序成功。对raw data中包含的接头信息和低质量碱基进行过滤,最终得到高质量的clean data,其总数据量为202 G,利用这些数据进行后续分析。
参考基因组(Lovell_Prunus_persica 2.0)[18]大小为230.26 Mb,将山桃杂交群体的测序序列与参考基因组序列比对,在全基因组范围内共检测到5 916 824个SNP,经过滤后,共得到2 023 619个高质量的SNP。
2.3 不同选择方式的抗蚜背景标记分布情况
由于在前期筛选高多态性SNP时最终得到775个SNP的变异信息[12],因此,为了保证3种方法的一致性,其余2种方法均将775作为筛选SNP数目的标准。3种方法筛选到的775个背景SNP在染色体上的相对位置如图1所示。
结果显示,以Pre-work SNP为依据筛选的775个SNP位点中,9个SNP位点未检测到变异信息,334个SNP位点在双亲中没有差异,剩余432个SNP在双亲中有差异,被认为是有效的差异位点。这432个SNP在第1—8染色体上的数目分别为71、59、58、48、38、73、34和33个。
以Random SNP为选择依据筛选的775个SNP位点中,146个SNP位点在双亲中没有差异,剩余630个SNP在双亲中有差异,有效差异位点在1—8号染色体上的数目分别为147、85、88、73、55、95、51和36个。
以Functional SNP为选择依据所筛选的775个SNP中,52个SNP位点在双亲中没有差异,剩余723个SNP在双亲中有差异,有效差异位点在1—8号染色体上的数目分别为153、112、100、96、78、9、87和88个。
2.4 山桃F2单株在不同背景标记下的双亲背景回复率
利用2.3筛选得到的SNP,计算F2单株在8条染色体上对双亲的回复率,如表1所示。
结果发现,以Pre-work SNP计算的‘橡皮油桃’和‘帚形山桃’平均回复率分别为55.62%和36.04%;而Random SNP在双亲的平均回复率分别为42.85%和25.79%;Functional SNP则分别为37.61%和29.61%。即3种方法均表明杂交后代中来自母本‘橡皮油桃’的遗传信息高于父本‘帚形山桃’。
分析不同染色体的亲本回复率,Pre-work SNP F2单株在6号染色体的‘橡皮油桃’平均回复率达到72.41%,变异系数为13.46%,在7号染色体的‘帚形山桃’回复率达到53.72%,变异系数为34.37%,6号与7号染色体分别为双亲回复率最高的染色体。Random SNP双亲平均回复率与Pre-work SNP结果一致,且6号染色体的橡皮油桃平均回复率达60%,7号染色体的帚形山桃背景回复率达40%。Functional SNP在F2代单株中双亲平均背景回复率与前两种有差异,‘橡皮油桃’背景回复率最高的是1号染色体,其次是6号染色体;而‘帚形山桃’背景回复率从高到低依次是4号、3号、7号染色体。该结果表明,不同染色体来自亲本的遗传背景不同,有的染色体更倾向母本,有的则更倾向父本。
本研究同时统计了F2各单株的‘橡皮油桃’背景标记回复率的频次分布,结果如图2所示,即3种方法都呈正态分布,但峰值所在的回复率有所不同。Functional SNP计算的回复率峰值较低,为30%— 40%;而Pre-work SNP和Random SNP计算的回复率峰值均为50%—60%。
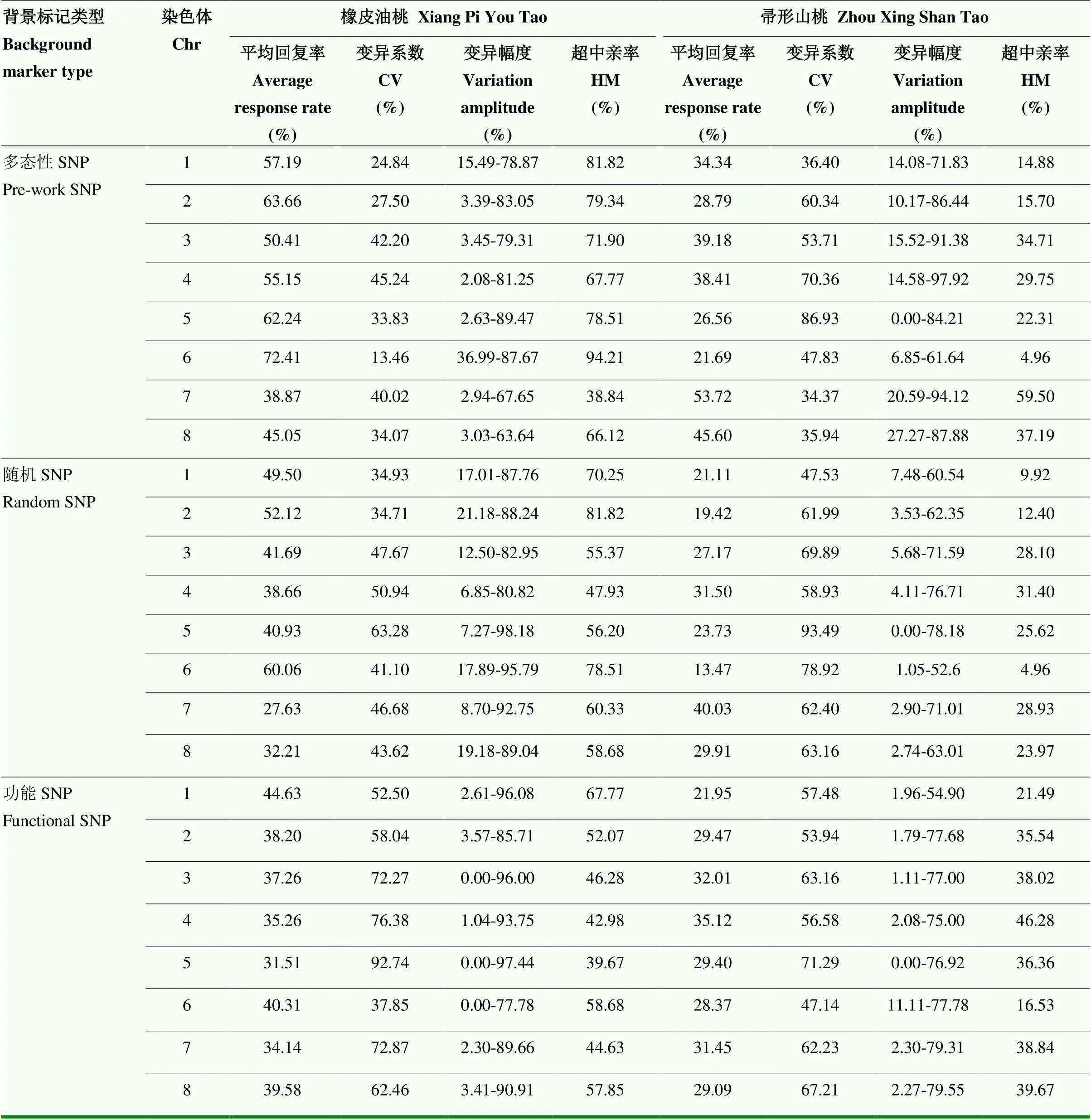
表1 F2单株在8条染色体上对双亲的回复率

图1 775个SNP标记在染色体上的分布情况

图2 F2代单株‘橡皮油桃’背景回复率频次分布
2.5 三种背景标记选择方法的重复性分析
此外,为探讨3种背景标记选择方法的重复性,本研究选择不同背景标记双亲回复率较高的前10株,分析这些单株在不同方法出现的情况。结果发现,在用Pre-work SNP筛选的‘橡皮油桃’回复率较高的前10株中,有2株在Random SNP中被鉴定到,在Functional SNP中同样仅鉴定出2株。而在Random SNP筛选的‘橡皮油桃’回复率较高的10株中,Pre-work SNP鉴定的仅2株,而Functional SNP鉴定的达到6株,即‘橡皮油桃’高回复率的单株在Random SNP与Functional SNP两种标记间的筛除率重复性较强,Pre-work SNP与Random SNP,以及Pre-work SNP与Functional SNP的重复性相当(图3-a)。同样地,在用Pre-work SNP筛选的‘帚形山桃’回复率较高的前10株中,有1株在Random SNP中被鉴定到,而在Functional SNP中未被鉴定到。在用Functional SNP筛选的‘帚形山桃’回复率较高的前10株中,有5株在Random SNP中被鉴定到,即‘帚形山桃’高回复率的单株在Random SNP与Functional SNP两种标记间的重复性强于Pre-work SNP与Random SNP、Pre-work SNP与Functional SNP(图3-b)。
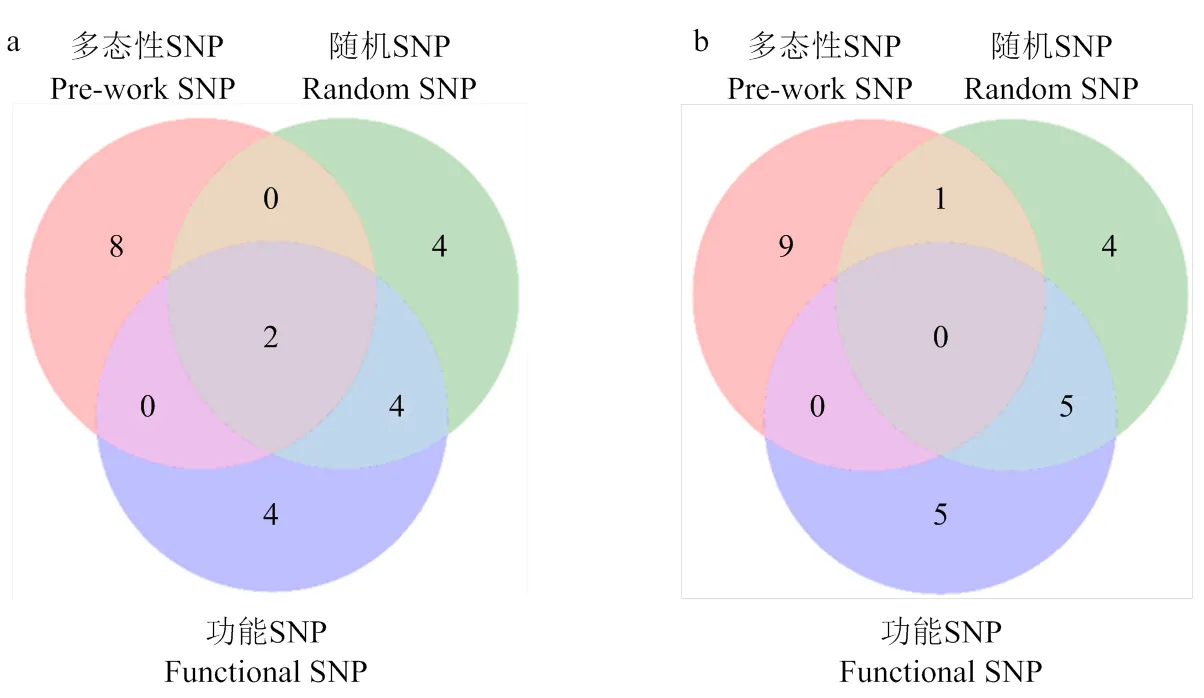
a:‘橡皮油桃’;b:‘帚形山桃’
2.6 三种背景标记选择方法的优劣分析
为比较3种背景标记选择方法的优劣,将回复率与表型进行相关性分析。由于背景标记回复率是用全基因组的SNP进行计算,而表型性状很难将所有的表型进行统计分析并得出一个整体的评价结果,因此,本研究仅选择育种家最关注的性状进行分析,以尽可能服务育种目标。为此,本研究评价杂交群体的单果重和可溶性固形物含量2个性状,同时将其与‘橡皮油桃’回复率进行联合分析,以此来衡量不同背景标记筛选方法的优劣。
首先,依据群体单株单果重的评价结果,从中分别选择大果(单果重大于30 g)与小果(单果重小于20 g)单株各10株,比较两类表型单株的‘橡皮油桃’背景回复率之间的差异(图4-a、b、c)。同样地,依据群体单株果实可溶性固形物含量的评价结果,从中分别选择可溶性固形物含量较低单株(SSC<10%)与较高单株(SSC>12%)各10株,比较两类表型单株的‘橡皮油桃’背景回复率之间的差异(图4-d、e、f)。
由图4可知,在以单果重作为评价指标时,“Pre-work SNP”筛选单株的‘橡皮油桃’回复率在两类群体中差异最大,其次为“Functional SNP”方法,而“Random SNP”筛选的单株回复率则在两类群体中无差异。而以可溶性固形物含量为评价指标时,3种背景标记筛选的单株虽然均表现为SSC高的单株,其‘橡皮油桃’的回复率也高,但回复率在两类群体间没有明显差异。
2.7 山桃F2群体优系筛选抗蚜单株示例
在依据“Pre-work SNP”与“Functional SNP”2种背景标记所筛选的‘橡皮油桃’回复率最高的10株后代中,有2株同时被筛选到(图3-a),分别为N20和N36。其中,N20在“Pre-work SNP”和“Functional SNP”中回复率分别为68.29%和52%,N36在两种背景标记中的回复率分别为71.99%和55.6%。
依据笔者课题组前期抗蚜分子标记研究成果[13],用InDel 2引物对扩增N20单株所得的PCR扩增产物在对应于Lovell基因组组装的Scaffold 3第24 761 660碱基处有9 bp的缺失,为TTTCCGGCC/---------,N36的PCR扩增产物没有缺失,认为N20携带抗蚜标记,N36不携带抗蚜标记。因此,N20是F2群体中最终筛选出的‘橡皮油桃’回复率最高且携带野生资源抗性标记的优异单株。
已知‘帚形山桃’单果重约11 g[23],而杂交单株N20单株的平均单果重为34.42 g,相比前者提高212.9%;N20的果实类型为毛桃,可溶性固形物含量为16.1%,单株表现抗蚜,抗性分级为1级,其标记分布如图5所示。
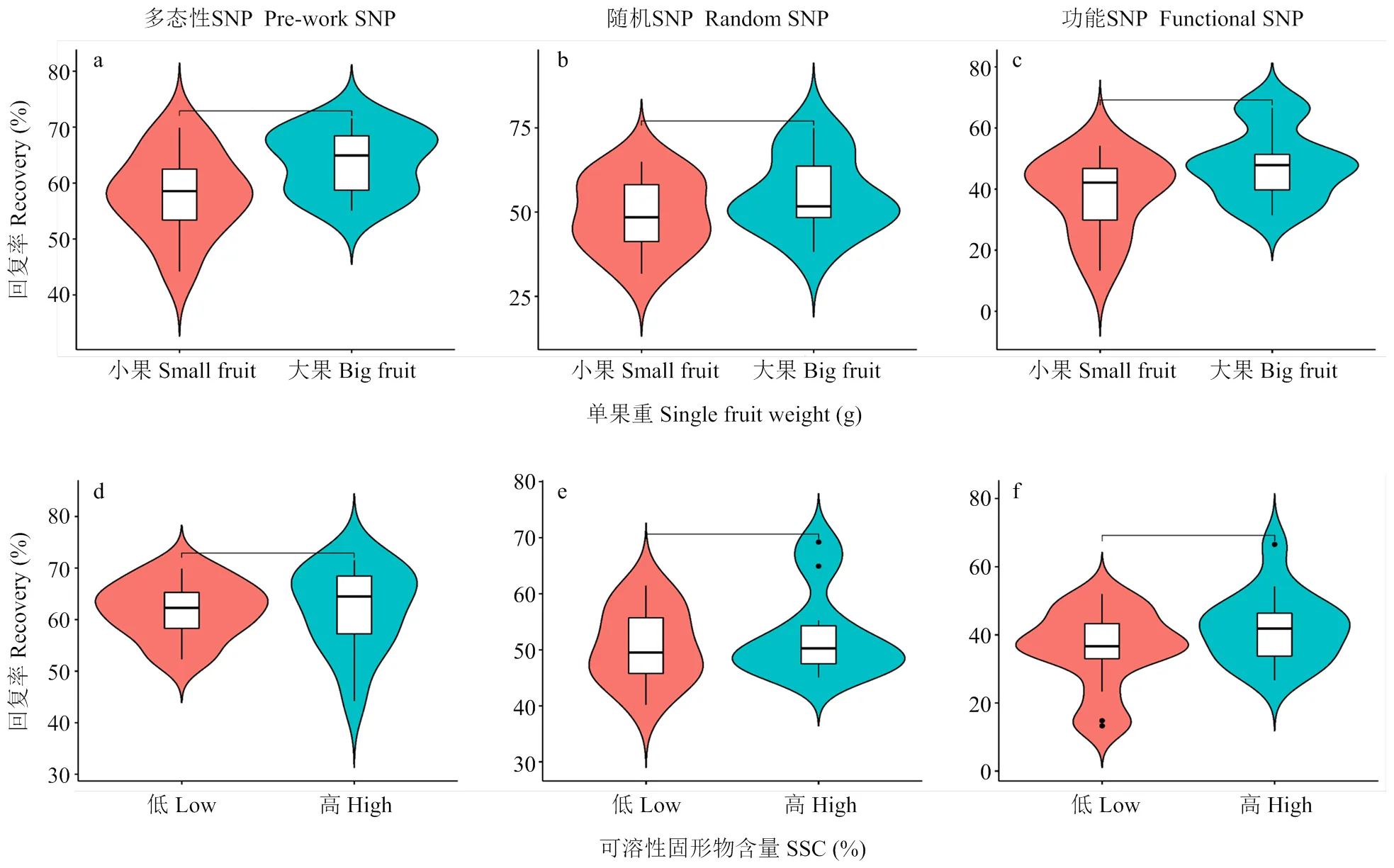
图4 极端差异表型单株‘橡皮油桃’背景回复率差异

图5 Pre-work SNP背景标记在N20染色体上的分布
3 讨论
桃蚜对桃树生长具有极其严重的危害,发掘与利用优异抗蚜种质资源,培育抗蚜品种、砧木,发掘抗蚜基因,是解决该问题的重要方法。在前期的研究中,鉴定出的抗蚜种质有寿星桃、碧桃、山桃[14,24]等,也分别开发了上述种质的抗蚜分子标记,发掘了相关候选基因[13,25-26],为桃抗蚜育种奠定了理论和方法基础。在作物的育种工作中,以分子标记为辅助手段,在苗期进行早期筛选,可以大大提高杂交后代轮回亲本的回复速度。本研究筛选出1份‘橡皮油桃’回复率高且携带抗蚜标记的种质,可以与‘橡皮油桃’继续回交,进一步提高其背景回复率,为提高‘橡皮油桃’的抗蚜性提供一种高效的方法。
3.1 前人研究所用标记类型
回交育种通常用于将有利农艺性状的基因从供体亲本转移到轮回亲本[27],WING等[28]提出的全基因组育种流程来源于FRISCH[29]“三步走”策略,即先通过优良品系与基因供体杂交得到F1代,继而以优良品系为轮回亲本对后续世代逐代回交,通过在目标位点两侧各设计1个分子标记用于负向选择以打破连锁累赘,最后挑选出回复率最高的单株。KARUNARATHNA[30]也认为,基因组背景选择结合标记前景辅助选择是一种有效的育种方法。然而,不同报道中,用于背景选择的分子标记筛选方法不尽相同。CHANDRAN等[31]为改良富含-胡萝卜素的自交系UMI1200+和UMI1230+,提高其赖氨酸和色氨酸含量,导入目标基因后,利用236个均匀分布在玉米基因组中的SSR标记进行背景选择。YU等[32]在完成500多个水稻全基因组测序后,共得到4 236 029个SNP,删除杂合基因型SNP后,继续在每100 kb随机选择2个SNP,最终筛选出5 102个SNP用于构建基因芯片RICE 6K,用于背景标记的筛选。YANG等[33]利用RICE 6K基因芯片作为背景标记,将广谱抗性基因导入丰39S以提高其稻瘟病抗性。本研究采用3种方法进行了比较分析,结果表明随机挑选SNP和筛选功能性SNP虽然二者重复率较高,但利用前期鉴定的高多态性SNP计算的背景回复率却与育种目标性状显示了较高的相关性,更适宜在桃上以单果重为目标进行后代的筛选。
3.2 背景标记优劣性分析的内在逻辑
本研究选择单果重和SSC与‘橡皮油桃’回复率的相关性作为衡量3种背景标记优劣性的评价方法。认为大果与高含量SSC性状来源于母本‘橡皮油桃’,然而,研究发现高单果重群体的‘橡皮油桃’回复率与低单果重群体有差异,但高SSC单株的‘橡皮油桃’回复率却与低SSC类群间没有明显差异。推测可能与单果重有主效基因,而SSC没有主效基因导致SSC的贡献既来自父本又来自母本有关。因此,当以微效多基因控制的性状作为育种目标时,采用背景选择的育种效率提升程度可能要低一些,需要相对更长的育种周期。
相比于准确性更好的Pre-work SNP,利用Random SNP选择的位点在染色体上的分布最为均匀,然而其准确性却较低。研究推测,利用Random SNP计算的回复率越高的单株,其在遗传背景上就更像亲本,但其标记的随机性导致其可能更多的与育种中“无用”的性状连锁,也决定了选择的“盲目性”。而Functional SNP的准确性不是最高,可能与导致表型多态性的功能性位点不仅包括本研究中的起始密码子缺失、翻译提前终止和终止缺失,也包含大量的非同义突变等有关。因此,在今后的研究中,可以尝试综合利用更多类型的功能性位点,甚至调控基因表达的启动子序列突变位点来进行综合分析,可能会得到理想的结果。
4 结论
本研究以前期抗蚜定位区间内的连锁标记为前景选择标记,以775个广泛分布于基因组的SNP为背景选择标记,分别计算不同背景标记选择方法的F2代单株的亲本背景回复率,发现该杂交后代的遗传背景更多来自母本。相对于其他两种方法(Random SNP、Functional SNP),利用多态性高的SNP(Pre-work SNP)进行背景选择显示了较高的选择效率。本研究筛选出一株母本背景回复率最高、携带抗蚜标记的单株N20,为后续桃的抗蚜性育种奠定了研究基础。
[1] 潘磊, 牛良, 鲁振华, 曾文芳, 崔国朝, 王志强. 桃树蚜虫的危害及其药剂防控. 果农之友, 2021(3): 37.
PAN L, NIU L, LU Z H, ZENG W F, CUI G C, WANG Z Q. Harm of peach aphid and its chemical control. Fruit Growers’ Friend, 2021(3): 37. (in Chinese)
[2] 柳强, 刘翠美, 王春燕. 果树农药污染的危害与解决措施. 农业工程技术, 2019, 39(20): 36.
LIU Q, LIU C M, WANG C Y. Harm of pesticide pollution in fruit trees and its solutions. Applied Engineering Technology, 2019, 39(20): 36. (in Chinese)
[3] EL-GENDY I, EL-BANOBI M I, VILLANUEVA-JIMÉNEZ J A. Bio-pesticides alternative diazinon to control peach fruit fly,(Saunders) (Diptera: Tephritidae). Egyptian Journal of Biological Pest Control, 2021, 31: 1-8.
[4] CRISAN L, BOROTA A, SUZUKI T, FUNAR-TIMOFEI S. An approach to identify new insecticides againststudy based on linear and non-linear regression techniques. Molecular Informatics, 2019, 38(8/9): 1800119.
[5] 牛良. 寿星桃抗蚜性鉴定及分子机制解析[D]. 武汉: 华中农业大学, 2019.
NIU L. Identification and molecular mechanism analysis of aphid resistance of Shouxing peach [D]. Wuhan: Huazhong Agricultural University, 2019. (in Chinese)
[6] 周洪昌. 玉米丝黑穗病分子标记辅助选择育种研究[D]. 长春: 吉林农业大学, 2011.
ZHOU H C. Study on molecular marker-assisted selection breeding of maize head smut [D]. Changchun: Jilin Agricultural University, 2011. (in Chinese)
[7] 冯艳霞. 果树育种中的新技术应用. 河北农业, 2022(7): 49-50.
FENG Y X. Application of new techniques in fruit tree breeding. Hebei Agriculture, 2022(7): 49-50. (in Chinese)
[8] 邓世峰, 王先如, 张安存, 陈次娥, 吴明. 分子标记辅助选择在我国水稻抗病育种中的研究进展. 江西农业, 2019(22): 40, 46.
DENG S F, WANG X R, ZHANG A C, CHEN C E, WU M. Research progress of molecular marker-assisted selection in rice disease resistance breeding in China. Jiangxi Nongye, 2019(22): 40, 46. (in Chinese)
[9] 杨大兵. 全基因背景分子选择改良水稻光温敏核不育系丰39S的病虫抗性[D]. 武汉: 华中农业大学, 2021.
YANG D B. Improvement of disease and pest resistance of photo- thermo sensitive genic male sterile line Feng 39S by whole gene background molecular selection [D]. Wuhan: Huazhong Agricultural University, 2021. (in Chinese)
[10] RAI A, MAHENDRU-SINGH A, RAGHUNANDAN K, KUMAR T P J, SHARMA P, AHLAWAT A K, SINGH S K, GANJEWALA D, SHUKLA R B, SIVASAMY M. Marker-assisted transfer ofgene to develop soft grain wheat cultivars. 3 Biotech, 2019, 9(5): 183-190.
[11] 赵雅楠. 无核抗寒葡萄胚挽救育种与分子标记辅助选择应用[D]. 杨凌: 西北农林科技大学, 2018.
ZHAO Y N. Embryo rescue breeding and molecular marker-assisted selection of seedless and cold-resistant grapes [D]. Yangling: Northwest A & F University, 2018. (in Chinese)
[12] GUAN L P, XU Q, CAO K, LI Y, ZHU G R, FANG W C, WANG X W, CHEN C W, GUO J, WANG Q, ZHAO Y L, WANG L R. Development of a 775 SNP array for peach based on whole-genome resequencing data, and assessment of the potential of its application. Scientia Horticulturae, 2021, 276: 109760.
[13] 王力荣, 王君秀, 李勇, 王新卫, 朱更瑞, 曹珂. 一组用于鉴定山桃杂交群体抗/感桃蚜性状的InDel标记及其应用. CN113186339A, 2021.
WANG L R, WANG J X, LI Y, WANG X W, ZHU G R, CAO K. A set of InDel markers for identification of peach aphid resistance/ susceptibility traits in a peach hybrid population and their applications, CN113186339A, 2021. (in Chinese)
[14] 王力荣, 朱更瑞, 方伟超, 左覃元, 韩立新. 桃种质资源对桃蚜的抗性评价. 果树学报, 2001, 18(3): 145-147.
WANG L R, ZHU G R, FANG W C, ZUO Q Y, HAN L X. Study on the resistance to peach aphid (Sulzer) of peach germplasm. Journal of Fruit Science, 2001, 18(3): 145-147. (in Chinese)
[15] 王力荣, 朱更瑞. 桃种质资源描述规范和数据标准. 北京: 中国农业出版社, 2005.
WANG L R, ZHU G R. Descriptors and Data Standard for Peach (L.). Beijing: China Agriculture Press, 2005. (in Chinese)
[16] LI H, DURBIN R. Fast and accurate short read alignment with Burrows-Wheeler transform. Bioinformatics, 2009, 25(14): 1754-1760.
[17] 陈凤珍, 李玲, 操利超, 严志祥. 四种常用的生物序列比对软件比较. 生物信息学, 2016, 14(1): 56-60.
CHEN F Z, LI L, CAO L C, YAN Z X. Comparison of four common biological sequence alignment tools. Chinese Journal of Bioinformatics, 2016, 14(1): 56-60. (in Chinese)
[18] VERDE I, JENKINS J, DONDINI L, MICALI S, PAGLIARANI G, VENDRAMIN E, PARIS R, ARAMINI V, GAZZA L, ROSSINI L, BASSI D, TROGGIO M, SHU S Q, GRIMWOOD J, TARTARINI S, DETTORI M T, SCHMUTZ J. The Peach v2.0 release: High- resolution linkage mapping and deep resequencing improve chromosome- scale assembly and contiguity. BMC Genomics, 2017, 18: 225.
[19] LI H. A statistical framework for SNP calling, mutation discovery, association mapping and population genetical parameter estimation from sequencing data. Bioinformatics, 2011, 27(21): 2987-2993.
[20] MCKENNA A, HANNA M, BANKS E, SIVACHENKO A, CIBULSKIS K, KERNYTSKY A, GARIMELLA K, ALTSHULER D, GABRIEL S, DALY M, DEPRISTO M A. The genome analysis toolkit: A MapReduce framework for analyzing next-generation DNA sequencing data. Genome Research, 2010, 20(9): 1297-1303.
[21] WANG K, LI M Y, HAKONARSON H. ANNOVAR: Functional annotation of genetic variants from high-throughput sequencing data. Nucleic Acids Research, 2010, 38(16): e164.
[22] DANECEK P, AUTON A, ABECASIS G, ALBERS C A, BANKS E, DEPRISTO M A, HANDSAKER B, LUNTER G, MARTH G, SHERRY S, MCVEAN G, DURBIN R. The variant call format and VCFtools. Bioinformatics, 2011, 27(15): 2156-2158.
[23] 王力荣, 朱更瑞, 方伟超. 中国桃遗传资源. 北京: 中国农业出版社, 2012.
WANG L R, ZHU G R, FANG W C. Peach Genetic Resource in China. Beijing: China Agriculture Press, 2012. (in Chinese)
[24] 曾梅, 韩立新, 高九思. 桃品种(系)抗桃蚜研究初报//华中三省(河南、湖北、湖南)昆虫学会2006年学术年会论文集, 2006: 116-118.
ZENG M, HAN L X, GAO J S. Preliminary report on peach aphid resistance in peach varieties (lines)//2006 Annual Academic Conference of the Entomological Society of Three Provinces in Central China, 2006: 116-118. (in Chinese)
[25] 张南南, 鲁振华, 崔国朝, 潘磊, 曾文芳, 牛良, 王志强. 基于SNP标记桃抗蚜性状的基因定位. 中国农业科学, 2017, 50(23): 4613-4621. doi: 10.3864/j.issn.0578-1752.2017.23.014.
ZHANG N N, LU Z H, CUI G C, PAN L, ZENG W F, NIU L, WANG Z Q. Gene mapping of aphid-resistant for peach using SNP markers. Scientia Agricultura Sinica, 2017, 50(23): 4613-4621. doi: 10.3864/j. issn.0578-1752.2017.23.014. (in Chinese)
[26] 瞿贵军, 林毅. 桃蚜关键抗性基因挖掘及抗蚜Cry蛋白预测. 华侨大学学报(自然科学版), 2023, 44(1): 94-103.
QU G J, LIN Y. Discovery of key resistance genes ofand prediction of anti-aphid cry proteins. Journal of Huaqiao University (Natural Science), 2023, 44(1): 94-103. (in Chinese)
[27] HASAN M M, RAFII M Y, ISMAIL M R, MAHMOOD M, RAHIM H A, ALAM M A, ASHKANI S, MALEK M A, LATIF M A. Marker-assisted backcrossing: A useful method for rice improvement. Biotechnology, Biotechnological Equipment, 2015, 29(2): 237-254.
[28] WING R A, PURUGGANAN M D, ZHANG Q F. The rice genome revolution: from an ancient grain to Green Super Rice. Nature Reviews Genetics, 2018, 19(8): 505-517.
[29] FRISCH M. Breeding strategies: Optimum design of marker-assisted backcross programs//Biotechnology in Agriculture and Forestry. Berlin/Heidelberg: Springer-Verlag, 2005: 319-334.
[30] KARUNARATHNA N L, PATIRANAGE D S R, HARLOFF H J, SASHIDHAR N, JUNG C. Genomic background selection to reduce the mutation load after random mutagenesis. Scientific Reports, 2021, 11: 19404.
[31] CHANDRAN S, PUKALENTHY B, ADHIMOOLAM K, MANICKAM D, SAMPATHRAJAN V, CHOCKLINGAM V, ESWARAN K, ARUNACHALAM K, JOIKUMAR MEETEI L, RAJASEKARAN R, MUTHUSAMY V, HOSSAIN F, NATESAN S. Marker-assisted selection to pyramid the() and β-carotene () genes in maize. Frontiers in Genetics, 2019, 10: 859.
[32] YU H H, XIE W B, LI J, ZHOU F S, ZHANG Q F. A whole-genome SNP array (RICE 6K) for genomic breeding in rice. Plant Biotechnology Journal, 2014, 12(1): 28-37.
[33] YANG D B, TANG J H, YANG D, CHEN Y, ALI J, MOU T M. Improving rice blast resistance of Feng39S through molecular marker-assisted backcrossing. Rice, 2019, 12: 70.
Background Selection and Comparison of Marker Superiority and Inferiority of Aphid-Resistant Seedlings in an Interspecific Cross Peach Population

1Zhengzhou Fruit Research Institute, Chinese Academy of Agricultural Sciences, Zhengzhou 450009;2Western Agricultural Research Center, Chinese Academy of Agricultural Sciences, Changji 831100, Xinjiang
【Objective】To establish a background selection system in peach, the seedlings contained aphid-resistance locus and high female parent recovery rate were screened from an F2population crossed by Xiang Pi You Tao peach (big fruit and susceptible to aphid) and Zhou Xing Shan Tao peach (small fruit and resistant to aphid). 【Method】Firstly, three methods were used to select background markers, including the high polymorphic single nucleotide polymorphism (SNP) obtained from the previous study (Pre-work SNP), SNP randomly selected in the whole genome (Random SNP), and functional SNP affecting the start and stop codon (Functional SNP). The number of final SNP selected by the above methods were 775. Then, using these SNPs, the parents recovery rate for all 121 individuals of the F2population were calculated, respectively. The repeatability of the selection methods was evaluated by comparing whether the top 10 seedlings with different selection markers were coincident or not. After completing the evaluation of aphid resistance, single fruit weight, and soluble solids content of F2population, 10 seedlings with extreme phenotypes for the single fruit weight and soluble solids content were selected, respectively. And the superiority and inferiority of different selection methods were estimated by comparing the significance of the differences in Xiang Pi You Tao recovery rates between the two types of phenotypes. Finally, the SNPs in the aphid-resistant location area were used as the foreground markers to screen the elite seedlings with high maternal genetic background and aphid resistance. 【Result】The background recovery rates of the F2seedlings which calculated by the three methods were 36.34%-71.99%, 31.75%-74.92%, and 4.51%-66.53%, respectively. Among the top 10 seedlings with high Xiang Pi You Tao recovery rates screened by the three background markers, Pre-work SNP and Random SNP had two duplicate single plants, and so do Pre-work SNP and Functional SNP, and there were 6 repetitive single plants in Random SNP and Functional SNP. This result indicated that the repeatability between the Random SNP and Functional SNP was the highest among all comparisons. When single fruit weight was selected as the breeding target, among the extreme phenotypic monocots, the three background markers, such as Pre-work SNP, Random SNP, and Functional SNP, had a significant Xiang Pi You Tao background recovery rate of 0.069, 0.26, and 0.092, respectively, which meant high relativity was found between the background recovery rate calculated by Pre-work SNP and their fruit weight, followed by Functional SNP, and Random SNP difference was not significant. When soluble solids content was selected as the target, the Xiang Pi You Tao background recovery rates among extreme phenotypic monocots were significant at 0.77, 0.65 and 0.31, respectively, and the differences among the three background markers were not significant. Finally, two individuals with high recovery rate of Xiang Pi You Tao peach were screened, including N20 and N36. Among them, N20 comprised the aphid-resistant markers, and this individual showed aphid resistance with an average fruit weight of 34.42 g and soluble solids content of 16.1%, which was considered to be the superior single strain of this population. 【Conclusion】In this study population, Pre-work SNP showed a stronger correlation between single fruit weight and Xiang Pi You Tao background recovery rates than Functional SNP and Random SNP, confirming the superiority of this background marker selection method, and the superior performance of N20 plants, which selected with this background marker in the target traits also supported this result. This study provided an idea of background selection and a method to judge the superiority and inferiority of different background markers in the study population, which could effectively improve the efficiency of resistance breeding in fruit crops.
peach; aphid-resistant; SNP; foreground selection; background selection

10.3864/j.issn.0578-1752.2023.15.013
2022-11-03;
2023-02-28
中国农业科学院科技创新工程专项(CAAS-ASTIP-2020-ZFRI)
刘苏宁,E-mail:lsn310@outlook.com。通信作者曹珂,E-mail:wyandck@126.com
(责任编辑 赵伶俐)

