基于COVID-19 相关基因的肝细胞癌分子分型及预后模型构建与验证
元喆悦,白易,童文,张晓雨,崔壮
(1.天津医科大学公共卫生学院,天津 300070;2.天津市第一中心医院肝胆胰外科,天津 300192;3.天津医科大学临床医学系,天津 300070)
肝细胞癌是全球发病率第五的肿瘤,死亡率却高达第二,且仍有上升趋势[1]。最近在中国进行的一项全国性研究显示,肿瘤患者感染新型冠状病毒肺炎(corona virus disease 2019,COVID-19)后,会较正常人群出现更为严重的临床症状[2]。此外免疫状态改变、炎症通路增加、宿主基因表达增加和表观遗传改变还会让肿瘤患者成为易感人群[3]。
新型冠状病毒(SARS-CoV-2)具有广泛的器官亲和性,并且可在肝脏和许多其他肺外器官中检测到其RNA 表达[4]。原位杂交显示,SARS-CoV-2 病毒粒子在血管管腔和门静脉内皮细胞中富集[5]。透射电镜观察到肝细胞细胞质中存在典型的SARSCoV-2 病毒颗粒,表现出明显的细胞膜功能障碍、线粒体肿胀和内质网扩张[6]。这些发现表明,SARSCoV-2 可能直接导致肝细胞病变。
COVID-19 相关基因是指与SARS-CoV-2 感染和COVID-19 疾病发展相关的基因。这些基因可以影响病毒的入侵和复制,调控宿主免疫应答以及改变个体对疫苗和药物的治疗反应。通过研究这些基因,可以更好的理解疾病的发病机制、寻找潜在的治疗靶点和开发个体化的治疗策略。如血管紧张素转换酶2(ACE2)是SARS-CoV-2 的主要受体,可介导病毒进入细胞[7]。肝脏单细胞RNA 测序分析显示,ACE2 在胆管细胞中的表达水平最高,其次是肝窦内皮细胞(LSECs)和肝细胞[8]。肝导管类器官培养显示,部分COVID-19 肝损伤可能是由于SARS-CoV-2 感染引起的胆管细胞直接损伤和胆汁酸积聚[9]。这些数据表明,肝脏是SARS-CoV-2 的潜在靶点,SARSCoV-2 直接与胆管细胞结合,导致胆管细胞损伤和胆管功能障碍,可能是肝损伤的机制之一[10-12]。
本研究基于COVID-19 相关基因对肝细胞癌进行分子亚型分类,并评估不同分子亚型与预后及临床特征的关系,以期为寻找肝细胞癌新的治疗靶点提供思路。
1 材料与方法
1.1 数据下载及相关基因集的来源 肝细胞癌患者组织的基因表达数据和临床随访信息分别从TCGA、HCCDB18 数据库(http://lifeome.net/database/hccdb/home.html)及Gene Expression Omnibus(GEO)数据库下载。从NCBI(https://www.ncbi.nlm.nih.gov/)以及genecards(https://www.genecards.org/)网站搜集到134 个COVID-19 相关的基因。
1.2 数据预处理 对TCGA-LIHC 的RNA-Seq 数据做如下处理:(1)去掉没有临床随访信息、生存时间、生存状态的样本。(2)将Ensembl ID 转换为基因名。(3)相同基因具有多个表达值的取均值。(4)过滤掉在样本中的表达量低于1 且占比高于50%的基因。
对GEO 的数据集做以下处理:(1)去掉没有临床随访信息、生存时间、生存状态的样本。(2)将探针转为基因名。(3)如一个探针对应多个基因,去除该探针。(4)相同基因具有多个表达值的取均值。
对HCCDB18 的RNA-Seq 数据做如下处理:去除没有临床随访信息、生存时间、生存状态、基因表达数据的样本。
3 组数据经预处理后,TCGA-LIHC 共有365 个样本;HCCDB18 数据集共有203 个样本;GSE14520共有221 个样本。
1.3 鉴定COVID-19 基因相关肝细胞癌分子亚型TCGA 表达谱数据去除所有样本中表达量小于1 且占比低于50%的基因后,有103 个COVID-19 相关基因。提取这些基因的表达量进行单因素Cox分析,以阈值P<0.05 进行过滤,得到预后相关基因。使用ConsensusClusterPlus(V1.48.0;参数:reps=100,pItem=0.8,pFeature=1,distance="euclidean")对TCGA 样本进行一致性聚类。D2 和欧氏距离分别作为聚类算法和距离度量。
1.4 分子亚型之间的免疫评分比较分析 分别使用R软件包StromalScore、ImmuneScore、ESTIMATEScore以及MCPcounter 评估10 种免疫细胞的评分。GSCA包的ssGSEA 方法计算28 种免疫细胞的评分。CIBERSSORT 包计算22 种免疫细胞的评分。
1.5 构建基于COVID-19 基因的预后风险模型
1.5.1 训练集和验证集划分 首先将TCGA 数据集中的365 个样本分成训练集和验证集,为避免随机分配偏差影响后续建模的稳定性,预先对所有样本进行100 次有放回随机分组,分组抽样按照训练集∶验证集=1∶1 比例进行。根据以下条件挑选最合适的训练集和验证集:(1)在年龄的分布、性别、随访时间以及患者死亡比例上两组相似。(2)随机分组的两个数据集基因表达谱聚类后,二分类样本数量接近。训练集和测试集样本使用χ2检验进行比较。将HCCDB18 数据集及GSE14520 数据集作为外部验证集。
1.5.2Lasso Cox回归分析 使用R 软件包glmnet进行lasso Cox回归分析,首先分析每个自变量的变化轨迹,再使用10-fold 交叉验证进行模型构建,分析每个lambda 下的置信区间,找到模型最优时候的目标基因数目。
1.6 风险评分与通路的关系 为了观察不同样本的风险得分与生物学功能的关系,选择这些样本对应的基因表达谱,使用R 软件包GSVA 进行单样本GSEA 分析,计算每个样本在不同功能上的得分即得到了每个功能对应各个样本的ssGSEA 评分,进一步计算这些功能与风险得分的相关性,选择相关性大于0.45 的功能进行聚类分析。
1.7 风险评分与临床特征构建列线图 利用TCGA全部数据集,整合风险评分和其他多因素变量构建列线图模型。并使用校正曲线评估模型的准确性。
2 结果
2.1 基于预后基因聚类3 个分子亚型 通过预后分析,得到28 个与预后相关的COVID-19 基因。基于这些基因,使用ConsensusClusterPlus 在K=3 时的分组,得到了3 个分子亚型(C1、C2、C3),见图1A。同时分析了28 个基因在不同亚型中的表达热图(图1B)。结果显示:无论在总生存时间还是在无进展生存时间方面,不同亚型之间均有差异,其中C2亚型预后最好(图1C、D)。
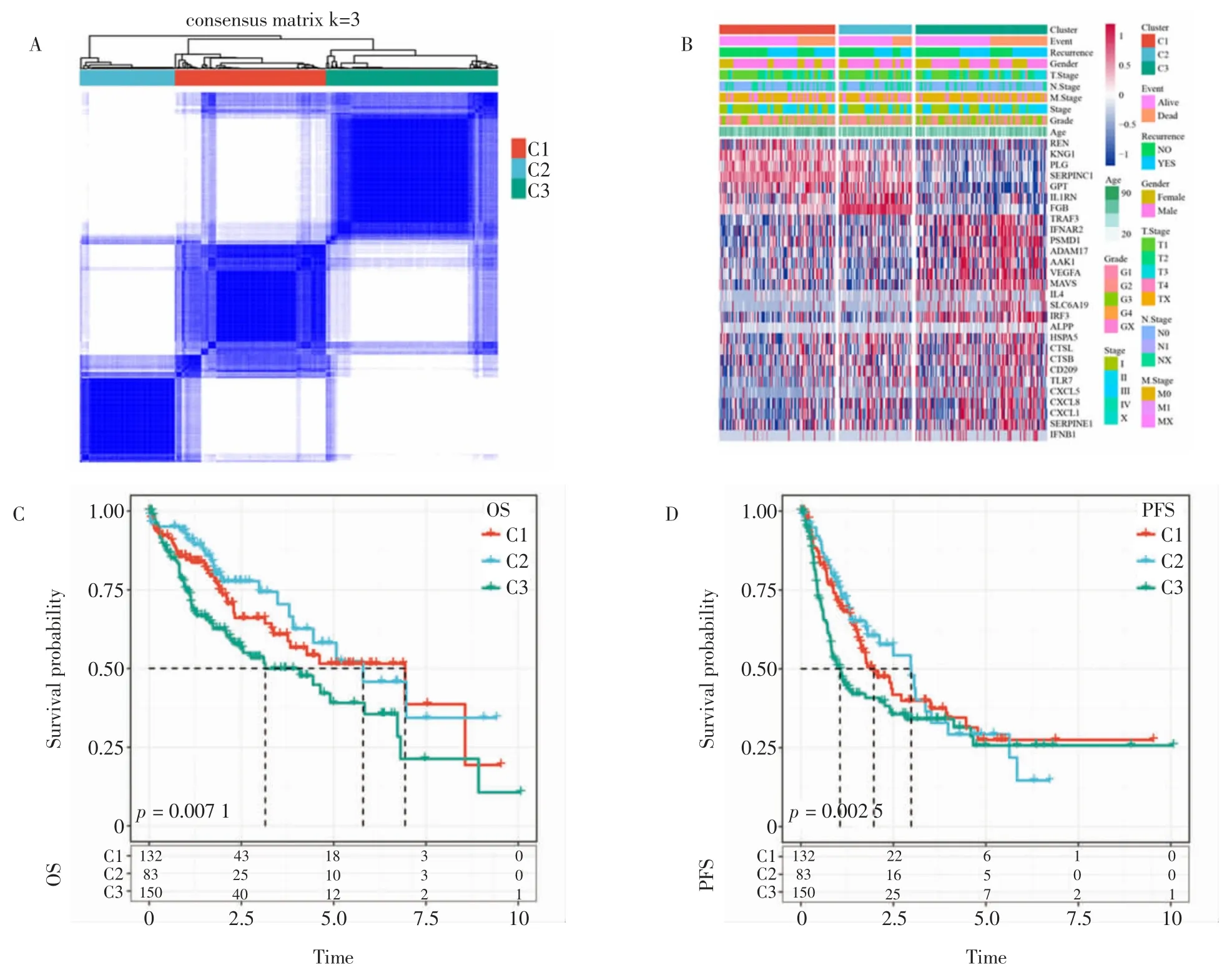
图1 基于ConsensusClusterPlus 包对肝细胞癌患者进行分型Fig.1 Hepatocellular carcinoma patients were typing based on the ConsensusClusterPlus package
2.2 比较分析不同分子亚型之间的免疫评分 分别使用StromalScore、ImmuneScore、ESTIMATEScore和MCPcounter R 包评估10 种免疫细胞的评分。GSCA 包的ssGSEA 方法计算28 种免疫细胞的评分。CIBERSSORT 包计算22 种免疫细胞的评分。结果显示,4 种方法的免疫评分中C2 亚型的免疫评分普遍高于C1 和C3 亚型(图2)。
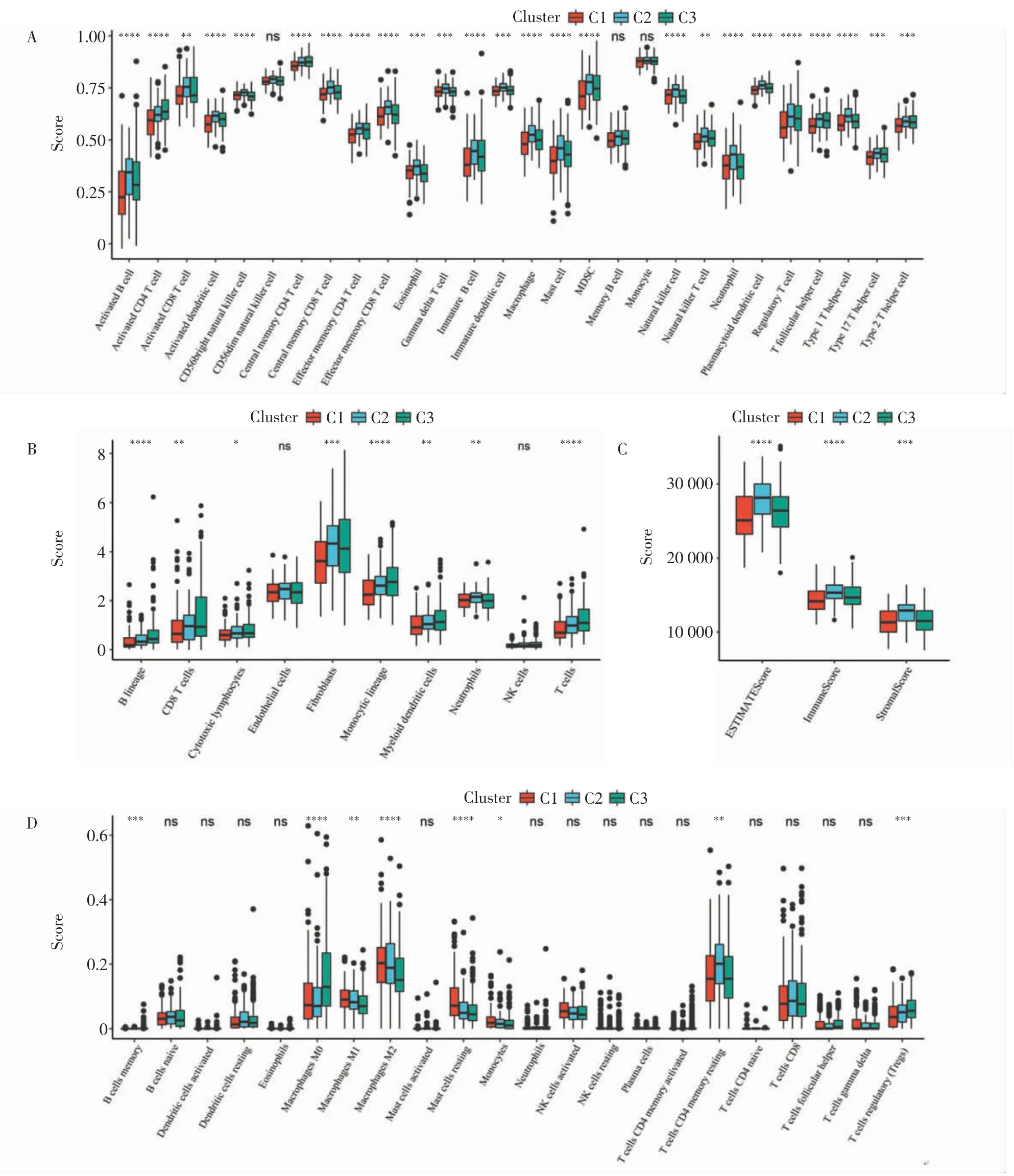
图2 肝细胞癌分子亚型之间免疫评分的比较Fig.2 Comparison of immune scores among molecular subtypes of hepatocellular carcinoma
2.3 构建基于COVID-19 基因的肝细胞癌预后模型
2.3.1 训练集样本随机分组 根据方法部分的分组方式,最终训练集数据共182 个样本,验证集数据共183 个样本,χ2检验结果表明本研究分组合理,组间无统计学差异(P>0.05)。使用训练集数据,针对每1 个COVID-19 基因以及生存数据利用R包survival coxph function 构建单变量Cox比例风险回归模型,选择P<0.05 作为阈值进行过滤,最后得到预后相关基因,分别为VEGFA、CD14、CD209、REN、PSMD1。
2.3.2 预后模型的构建与评估 使用R 软件包glmnet 进行lasso Cox回归分析,对这6 个基因进一步降维,以减少风险模型的基因数量。如图3A 所示,首先分析每个自变量的变化轨迹,可以看出随着lambda 的逐渐增大,自变量系数趋于0 的个数也逐渐增多,使用10-fold 交叉验证进行模型构建,分析每个lambda 下的置信区间。如图3B 所示,当lambda=0.012 时模型达到最优,选择对应的5 个基因进行后续分析。对筛选的5 个基因进行多因素Cox分析,并计算每一个基因的风险系数,并得到风险得分的公式:风险得分=(0.227×VEGFA)-(0.125×CD14)+(0.208×CD209)+(-0.124×REN)+(0.034×PSMD1)。
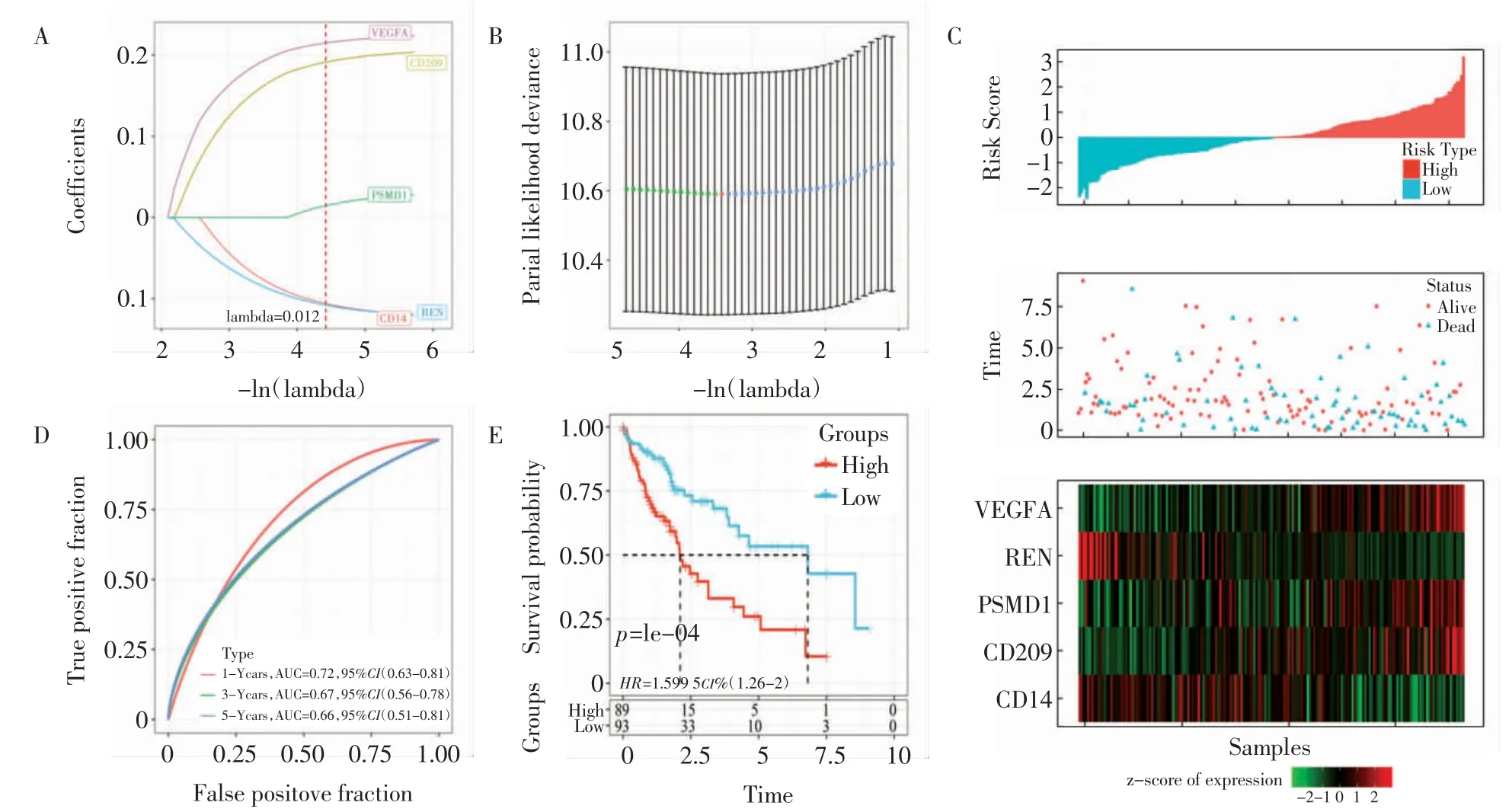
图3 使用lasso 回归算法构建预后模型Fig.3 Constructing a prognostic model using lasso regression algorithm
如图3C 所示,根据样本的表达水平分别计算每个样本的风险评分,并绘制样本的风险评分分布。此外,使用R 包timeROC 对风险评分用于预后分类的受试者工作特征(ROC)曲线进行分析,如图3D所示,通过分别分析1、3、5 年生存率的预后预测效率,可以看出该模型具有较高的曲线下面积(AUC);最后对风险评分进行zscore 分析,将样本划分为高风险组(n=89,风险评分>0)和低风险组(n=93,风险评分<0)。此外,通过绘制KM 生存曲线可以看出高风险组和低风险组存在显著差异(图3E,P<0.05)。
2.3.3 内部数据集验证5 种基因风险评分的鲁棒性 TCGA 的验证集采用与训练集相同的模型和相同的系数,根据样本的表达水平分别计算风险得分,并绘制RiskScore 分布(图4A),由图可见,风险得分高的样本具有更差的预后。1、3、5 年的预后ROC 分析如图4B 所示。最后对风险得分进行zscore 转化,将大于零的样本划分为高风险组,小于零的样本划至低风险组,并绘制KM 曲线,可以看到显著的生存差异(图4C,P<0.001)。
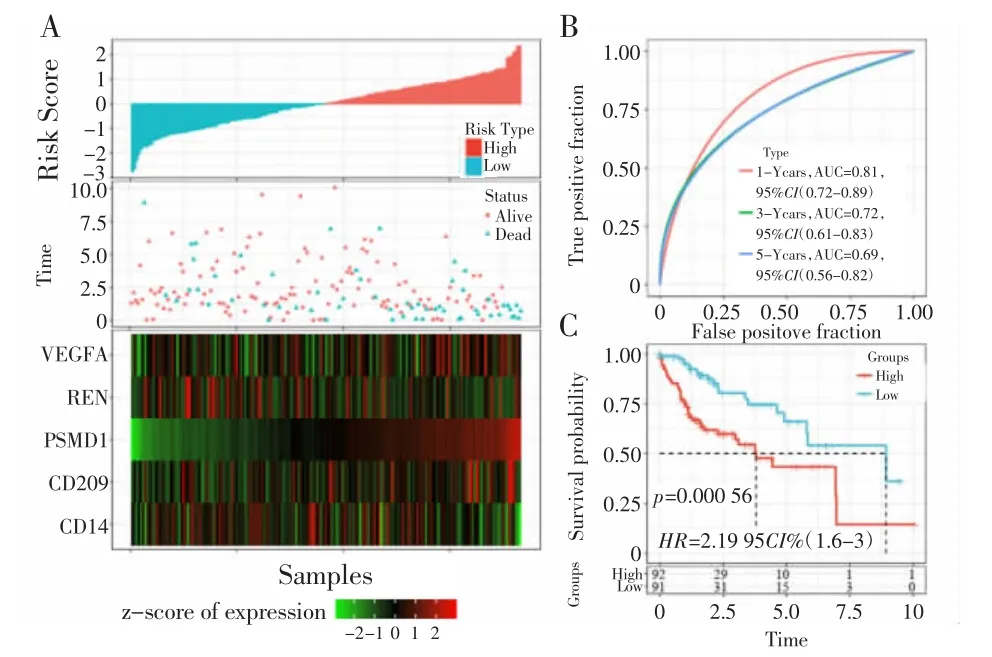
图4 内部数据集验证预后模型的鲁棒性Fig.4 Internal data sets validated the robustness of the prognostic model
2.3.4 外部数据集验证5 种基因风险评分的鲁棒性 在外部验证集GSE14520 和HCCDB18 中采用与训练集相同的模型和相同的系数。同样根据样本的表达水平分别计算风险得分,并绘制样本的风险得分分布。如图5A 所示,风险得分高的样本预后更差。该模型在GSE14520 集合中的1、3、5 年的预后预测分类效率如图5B 所示;对风险得分进行zscore 转化,将大于零的样本定义为高风险组(n=101),小于零的样本定义为低风险组(n=120),生存分析显示高风险组患者预后显著劣于低风险组(图5C,P<0.001)。
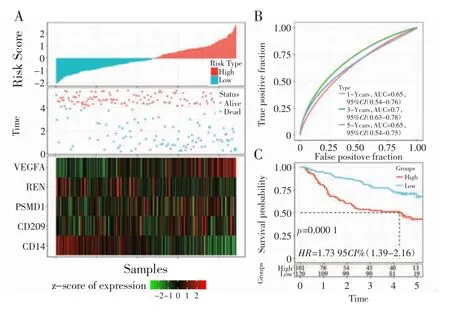
图5 外部数据集GSE14520 验证预后模型的鲁棒性Fig.5 External data set GSE14520 verified the robustness of the prognostic model
独立验证数据集HCCDB18 的风险得分分布如图6A 所示,也得到了相似的结果。进一步使用timeROC 对风险得分进行预后分类的ROC 分析,因为该数据集中,5 年生存样本太少,因此只分析了1、3、4 年的预后预测分类效率(图6B);最后对风险得分进行zscore 转化,发现高风险组(n=95)和低风险组(n=108)间具有统计学差异(图6C,P<0.05)。
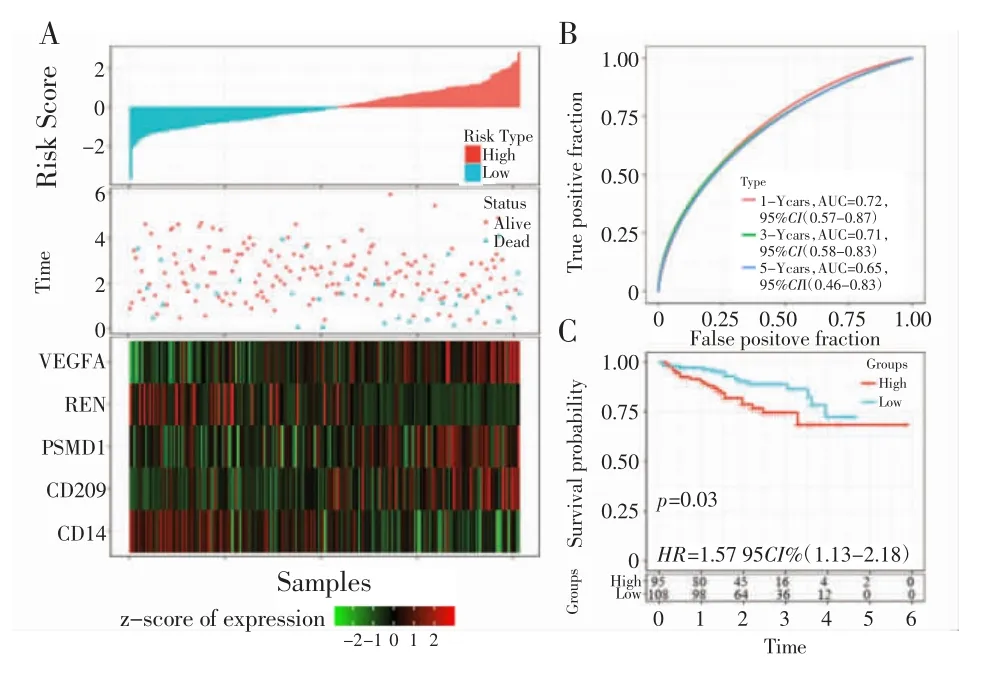
图6 外部数据集HCCDB18 验证预后模型的鲁棒性Fig.6 External dataset HCCDB18 verified the robustness of the prognostic mode
2.4 风险模型与肝细胞癌患者临床特征的预后分析 基于血管内皮生长因子α(VEGFA)、CD14、CD209、REN及PSMD1构建的5 基因风险评分模型的临床亚组生存分析显示,该预测模型可以显著区分年龄、性别、T 分期、N0 分期、M0 分期、Ⅰ+Ⅱ期、分级、复发亚组的预后(图7,P<0.05)。
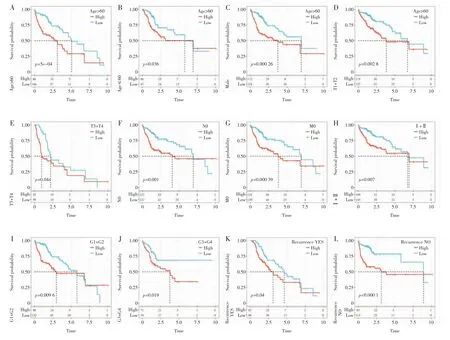
图7 预后模型在不同临床特征上的表现Fig.7 The performance of the prognostic model on different clinical features
2.5 构建基于风险评分的列线图 使用多因素Cox回归分析5 基因特征模型在TCGA 数据集中的临床独立性,结果显示,风险评分(图8A,HR=1.8,95%CI:1.23~2.65,P=0.003)与生存显著相关。根据风险评分构建了列线图模型(图8B)。校准曲线证明该模型具有准确的预测性能(图8C)。
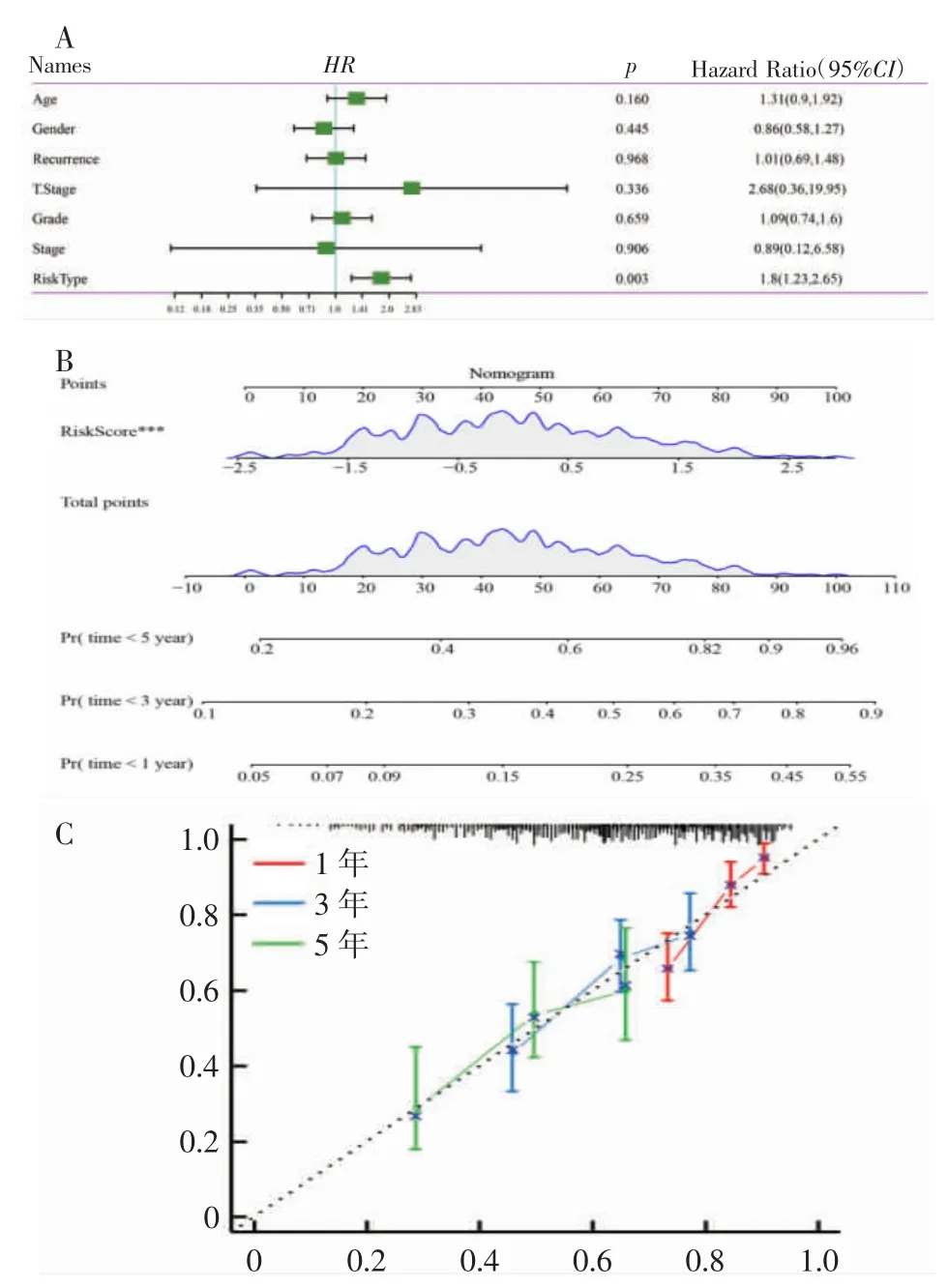
图8 基于5 基因预后模型构建列线图及其预测表现的验证Fig.8 Construction of a nomogram based on a 5-gene prognostic model and validation of its predictive performance
3 讨论
由于肝细胞癌的异质性,肝细胞癌患者可能存在与预后相关的不同分子亚型,考虑到肝细胞癌有限的治疗获益和极差的预后,迫切需要建立新的预后模型。本研究基于28 个COVID-19 预后相关的基因对TCGA 的365 个肝细胞癌样本进行分子分型,发现可以将这些样本分为3 个亚型,且具有不同的临床特征和预后结局。
近年来,关于肝细胞癌预后模型的研究越来越多,但基于COVID-19 相关基因对肝细胞癌预后进行评估尚无相关报道。本研究基于COVID-19 相关分子分型建立了一个新的5 基因模型(包括VEGFA、CD14、CD209、REN、PSMD1)用于肝细胞癌预后预测,并采用内部和外部验证的方法,在多个数据库中得到了验证。其中VEGFA 在血管生成过程中起关键作用[13]。而血管生成是恶性肿瘤发生、发展的重要过程,与肿瘤的进展和转移有关。肿瘤细胞可以上调VEGFA 的表达,促进肿瘤血管生成[14]。研究表明,增加VEGFA 在肝细胞癌中的表达,可抑制肿瘤细胞的增殖和迁移[15-18]。CD14 抗原是一种糖基磷脂酰肌醇连接的糖蛋白,在Toll 样受体(TLR)的信号通路中起着至关重要的作用[19]。CD14 在膀胱癌细胞中的表达可促进细胞因子产生和肿瘤生长[20]。CD14 阳性巨噬细胞的大量浸润预示着乳腺癌的早期复发[21]。研究提示,CD14 多态性可能增加幽门螺杆菌感染者患胃癌的风险[22]。在肝细胞癌细胞中,CD14 的过度表达可以减少肝细胞癌细胞的凋亡,抑制CD14 表达可抑制脂多糖/TLR4 信号转导,从而明显减轻肝细胞癌的炎症和进展[23]。这些研究提示CD14 可能是一个潜在的侵袭性生物标志物。DC-SIGN(CD209)是树突状细胞特异性的C 型凝集素超家族受体,在感染、树突状细胞迁移和T 细胞激活的初始步骤中具有模式识别受体的功能[24]。一些证据表明DC-SIGN(CD209)和传染病之间存在联系,例如登革热、结核病和艾滋病和川崎病等[25-27],并且CD209L 和CD209 在SARS-CoV-2 靶器官中广泛表达,可能参与感染和致病过程[28]。在肿瘤领域[29],DC-SIGN(CD209)还通过调节Janus 激酶2/信号转导和转录激活因子3 信号通路介导胃癌进展[30]。LSIGN(CD209L)和DC-SIGN(CD209)介导丙型肝炎病毒对肝细胞的反复感染,而持续感染丙型肝炎病毒可能发展为肝炎或肝硬化,甚至引起肝细胞癌[31]。有证据显示,REN 基因与终末期肾病相关[32],但尚无肿瘤方面研究。PSMD1 和PSMD2 都是蛋白酶体26S 的重要亚基[33]。许多研究也报道了PSMD1 在细胞增殖中的作用。例如,PSMD1 在未分化的甲状腺癌组织中表达上调[28],可以调节乳腺癌细胞的生长,在乳腺癌细胞对三苯氧胺耐药的发展过程中起重要作用[34]。肿瘤内表达PSMD1 是胃癌患者无病生存率和总生存期的独立预测因子[35]。此外,PSMD1 通过促进细胞内脂滴的积累而促进肝细胞性肝细胞癌细胞系的增殖,有望成为一个新的治疗靶点[36]。本研究构建预测模型中的大部分基因参与了肿瘤的发生、发展过程,并且与肝细胞癌肿瘤细胞的生长、转移或侵袭密切相关,这些发现为进一步研究肝细胞癌的预后和寻找晚期肝细胞癌治疗的分子靶点提供了新的视角。
本研究也存在一定局限性。首先,这些结论基于单一平台,并且是回顾性研究,因此还需要来自不同中心和不同平台的数据来进一步测试评估模型的性能。其次,由于样本数量有限,可能会导致选择偏倚。因此建议开展大规模、多中心、前瞻性的研究来验证本研究结果,并对本研究中鉴定的5 种基因进行更深入的细胞实验和动物研究,以确保该模型在肝细胞癌预后和治疗价值方面的作用。

