基于SSR标记的紫花风铃木群体遗传多样性分析
黄稚清, 吴林源, 高筱钰, 丁释丰, 冯志坚*, 秦新生, 刘奕彤,肖观康
基于SSR标记的紫花风铃木群体遗传多样性分析
黄稚清1,2, 吴林源1,3, 高筱钰1, 丁释丰1, 冯志坚1*, 秦新生1, 刘奕彤2,肖观康1
(1. 华南农业大学林学与风景园林学院,广州 510642;2. 广东生态工程职业学院生态工程学院,广州 510520;3. 广东职业技术学院轻工与材料学院,广东 佛山 528041)
为揭示紫花风铃木()的群体遗传变异特征,对广东省6市12个群体72份种质材料进行遗传多样性和群体遗传结构分析。结果表明,9对引物共扩增出123个等位基因位点,引物的平均多态信息量为0.754,具有较高的多态性。12个群体均具有较高的遗传多样性,群体间平均有效等位基因数为3.272个,平均Shannon指数为1.159。AMOVA分析表明群体间遗传分化程度相对较低,群体内遗传分化程度较高。群体的总体遗传分化系数为0.077,处于中等程度。基于Structure分析、主坐标分析和NJ聚类分析均可将12个群体分为2大类群,分组结果具有一定相似性,表明供试紫花风铃木群体遗传结构较为简单。这为紫花风铃木优良种质资源的利用、遗传变异和科学育种提供了理论参考。
紫花风铃木;遗传多样性;SSR标记;遗传结构
紫花风铃木()是紫葳科(Bignoniaceae)风铃木属落叶乔木,原产于南美和中美地区,可作为用材树种和观赏树种。盛花期时观赏效果极佳,花量大,花色多样[1–3]。自20世纪70年代引入我国,紫花风铃木常被用作行道树、庭院树、园林观花树种且表现良好[4–5]。但目前, 我国紫花风铃木园林应用具有如下问题:一是引种来源较为复杂,前人研究已发现我国园林栽培种中存在紫花风铃木和红花风铃木的杂交种[6]以及种质资源和命名混乱的情况[7]。二是紫花风铃木良种选育工作比较滞后,园林应用中可观测到不同个体在观赏效果上的差异,有的个体花量小、花色不显著, 群植时花期不统一。因此亟待开展紫花风铃木的遗传多样性研究,有助于推动繁育和保存紫花风铃木优质种质资源工作的开展,同时为优质种质创新提供支撑,提升景观观赏效果。
目前关于紫花风铃木的研究主要集中在栽种、观赏性、抗逆性等研究[8–14],关于分子水平遗传多样性在风铃木类中开展较多,单以紫花风铃木作为研究对象开展研究较少。对风铃木类植物的DNA分子水平遗传多样性的探究,已采用多种分子标记技术对风铃木类植物进行分类与鉴别[6–7]和遗传多样性差异分析,如单核苷酸多态性(single nucleotide polymorphism, SNP)位点结合测序基因分型技术(genotyping-by-sequencing, GBS)、叶绿体L-F、C序列分子遗传分析、简单重复序列(simplesequence repeats, SSR)分子标记[15]和区间简单重复序列(inter simple sequence repeats, ISSR)分子标记[16]等。
在分子标记技术中,SSR分子标记技术因具有高灵活性、成本低、易推广、多态性丰富、重复性好的优势而得到广泛应用[18–19]。SSR又叫微卫星DNA,SSR的产生是在DNA复制或修复过程中DNA滑动和错配或者有丝分裂、减数分裂姐妹染色单体不均等交换的结果,具有较高的物种特异性[20–21]。SSR分子标记技术目前已广泛应用于植物的种群遗传学、遗传图谱构建、指纹分析等研究领域[19–21]。当前国内还没有针对紫花风铃木进行基于SSR标记的群体遗传多样性研究, 因此本文利用SSR分子标记技术对广东省内6市12个群体的紫花风铃木进行遗传多样性分析研究, 探究其遗传多样性丰富度、遗传结构以及类群的划分,为紫花风铃木优良种质资源的保存和优质繁育提供理论依据。
1 材料和方法
1.1 材料
紫花风铃木()材料采自广东省6市12个群体,共72份,来源于同一个采样地的样本为1个群体(表1)。于2020—2021年采集样本,采集新鲜叶片后,放入-80 ℃冰箱保存。
1.2 DNA提取与引物扩增
风铃木叶片中含有较多多糖和多酚物质[22],为减少DNA提取时多糖和多酚的干扰,参考周仙莉等[23]的改良CTAB法提取DNA, 然后用1.5%琼脂糖凝胶电泳和核酸检测仪检测DNA质量和浓度, 后保存于4 ℃冰箱中待用。
选取同属植物的30对引物(mTb002~ mTb052)[24]和紫葳科印叶藤属植物的28对引物(Stiz2~Stiz45)[25],共计58对引物进行聚合酶链反应(polymerase chain reaction, PCR)扩增。所用引物由生工生物工程(上海)股份有限公司提供。随机选取8个样本进行引物的特异性和多态性筛选,最终筛选出9对杂带较少、主带清晰、多态性较好、条带差异明显的SSR引物进行后续实验(表2)。
采用荧光标记法进行基因分型,在每对引物的5′端标记荧光染料。PCR反应体系为30L,包含2×SanTaq PCR Master Mix 15L,正、反向引物(10mol/L)各1L,模板DNA (50 ng/L) 1L, ddH2O 12L。PCR扩增程序为95 ℃预变性5 min,然后95 ℃变性30 s,57 ℃复性90 s,72 ℃延伸30 s, 共28个循环,最后72 ℃延伸30 min,扩增产物于4℃保存备用。

表1 紫花风铃木群体信息

表2 紫花风铃木的SSR引物
1.3 数据整理与分析
利用Genemaker v2.2.0软件对基因分型结果进行判读,按引物预期扩增长度范围读出符合条件的数值并进行整理。利用GenAlEx 6.5进行遗传多样性统计、分子变异方差检验分析(analysis of molecular variance, AMOVA)和主坐标分析(principal coordinates analysis, PCoA)。利用Structure 2.3.4软件对群体进行遗传结构分析,并用Structure Harvester计算△K和LnP(D),从而计算出最佳群体数目(K值)。利用PowerMaker 3.25计算多态性信息量(polymorphism information content, PIC)和计算Nei’s遗传距离和遗传一致度,并使用MEGA X 10.1进行邻接聚类分析(neighboring-joining clustering analysis, NJ聚类)。
2 结果和分析
2.1 群体遗传多样性分析
2.1.1 SSR位点多态性
从表3可见,9对引物在72份材料中检测出123个等位基因,观测等位基因数(N)和有效等位基因数(N)分别为6.000~22.000和2.317~12.314,均值分别为13.667和6.072。Shannon指数()为1.133~2.519,平均为1.964,遗传多样性较为丰富。平均观测杂合度(H)和期望杂合度(H)分别为0.310和0.775。基因流(N)为0.519~1.611,平均为0.941。
2.1.2 群体遗传多样性分析
从表4可见,群体间的平均N为3.272个,其中jm群体的最大,hd群体最小;为0.448~ 1.630,平均为1.159,最大、最小的群体分别为jm和hd,与N的一致;H为0.292~0.734,H为0.222~ 0.389。

表3 9对SSR引物的遗传多样性参数

表4 紫花风铃木的群体遗传多样性
sf、fs、zc、ns、th、ld、hn、jl、xm、jm、hd、zs见表1;N、N、、H、H见表3; 下同
sf, fs, zc, ns, th, ld, hn, jl, xm, jm, hd, zs see Table 1. N,N,,H,Hsee Table 3. The same below
2.1.3 群体遗传分化和AMOVA分析
AMOVA分析表明(表5),紫花风铃木群体间的遗传变异仅占8%,而大部分遗传变异发生在群体内(92%),说明紫花风铃木群体间结构弱,群体间遗传分化程度相对较低,群体内遗传分化程度相对较高。从表6可见,紫花风铃木群体间的分化系数(st)为0.077,居于0.050~0.150,表明所有样本的遗传分化有7.7%来自群体间,有92.3%来自群体内部,其结果与反映的结果一致。来自群体内近交系数(is)是0.574,群体间近交系数(it)为0.607。

表5 紫花风铃木群体的AMOVA分析
2.2 遗传结构分析
2.2.1 群体结构分析
用Structure软件中基于贝叶斯聚类方法对紫花风铃木群体的遗传结构进行初步分析(图1),当K=2时最佳。此时可将12个紫花风铃木群体分为2个不同类群(图2)。其中类群1包括sf、ld、zc、zs共4个群体,彼此之间遗传背景较为相似,尤其是ld和zs结合前文简要收集的形态学信息也观察到这2个群体表型性状较接近;类群2包括ns、th、hn、jl、xm、jm、hd和fs共8个群体。
2.2.2 群体主坐标分析(PCoA)
主坐标分析大致将紫花风铃木群体分为2个类群,类群A有sf、ld、zs、zc群体,类群B有ns、th、hn、hd、fs群体,而jm、jl、xm群体的个体分散在A、B类群中,但大多数个体分布在类群B中(图3)。主坐标分析中个体位置越近说明亲缘关系较近,紫花风铃木不同群体的个体间存在相互交叉, 说明来自不同群体的个体间可能存在较近的亲缘关系。主坐标分析结果与Structure结构分析的结果基本一致,在Structure结构分析中jm、jl、xm则与ns、th、hn、hd、fs群体归属一类。但结合形态学特征来看,类群与形态特征之间并没有明显的关系,不同形态特征的群体被分为同一类。

图1 基于△K=mean(|L*(K)|)/sd(L(K))准则选择K值

图2 紫花风铃木群体的遗传结构图
2.2.3 群体聚类分析
应用NJ聚类法对12个紫花风铃木群体基于Nei’s遗传距离进行群体聚类分析,可划分为2个类群(图4),第一类包括sf、ld、zs、zc、xm、jl、jm群体,来自佛山、广州、中山、深圳和江门;第二类包括ns、th、hn、hd、fs,来自广州、惠州和佛山。这与Structure结构分析和主坐标分析的结果相近,均可被分为2个群体。xm、jl、jm这3个群体在不同的分析方法中归在不同的类群里,说明紫花风铃木遗传距离与地理距离之间可能无显著相关性。
3 结论和讨论
生物的遗传多样性是物种多样性和生态系统多样性的基础,也是生物多样性的基础。一般情况下,狭义遗传多样性侧重于描述物种之间的遗传变异。丰富的遗传多样性是物种适应环境变化的物质基础, 物种为了适应环境变化而进化, 遗传变异与进化相伴相生。变异的丰富度越大意味着物种的遗传多样性越高, 表明物种适应能力越强。评价物种遗传多样性水平,可以从形态学或DNA分子等角度切入研究[26–27],而随着分子标记技术的发展, 其在物种鉴定、种群遗传多样性研究及物种起源等方面得到了广泛的应用[28]。相比于使用形态学评价遗传多样性,DNA分子水平分析受环境影响较小,稳定性高,结果更为可靠[29]。而群体遗传多样性主要分析和把握群体内和群体间的遗传多样性分布情况,可以用来衡量种群的数量变化、遗传变异、生态习性等情况[26–27,30–31]。

图3 紫花风铃木群体的主坐标分析。黄色和蓝色圆圈分别为类群A和类群B。
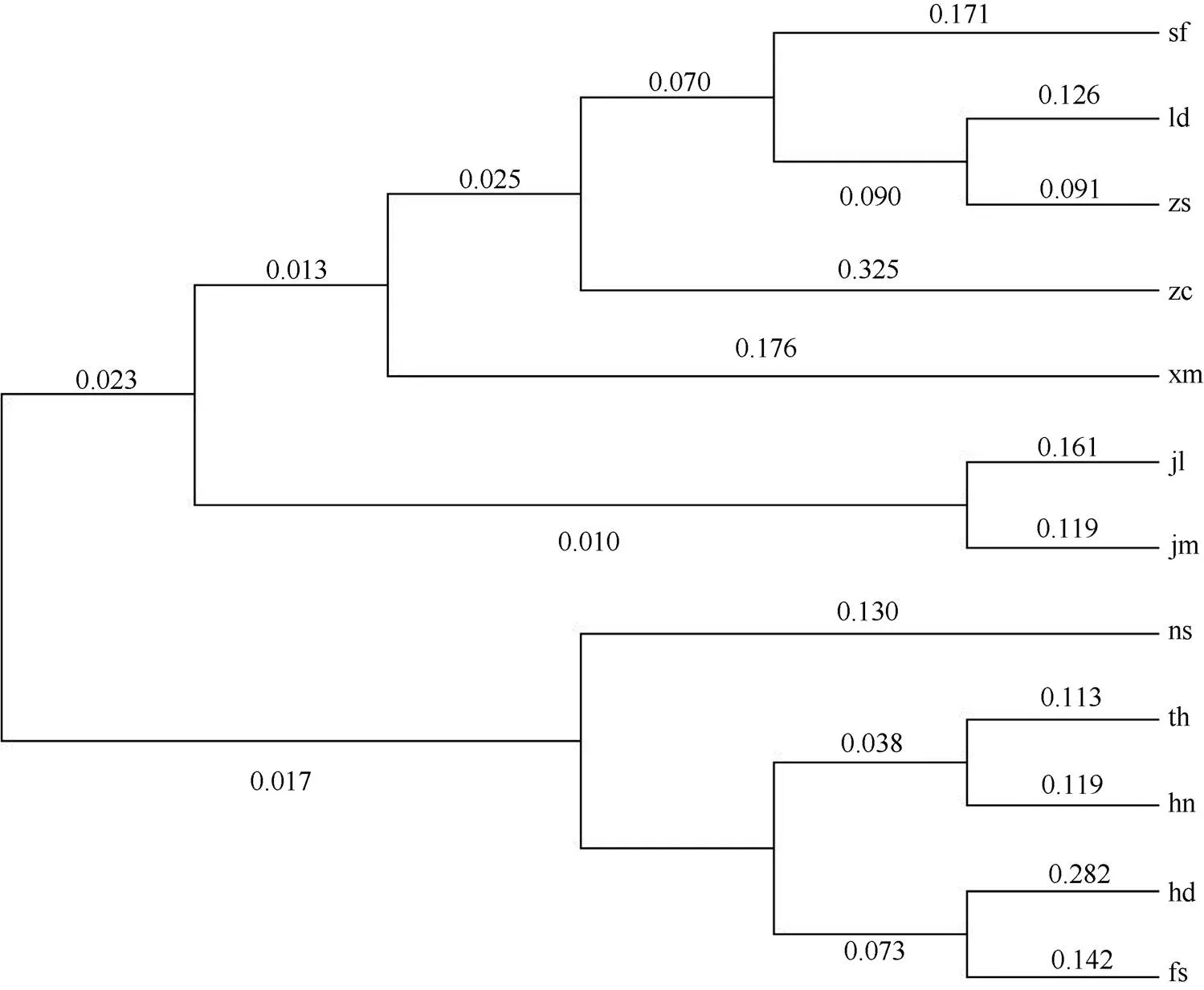
图4 基于Nei’s遗传距离的紫花风铃木群体的NJ聚类分析
对紫花风铃木遗传多样性开展DNA分子水平研究,能为科学利用和培育优良紫花风铃木品种提供更多的参考。目前已经有多种分子标记技术在风铃木类植物中应用于遗传多样性研究,叶绿体L-F和C序列标记和SNP分子标记用于对风铃木类树种进行分子水平的鉴种和分类,纠正采集样本中风铃木类树种同名异物或异物同名的问题,同时也表明紫花风铃木遗传多样性较为丰富,种下变异较多[6–7]。Collevatti等[15]对分布在巴西Altamiro de Moura Pacheco生态公园的4种风铃木植物进行SSR分子标记研究,采集了75个紫花风铃木的样本,平均H和H分别为0.857和0.703,结合种群结构分析表明紫花风铃木具有较高的遗传多样性, 但多态性水平较低。Alves等[16]采用8对ISSR引物对巴西Açu国家森林公园内30个紫花风铃木样本进行研究,共检测出62个等位基因位点,为0.34~ 0.49,平均为0.43,表明ISSR标记的多态性为中等,能有效反映紫花风铃木的遗传多样性。同时,当Nei’s遗传距离为0.70时可将30份样本划分成12个群体,但Shannon指数仅为0.52,说明多样性指数较低。
一般认为>0.5时该标记是高度多态的;为0.25~0.5时具有中度多态;<0.25时为低多态性的[32]。本研究中9对引物的为0.529~0.913,说明具有高度多态性,比Collevatti等[15]基于SSR分子标记和Alves等[16]基于ISSR分子标记的紫花风铃木样本遗传多样性分析的多态性较高,说明筛选的9对引物可以用于对紫花风铃木群体的遗传多样性研究。
本文采用9对SSR引物对来自12个群体的72份紫花风铃木样本进行遗传多样性分析,Shannon多样性指数为1.964,H和H分别为0.775和0.310,表明紫花风铃木具有丰富的遗传多样性,采集自深圳和江门的样本群体遗传多样性相对较高,但均比Collevatti等[15]研究中的H和H偏小。H明显大于H,说明供试紫花风铃木样本受到人为选择或近交等因素的影响较大。李蓉蓉[6]的研究也表明,目前园林苗圃中存在紫花风铃木和红花风铃木的杂交个体,这值得进一步去探究园林应用中的紫花风铃木的杂交现象。
物种的群体遗传结构受地理位置、生境环境等多种因素的影响。Ayala[33]对群体的遗传分化程度进行了划分,st为0~0.05时群体间遗传分化很小, 可不考虑;st为0.05~0.15时群体间存在中等程度的遗传分化;st为0.15~0.25时群体间遗传分化较大;st大于0.25表明群体间有很大的遗传分化。本研究中12个紫花风铃木群体的st为0.077,表明群体的遗传分化处于中等程度。AMOVA分析表明,紫花风铃木遗传变异仅有8%来自群体间,而大部分遗传变异发生在群体内。而群体的基因流为0.941,小于1。本研究中的紫花风铃木群体处于中等水平遗传分化和低基因流,这主要与紫花风铃木的引种与栽培繁殖方式有关。目前我国园林栽培应用的紫花风铃木引种来源不一,情况较为复杂。本研究选取的紫花风铃木样本多栽种于道路或城市绿地中,每个群体的个体构成受到人为因素影响较大,并不是以自然更新为主形成的自然群体,群体遗传分化规律性较弱。人们更侧重选择观赏效果优良的个体栽种在一起,因此同一样地采集的紫花风铃木可能有多个引种来源。再加上园林应用中为了加快优良紫花风铃木繁殖速度,常常采用嫁接、扦插等无性繁殖[10]手段,更延缓了紫花风铃木遗传分化速度。此外,由于本研究在收集样本阶段没有记录样本对应植株是否为实生苗还是无性繁殖苗,因此若样本中有来自实生苗个体组成的群体,其在苗圃繁育阶段遗传分化也可能受到紫花风铃木传粉方式的影响。紫花风铃木授粉存在同株同花合子后自交不亲和的现象,但可以进行同株异花传粉自交[34],紫花风铃木簇生花相和花量大的特点也利于促进同株异花传粉自交。其次,紫花风铃木的花结构决定了其主要依靠具有长距离飞行能力且具有长喙的昆虫进行传粉[35],因此对异株传粉也有一定的限制。
依据Structure结构分析、主坐标分析以及聚类分析,均可将12个群体大致分为2大类群,相互补充,但与本研究中简要提及的各群体形态学上的特征分类并没有完全对应,因此日后对紫花风铃木开展遗传多样性分析可以将分子研究结合更多细致的形态学指标如雌雄蕊的细部结构特征,进行更加全面的遗传多样性分析。这3种方法中,来自深圳的xm、jl以及江门的jm这3个群体在不同方法中被归属于不同的类群,在主坐标分析中还呈现散布的特征,总体而言分布规律并不显著。推测这3个群体的个体存在种间杂交的可能,值得进一步探究。这也表明紫花风铃木群体结构与地理分布并没有完全统一,很可能还是因为不同地区的引种来源情况复杂的问题。本研究的不足之处在于选取的样本数量相对较少,且集中在广东。今后对紫花风铃木开展遗传多样性研究和选育工作时,要进一步扩充取样范围和取样数量,采集不同地区栽培种或有条件获取原产地的样本,为紫花风铃木的优质种质资源收集、科学育种等提供更多有利的遗传学数据。
[1] JUSTINIANO M J, FREDERICKSEN T S, NASH D. Ecología y Silvicultura de Especies Menos Conocidas: Tajibos o Lapachos (spp. Gomes ex A.P. de Candolle) Bignoniaceae [M]. Santa Cruz: Editora El País, 2000: 8.
[2] LARRABURU E E, LLORENTE B E.enhances invitro rhizogenesis of(pink lapacho) in different culture media [J]. Ann For Sci, 2015, 72(2): 219– 229. doi: 10.1007/s13595-014-0418-9.
[3] SILVA-JUNIOR O B, GRATTAPAGLIA D, NOVAEs E, et al. Genome assembly of the Pink Ipê (, Bignoniaceae), a highly valued, ecologically keystone Neotropical timber forest tree [J]. Gigascience, 2018, 7(1): 1–16. doi: 10.1093/gigascience/gix125.
[4] ZHANG J, LI R R, MENG J X, et al. Phenotypic variation and genetic diversity of leaves traits ofand(Bignoniaceace) in China [J]. Bull Bot Res, 2021, 41(6): 851–861. [张捷, 李蓉蓉, 孟景祥, 等. 我国风铃木类植物叶性状表型变异与遗传多样性研究 [J]. 植物研究, 2021, 41(6): 851–861. doi: 10.7525/j.issn.1673-5102. 2021.06.002.]
[5] CAI D C, FANG L, SHANG X H, et al. Research status and deve- lopment prospects of[J]. Eucalypt Sci Technol, 2016, 33(3): 46–50. [蔡德才, 方良, 尚秀华, 等. 风铃木研究现状及开发利用前景 [J]. 桉树科技, 2016, 33(3): 46–50. doi: 10.3969/j.issn.1674-3172. 2016.03.009.]
[6] LI R R. Identification and genetic diversity ofandspecies in coastal areas of south China [D]. Harbin: North- east Forestry University, 2021. [李蓉蓉. 华南沿海风铃木类植物鉴定与遗传多样性研究 [D]. 哈尔滨: 东北林业大学, 2021. doi: 10. 27009/d.cnki.gdblu.2021.000586.]
[7] JIN H X, DAI J H, HUANG G L, et al. Germplasm resources ofandin south China: Morphology and mole- cular identification [J]. Chin Agric Sci Bull, 2019, 35(7): 36–41. [金海湘, 戴金宏, 黄桂莲, 等. 华南风铃木类植物种质资源的形态与分子鉴定 [J]. 中国农学通报, 2019, 35(7): 36–41. doi: 10.11924/j.issn. 1000-6850.casb18100035.]
[8] SHANG X H, GAO L Q, ZHANG P J, et al. Three kinds ofcuttings [J]. Eucalypt Sci Technol, 2016, 33(1): 38–42. [尚秀华, 高丽琼, 张沛健, 等. 3种风铃木扦插繁殖技术研究 [J]. 桉树科技, 2016, 33(1): 38–42. doi: 10.13987/j.cnki.askj.2016.01.008.]
[9] ZHANG J. Study on flowering traits and ornamental value ofandspecies [D]. Beijing: Chinese Academy of Forestry, 2016. [张静. 风铃木类植物开花性状与观赏价值研究 [D]. 北京: 中国林业科学研究院, 2016.]
[10] WU L Q. Introduction, cultivation and application of[J]. Guangdong Agric Sci, 2010, 37(7): 77–79. [武丽琼. 红花风铃木的引种栽培及应用 [J]. 广东农业科学, 2010, 37(7): 77– 79. doi: 10.16768/j.issn.1004-874x.2010.07.081.]
[11] MENDONÇA A M C, LIRA J M S, VILELA A L D, et al. High aluminum concentration and initial establishment of: Clues about an Al non-resistant species in Brazilian Cerrado [J]. J For Res, 2020, 31(6): 2075–2082. doi: 10.1007/s11676- 019-01033-5.
[12] MARTINS J R, EDVALDO A A S, ALVARENGA A A, et al. Seedling survival of(Mart ex DC) Mattos in a semi-arid environment through modified germination speed and post- germination desiccation tolerance [J]. Braz J Biol, 2015, 75(4): 812– 820. doi: 10.1590/1519-6984.23413.
[13] GAI A P C, DOS SANTOS D S, VIEIRA E A. Effects of zinc excess on antioxidant metabolism, mineral content and initial growth of(Mart. ex DC.) Mattos and(Ridl.) Sandwith [J]. Environ Exp Bot, 2017, 144: 88–99. doi: 10.1016/j.envexpbot.2017.09.006.
[14] PESSOA J L, FREIRE A L O, COSTA A S. Trocas gasosas de plantas de(Mart. ex DC.) Mattos submetidas ao déficit hídrico e posterior reidratação [J]. Rev Ciên Agrovet, 2017, 16(3): 269–276. doi: 10.5965/223811711632017269.
[15] COLLEVATTI R G, ESTOLANO R, RIBEIRO M L, et al. High genetic diversity and contrasting fine-scale spatial genetic structure in four seasonally dry tropical forest tree species [J]. Plant Syst Evol, 2014, 300(7): 1671–1681. doi: 10.1007/s00606-014-0993-0.
[16] PIMENTA J M A, FELIX F C, DE ARAÚJO J S O, et al. Selection of ISSR molecular primers for studies of genetic diversity in(Mart. ex DC.) Mattos [J]. Rev Caatinga, 2022, 35(1): 231–238. doi: 10.1590/1983-21252022v35n124rc.
[17] JIANG C H, WANG Y Y, SUN Y H. Application advance of molecular marker techniques of SSR and ISSR [J]. Chin Tob Sci, 2007, 28(2): 1–5. [蒋彩虹, 王元英, 孙玉合. SSR和ISSR标记技术应用进展 [J]. 中国烟草科学, 2007, 28(2): 1–5. doi: 10.3969/j.issn.1007-5119.2007. 02.001.]
[18] LI X, TIAN H L, WANG F G, et al. Comparison of SSR and SNP markers in maize varieties genuineness identification [J]. Mol Plant Breed, 2014(5): 1000–1004. [李雪, 田红丽, 王凤格, 等. SSR和SNP两种标记技术在玉米品种真实性鉴定中的比较分析 [J]. 分子植物育种, 2014(5): 1000–1004. doi: 10.13271/j.mpb.012.001000.]
[19] LUO Q, YU Y P, LU Y F, et al. Analysis of genetic relationship of wildspecies based on SSR marker [J]. N Hort, 2017(19): 111–117. [罗清, 於艳萍, 卢业飞, 等. 基于SSR标记的杜鹃品种亲缘关系分析 [J]. 北方园艺, 2017(19): 111–117. doi: 10.11937/bfyy. 20170663.]
[20] LIN L, LIN L J, ZHU Z Y, et al. Construction of SSR fingerprint and genetic diversity analysis of 93 maple germplasm resources [J]. Mol Plant Breed, 2022, 20(4): 1250–1263. [林立, 林乐静, 祝志勇, 等. 93份槭树种质资源的SSR指纹图谱构建与遗传多样性分析 [J].分子植物育种, 2022, 20(4): 1250–1263. doi: 10.13271/j.mpb.020. 001250.]
[21] CHEN Y L, LUO Y S, TAO X H. The genetic relationship of two new colorful sweetcultivars revealed by AFLP and SSR markers [J]. Agric Sci Technol, 2019, 20(4): 40–48. doi: 10. 16175/j.cnki.1009-4229.2019.04.006.
[22] DE ABREU M B, TEMRAZ A, VASSALLO A, et al. Phenolic glycosides fromand[J]. Phytochem Lett, 2014, 7: 85–88. doi: 10.1016/j.phytol.2013.10.006.
[23] ZHOU X L, TIAN X, SHEN N D, et al. Study on extraction method of total DNA from[J]. Mol Plant Breed, 2019, 17(7): 2217– 2222. [周仙莉, 田霞, 沈宁东, 等. 藏茴香总DNA提取方法探究 [J]. 分子植物育种, 2019, 17(7): 2217–2222. doi: 10.13271/j.mpb.017. 002217.]
[24] MORILLO E, BUITRON J, LIMONGI R, et al. Characterization of microsatellites identified by next-generation sequencing in the Neo- tropical tree(Bignoniaceae) [J]. Appl Plant Sci, 2016, 4(5): 1500135. doi: 10.3732/apps.1500135.
[25] BEYER M, NAZARENO A G, LOHMANN L G. Using genomic data to develop chloroplast DNA SSRs for the Neotropical liana(Bignonieae, Bignoniaceae) [J]. Appl Plant Sci, 2017, 5(10): 1700061. doi: 10.3732/apps.1700061.
[26] LIU X H, LI Y G, ZHAO X, et al. Phenotypic diversity in natural populations of[J]. For Res, 2011, 24(6): 694–700. [柳新红, 李因刚, 赵勋, 等. 白花树天然群体表型多样性研究 [J]. 林业科学研究, 2011, 24(6): 694–700. doi: 10.13275/j.cnki.lykxyj. 2011.06.013.]
[27] LI H L, LI X, PANG H Y. Phenotypic trait diversity of wildfruit in Heilongjiang Province [J]. J W China For Sci, 2022, 51(2): 20–26. [李红莉, 李雪, 逄宏扬. 黑龙江野生毛榛果实表型性状的多样性研究 [J]. 西部林业科学, 2022, 51(2): 20–26. doi: 10.16473/j.cnki.xblykx1972.2022.02.004.]
[28] MAURIZIO R, PAUL D R. Applications of molecular markers in plant conservation [M]// HENRY R J. Molecular Markers in Plants. Hoboken, NJ: John Wiley & Sons, 2012: 81–98. doi: 10.1002/9781118473023. ch6.
[29] GROVER A, SHARMA P C. Development and use of molecular markers: Past and present [J]. Crit Rev Biotechnol, 2016, 36(2): 290– 302. doi: 10.3109/07388551.2014.959891.
[30] SONG Y, ZHANG X R, LI Z, et al. Genetic diversity and genetic structure analysis ofDC. based on SSR molecular marker [J]. Acta Pharm Sin, 2022, 57(4): 1193–1202. [宋芸, 张鑫瑞, 李政, 等. 基于SSR分子标记的柴胡遗传多样性与遗传结构分析 [J]. 药学学报, 2022, 57(4): 1193–1202. doi: 10.16438/j.0513-4870. 2021-1479.]
[31] YANG J L, ZHAO Y J, HUANG G F, et al. Genetic diversity analysis of ancient tea plants in Xishuangbanna area [J]. Bot Res, 2018, 7(4): 442–452. [杨金玲, 赵艳娟, 黄光福, 等. 西双版纳地区古茶树遗传多样性分析 [J]. 植物学研究, 2018, 7(4): 442–452. doi: 10.12677/br. 2018.74054.]
[32] SHARMA P, MEHTA G, SHEFALI, et al. Development and validation of heat-responsive candidate gene and miRNA gene based SSR markers to analysis genetic diversity in wheat for heat tolerance breeding [J]. Mol Biol Rep, 2021, 48(1): 381–393. doi: 10.1007/ s11033-020-06059-1.
[33] AYALA F J. Evolutionary genetics: Wright’s treatise continued [J]. Science, 1977, 196(4295): 1191–1192. doi: 10.1126/science.196.4295. 1191.
[34] BITTENCOURT JÚNIOR N S. Evidence for post-zygotic self- incompatibility in(Bignoniaceae) [J]. Plant Reprod, 2017, 30(2): 69–79. doi: 10.1007/s00497-017-0300-7.
[35] SCHLINDWEIN C, WESTERKAMP C, CARVALHO A T, et al. Visual signalling of nectar-offering flowers and specific morphological traits favour robust bee pollinators in the mass-flowering tree(Bignoniaceae) [J]. Bot J Linn Soc, 2014, 176(3): 396–407. doi: 10.1111/boj.12212.
Genetic Diversity Analysis ofPopulations by SSR Markers
HUANG Zhiqing1,2, WU Linyuan1,3, GAO Xiaoyu1, DING Shifeng1, FENG Zhijian1*, QIN Xinsheng1, LIU Yitong2, XIAO Guankang1
(1. College of Forestery and Landscape Architecture, South China Agricultural University,Guangzhou 510642, China; 2. College of Ecological Engineering, Guangdong Eco-Engineering Polythenic, Guangzhou 510520, China; 3. School of Light Industry and Materials, Guangdong Vocational and Technical College, Foshan 528041, Guangdong, China)
,introduced from South America and Central America, had widely used as ornamental tree in southern China, which had rich germplasm resources. However, the resources of introduction is complex while the work of improved variety breeding is lagging. In order to reveal genetic variation ofpopulations, the genetic diversity and population genetic structure of 72 samples from 12 populations collected from 6 cities in Guangdong Province were analyzed. The results showed that a total of 123 alleles were amplified by 9 pairs of SSR primers with average PIC of 0.754, indicating that thegermplasm had high genetic diversity. All 12 populations ofalso had high genetic diversity, the average effective alleles between populations was 3.272, and the average Shannon information index was 1.159. AMOVA analysis showed that genetic differentiation among populations was relatively low, while that within populations was high. The overall genetic differentiation coefficient of populations was 0.077, which was in the medium degree. Based on the Structure analysis, PCoA analysis and NJ clustering analysis, 12 populations ofcould be divided into 2 groups, and the grouping results had certain similarity, indicating that the genetic structure of tested populations was not complex. These would provide a theoretical basis for the utilization of germplasm resources, genetic variation and scientific breeding for
; Genetic diversity; SSR marker; Genetic structure
10.11926/jtsb.4697
2022-07-07
2022-11-11
国家自然科学基金项目(31870699);广东省林业科技创新项目(2018KJCX030)资助
This work was supported by the National Natural Science Foundation of China (Grant No. 31870699), and the Project for Forestry Science and Technology Innovation in Guangdong (Grant No. 2018KJCX030).
黄稚清(1995年生),女,助理实验员,从事野生植物资源利用与园林植物应用研究。E-mail: starlight0715@foxmail.com
* 通讯作者 Corresponding author. E-mail: fengzj@scau.edu.cn

