基于切片关联信息的慢性阻塞性肺疾病CT诊断
梁宇辰,蔡 念,欧阳文生,谢依颖,王 平
(1.广东工业大学 信息工程学院, 广东 广州 510006;2.广州医科大学附属第一医院 肝胆外科, 广东 广州 510120)
慢性阻塞性肺疾病(Chronic Obstructive Pulmonary Disease,COPD) 是一种常见的全球性呼吸疾病,已成为全球的第三大死因[1]。近些年,我国的COPD患病率也呈现不断上升趋势,且死亡率高于全球平均水平[2-3]。COPD患者通常会出现气道严重阻塞现象,进一步导致呼吸困难,甚至存在肺心病、呼吸衰竭等风险[4]。目前,主要采用CT图像评估[5-6]鉴别COPD肺部异常,这需要医生根据COPD患者的上百张CT序列图片中的病灶气泡占比情况进行主观分析,耗费医生大量精力和时间,也给医生带来视觉疲劳并影响主观评估[7]。
近几年,随着深度学习尤其是卷积神经网络(Convolutional Neural Network, CNN)的高速发展,CNN已经被广泛应用于CT图像处理,以辅助肺部疾病诊断。Shah等[8]使用VGG-19对新型冠状病毒肺炎(Corona Virus Disease 2019 ,COVID-19) 进行诊断。Polat等[9]在Inceptionv3模型上使用迁移学习,从患者的单张胸部CT图像获取病灶信息识别COPD的严重程度。虽然上述方法在肺部疾病辅助诊断上取得不错的效果,但是这些方法只考虑了部分CT切片图像内的局部病变肺泡特征,没有考虑CT切片图像之间的关联信息,这将不利于肺部病灶区病变肺泡识别[10]。
Xu等[11]从胸部CT序列中随机选出8张CT图像,采用AlexNet网络对每张CT图像进行特征提取,然后采用SVM进行分类实现COPD诊断。可是,该方法只对单张CT图像进行单独分析,虽然SVM对8张CT图像进行了分类,但是本质上仍未考虑这8张CT切片之间的关联信息。Ahmed 等[12]将VoxResNet拓展到3D卷积结构,通过多个残差3D卷积结构保留尽可能多的空间信息对CT序列图像进行处理来诊断COPD。Varchagall等[13]使用3D ResNet提取CT图像病灶空间特征并使用迁移学习进行肺癌诊断。Kienzle等[14]将ConvNeXt网络拓展到3维结构构成3D ConvNeXt网络对COVID-19进行诊断。这些网络都是采用3D卷积方法对CT序列图像进行处理,但是3D卷积难以提取病灶区细微肺泡特征,影响网络的分类效果。Wu等[15]从CT序列图像中提取气管树图像和9张3D肺部图像等其他模态信息作为ResNet26的网络输入,从而实现COPD辅助诊断。可是,该方法受制于气管树3D形态提取的精准性,缺乏COPD辅助诊断的便捷性。Kollias等[16]使用CNN和RNN分别获取局部病变肺泡的图像特征和全局切片间的关联信息,构建了一种MIA-COVID-19网络对COVID-19进行诊断。Humphries等[17]结合CNN与长短时记忆(Long-Short Term Memory, LSTM)对COPD进行诊断。上述两个网络可以同时学习病灶图像局部特征和切片之间全局关联信息。但是,它们直接对上百张CT切片图像进行特征关联信息提取,忽视了不同距离的CT切片图像之间的关联性是不同的。
本文提出一种基于CT切片图像关联信息的深度网络,辅助诊断COPD。将COPD患者胸部CT切片序列分为若干组作为整个网络的网络分支的输入,同时提取组内切片之间的局部关联信息和各切片内的病灶图像局部特征。为了提高各网络分支的病灶图像局部特征提取能力,融入ConvNeXt提出一种增强的多头卷积注意力模块。
1 方法
1.1 网络结构
基于切片关联信息的COPD诊断网络输入COPD患者的胸部CT图像序列,输出诊断结果。网络由多个局部切片关联信息提取分支和一个全局的切片关联信息提取阶段构成。图1展示了3个分支组成的网络结构示意图。局部切片关联信息提取分支由Conv STEM模块、En-MHCA模块、Down Sample模块和Ef-Transformer模块构成,主要提取组内CT切片之间的局部关联信息和组内CT切片病灶区的局部图像特征。将每个分支的特征信息以N切片数的维度进行二次拼接组成局部特征序列输入到全局切片关联信息提取阶段,该阶段主要由BiLSTM[18]构成,提取各分支之间的切片之间的全局关联信息。最后,级联一个多层感知器(Multi-Layer Perceptron,MLP)进行COPD诊断。

图1 本文提出的深度网络架构Fig.1 The framework of the proposed deep network
1.2 局部切片关联信息提取分支
肺部病变肺泡是COPD患者的典型病理特征,是COPD诊断重要依据。根据合作医生提供的先验知识[19],COPD病变肺泡通常在3至4张CT切片中就能观察到完整的病变肺泡结构;在一些切片数量较多的情况中,病变肺泡可能分布在10张左右CT切片中。因此,本文提出对COPD患者胸部CT序列图像进行合理分组,各分组可以提取组内CT切片之间的局部关联信息和组内CT切片病灶区的局部图像特征。
将COPD患者CT序列数据划分为若干组,每组内有N张切片,每张CT切片图像维度为(C,W,H) ,其中C代表特征通道数,W代表图像宽度,H代表图像高度。本文中,N经验选择为10,后续消融实验将验证此选择。因此,分组后的序列图像组数由患者CT序列的切片总数决定,如患者CT序列为400张CT切片,则分组数为40组。
将各组CT切片作为各局部切片关联信息提取分支的输入,其结构如图2所示。其中,Conv STEM模块用于低维特征提取,2个En-MHCA模块和2个Down Sample下采样模块用于提取高维局部病灶特征。提取到的高维特征按N切片数的维度拼接为特征序列输入到Ef-Transformer模块。图2中仅展示了3张图像的特征拼接情况。在Ef-Transformer模块中,特征序列通过自注意力[20]获取组内切片之间的局部关联信息和局部病灶图像特征。

图2 局部切片关联信息提取分支的结构Fig.2 The structure of the branch for local slice correlated information extraction
1.3 Conv STEM模块
Conv STEM模块的结构如图3所示,包含一组由Depth Wise卷积层和Point Wise卷积层构成的深度可分离卷积层、Batch Normal归一化层、Max Pool层和ReLU层,数学表示为

图3 Conv STEM的结构Fig.3 The structure of the Conv STEM
式中:x1为输入特征,M代表Max Pool层,R代表ReLU激活函数层,BN代表Batch Normal归一化层,PWC 代 表Point Wise卷积层, D WC代表Depth Wise卷积层。
1.4 Down Sample模块
Down Sample模块的结构如图4所示,主要由1个Convolution层和1个Max Pool层构成,其数学表示为
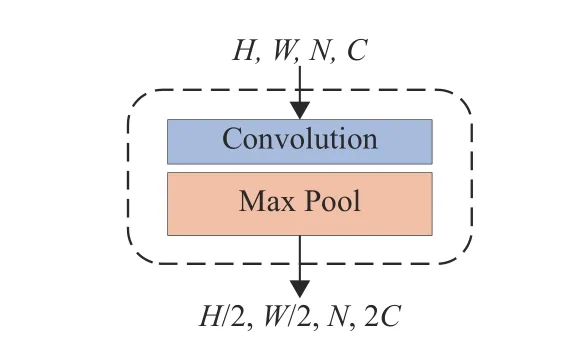
图4 下采样模块Fig.4 Down sampling module
式中:x2为该模块的输入,Conv() 代表卷积层操作。在图4中,H、W和C分别为特征映射图的高、宽和通道数,N为切片数。
1.5 En-MHCA模块
受ConvNeXt[21]的启发,提出了一个增强的多头卷积注意力模块(Enhanced Multi Head Convolutional Attention, En-MHCA) 。与传统的多头卷积注意力模块(Multi Head Convolutional Attention, MHCA)[22]不同的是,En-MHCA融合ConvNeXt进行特征提取和非线性拟合,去除了只有非线性拟合能力的MLP结构,因此En-MHCA本质上是一个倒置的多头卷积注意力结构。传统MHCA仅使用MHCA层进行特征学习,而且MLP结构仅有非线性拟合能力。相比之下, En-MHCA包含了2个拥有特征学习能力的残差结构(即ConvNeXt和MHCA),具有更强的特征提取能力。
如图5所示,ConvNeXt由1个Depth Wise卷积层、2个Point Wise卷积层以及残差结构构成。Depth Wise卷积层是一个大小为3、步长为1且分组数和通道数一致的卷积层。Point Wise卷积层是一个大小为1、步长为1的通道转换卷积层。经过1个Depth Wise卷积层、2个Point Wise卷积层后输出的特征映射图和ConvNeXt输入特征映射图进行一次残差结构相加。

图5 增强的多头卷积注意力模块Fig.5 Enhanced multi-head convolutional attention module
ConvNeXt输出的特征映射图作为MHCA输入。MHCA主要由1个3 ×3分组卷积层、1个1×1卷积层、1个Batch Normal层和1个ReLU层构成,其数学表示为
式中:x3为 输入特征映射图,R1为ConvNeXt输出特征映射图,R2为MHCA输出特征映射图。
1.6 Ef-Transformer模块
为了更好地获取组内切片间的局部关联信息,在分支中采用Ef-Transformer 模块[23]提取特征。如图6所示,特征映射图经过1个Layer Norm层,再输入到E-MHSA(Efficient Multi-head Self-attention)。EMHSA输出的特征映射图和Ef-Transformer输入的初始特征映射图通过一个残差连接进行相加得到组内切片间的关联信息S1。 随后,S1经过1个Layer Norm层和1个MLP后,再与S1执行一次残差连接相加。Ef-Transformer模块的计算过程可以数学表示为

图6 Ef-Transformer模块Fig.6 The Ef-Transformer module
式中:x4为输入特征映射图,EMHSA代表多头自注意力模块操作,LN代表LayerNorm正则化层操作,S1为 一次残差连接的输出特征,S2为第二次残差连接的输出特征。
1.7 BiLSTM模块
因为不同COPD患者的CT切片数是不一致的,所以各患者CT切片序列的分组数也有所不同。故采用BiLSTM多单元模块来提取各组之间的切片全局关联信息。BiLSTM结构如图7所示,可以数学表示为


图7 BiLSTM模块Fig.7 The BiLSTM module
1.8 损失函数
采用交叉熵损失函数优化提出的深度网络参数,数学表达为
式中:yi为 COPD真实诊断标签,pi为模型根据数据诊断的结果,M表示训练集的样本数,i表示当前样本序号。
2 实验
2.1 数据集及训练环境配置
本文实验数据均由广州医科大学第一附属医院提供,共收集了161例COPD患者CT序列,每例数据包含130到400之间的CT切片图像。因此,数据集总共包含43 140张CT切片图像。每张切片图像的分辨率是512×512,像素平均间距为0.68 mm,切片平均厚度为1.143 mm。由于患者隐私等协议条款,本文不能公开这些影像数据。
训练集由110例共30 190张CT切片构成,测试集由51例共12 950张CT切片构成,数据标注均由经验丰富的医生完成。
本文实验皆在一台配置为NVIDIA RTX A6000 48 GB GPU和Inter Xeon(R) Gold 5218R 2.10GHz CPU的工作站上完成。模型训练和测试代码都是基于PyTorch 1.6(python3.8)深度学习框架编程实现,其训练初始化参数如表1所示。

表1 模型训练初始化参数Table 1 The initialization parameters of the model in training
评估标准有准确率(Accuracy, ACC)、灵敏度(Sensitivity, SEN)、特异性(Specificity, SPE)和受试者工作特征(Receiver Operating Characteristic, ROC)曲线下的面积值(Area Under Curve, AUC) 。其中,AUC的值由ROC曲线与坐标围成的区域面积所得。ACC、SEN、SPE的计算公式为
式中:TP表示真阳性,真实标签为COPD患者,模型预测为COPD患者;FP表示假阳性,真实标签为COPD患者,模型预测为非COPD患者;TN表示真阴性,真实标签为非COPD患者,模型预测为非COPD患者;FN表示假阴性,真实标签为非COPD患者,模型预测为COPD患者。
2.2 消融实验
(1) 不同模块对网络性能的影响。本文通过相关模块的消融实验说明网络中相关模块对网络诊断性能的影响,Ef-Transformer缩写为Ef-Trans,数据如表2所示。

表2 不同特征提取模块的对比Table 2 Comparisons of different feature extraction modules
从表2可以看到,当只使用Ef-Trans时,网络只获取了组内切片间的局部关联信息,而忽视了组间切片的全局关联信息,从而导致网络诊断性能最差。而当只采用BiLSTM时,网络的诊断准确率等性能要比只使用Ef-Trans有大幅度的提升,这说明组间的长程关联性对COPD诊断具有重要指导价值。当同时使用BiLSTM和Ef-Trans时,网络性能相比于只使用BiLSTM时的网络略微有所提升,这说明组间的切片全局关联信息和组内的切片局部关联信息的有机融合能提升COPD患者的CT序列分类效果。从最后一行数据来看,提出的增强多头卷积注意力模块与普通的多头卷积主力模块相比,网络的诊断准确率等性能有所提升。
(2) 不同分组切片参数对网络性能的影响。
为了探索不同的分组切片数对网络性能的影响,本文做了对比实验。如表3所示,当分组切片数为10时,网络的AUC指标表现更好。这是因为过少的分组切片数会造成网络无法获取完整的结构关联信息,而过多的分组切片数提供了冗余的切片关联信息。

表3 不同分组切片参数的对比实验Table 3 Comparative experiments with different grouping of slicing parameters
2.3 对比实验
为了验证提出模型的效果,将其与现有的一些基于CT图像的肺部疾病深度学习方法进行对比,这些方法分别是VGG-19[8]、3D VoxResNet[12]、DCT-MIL[11]、MIA-COV19D[16]、3D ResNet[13]、CNN+LSTM[17]和3D ConvNeXt[14],结果如表4所示。

表4 不同深度学习方法的对比Table 4 Comparisons among different deep learning methods
从表4可以看出,VGG-19网络在提取CT图像特征过程中,网络深度的增加导致病变肺泡特征信息丢失,且VGG-19网络也未考虑切片之间的关联信息,因此获得非常差的COPD诊断性能。
3D ResNet和3D VoxResNet都通过残差结构缓解因网络深度不断加深而丢失特征的现象,因此获得比VGG-19网络更好的COPD诊断性能。3D ConvNeXt网络使用的3D ConvNeXt模块具有很强的空间提取能力,因此获得了高达92.91%的AUC值。虽然这3种3D网络都较好地实现了COPD辅助诊断,但是它们仅使用CT图像内的局部空间特征,忽视了切片间的关联信息,因此难以获取病变肺泡的整体形貌信息,不同程度地影响网络诊断性能。MIACOV19D和CNN+LSTM采用CNN提取切片内的局部细节信息,分别采用RNN和LSTM提取CT序列的全局关联信息,因此分别获得了87.37%和83.22%的AUC值。但是,这两种方法在提取CT序列的全局关联信息时未考虑不同距离切片之间的关联程度。DCTMIL采用AlexNet迁移特征,并采用多实例实习策略分析随机选取的8张连续CT切片图像,并考虑了邻近切片之间的关联性,因此也获得了82.53%AUC值。但是,DCT-MIL忽视了整个CT序列的长程关联关系,导致其诊断性能仍然较低。本文所提出的深度网络结合了组间切片的全局关联信息和组内切片的局部关联信息,同时融合了切片图像病灶区的局部图像特征,因此获得了最佳的COPD辅助诊断效果,准确率达到92.15%,敏感度达到94.17%,特异性达到91.17%,AUC达到95.33%。图8为不同深度学习方法的ROC曲线图。如图8所示,本文网络的ROC曲线包裹面积最大。

图8 ROC曲线图Fig.8 The ROC curves
3 结论
本文提出一种基于切片关联信息的深度网络,应用于COPD辅助诊断。采用分组方式,融合ConvNeXt和MHCA提出增强的多头卷积注意力模块(En-MHCA),提取组内CT切片间的局部关联信息和CT切片病灶区的局部图像特征。采用BiLSTM提取组间CT切片的全局关联信息。实验结果表明,提出的深度网络比多个现有深度网络具有更好的诊断性能,准确率、敏感度、特异性和AUC分别达到92.15%、94.17%、91.17%和95.33%。

