CRISPR/Cas9系统在畜禽遗传改良中研究进展
鲍艳春,戴伶俐,刘在霞,马凤英,王宇,刘永斌,谷明娟,娜日苏,张文广,6
综 述
CRISPR/Cas9系统在畜禽遗传改良中研究进展
鲍艳春1,2,戴伶俐2,3,刘在霞1,2,马凤英1,2,王宇4,刘永斌5,谷明娟1,2,娜日苏1,2,张文广1,2,6
1. 内蒙古农业大学动物科学学院,呼和浩特 010018 2. 内蒙古自治区农业基因组大数据工程研究中心,呼和浩特 010018 3. 内蒙古自治区农牧业科学院兽医研究所,呼和浩特 010031 4. 内蒙古农业大学兽医学院,呼和浩特 010018 5. 内蒙古大学生命科学学院,呼和浩特 010021 6. 内蒙古农业大学生命科学学院,呼和浩特 010018
CRISPR/Cas9基因编辑技术作为一种高效的基因组编辑方法,在畜牧业遗传改良领域得到了广泛的应用。该技术以高效、精准的特点,为畜牧业发展带来了一场革命。目前,基于CRISPR/Cas9的基因敲除、基因敲入和基因修饰等已被广泛应用,实现了对畜禽物种的重要生产性状进行精准改良。本文介绍了CRISPR/Cas9技术的工作原理及发展历程,重点介绍了该技术在畜禽肌肉生长发育、绒毛纤维生长、乳品质成分、抗病育种以及动物福利中的研究进展,旨在为更深入地了解CRISPR/Cas9技术在畜禽基因编辑上的应用提供参考。
CRISPR/Cas9;基因编辑;畜禽;遗传改良;经济性状
基因编辑是一种极具发展前景的生物工程技术,通过人为方式在有机体的基因组特定位置上删除、添加或改变碱基,以达到编辑基因组的目的。理想的基因组编辑方法需要高效并准确地改变基因组序列,并尽量减少或避免产生脱靶效应[1]。随着锌指核酸内切酶(zinc-finger endonuclease,ZFN)[2]、类转录激活因子效应物核酸酶(transcription activator like effector nuclease,TALEN)[3]和成簇规律间隔短回文重复序列及其相关系统(clustered regularly inter spaced short palidromic repeats/CRISPR-associated protein 9,CRISPR/Cas9)[4,5]等基因编辑技术的出现,为应对人类对蛋白质日益增长的需求问题提供了潜在的解决途径。与ZFN和TALEN不同,CRISPR/Cas9系统具有成本低廉、操作简单、高效以及可对多个靶基因进行同时编辑等优势,目前已被广泛应用于分子育种、疾病防治、遗传改良和制药等领域[6~11]。
近年来CRISPR/Cas9技术在畜牧业方面的应用带来了诸多突破,包括生成转基因动物、改良基因组特征、提高动物抗病能力以及改善农产品的品质等。科研人员通过对特定基因进行编辑和调控,增强了动物的生产潜力和生态适应性。此外,该技术的可靠性也在不断取得进展,例如通过碱基修饰来实现单碱基突变的插入,而无需依赖复杂的DNA供体。这意味着CRISPR/Cas9技术的应用更加简便和高效。CRISPR/Cas9技术的快速发展极大地推动了畜禽养殖领域的革新和发展,为未来的畜牧业发展提供了更广阔的应用前景。本文对CRISPR/Cas9技术的工作原理及进展进行了概述,着重介绍了目前该技术在主要家养动物基因组中的应用进展,为CRISPR/Cas9技术在畜禽遗传改良的研究提供参考依据。
1 CRISPR/Cas9技术发展历程和原理
日本科学家在1987年对大肠杆菌()的基因进行研究时,首次在位于基因3′端侧翼区发现了5个高度同源的特殊结构,由29个核苷酸序列排列成的重复序列组成。由于当时在原核生物中还未发现与这些序列同源的序列,因此对这些序列的生物意义是未知的[12]。1991年,Pavletich等[13]通过解析Zif268蛋白与DNA结合的晶体结构,首次揭示了锌指蛋白与DNA识别的分子机制。随后,1996年Kim等[14]首次成功制造了嵌合型核酸内切酶——锌指核酸酶。2002年,CRISPR首次被正式命名,并发现这种新型的DNA序列家族在细菌和古细菌中广泛存在[15],具有抵御病毒或外源物质入侵的功能,其存在形式及作用机理均不相同。同年,利用ZFN技术成功地在果蝇()中实现了基因的诱变[16,17]。之后,Barrangou等[18]确认CRISPR/Cas系统是一种原核生物特有的天然适应性防御机制。2010年,Christian等[19]将TALE蛋白和酶嵌合创造出了一种新的基因组编辑技术——TALEN。直到2011年,CRISPR/Cas9的作用机制才被揭示。当病毒首次入侵机体时,细菌会将外源基因的一段序列整合到自身CRISPR序列的间隔区;在病毒再次入侵时,CRISPR序列会转录生成前体crRNA(pre-crRNA),pre-crRNA通过加工形成含有与外源基因匹配序列的crRNA,该crRNA与病毒基因组的同源序列识别后,介导Cas蛋白结合并且对其进行切割,从而保护机体免受病毒的入侵[20,21](图1)。2013年,在哺乳动物细胞中成功实现了II型Cas9系统进行基因编辑的研究[22]。由于传统的基因筛选方法(如RNAi)在哺乳动物细胞中进行基因敲除较为困难,因此,Koike-Yusa等[23]于2014年首次将全基因组CRISPR/ Cas9应用于小鼠()胚胎干细胞中,成功鉴定出一些与抗毒素相关的基因。2016年,Komor等[24]发现传统的CRISPR/Cas9通常需要通过引发双链断裂(double strand break,DSB)来实现DNA修饰。然而,DSB可能引发细胞的修复机制,并导致不可预测的变异。因此,该团队开发了一种新方法,称为碱基编辑器(base editing,BE),旨在精确修改基因组DNA的单个碱基。具体而言,通过使用Cas9蛋白和特定的单链RNA引导子,将化学修饰核苷酸修饰酶(cytidine deaminase)定向到目标碱基上,并将目标碱基从胞嘧啶(C)改变为嘧啶(T)。这项技术为CRISPR/Cas9的发展提供了一个创新性突破。在此基础上,该团队又开发了一种名为“Target-AID”的单核苷酸编辑系统,它通过引入特定的修饰酶,能够直接将一种碱基转化为另一种碱基。Anzalone等[25]设计了适用于特定目标序列的编辑引导RNA,将Target-AID定向到目标碱基上,再使用Target-AID修饰酶将目标碱基替换为编辑后的碱基。由于传统的CRISPR/Cas9系统通常需要数个小时完成基因编辑,Liu等[26]提出了一种新策略,即使用笼状RNA策略(caged RNA,cgRNA)。该策略在RNA序列中添加光敏基团,限制了Cas9酶的功能,直到引入光源。光源激活后,光敏基团解离,使得Cas9酶能够在几秒内快速切割目标DNA。这种新方法被称为超快速CRISPR基因编辑方法(very fast CRISPR,vfCRISPR)。这一研究成果为CRISPR/Cas9领域带来了变革性的科学进展。目前,科研人员仍在努力研发更为高效且具有更低脱靶率的基因编辑技术(图2)。
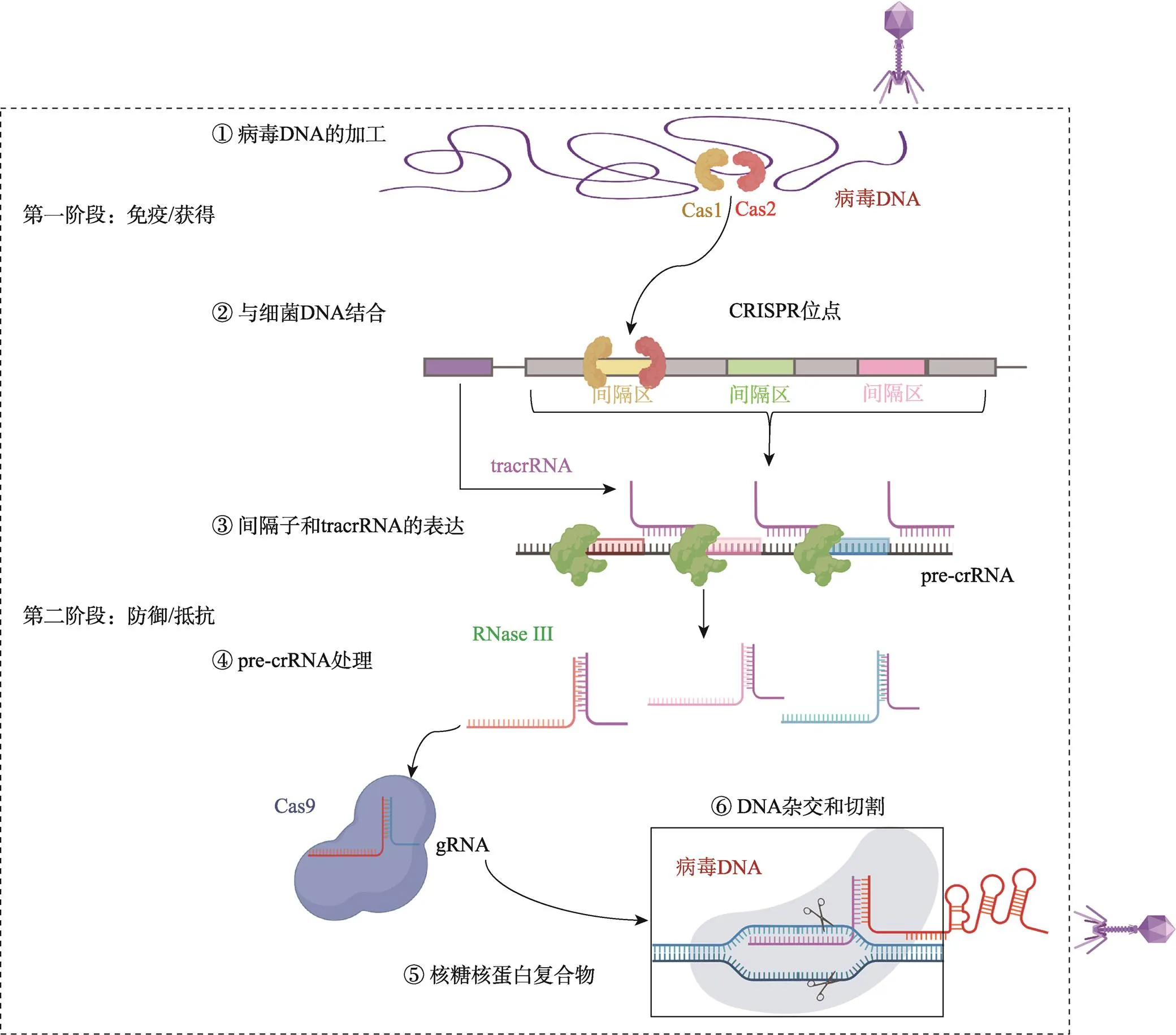
图1 噬菌体CRISPR/Cas9适应性免疫系统
CRISPR基因序列主要由前导序列(leader)、重复序列(repeat)和间隔序列(spacer)构成。前导序列位于CRISPR基因上游,被认为是CRISPR的启动子,其富含AT碱基[17]。重复序列的长度约为20~50 bp,包括5~7 bp回文序列[27],其转录产物能够形成发卡结构,从而稳定RNA的二级结构[20]。间隔序列是由细菌捕获的外源性DNA序列[15]。目前,在细菌免疫防御中根据Cas蛋白的作用将CRISPR系统分为两大类,又分为6种不同类型(I~VI),并且子类型的数目还在持续增长[17]。I类系统包括的I、III和IV型系统的crRNA效应复合物由多个亚基组成,而II类系统包括的II型、V型和VI系统的crRNA效应复合物仅由单亚基组成[28,29]。Cas9蛋白是II类系统所需的唯一蛋白质,Cas9与crRNA-tracrRNA双链体结合,可用于高效的基因组编辑。这种双RNA结构与Cas9蛋白形成RNP复合体,识别与crRNA互补的PAM序列,将DNA双链解链,crRNA将与互补链杂交,而另一条DNA链则保持游离状态。之后,Cas9蛋白负责准确切割目标PAM序列,再切割与crRNA互补的DNA单链,最终在Cas9的作用下DNA发生DSB,导致切割位点上的小片段序列的插入和/或缺失[30],此外,它还可以通过机体的同源定向修复机制导致外源DNA的表达沉默,从而实现对病毒的防御[5,11,15,31]。Yu等[32]将CRISPR/Cas9系统成功运用于基因组编辑,借助其在病毒防御中的原理,为基因编辑和研究领域带来了新的可能性和机遇。主要流程如下:(1)构建相关基因的sgRNA文库,并将sgRNA整合到慢病毒中,制备慢病毒文库;(2)使用低感染度感染细胞,使sgRNA整合到细胞基因组中。随着基因组DNA的复制,sgRNA被复制并传递给细胞后代。通过抗生素或其他特定筛选条件,筛选出感染病毒的细胞;(3)根据特定的表型需求,如耐药性、增殖能力、存活能力和带有特定标记基因等,选择适当的细胞;(4)提取细胞核基因组,准备进行下一步的建库。通过高通量测序对建库的细胞核基因组进行测序;(5)通过各种生物信息学分析方法,从测序数据中获取目标基因的相关信息,进一步研究和理解其功能(图3)。
2 CRISPR/Cas9技术在畜禽遗传改良中的应用
近年来,基因组编辑技术CRISPR的迅速发展使得该技术在畜禽的多方面应用提供了可能性,通过CRISPR/Cas9技术,畜禽肌肉生长可以得到促进,这使得肉类产量得到提高。绵羊毛和山羊绒的质量也得到改良,使得纤维产品更具商业价值。此外,通过编辑动物基因,牛奶和羊奶的品质可以得到改善,从而提供更健康和高营养价值的乳制品。该技术还有助于提高畜禽的抗病能力,减少疾病对养殖业的影响,进而提高养殖效益。此外,通过增强畜禽的生长和健康状况,CRISPR/Cas9技术也有助于改善动物福利。

图2 基因编辑技术发展历程图
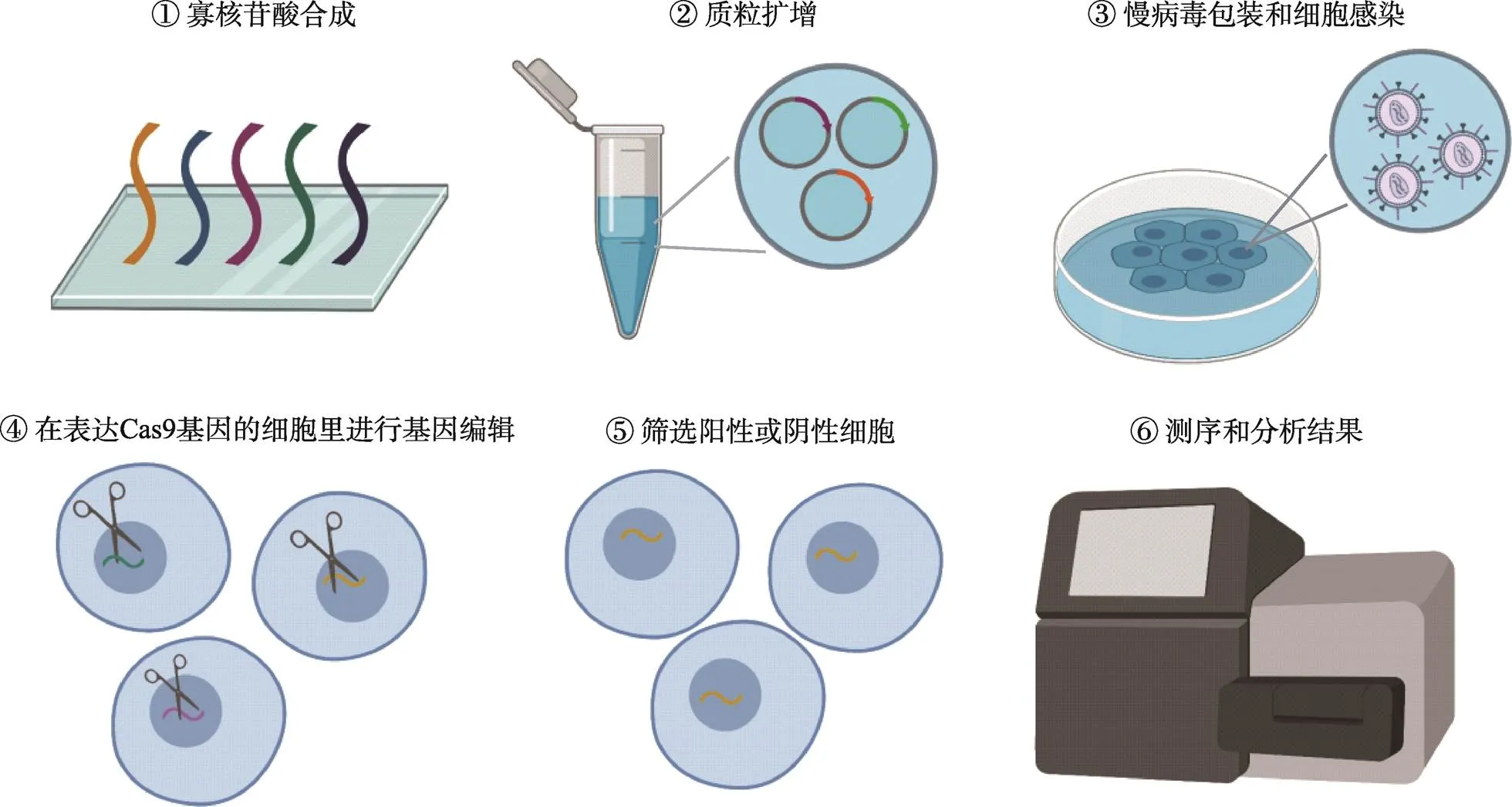
图3 CRISPR/Cas9系统工作的主要流程
2.1 在畜禽肌肉生长和发育上的研究进展
肌肉抑制素(myostatin,MSTN)是一种肌肉生长抑制因子,它对肌肉的生长和发育起到负调控的作用。基因突变、缺失或敲除可以导致肌肉细胞增殖和分化的增加,从而促进肌肉的生长和发育[33]。通过CRISPR/Cas9基因编辑以及优化Cas9:sgRNA (向导RNA)传递系统,成功实现了绵羊()的双等位基因敲除,与野生型(WT)绵羊相比,敲除绵羊具有更大的肌肉质量和肌肉纤维直径,但其肉质品质和口感等方面未受到影响[34]。同样,在牛()中也实现将sgRNA和Cas9的合成mRNA体外显微注射到受精牛胚胎,出生健康犊牛的突变率高达99.9%,表现出双等位基因突变和双肌肉表型[35]。在山羊()中也有各项研究表明,抑制基因的表达,可促进双肌肉表型的形成[36~41]。在猪()的研究中,Li等[42]使用特异性靶向编辑技术,通过设计特定的sgRNA,对两广小花猪胚胎中的MSTN信号肽进行基因编辑。编辑后的小猪与未编辑的小猪相比,显示出明显增加的肌肉量,同时其发育和健康状态与未编辑的小猪无明显差异。在鸡()中,使用D10A-Cas9 nickase介导的方法,选择性地引入DSB,从而生成了基因敲除的鸡群,这些敲除突变鸡的肌肉生长显著增加,生长和繁殖均正常[43]。在马()中,一般会通过编辑基因来增强肌肉生长和改善运动表现[44]。
CRISPR/Cas9基因编辑技术除对基因进行编辑之外,还在肌原细胞分化蛋白1(myogenic differentiation 1,MYOD1)[45]、胰岛素样生长因子1(insulin-like growth factor 1,IGF1)[46]、胰岛素样生长因子2(insulin-like growth factor 2,IGF2)[47,48]和抗肌肉生长因子抗体(follistatin,FST)[49]等编码与肌肉发育或分化相关蛋白的基因功能调控上得到了应用。
过氧化物酶增殖物激活受体(peroxisome proliferator-activated receptor,PPAR)是调控脂肪细胞分化的关键因子,可通过激活脂肪细胞分化调节因子(如FABP4和CCAAT增强子结合蛋白)以及增强和基因的表达来促进脂肪细胞分化,从而调控脂肪沉积。Gu等[50]通过随机插入和CRISPR/Cas9转基因克隆程序在两个猪模型中成功地肌肉特异性过表达了过氧化物酶增殖物激活受体γ (peroxisome proliferator-activated receptor gamma,PPARγ),该编辑猪肌内脂肪含量显著增加,瘦肉比例无变化。此外,在肉牛中成功地实现了对基因的位点定向突变的同时进行了位点的定向敲入,突变对生长性状和肌肉发展产生了显著的影响[51],而敲入对这些性状的影响较小[52]。
2.2 在绒毛生长发育中的研究进展
绵羊和山羊作为重要的畜禽资源,为人类提供了用于生产各种服装和纺织品的宝贵的纤维来源。成纤维细胞生长因子5 (fibroblast growth factor 5,FGF5)是影响羊毛长度的主要因子。目前,通过CRISPR/Cas9技术成功生成了敲除绵羊。该敲除绵羊所有细胞系都存在基因突变,同时还检测到5、13和33个碱基对的基因缺失,这些结果导致FGF5蛋白高级结构发生变化,影响其正常功能,从而引起敲除绵羊的羊毛长度明显长于WT绵羊[53]。另外,Li[54]等和Wang等[55]的研究也确认了这一事实,即CRISPR/Cas9介导的基因缺失不仅导致其活性的丧失,还能促进绵羊毛和山羊绒的生长,从而增加它们的长度和产量。刺鼠信号蛋白(agouti signaling protein,ASIP)是调节皮肤和毛发色素的蛋白,其主要功能是调控皮肤和毛囊中色素产生。Zhang等[56]成功地干扰了中国美利奴羊ASIP的正常功能,导致羊毛的颜色发生了变化,这一突破为产生具有理想羊毛颜色的品种培育提供了机会。
有研究表明,在位点敲入血管内皮生长因子(vascular endothelial growth factor,VEGF),并使用和的组合能够显著提高CRISPR/Cas9介导的同源定向修复效率,通过该技术获得了一只在位点整合了基因的具有突出绒毛性能的绒山羊[57]。此外,通过将胸腺素4 (thymosin β4,Tβ4)基因特异性插入山羊趋化因子受体(chemokine receptor 5,CCR5)基因位点,培育出了高产绒山羊,羊绒产量提高了74.5%[58],细度和质量不受影响。在双基因敲除绒山羊模型中,Wang等[59]使用CRISPR/Cas9技术,在受精卵中引入了设计好的特异性靶向RNA和DNA片段。这些靶向RNA和DNA片段与目标基因的特定区域匹配,并促使CRISPR/Cas9系统发生基因编辑作用,成功地敲除了基因和基因,这使得绒山羊的肌肉发育得到显著改善,并且观察到了更多的次级毛囊的形成和更长的羊绒纤维,从而提高了绒山羊的商业价值和经济效益。
2.3 在乳品质改良上的应用进展
人体对牛奶或羊奶过敏主要是由于人体免疫系统对奶中的蛋白质产生异常免疫反应所造成的。β-乳球蛋白(beta-lactoglobulin,BLG)被认为是一种重要的致敏性物质,因此可以通过基因编辑技术对乳品质进行改善。科研人员使用CRISPR/Cas9基因编辑技术,通过对牛的基因进行修改来改变牛奶的成分和性质。例如:Alessio等[60]将引入转座子中,催化合成ω-3和ω-6酸,然后,通过CRISPR/ Cas9技术,实现靶向敲除BLG蛋白,成功地修改了牛的基因,提高了牛奶中的脂肪和蛋白质含量。Silaeva等[61]着重于减少牛奶中引起过敏的成分,在BLG中引入双链gap,从而产生了过敏原含量更低的牛奶。Singina等[62]在牛胚胎成纤维细胞中成功获得了敲除BLG蛋白和β-乳球蛋白样蛋白基因细胞集落,未来将被用于生产缺乏BLG蛋白的牛。在山羊上,将Cas9 mRNA和sgRNA共注射到胚胎中生成BLG蛋白敲除山羊,发现在双链sgRNA引导的靶向实验的编辑效率比单链sgRNA低,并且基因以及其他乳蛋白编码基因在敲除山羊乳腺中的相对表达显着降低,此外,大部分编辑的胎儿是嵌合体,表明编辑效果可以在多个组织中实现[63]。
山羊奶含有丰富的不饱和脂肪酸,是合成乳脂的必需因子。而硬脂酰辅酶A去饱和酶1 (stearoyl- CoA desaturase 1,SCD1)是催化单不饱和脂肪酸合成的关键酶,对乳脂代谢至关重要。Tian等[64]使用CRISPR/Cas9技术在山羊乳腺上皮细胞(goat mammary epithelial cells,GMEC)中敲除了SCD1酶,导致三酰甘油、胆固醇含量降低和脂肪酸的去饱和酶指数减少,但对其他乳汁成分没有影响。通过乳脂质组学分析发现,SCD1敲除降低了三酰甘油和二酰甘油水平,并在甘油脂和甘油磷脂代谢途径中引起了差异。此外,Tian等[65]还揭示了SCD1通过影响脂质代谢基因表达和脂质代谢途径来调控山羊乳脂和不饱和脂肪酸的合成。MicroRNA(miRNA)可通过调节脂肪酸代谢途径中的关键基因来调控乳腺细胞的脂肪酸代谢,敲除和后,可以上调靶基因,同时上调并影响脂肪酸代谢关键基因的表达。这些作用有助于调节脂滴、甘油三酯和胆固醇的合成,并对C18:0、C18:2以及C20多不饱和脂肪酸的含量产生了影响[66~68]。敲除GMEC中的ASIP蛋白也可以促进中长链脂肪酸合成增加,而敲除丙二酰/乙酰转移酶(malonyl/ acetyltransferase,MAT)降低甘油三酯和中链脂肪酸的含量。总体而言,这些因子在调节GMEC的脂质代谢中起着关键作用,这可能会进一步影响山羊奶中脂质的成分。
2.4 在畜禽抗病力研究中的应用
全球范围内畜禽传染病会降低动物生产能力和质量,导致农业经济巨大损失。宿主可以通过特定的基因编码的抗微生物肽或蛋白质,直接破坏或抑制病原微生物,或者阻断病原微生物进入宿主细胞所需的受体。此外,还可以通过干扰病原微生物的生命周期过程,阻碍正常生存和复制,从而降低其对宿主的伤害程度。这两种策略都是宿主自身的机制来保护自己免受感染。因此,如何从源头上进行疾病的预防是目前农牧业面临的关键问题[69]。
黑色素瘤分化相关蛋白5 (melanoma differentiation-associated protein 5,MDA5)、线粒体抗病毒信号(mitochondrial antiviral signaling protein,MAVS)蛋白和干扰素基因刺激因子(stimulator of interferon genes,STING)均为细胞对抗病原微生物感染相关的蛋白质,它们在细胞内形成一个信号传递网络,促进免疫反应的激活和病原微生物的清除。鸡的先天免疫系统具有两种主要的病原体识别受体,即Toll样受体3(toll-like receptor 3,TLR3)和MDA5,通过敲除鸡DF-1成纤维细胞中和MDA5蛋白,与WT细胞相比,发现双敲除细胞中AOAV-1病毒的复制率明显提高[70]。粘病毒抵抗基因(myxovirus resistance,Mx)被证明具有广泛的抗病毒和GTP酶活性。为了研究鸡对新城疫病毒(Newcastle disease virus,NDV)感染的影响,使用CRISPR/Cas9基因编辑系统构建了敲除基因的DF-1细胞系。发现敲除的细胞中NDV的病毒滴度更高,并促进了一些免疫因子的表达。表明在防止病毒侵入中发挥了重要的作用[71]。此外,敲除基因后,牛乳腺上皮细胞对鸟分枝杆菌副结核病(subsp.,MAP)菌株细胞溶解物的炎症反应减弱,表现出更低的炎症因子的产生,这暗示在MAP感染诱导的牛乳腺炎症反应中发挥重要作用,可能对抗MAP菌株感染具有潜在保护作用[72]。敲除也影响牛乳腺上皮细胞的形态、增殖、迁移和β-酪蛋白分泌,能够减少乳腺炎的发生[73]。对患有糖原分支酶缺乏症(glycogen branching enzyme deficiency,GBED)的马进行基因组筛查,检测到这些马在GBE1基因有一个点突变,随后,使用CRISPR/ Cas9系统来针对这个点突变进行修复,成功地恢复了基因的正常表达和功能,并实现了对GBED马的治疗,这项研究也暗示,CRISPR/Cas9系统是对畜禽遗传疾病进行研究和治疗的有效工具[74]。
猪β-防御素2(porcine beta-defensin 2,PBD-2)是一种重要的天然免疫分子,具有广谱的抗菌活性和免疫调节功能。研究人员使用CRISPR/Cas9和Cre/loxP系统生成无标记的PBD-2敲入猪的基因座,再通过体细胞核移植产生克隆仔猪,发现PBD-2在转基因仔猪不同组织中的表达水平显著高于WT仔猪,并且克隆猪的猪耳成纤维细胞的抗菌特性显著增加[75]。在一项关于非洲猪瘟病毒(African swine fever virus,ASFV)研究中,发现了5种猪干扰素诱导的跨膜蛋白(SwIFITM1a、-1b、-2、-3和-5)敲除增强了ASFV的复制,这表明SwIFITMs对ASFV具有强烈的抗病毒作用[76]。自然抵抗相关巨噬细胞蛋白1 (natural resistance-associated macrophage protein 1,NRAMP1)在抗菌免疫中发挥重要作用。通过使用Cas9剪切酶的单个活性位点,在牛的基因组中插入NRAMP1蛋白,显著降低离靶效应并提高基因编辑的精确性,获得了具有强抗结核病的转基因牛[77]。利用CRISPR/Cas9技术建立组蛋白脱乙酰酶9(histone deacetylases 9,HDAC9)敲除BHK-21细胞,发现在口蹄疫病毒感染后各病毒相关指标均增长,这说明HDAC9在宿主抗病毒先天免疫应答中有至关重要的作用[78]。
马立克病病毒(Marek’s disease virus,MDV)是家禽中一种常见的传染性疾病,对家禽产业造成了严重的经济损失。通过敲除MDV关键基因,研究人员成功阻断了MDV的复制和传播,从而有效抑制了马立克病病毒的感染。这项研究为控制马立克病的传播和疫苗开发提供了新的思路和方法[79]。在牛病毒性腹泻病(bovine viral diarrhoea virus,BVDV)方面,一篇研究探讨了黄病毒科瘟病毒属内不同病毒在宿主细胞中的进入机制。该报道显示,牛补体调节蛋白46 (complement regulatory protein 46,CD46BOV)在不同病毒中具有不同的作用。CD46BOV作为HoBi样瘟病毒(HoBi-like pestiviruses,HoBiPeV)的主要细胞进入因子,而不是起源于肯尼亚的长颈鹿瘟病毒(giraffe pestivirus,GPeV)。此外,他们还发现,、、和的病毒分离株能够通过使用硫酸乙酰肝素进入宿主细胞来适应细胞培养条件。这表明不同的牛瘟病毒使用不同的宿主细胞进入机制[80]。伪狂犬病病毒(pseudorabies virus,PRV)是一种嗜神经病毒,可引起猪的神经系统疾病。在PRV进入宿主细胞的过程中,血小板反应蛋白3 (thrombospondin 3,THBS3)起着重要作用。THBS3是一种新的PRV的共受体,可以与PRV的包膜糖蛋白相互作用,启动PRV进入细胞的过程。通过在不同细胞中敲除或过表达THBS3来验证其在PRV感染中的作用,结果显示THBS3通过与PRV的特定蛋白相互作用,促进了病毒与宿主细胞的结合和膜融合[81]。
通过多基因编辑可以使家畜对多种主要病毒具有抗性。猪繁殖与呼吸综合征病毒(porcine reproductive and respiratory syndrome virus,PRRSV)和传染性肠胃炎病毒(transmissible gastroenteritis virus,TGEV)是两种对全球养猪产业造成重大经济损失的高传染性病毒。对CD163和pAPN进行双基因敲除的猪,发现DKO猪对PRRSV和TGEV具有完全耐药性。在肉质和繁殖性能方面,野生型和双基因敲除猪没有差异,并且敲除猪对猪德尔塔冠状病毒(porcine deltacoronavirus,PDCoV)的易感性也降低了[82]。
2.5 在动物福利改善中的研究进展
执行动物福利的必要性是基于人们对动物的道德责任和尊重。动物是有感知和情感的生物,应该受到适当的关爱和保护。随着现代畜牧业的发展,基因编辑技术可以避免牲畜遭受一些比如犊牛去角、雄性阉割、奶牛去尾、堕胎或剔除不需要的性别等不必要的痛苦[83]。
安格斯牛的1号染色体上P等位基因的变异由212 bp的DNA重复序列组成,取代了无角位点的10 bp序列导致了其自然无角。Hennig等[84]通过CRISPR/Cas9技术采用双sgRNA删除了一个包括在无角位点缺失的10 bp的133 bp区域,随后进行133 bp缺失胚胎的移植,最终获得了缺失133 bp的双等位基因胎儿,但均显示出角芽发育,因此还需进一步研究寻找其他引发无角表型的因素。
通常,为了削除腥臭味,用于猪肉供应的雄性仔猪会进行手术阉割。然而,这可能导致雄性仔猪的感染和身体的痛苦。Flórez等[85]将KiSS-1转移抑制因子(KiSS-1 metastasis suppressor,KISS1)作为目标,通过使用CRISPR/Cas9技术,在猪的基因组中编辑了基因,成功地导致其功能受损,编辑后的猪表现出下丘脑性性腺功能减退症(hypogonadotropic hypogonadism)的特征,即性腺功能不正常。该研究为提供一种无需进行去势的方法来控制猪的繁殖特征提供了可能性。
热应激可能导致动物的脱水、电解质紊乱、增加患病风险等健康问题,还可能导致如减少活动和出现不安行为等行为改变,并且导致如生长速度减缓、产蛋量下降。因此,亟需采取相应的措施解决热应激对家养动物的影响,从而保障健康、行为和生产能力。催乳素受体(prolactin receptor,PRLR)和热休克蛋白(heat shock 70 kDa protein 1-like,HSPA1L)的突变可赋予牛良好的体温调节和细胞保护能力。通过基因编辑技术将这些特定基因转移给无法适应高温气候的品种,有望减少热应激对牛类生产的影响[86]。
3 结语与展望
尽管CRISPR/Cas9技术在基因组编辑领域的应用被认为是一项具有巨大潜力的革命性进展,但在学术领域和公众之间仍存在分歧,这些分歧包括技术应用的安全性和可靠性问题。首先,安全性方面的问题主要涉及到对编辑动物基因组的全面评估。这包括检查编辑基因对动物本身以及周围环境的潜在影响,并评估潜在的风险。此外,还需要关注导入基因组的稳定性和遗传多样性的维持,以避免不可逆的遗传塑造和遗传缺陷。通过全面的安全评估,可以确保编辑动物在健康和繁殖能力方面与非编辑群体一致,并减少任何可能的不良遗传影响。其次,可靠性方面的问题涉及到技术本身的精确性和效率。CRISPR/Cas9技术在基因编辑方面具有准确性和高效性的优点,但仍需继续改进。除此之外,也需要集中精力解决编辑技术中的潜在误差和副作用,以提高技术的可靠性和预测性。CRISPR/Cas9技术在基因编辑畜禽动物中未来发展的重要方向为进一步研究基因功能和基因网络的理解,以及CRISPR/ Cas9与其他基因编辑技术的结合。这些方向将有助于进一步拓宽畜禽遗传改良的可能性。总体而言,随着CRISPR/Cas9基因编辑技术的不断发展和改进,相信它将在畜牧业遗传改良中发挥越来越重要的作用,并为提高畜禽生产效率和质量做出贡献。
[1] Singh P, Ali SA. Impact of CRISPR-Cas9-based genome engineering in farm animals., 2021, 8(7): 122.
[2] Bibikova M, Beumer K, Trautman JK, Carroll D. Enhancing gene targeting with designed zinc finger nucleases., 2003, 300(5620): 764.
[3] Hockemeyer D, Wang HY, Kiani S, Lai CS, Gao Q, Cassady JP, Cost GJ, Zhang L, Santiago Y, Miller JC, Zeitler B, Cherone JM, Meng XD, Hinkley SJ, Rebar EJ, Gregory PD, Urnov FD, Jaenisch R. Genetic engineering of human pluripotent cells using TALE nucleases., 2011, 29(8): 731–734.
[4] Doudna JA, Charpentier E. Genome editing. The new frontier of genome engineering with CRISPR-Cas9., 2014, 346(6213): 1258096.
[5] Sander JD, Joung JK. CRISPR-Cas systems for editing, regulating and targeting genomes., 2014, 32(4): 347–355.
[6] Baliou S, Adamaki M, Kyriakopoulos AM, Spandidos DA, Panayiotidis M, Christodoulou I, Zoumpourlis V. CRISPR therapeutic tools for complex genetic disorders and cancer (Review)., 2018, 53(2): 443–468.
[7] Kruminis-Kaszkiel E, Juranek J, Maksymowicz W, Wojtkiewicz J. CRISPR/Cas9 technology as an emerging tool for targeting amyotrophic lateral sclerosis (ALS)., 2018, 19(3): 906.
[8] Belk JA, Yao W, Ly N, Freitas KA, Chen YT, Shi QM, Valencia AM, Shifrut E, Kale N, Yost KE, Duffy CV, Daniel B, Hwee MA, Miao Z, Ashworth A, Mackall CL, Marson A, Carnevale J, Vardhana SA, Satpathy AT. Genome-wide CRISPR screens of T cell exhaustion identify chromatin remodeling factors that limit T cell persistence., 2022, 40(7): 768–786.
[9] Ferrari G, Thrasher AJ, Aiuti A. Gene therapy using haematopoietic stem and progenitor cells., 2021, 22(4): 216–234.
[10] Nuñez JK, Lee ASY, Engelman A, Doudna JA. Integrase-mediated spacer acquisition during CRISPR-Cas adaptive immunity.e, 2015, 519(7542): 193–198.
[11] Makarova KS, Koonin EV. Annotation and classification of CRISPR-Cas systems., 2015, 1311: 47–75.
[12] Ishino Y, Shinagawa H, Makino K, Amemura M, Nakata A. Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product., 1987, 169(12): 5429–5433.
[13] Pavletich NP, Pabo CO. Zinc finger-DNA recognition: crystal structure of a Zif268-DNA complex at 2.1 A., 1991, 252(5007): 809–817.
[14] Kim YG, Cha J, Chandrasegaran S. Hybrid restriction enzymes: zinc finger fusions to Fok I cleavage domain., 1996, 93(3): 1156–1160.
[15] Horvath P, Barrangou R. CRISPR/Cas, the immune system of bacteria and archaea., 2010, 327(5962): 167– 170.
[16] Jansen R, van Embden JDA, Gaastra W, Schouls LM. Identification of genes that are associated with DNA repeats in prokaryotes., 2002, 43(6): 1565–1575.
[17] Bibikova M, Golic M, Golic KG, Carroll D. Targeted chromosomal cleavage and mutagenesis in Drosophila using zinc-finger nucleases., 2002; 161(3): 1169–1175.
[18] Barrangou R, Fremaux C, Deveau H, Richards M, Boyaval P, Moineau S, Romero DA, Horvath P. CRISPR provides acquired resistance against viruses in prokaryotes., 2007, 315(5819): 1709–1712.
[19] Christian M, Cermak T, Doyle EL, Schmidt C, Zhang F, Hummel A, Bogdanove AJ, Voytas DF. Targeting DNA double-strand breaks with TAL effector nucleases., 2010, 186(2): 757–761.
[20] Deltcheva E, Chylinski K, Sharma CM, Gonzales K, Chao YJ, Pirzada ZA, Eckert MR, Vogel J, Charpentier E. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase III., 2011, 471(7340): 602–607.
[21] Li TM, Du B. CRISPR-Cas system and coevolution of bacteria and phages., 2011, 33(3): 213–218.
[22] Mali P, Yang LH, Esvelt KM, Aach J, Guell M, DiCarlo JE, Norville JE, Church GM. RNA-guided human genome engineering via Cas9., 2013, 339(6121): 823–826.
[23] Koike-Yusa H, Li YL, Tan EP, Velasco-Herrera MDC, Yusa K. Genome-wide recessive genetic screening in mammalian cells with a lentiviral CRISPR-guide RNA library., 2014, 32(3): 267–273.
[24] Komor AC, Kim YB, Packer MS, Zuris JA, Liu DR. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage., 2016, 533(7603): 420–424.
[25] Anzalone AV, Randolph PB, Davis JR, Sousa AA, Koblan LW, Levy JM, Chen PJ, Wilson C, Newby GA, Raguram A, Liu DR. Search-and-replace genome editing without double-strand breaks or donor DNA., 2019, 576(7785): 149–157.
[26] Liu Y, Zou RS, He SX, Nihongaki Y, Li XG, Razavi S, Wu B, Ha T. Very fast CRISPR on demand., 2020, 368(6496): 1265–1269.
[27] Kunin V, Sorek R, Hugenholtz P. Evolutionary conservation of sequence and secondary structures in CRISPR repeats., 2007, 8(4): R61.
[28] Jinek M, Chylinski K, Fonfara I, Hauer M, Doudna JA, Charpentier E. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity., 2012, 337(6096): 816–821.
[29] Garneau JE, Dupuis MÈ, Villion M, Romero DA, Barrangou R, Boyaval P, Fremaux C, Horvath P, Magadán AH, Moineau S. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA., 2010, 468(7320): 67–71.
[30] Rouet P, Smih F, Jasin M. Introduction of double- strand breaks into the genome of mouse cells by expression of a rare-cutting endonuclease., 1994, 14(12): 8096–8106.
[31] Marraffini LA, Sontheimer EJ. Self versus non-self discrimination during CRISPR RNA-directed immunity., 2010, 463(7280): 568–571.
[32] Yu JSL, Yusa K. Genome-wide CRISPR-Cas9 screening in mammalian cells., 2019, 164–165: 29–35.
[33] Ge LX, Dong XC, Gong XT, Kang J, Zhang Y, Quan FS. Mutation in myostatin 3'UTR promotes C2C12 myoblast proliferation and differentiation by blocking the translation of MSTN., 2020, 154: 634–643.
[34] Zhou SW, Kalds P, Luo Q, Sun KX, Zhao XE, Gao YW, Cai B, Huang SH, Kou QF, Petersen B, Chen YL, Ma BH, Wang XL. Optimized Cas9: sgRNA delivery efficiently generates biallelic MSTN knockout sheep without affecting meat quality., 2022, 23(1): 348.
[35] Gim GM, Kwon DH, Eom KH, Moon J, Park JH, Lee WW, Jung DJ, Kim DH, Yi JK, Ha JJ, Lim KY, Kim JS, Jang G. Production of MSTN-mutated cattle without exogenous gene integration using CRISPR-Cas9., 2022, 17(7): e2100198.
[36] Lyu M, Wang X, Meng XY, Qian HR, Li Q, Ma BX, Zhang ZY, Xu K. chi-miR-487b-3p inhibits goat myoblast proliferation and differentiation by targeting IRS1 through the IRS1/PI3K/Akt signaling pathway., 2021, 23(1): 115.
[37] Zhang J, Liu J, Yang WL, Cui ML, Dai B, Dong YH, Yang J, Zhang XM, Liu DJ, Liang H, Cang M. Comparison of gene editing efficiencies of CRISPR/Cas9 and TALEN for generation of MSTN knock-out cashmere goats., 2019, 132: 1–11.
[38] Wang X, Niu Y, Zhou J, Zhu H, Ma B, Yu H, Yan H, Hua J, Huang X, Qu L, Chen Y. CRISPR/Cas9-mediated MSTN disruption and heritable mutagenesis in goats causes increased body mass., 2018, 49(1): 43–51.
[39] Guo RH, Wan Y,J Xu D, Cui LB, Deng MT, Zhang GM, Jia RX, Zhou WJ, Wang Z, Deng KP, Huang MR, Wang F, Zhang YL. Generation and evaluation of Myostatin knock-out rabbits and goats using CRISPR/Cas9 system., 2016, 6: 29855.
[40] Li C, Zhou SW, Li Y, Li GW, Ding YG, Li L, Liu J, Qu L, Sonstegard T, Huang XX, Jiang Y, Chen YL, Petersen B, Wang XL. Trio-based deep sequencing reveals a low incidence of off-target mutations in the offspring of genetically edited goats., 2018, 9: 449.
[41] He ZY, Zhang T, Jiang L, Zhou MY, Wu DJ, Mei JY, Cheng Y. Use of CRISPR/Cas9 technology efficiently targetted goat myostatin through zygotes microinjection resulting in double-muscled phenotype in goats., 2018, 38(6): BSR20180742.
[42] Li RQ, Zeng W, Ma M, Wei ZX, Liu HB, Liu XF, Wang M, Shi X, Zeng JH, Yang LF, Mo DL, Liu XH, Chen YS, He ZY. Precise editing of myostatin signal peptide by CRISPR/Cas9 increases the muscle mass of Liang Guang Small Spotted pigs., 2020, 29(1): 149– 163.
[43] Kim GD, Lee JH, Song SM, Kim SW, Han JS, Shin SP, Park BC, Park TS. Generation of myostatin-knockout chickens mediated by D10A-Cas9 nickase., 2020, 34(4): 5688–5696.
[44] Moro LN, Viale DL, Bastón JI, Arnold V, Suvá M, Wiedenmann E, Olguín M, Miriuka S, Vichera G. Generation of myostatin edited horse embryos using CRISPR/Cas9 technology and somatic cell nuclear transfer., 2020, 10(1): 15587.
[45] Zhou D, Wang Y, Yang R, Wang F, Zhao ZH, Wang X, Xie LL, Tian XZ, Wang GZ, Li B, Gong Y. The MyoD1 promoted muscle differentiation and generation by activating CCND2 in Guanling cattle., 2022, 12(19): 2571.
[46] Roberston MJ, Raghunathan S, Potaman VN, Zhang F, Stewart MD, McConnell BK, Schwartz RJ. CRISPR- Cas9-induced IGF1 gene activation as a tool for enhancing muscle differentiation via multiple isoform expression., 2020, 34(1): 555–570.
[47] Zou HY, Yu DW, Yao S, Ding FR, Li JL, Li L, Li X, Zhao SJ, Pang YW, Hao HS, Du WH, Zhao XM, Dai YP, Zhu HB. Efficient editing of the ZBED6-binding site in intron 3 of IGF2 in a bovine model using the CRISPR/Cas9 system., 2022, 13(7): 1132.
[48] Xiang GH, Ren JL, Hai T, Fu R, Yu DW, Wang J, Li W, Wang HY, Zhou Q. Editing porcine IGF2 regulatory element improved meat production in Chinese Bama pigs., 2018, 75(24): 4619–4628.
[49] Li MJ, Tang XC, You WN, Wang YB, Chen YW, Liu Y, Yuan HM, Gao C, Chen X, Xiao ZW, Ouyang HS, Pang DX. Erratum: HMEJ-mediated site-specific integration of a myostatin inhibitor increases skeletal muscle mass in porcine., 2021, 26: 49–62.
[50] Gu H, Zhou Y, Yang JZ, Li JN, Peng YX, Zhang X, Miao YL, Jiang W, Bu GW, Hou LM, Li T, Zhang L, Xia XL, Ma ZY, Xiong YZ, Zuo B. Targeted overexpression of PPARγ in skeletal muscle by random insertion and CRISPR/Cas9 transgenic pig cloning enhances oxidative fiber formation and intramuscular fat deposition., 2021, 35(2): e21308.
[51] Zhao YF, Yang L, Su GH, Wei ZY, Liu XF, Song LS, Hai C, Wu D, Hao ZT, Wu YX, Zhang L, Bai CL, Li GP. Growth traits and sperm proteomics analyses of myostatin gene-edited Chinese Yellow Cattle., 2022, 12(5): 627.
[52] Ge LX, Kang J, Dong XC, Luan DJ, Su GH, Li GP, Zhang Y, Quan FS. Myostatin site-directed mutation and simultaneous PPARγ site-directed knockin in bovine genome., 2021, 236(4): 2592–2605.
[53] Hu R, Fan ZY, Wang BY, Deng SL, Zhang XS, Zhang JL, Han HB, Lian ZX. RAPID COMMUNICATION: generation of FGF5 knockout sheep via the CRISPR/Cas9 system., 2017, 95(5): 2019–2024.
[54] Li WR, Liu CX, Zhang XM, Chen L, Peng XR, He SG, Lin JP, Han B, Wang LQ, Huang JC, Liu MJ. CRISPR/ Cas9-mediated loss of FGF5 function increases wool staple length in sheep., 2017, 284(17): 2764– 2773.
[55] Wang XL, Cai B, Zhou JK, Zhu HJ, Niu YY, Ma BH, Yu HH, Lei AM, Yan HL, Shen QY, Shi L, Zhao XE, Hua JL, Huang XX, Qu L, Chen YL. Disruption of FGF5 in cashmere goats using CRISPR/Cas9 results in more secondary hair follicles and longer fibers., 2016, 11(10): e0164640.
[56] Zhang XM, Li WR, Liu CX, Peng XR, Lin JP, He SG, Li XJ, Han B, Zhang N, Wu YS, Chen L, Wang LQ, MaYila, Huang JC, Liu MJ. Alteration of sheep coat color pattern by disruption of ASIP gene via CRISPR Cas9., 2017, 7(1): 8149.
[57] Hu X, Hao F, Li XC, Xun ZY, Gao Y, Ren BX, Cang M, Liang H, Liu D. Generation of VEGF knock-in cashmere goat via the CRISPR/Cas9 system., 2021, 17(4): 1026–1040.
[58] Li XC, Hao F, Hu X, Wang H, Dai B, Wang X, Liang H, Cang M, Liu DJ. Generation of Tβ4 knock-in cashmere goat using CRISPR/Cas9., 2019, 15(8): 1743–1754.
[59] Wang XL, Yu HH, Lei AM, Zhou JK, Zeng WX, Zhu HJ, Dong ZM, Niu YY, Shi BB, Cai B, Liu JW, Huang S, Yan HL, Zhao XE, Zhou GX, He XL, Chen XX, Yang YX, Jiang Y, Shi L, Tian X, Wang YJ, Ma BH, Huang XX, Qu L, Chen YL. Generation of gene-modified goats targeting MSTN and FGF5 via zygote injection of CRISPR/Cas9 system., 2015, 5: 13878.
[60] Alessio A, Pericuesta E, Llamas-Toranzo I, Forcato D, Fili A, Liaudat C, Rodriguez N, Kues W, Bermejo-Álvarez P, Bosch P. 203 genome modifications by sleeping beauty transposition and CRISPR/Cas9 to improve cow milk composition for human consumption., 2018, 30(1): 242.
[61] Silaeva YY, Kubekina MV, Bruter AV, Isaeva AG, Koshchaev AG. Gene editing CRISPR/Cas9 system for producing cows with hypoallergenic milk on the background of a beta-lactoglobulin gene knockout., 2020, 176: 01006.
[62] Singina GN, Sergiev PV, Lopukhov AV, Rubtsova MP, Taradajnic NP, Ravin NV, Shedova EN, Taradajnic TE, Polejaeva IA, Dozev AV, Brem G, Dontsova OA, Zinovieva NA. Production of a cloned offspring and CRISPR/Cas9 genome editing of embryonic fibroblasts in cattle., 2021, 496(1): 48–51.
[63] Zhou WJ, Wan YJ, Guo RH, Deng MT, Deng KP, Wang Z, Zhang YL, Wang F. Generation of beta-lactoglobulin knock-out goats using CRISPR/Cas9., 2017, 12(10): e0186056.
[64] Tian HB, Luo J, Zhang ZF, Wu J, Zhang TY, Busato S, Huang L, Song N, Bionaz M. CRISPR/Cas9-mediated stearoyl-CoA desaturase 1 (SCD1) deficiency affects fatty acid metabolism in goat mammary epithelial cells., 2018, 66(38): 10041–10052.
[65] Tian HB, Niu HM, Luo J, Yao WW, Chen XY, Wu J, Geng YN, Gao WC, Lei AM, Gao ZM, Tian X, Zhao X, Shi HP, Li C, Hua JL. Knockout of stearoyl-CoA desaturase 1 decreased milk fat and unsaturated fatty acid contents of the goat model generated by CRISPR/Cas9., 2022, 70(13): 4030–4043.
[66] Huang L, Tian HB, Luo J, Song N, Wu J. CRISPR/Cas9 based knockout of miR-145 affects intracellular fatty acid metabolism by targeting INSIG1 in goat mammary epithelial cells., 2020, 68(18): 5138– 5146.
[67] Huang L, Luo J, Gao WC, Song N, Tian HB, Zhu L, Jiang QM, Loor JJ. CRISPR/Cas9-induced knockout of miR-24 reduces cholesterol and monounsaturated fatty acid content in primary goat mammary epithelial cells., 2022, 11(14): 2012.
[68] Huang L, Luo J, Song N, Gao WC, Zhu L, Yao WW. CRISPR/Cas9-mediated knockout of miR-130b affects mono- and polyunsaturated fatty acid content via PPARG-PGC1α axis in goat mammary epithelial cells., 2022, 23(7): 3640.
[69] Gao F, Li P, Yin Y, Du XG, Cao GS, Wu S, Zhao YF. Molecular breeding of livestock for disease resistance., 2023, 587: 109862.
[70] Lee CW, Kc M, Ngunjiri JM, Ghorbani A, Lee K. TLR3 and MDA5 knockout DF-1 cells enhance replication of avian orthoavulavirus 1., 2023, 67(1): 94–101.
[71] Wang L, Xue Z, Wang JP, Jian YW, Lu HZ, Ma HD, Wang SS, Zeng WX, Zhang T. Targeted knockout of Mx in the DF-1 chicken fibroblast cell line impairs immune response against Newcastle disease virus., 2023, 102(9): 102855.
[72] Shandilya UK, Sharma A, Mallikarjunappa S, Guo J, Mao Y, Meade KG, Karrow NA. CRISPR-Cas9-mediated knockout of TLR4 modulates Mycobacterium avium ssp. paratuberculosis cell lysate-induced inflammation in bovine mammary epithelial cells., 2021, 104(10): 11135–11146.
[73] Wang H, Wang XX, Li XR, Wang QW, Qing SZ, Zhang Y, Gao MQ. A novel long non-coding RNA regulates the immune response in MAC-T cells and contributes to bovine mastitis., 2019, 286(9): 1780–1795.
[74] Pinzon-Arteaga C, Snyder MD, Lazzarotto CR, Moreno NF, Juras R, Raudsepp T, Golding MC, Varner DD, Long CR. Efficient correction of a deleterious point mutation in primary horse fibroblasts with CRISPR-Cas9., 2020, 10(1): 7411.
[75] Huang J, Wang AT, Huang C, Sun YF, Song BX, Zhou R, Li L. Generation of marker-free pbd-2 knock-in pigs using the CRISPR/Cas9 and Cre/loxP systems., 2020, 11(8): 951.
[76] Cai SQ, Zheng ZZ, Cheng JJ, Zhong LT, Shao R, Zheng FY, Lai ZY, Ou JJ, Xu L, Zhou P, Lu G, Zhang GH. Swine interferon-inducible transmembrane proteins potently inhibit African swine fever virus replication., 2022, 13: 827709.
[77] Gao YP, Wu HB, Wang YS, Liu X, Chen LL, Li Q, Cui CC, Liu X, Zhang JC, Zhang Y. Single Cas9 nickase induced generation of NRAMP1 knockin cattle with reduced off-target effects., 2017, 18(1): 13.
[78] Hou ST, Wang XW, Ren SH, Meng XL, Yin XP, Zhang J, Tarasiuk K, Pejsak Z, Jiang T, Mao RQ, Zhang YG, Sun YF. Knockout of HDAC9 gene enhances foot-and-mouth disease virus replication., 2022, 13: 805606.
[79] Hagag IT, Wight DJ, Bartsch D, Sid H, Jordan I, Bertzbach LD, Schusser B, Kaufer BB. Abrogation of Marek’s disease virus replication using CRISPR/Cas9., 2020, 10(1): 10919.
[80] Leveringhaus E, Cagatay GN, Hardt J, Becher P, Postel A. Different impact of bovine complement regulatory protein 46 (CD46bov) as a cellular receptor for members of the speciesand., 2022, 11(1): 60–72.
[81] Pan YD, Guo LJ, Miao Q, Wu L, Jing ZY, Tian J, Feng L. Association of THBS3 with glycoprotein D promotes pseudorabies virus attachment, fusion, and entry., 2023, 97(2): e0187122.
[82] Xu K, Zhou YR, Mu YL, Liu ZG, Hou SH, Xiong YJ, Fang LR, Ge CL, Wei YH, Zhang XL, Xu CJ, Che JJ, Fan ZY, Xiang GM, Guo JK, Shang HT, Li H, Xiao SB, Li JL, Li K. CD163 and pAPN double-knockout pigs are resistant to PRRSV and TGEV and exhibit decreased susceptibility to PDCoV while maintaining normal production performance., 2020, 9: e57132.
[83] Menchaca A. Sustainable food production: the contribution of genome editing in livestock., 2021, 13(12): 6788.
[84] Hennig SL, Owen JR, Lin JC, McNabb BR, Van Eenennaam AL, Murray JD. A deletion at the polled PClocus alone is not sufficient to cause a polled phenotype in cattle., 2022, 12(1): 2067.
[85] Flórez JM, Martins K, Solin S, Bostrom JR, Rodríguez- Villamil P, Ongaratto F, Larson SA, Ganbaatar U, Coutts AW, Kern D, Murphy TW, Kim ES, Carlson DF, Huisman A, Sonstegard TS, Lents CA. CRISPR/Cas9- editing of KISS1 to generate pigs with hypogonadotropic hypogonadism as a castration free trait., 2022, 13: 1078991.
[86] Hansen PJ. Prospects for gene introgression or gene editing as a strategy for reduction of the impact of heat stress on production and reproduction in cattle., 2020, 154: 190–202.
Progress on CRISPR/Cas9 system in the genetic improvement of livestock and poultry
Yanchun Bao1,2, Lingli Dai2,3, Zaixia Liu1,2, Fengying Ma1,2, Yu Wang4, Yongbin Liu5, Mingjuan Gu1,2, Risu Na1,2, Wenguang Zhang1,2,6
CRISPR/Cas9 gene editing technology, as a highly efficient genome editing method, has been extensively employed in the realm of animal husbandry for genetic improvement. With its remarkable efficiency and precision, this technology has revolutionized the field of animal husbandry. Currently, CRISPR/Cas9-based gene knockout, gene knock-in and gene modification techniques are widely employed to achieve precise enhancements in crucial production traits of livestock and poultry species. In this review, we summarize the operational principle and development history of CRISPR/Cas9 technology. Additionally, we highlight the research advancements utilizing this technology in muscle growth and development, fiber growth, milk quality composition, disease resistance breeding, and animal welfare within the livestock and poultry sectors. Our aim is to provide a more comprehensive understanding of the application of CRISPR/Cas9 technology in gene editing for livestock and poultry.
CRISPR/Cas9; gene editing; livestock and poultry; genetic improvement;economic traits
2024-01-17;
2024-02-25;
2024-02-29
内蒙古自治区自然科学基金项目(编号:2021ZD05),内蒙古自治区科技计划项目(编号:2021GG0008),内蒙古自治区科技重大专项项目(编号:2021ZD0009),内蒙古农业大学动物科学学院高水平成果培育专项(编号:BZX202201)和内蒙古自治区直属高校基本科研业务费(编号:BR221024)资助[Supported by the Natural Science Foundation of Inner Mongolia Autonomous Region (No. 2021ZD05), the Science and Technology Plan Project of Inner Mongolia Autonomous Region (No. 2021GG0008), the Major Science and Technology Project of Inner Mongolia Autonomous Region (No. 2021ZD0009), the High Level Achievement Cultivation Project of College of Animal Science of Inner Mongolia Agricultural University (No. BZX202201) and the Basic Research Expenses of Universities Directly of Inner Mongolia Autonomous Region (No. BR221024)]
鲍艳春,博士研究生,专业方向:动物遗传育种与繁殖。E-mail: byc107054@163.com
谷明娟,博士,讲师,研究方向:肉牛分子育种。E-mail: gmj0119@yeah.net
娜日苏,博士,副教授,研究方向:牛羊遗传育种与繁殖。E-mail: narisu@swu.edu.cn
张文广,博士,教授,研究方向:数量基因组学与生物信息学。E-mail: actgnmbi@aliyun.com
10.16288/j.yczz.24-021
(责任编委: 姜雨)