陆地棉GhRTE基因家族的鉴定与耐盐性分析
徐凯祥 焦玚 王帅帅 陈全家 郑凯


摘要:RTE(reversion-to-ethylene)是一种植物乙烯响应的负调节因子,参与植物生长发育并在非生物胁迫的逆境中起着重要作用。通过对陆地棉(Gossypium hirsutum)RTE家族成员的鉴定及其在盐胁迫下的表达模式进行解析,为进一步探索棉花RTE家族成员的功能提供一定的研究指导。通过棉花全基因组数据,鉴定棉花RTE家族成员,以陆地棉为主要研究对象,结合海岛棉(Gossypium barbadense)、亚洲棉(Gossypium arboreum)、雷蒙德氏棉(Gossypium raimondii)等植物的RTE基因家族成员进行物种内的进化分析。同时,利用转录组学方法,研究陆地棉RTE家族基因的表达量及其对盐胁迫的应答规律。结果表明,棉花RTE蛋白家族共鉴定出24个RTE基因,其中来自陆地棉和海岛棉的各有8个,有4个来自雷蒙德氏棉,4个来自亚洲棉。系统发育树分析将24个RTE基因分成3个亚族,GhRTE基因同一亚族成员间具有相似的基因结构和保守基序。GhRTE启动子区包含了许多与植物激素反应、生长发育和逆境应答密切相关的顺式调控因子。通过对转录组数据分析以及荧光定量分析,初步验证该家族GhRTE6基因可能在棉花生长发育以及盐胁迫响应中发挥作用,可为阐明GhRTE基因耐盐的分子机制提供理论依据。
关键词:陆地棉;RTE基因家族;非生物胁迫;表达特性分析;耐盐性
中图分类号:S562.01 文献标志码:A
文章编号:1002-1302(2024)06-0052-09
收稿日期:2023-05-29
基金项目:新疆维吾尔自治区重大科技专项(编号:2022A03004-2)。
作者简介:徐凯祥(1995—),男,甘肃兰州人,硕士研究生,研究方向为棉花耐盐性。E-mail:965762115@qq.com。
通信作者:陈全家,博士,教授,博士生导师,从事棉花遗传育种研究,E-mail:chenqjia@126.com;郑凯,博士,副教授,从事棉花种质资源优异基因挖掘和分子育種研究,E-mail:zhengkai555@126.com。
乙烯是人类迄今发现的唯一一种植物气体激素,它在植物的生长发育过程中起着十分关键的作用。乙烯与受体结合后,可观察到对种子萌发、干细胞分裂、细胞伸长和分化、根毛生长、幼苗、性别决定、果实成熟、果实衰老、果实脱落以及对盐、干旱、洪水、低温胁迫等方面的各种反应及影响[1]。RTE是植物乙烯反应的负调节因子,参与植物生长发育并在非生物胁迫的逆境中起着重要作用。RTE基因在高等真核生物中广泛表达,而在低等真核生物和原核生物中不表达。前人通过遗传学分析及转基因技术,对拟南芥中RTE的功能进行了进一步研究,结果表明,RTE基因的过表达导致了乙烯的抗性;然而,当乙烯受体ETR(ethylene-response)基因被敲除,该基因过表达后,植株就会丧失对乙烯的抗性,这表明乙烯受体与RTE之间存在着紧密的相互作用。通过构建RTE和受体功能缺失的多突变体,发现RTE可能在乙烯信号通路中起负调控作用,并且RTE可能对ETR起正调控作用[2]。然而在陆地棉中,关于棉花RTE基因家族成员相关的生物信息及功能特性鲜有报道。
棉花是一种重要的纤维作物,也是一种以商业规模将纤维转化为织物的经济作物[3]。盐胁迫对植物生长发育的影响贯穿于植物个体发育的整个过程,但幼苗期被认为是最脆弱的时期之一[4-5]。此外,虽然棉花被认为是中等耐盐的,临界值为7.7 dS/m,但其生长在苗期受到严重影响,从而降低了产量[6]。新疆是我国最大的棉产地,2022年,新疆棉花产量539.1 万t,占全国总量的90.2%,比2021年增加26.2 万t,增长5.1%[7],新疆棉花生产能够直接影响我国的棉花生产。据有关数据统计,新疆盐碱土地面积约占我国盐碱土地总面积的22.01%,达到 2 181.4 万hm2。在新疆地区407 万hm2耕地中,受盐渍化影响的耕地达122.88 万hm2,极大程度影响了棉花的种植[8-9]。提高棉花的耐盐性是改善土壤盐碱化的必要方法。
1 材料与方法
1.1 植物材料
本研究所用陆地棉耐盐材料CQJ-5和敏盐材料新陆早49号(XLZ-49)均由新疆作物遗传改良与种质创新重点实验室提供[10-11]。将形状饱满的棉种播种于装满蛭石与花土体积比1 ∶1混合的营养钵中,每个营养钵放入3~4粒种子,于培养室中进行培养,温度为 25 ℃,光、暗周期为12 h光照、12 h黑暗。待长到3叶1心期,选取真叶生长一致的幼苗,展开盐胁迫处理,将幼苗根系浸入150 mmol/L NaCl溶液中,分别于0 、3 、6 、12 、24、48 h后,取棉花幼苗的根、茎、叶部用于分析GhRTE6基因在盐胁迫条件下的表达水平。以上样品选取后迅速用液氮冷冻,贮藏于 -80 ℃ 冰箱备用[12]。
1.2 棉花RTE家族基因鉴定与蛋白理化性质分析
本试验首先从CottonGen数据库(https://www.cottongen.org/)中获取棉花的全基因组和蛋白组学数据,并下载GFF3注释文件[13]。以从NCBI-CDD数据库获取的RTE蛋白保守结构域(PF05608)为切入点,借助于隐马尔可夫模型,并基于Hmmsearch搜索结果,通过本地数据库BLAST比对,获取棉花全基因组RTE蛋白质的序列信息,并对其进行生物信息学分析,进而对已知的RTE蛋白质进行功能分析。通过ExPASy网站上的ProtParam在线工具,分析棉花RTE基因编码的蛋白质序列的分子量(MW)、等电点(pI)等物理化学性质,并通过WoLF PSORT(https://wolfpsort.hgc.jp/)进行亚细胞定位[14-15]。
1.3 进化树、同源性分析
采用MEGA-X软件对陆地棉、海岛棉、亚洲棉、雷蒙德氏棉等棉花品种的RTE蛋白质序列进行多序列比对,并建立进化树[16]。用名为Evolview v3的在线网站对其进行美化[17]。根据棉花的基因组和染色体数据库CottonFGD,利用TBtools软件显示出RTE蛋白家族成员在棉花染色体上的同源性[18]。通过CottonGen数据库、BLAST软件、TBtools软件和MCScanX等工具,对在陆地棉内部A、D基因亚组之间以及四大棉种之间的RTE蛋白家族同源性,进行了比对和共线性分析,并通过TBtools进行可视化分析后用Adobe Illustrator CS6进行美化修饰[19-22]。
1.4 染色体定位及基因结构分析
借助TBtools软件将GhRTE基因染色体的定位信息从已下载的GFF3注释文件中提取出来,之后进行可视化。利用MEME程序分析RTE家族成员的Motif基序(最大发现数设置为15)[23],并使用TBtools软件对其进行可视化分析。
1.5 顺式作用元件预测
利用TBtools软件,从已下载的基因组文件中提取陆地棉RTE基因起始密码子上游2 000 bp序列,将这些序列视为启动子区,并利用PlantCARE数据库(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)对其进行分析,以此来预测GhRTE基因上游启动子区可能存在的顺式作用元件[15,24]。
1.6 陆地棉RTE基因RNA-seq表达模式分析
从NCBI SRA网站(https://www.ncbi.nlm.nih.gov/sra)下载陆地棉各器官的转录组数据(根、茎、叶、雌蕊、雄蕊、花萼、花瓣和花托)和非生物胁迫处理的转录组数據(基因组测序计划编号为PRJNA248163)。参考基因组文件TM-1_genome_ZJU_v2.1,对原始数据进行转录组标准分析,获得表达数据[25-26]。表达数据标准化为lg(TPM+1),通过TBtools软件绘制表达量图。本研究的基因结构图、顺式作用元素图和热图均由TBtools软件完成,然后用Adobe Illustrator CS6进行美化和调整。
1.7 RNA提取及qRT-PCR分析
利用CottonFGD数据库下载陆地棉RTE基因编码序列(CDS)信息,利用DNAMan (Lynnon Corporation,Canada)软件实现对相关基因特异性荧光定量引物的设计。根据生产商提供的说明书,使用RNAprep Pure多糖多酚的总RNA提取试剂盒[天根生化科技(北京)有限公司]从幼苗中提取总RNA,并使用abm反转录试剂盒进行合成,以获得第1条cDNA。通常选择棉花Ubiquitin7(UBQ7)作为内参基因,利用罗氏Light Cycler荧光定量PCR仪,对在不同部位各个胁迫处理时期的RTE基因进行 qRT-PCR扩增(3次技术重复),相关基因相对表达量分析采用2-ΔΔCT法[27]。最后选择GraphPad Prism version 9.0.0 for Windows对数据进行可视化处理,完成后用Adobe Illustrator CS6进行美化调整。
2 结果与分析
2.1 棉花RTE基因的鉴定和分析
通过本地BLAST数据库和NCBI-CDD数据库对棉花RTE蛋白家族进行筛选验证,共鉴定出24个RTE基因,其中陆地棉和海岛棉均包含8个基因,雷蒙德氏棉4个、亚洲棉4个(表1)。如表1所示,陆地棉RTE基因编码区长度范围为684~762 bp,氨基酸长度范围在227~253 aa之间,基因编码的蛋白质分子量范围为25.84~28.62 ku,等电点(pI)介于5.86~7.09之间;海岛棉RTE基因编码区长度为 468~762 bp,氨基酸长度在155~253 aa之间,基因编码的蛋白质分子量范围为17.92~28.64 ku,等电点介于4.99~7.09之间;雷蒙德氏棉RTE基因编码区长度为588~762 bp,氨基酸长度在195~253 aa 之间,基因编码的蛋白质分子量范围为22.38~28.57 ku,等电点介于4.94~7.09之间;亚洲棉RTE基因编码区长度为684~762 bp,氨基酸长度在227~253 aa之间,基因编码的蛋白质分子量范围 25.83~28.54 ku,等电点介于5.27~7.01之间。预测亚细胞的定位结果显示,棉花中的24个RTE编码蛋白基本都位于细胞质或质膜中。RTE基因家族成员的理化性质和亚细胞定位结果大致相同,这也预示该基因家族在棉花生长发育信号传递过程中扮演着重要角色。
2.2 棉花RTE基因染色体定位与命名
将棉花RTE基因进行染色体结构定位分析,结果如图1所示,4个棉花品种RTE基因在染色体上呈不均匀分布。陆地棉和海岛棉的A01、A02、A03、A04、A05、A06、A09、A12、A13、D01、D02、D03、D04、D05、D06、D09、D12、D13染色体均无RTE基因分布,A07、A08、A10、A11、D07、D08、D10和D11染色体各有1个基因分布。亚洲棉中Chr07、Chr08、Chr10和Chr11染色体各有1个基因分布,其他染色体上均无RTE基因。雷蒙德氏棉中Chr01、Chr04、Chr07和Chr11染色体各有1个基因,其他染色体上均无RTE基因。陆地棉所含的RTE基因分布于8条不同的染色体上,其中A亚组染色体上有4个,D亚组染色体上有4个,海岛棉与之相同。雷蒙德氏棉4个RTE基因分布于4条不同的染色体上,亚洲棉与之相同。说明该基因家族在进化过程中比较稳定。之后根据基因在染色体上的排列位置,将它们命名为GhRTE1~GhRTE8、GbRTE1~GbRTE8、GaRTE1~GaRTE4、GrRTE1~GrRTE4。
2.3 棉花RTE基因家族系统进化分析
采用邻接法对棉花RTE基因进行聚类,并建立系统发育树。从图2可以看出,24个RTE基因可以被划分为3个亚族,分别用Group1、Group2和Group3表示。Group1成员最多有12个,其中陆地棉和海岛棉均有4个,雷蒙德氏棉和亚洲棉均有2个。其次,Group2和Group3成员个数相同,均为6个,Group2中陆地棉和海岛棉成员均有2个,雷蒙德氏棉和亚洲棉成员均有1个。Group3中陆地棉和海岛棉成员均有2个,雷蒙德氏棉和亚洲棉均有1个。结合图1可以发现,陆地棉和海岛棉在染色体相同位置上的RTE基因都紧密地聚集在了同一个亚族中,另外,每一个亚族中,与雷蒙德氏棉、亚洲棉相比,陆地棉、海岛棉所含的基因数量均是它们的2倍,这与棉属的演化规律相吻合。
2.4 陆地棉RTE基因同源性分析
同源是指在演化过程中具有相同祖先血统的支系间的演化关系。在不同的物种中,源自于同一个祖先的基因被叫作直系同源基因,而在相同的物种中,因为基因复制而分离出来的同源基因就是旁系同源基因[28]。为了解棉花RTE基因家族的同源进化关系,对陆地棉、海岛棉、亚洲棉以及雷蒙德氏棉进行分析,同时也可以了解到8个GhRTE在二倍体棉和四倍体棉中的进化联系。陆地棉各染色体之间有4对直系同源基因,A亚组和D亚组出现了加倍复制,并且不同亚组之间有更紧密的进化关系(图3)。陆地棉与亚洲棉之间有12对直系同源基因,与雷蒙德氏棉之间有12对直系同源基因,与海岛棉之间有8对直系同源基因(图4),说明彼此之间具有极密切的进化关系,且这些基因在不同棉花中可能具有相同的功能。
2.5 陆地棉RTE蛋白保守基序及对应的基因结构分析
本研究分析了陆地棉中8个RTE蛋白的序列特征,发现其结构域保守程度较高,将从RTE蛋白序列中找到的12个保守基序依次命名为Motif1~Motif12,8个GhRTE蛋白均含有Motif1~Motif7和Motif9(图5-A)。Motif11基序只存在于GhRTE1与GhRTE5中。Motif10基序只存在于GhRTE2与GhRTE6中。Motif12基序只存在于GhRTE3与GhRTE7中。而GhRTE1、GhRTE5、GhRTE2和GhRTE6中均含有Motif8基序。GhRTE家族8个基因均有2个外显子(图5-B)。从基因长度方面来看,具有不同长度的基因,其编码序列是相似的。这表明,在进化的过程中,RTE基因的内含子长度
发生了较大的改变,这就有可能导致不同的功能。
2.6 陆地棉RTE基因家族的启动子分析
对陆地棉启动子的顺式元件进行分析,可为研究基因的组织特异性或应激性表达模式提供思路和帮助。由图6可知,在8个陆地棉RTE基因中鉴定到的顺式作用元件中,有41个与激素有关,例如水杨酸响应元件(TCA-element,SARE)、脫落酸响应元件(ABRE)、赤霉素响应元件(GARE-motif,TATC-box,P-box)、生长素响应元件(TGA-element,AuxRR-core)、茉莉酸甲酯(MeJA)响应元件(TGACG-motif,CGTCA-motif)等[29]。此外,还发现了20个逆境应答元件,如低温反应响应元件(LTR)、防御和应激响应元件(TC-rich repeats)、干旱胁迫响应元件(MBS),以及1个类黄酮合成响应元件(MBSI)。8个GhRTE基因含有4~10个不同的顺式作用元件,其中以脱落酸和茉莉酸酯相关的顺式作用元件的含量最高。据研究,脱落酸与植株抗逆性显著相关,茉莉酸甲酯与植物生长发育以及抗逆性相关。其中GhRTE1、GhRTE4和GhRTE5基因所含脱落酸、茉莉酸甲酯顺式元件数量为3个,GhRTE6和GhRTE8基因所含脱落酸、茉莉酸甲酯顺式元件数量为4个。说明陆地棉RTE基因家族可能在植物生长发育以及在非生物胁迫逆境中发挥着较强作用。
2.7 陆地棉RTE基因家族的表达分析
为了明确RTE家族基因在陆地棉中的组织表达特异性,以及它们对盐胁迫的响应规律,利用转录组数据,借助TBtools软件对8个GhRTE基因在棉花根、茎、叶、花瓣、花托、花萼等组织进行了特异性表达分析。在正常的生长环境下,这些基因在陆地棉的不同部位表现出了不同的表达模式:GhRTE1、GhRTE2、GhRTE4、GhRTE5、GhRTE6和GhRTE8基因在不同组织中的表达有很大的差异,并且它们的表达量比较高,而其他基因的表达量则比较低,甚至不表达,暗示这些基因可能在陆地棉的生长发育中起重要作用(图7-A)。同时分析在不同时期盐胁迫下的陆地棉RTE家族基因表达情况,发现GhRTE2、GhRTE5和GhRTE6在盐胁迫下的各个时间阶段中表现出高表达趋势(图7-B),且在花托和花萼这类营养器官中的表达量高于其他组织。由此可推测出GhRTE2、GhRTE5和GhRTE6可能在棉花的生长发育阶段以及在非生物胁迫响应中发挥重要作用。
2.8 qRT-PCR分析
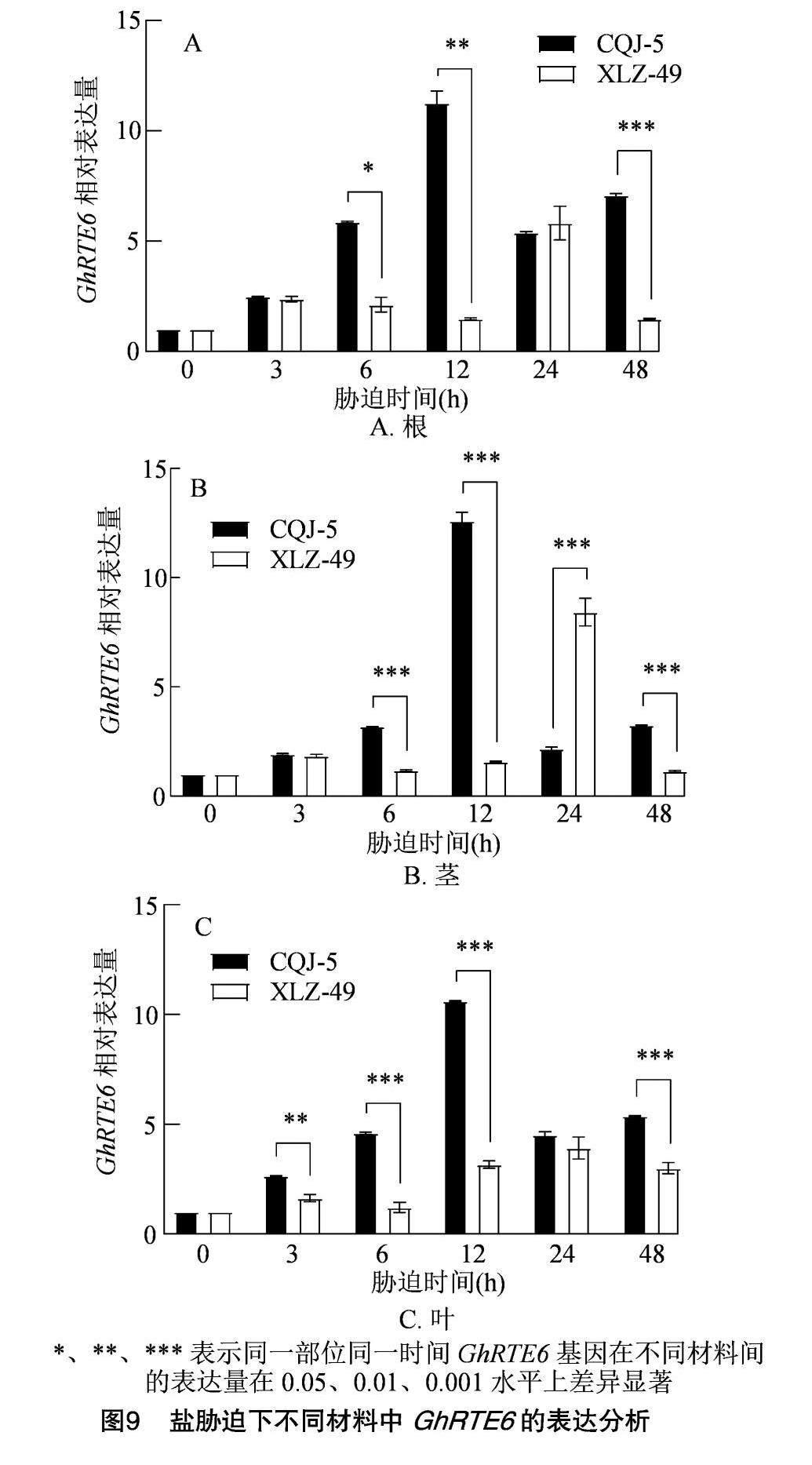
通过启动子区相关顺式作用元件(图6)以及转录组表达(图7)分析结果,选取盐胁迫下表达变化明显且抗逆性相关顺式作用元件交集最多的GhRTE基因,最终挑选GhRTE6基因进行进一步研究。分别对耐盐棉花材料CQJ-5以及敏盐材料新陆早49号进行72 h盐胁迫处理(图8)。根据GhRTE6基因序列设计引物(表2)并利用qRT-PCR分析发现,耐盐材料CQJ-5经盐胁迫处理 12 h 后,GhRTE6基因在茎部的表达量较高,而在叶片和根部的表达量则相对较低;敏盐材料新陆早49在胁迫后24 h时,在茎中表达水平较高,而在根和叶中表达水平相对较低(图9)。从总体观察发现,GhRTE6基因在耐盐材料中的表达量高于敏盐材料。
3 讨论与结论
RTE基因在多种植物中的功能分析已见报道,但关于RTE基因家族分析研究却极少,在棉花中从未有过基因家族鉴定及功能分析。本研究鉴定出陆地棉8个、海岛棉8个、雷蒙德氏棉4个和亚洲棉4个,共24个棉花RTE家族成员,进行进化关系解析将其分为3个亚族,每个亚族中的陆地棉和海岛棉在染色体相同位置上的RTE基因都紧密地聚集在了一起,而且每个亚组中陆地棉和海岛棉的基因数量都是雷蒙德氏棉和亚洲棉的2倍左右,符合棉花物种的进化关系。本研究中棉花RTE基因家族成员编码的蛋白通过亚细胞定位预测多数位于质膜上,少数位于细胞质中,这与前人的研究结果[2]较为一致。
前人的研究表明,RTE家族包括2个类群:RTE1组和RTH(RTE1-homolog)组[23,30-32]。RTE1可以正調控乙烯受体ETR1,并通过ETR1的N端跨膜域对乙烯信号进行调控,番茄中的GR(green-ripe)和GRL1(green-ripe like 1)是拟南芥中的AtRTE1的同源基因,对RTE1/GR/GRL1基因进行过表达,可以提高乙烯受体活性,进而降低植株对乙烯的敏感性[31-33]。拟南芥中的AtRTE1也可以结合生长素调节基因(ARGOS)参与调控器官大小,它的保守的TPT区使其对乙烯的敏感性下降[33-34],ARGOS基因的过表达能够提高拟南芥和玉米的耐旱性。本研究通过顺式作用元件预测得出陆地棉RTE基因家族启动子区包含多个生长发育激素以及逆境响应调控元件,其中GhRTE1、GhRTE4、GhRTE5、GhRTE6和GhRTE8基因所含脱落酸、茉莉酸甲酯顺势元件数量最多,且GhRTE1、GhRTE4和GhRTE5基因含有抵御胁迫相关的顺式作用元件,说明RTE基因家族可能也参与了棉花的生长发育并调控其他酶类合成以应对逆境胁迫。
对RTE家族中不同组织及逆境条件下不同基因表达量分析发现,GhRTE1、GhRTE2、GhRTE4、GhRTE5、GhRTE6和GhRTE8基因的表达量明显高于其他基因,表明它们在棉花生长发育中起着重要的作用。同时分析在不同时期盐胁迫下的陆地棉RTE家族基因表达情况,结果表明,GhRTE2、GhRTE5和GhRTE6在不同的盐分胁迫时期表现出了较高的表达趋势,在12~48 h之间表达量较高,其余基因受非生物胁迫后表达量低甚至不表达。GhRTE6基因在棉花受到盐胁迫处理下,随着胁迫时间的延长,在不同的陆地棉材料中做出了显著性应答。在耐盐材料中,GhRTE6基因在胁迫12 h后表达量较高,而在敏盐材料中GhRTE6在胁迫24 h后表达量较高,且都在茎中高表达,这说明GhRTE6在敏盐材料中对于盐胁迫的响应比耐盐材料中慢,证明GhRTE6对盐胁迫具有一定的调控作用。由此可推测出GhRTE6可作为棉花响应非生物胁迫的关键基因进一步研究其功能特性。
本研究从陆地棉、海岛棉、雷蒙德氏棉以及亚洲棉等4个棉花品种中,发现了24个RTE基因家族成员,并在进化树上将它们划分为3个亚族,依据同源性关系可推断出陆地棉与海岛棉、亚洲棉以及雷蒙德氏棉之间的进化关系,发现同族成员中个别基因保守基序不同且相似长度基因其外显子的数量和长度也有所不同,故其功能可能存在一定的差异。通过顺式作用元件分析发现,陆地棉RTE基因启动子区含有多个生长发育激素和逆境响应元件,说明其可能在植物生长发育及抵御非生物胁迫逆境中发挥着较大作用。本研究通过转录组表达模式分析发现,陆地棉RTE基因在不同组织和非生物胁迫下的表达量存在差异,并且qRT-PCR结果证明GhRTE6对盐胁迫具有一定的调控作用。本研究结果可为今后探索棉花RTE基因的功能及分子机制提供参考。
参考文献:
[1]李建,黄琳丽. 乙烯在植物抗性反应中的作用[J]. 生物化工,2020,6(6):140-142.
[2]周欣,张杰,文光. RTE1:拟南芥乙烯受体ETR1的调节子[C]//2006年中国植物逆境生理生态与分子生物学学术研讨会论文摘要汇编.上海:中国植物生理学会,2006.
[3]Lacape J M,Nguyen T B,Courtois B,et al. QTL analysis of cotton fiber quality using multiple Gossypium hirsutum×Gossypium barbadense backcross generations[J]. Crop Science,2005,45(1):123-140.
[4]Chartzoulakis K,Klapaki G. Response of two greenhouse pepper hybrids to NaCl salinity during different growth stages[J]. Scientia Horticulturae,2000,86(3):247-260.
[5]张超,马晓丽,卢晓峰,等. 盐分胁迫下土施甲哌钅翁对棉苗叶片生理和根系形态的影响[J]. 江苏农业科学,2022,50(22):81-86.
[6]Sexton P D,Gerard C J. Emergence force of cotton seedlings as influenced by salinity[J]. Agronomy Journal,1982,74(4):699-702.
[7]熊丽. 2022年全国棉花产量增长4.3%[N]. 经济日报,2022-12-27(4).
[8]石婧,黄超,刘娟,等. 脱硫石膏不同施用量对新疆盐碱土壤改良效果及作物产量的影响[J]. 环境工程学报,2018,12(6):1800-1807.
[9]王俊铎,黎玉华,龚照龙,等. 棉花耐盐性研究进展[J]. 棉花科学,2021,43(5):3-10.
[10]范蓉,郭亚萍,赵柯柯,等. 旱、盐胁迫下棉花3个转录因子基因的表达与生理指标相关性分析[J]. 生物学杂志,2021,38(4):86-91.
[11]郑巨云,曾辉,王俊铎,等. 陆地棉品种资源萌发期耐盐性的隶属函数法评价[J]. 新疆农业科学,2018,55(9):1579-1592.
[12]王亦学,董艳辉,张欢欢,等. 陆地棉胁迫相关蛋白基因GhSAP8的克隆及其耐盐性分析[J]. 农业生物技术学报,2020,28(7):1230-1239.
[13]Zhu T,Liang C Z,Meng Z G,et al. CottonFGD:an integrated functional genomics database for cotton[J]. BMC Plant Biology,2017,17(1):101-113.
[14]Wilkins M R,Gasteiger E,Bairoch A,et al. Protein identification and analysis tools in the ExPASy server[J]. Methods in Molecular Biology,1999,112:531-552.
[15]陈琴,李多露,赵杰银,等. 陆地棉UDPGP基因家族的鉴定及抗旱性分析[J]. 棉花学报,2022,34(1):12-22.
[16]Kumar S,Stecher G,Li M,et al. MEGA X:molecular evolutionary genetics analysis across computing platforms[J]. Molecular Biology and Evolution,2018,35(6):1547-1549.
[17]Subramanian B,Gao S H,Lercher M J,et al. Evolview v3:a webserver for visualization,annotation,and management of phylogenetic trees[J]. Nucleic Acids Research,2019,47(W1):W270-W275.
[18]Chen C J,Chen H,Zhang Y,et al. TBtools:an integrative toolkit developed for interactive analyses of big biological data[J]. Molecular Plant,2020,13(8):1194-1202.
[19]Yu J,Jung S,Cheng C H,et al. CottonGen:a genomics,genetics and breeding database for cotton research[J]. Nucleic Acids Research,2014,42(D1):D1229-D1236.
[20]ODriscoll A,Belogrudov V,Carroll J,et al. HBLAST:parallelised sequence similarity:a Hadoop MapReducable basic local alignment search tool[J]. Journal of Biomedical Informatics,2015,54:58-64.
[21]Wang Y P,Tang H B,Debarry J D,et al. MCScanX:a toolkit for detection and evolutionary analysis of gene synteny and collinearity[J]. Nucleic Acids Research,2012,40(7):e49.
[22]陳琴,李多露,高文举,等. 陆地棉APX基因家族鉴定及抗旱性分析[J]. 农业生物技术学报,2021,29(10):1894-1903.
[23]Bailey T L,Boden M,Buske F A,et al. MEME SUITE:tools for motif discovery and searching[J]. Nucleic Acids Research,2009,37:W202-W208.
[24]Lescot M,Déhais P,Thijs G,et al. PlantCARE,a database of plant cis-acting regulatory elements and a portal to tools for in silico analysis of promoter sequences[J]. Nucleic Acids Research,2002,30(1):325-327.
[25]Zhang T Z,Hu Y,Jiang W K,et al. Sequencing of allotetraploid cotton (Gossypium hirsutum L. acc. TM-1) provides a resource for fiber improvement[J]. Nature Biotechnology,2015,33(5):531-537.
[26]Livak K J,Schmittgen T D.Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCTmethod[J]. Methods,2001,25(4):402-408.
[27]張岚,程琦,梁士辰,等. 棉花UGPase基因鉴定与生物信息学分析[J]. 棉花学报,2021,33(4):337-346.
[28]Tan Y Y,Liu J X,Huang F,et al. PhGRL2 protein,interacting with PhACO1,is involved in flower senescence in the petunia[J]. Molecular Plant,2014,7(8):1384-1387.
[29]张朵朵,林丽梅,国红玉,等. 人参与三七鲨烯环氧酶家族的适应性进化[J]. 华北理工大学学报(自然科学版),2022,44(1):98-106.
[30]Dong C H,Jang M,Scharein B,et al. Molecular association of the Arabidopsis ETR1 ethylene receptor and a regulator of ethylene signaling,RTE1[J]. The Journal of Biological Chemistry,2010,285(52):40706-40713.
[31]Barry C S,Giovannoni J J. Ripening in the tomato Green-ripe mutant is inhibited by ectopic expression of a protein that disrupts ethylene signaling[J]. Proceedings of the National Academy of Sciences of the United States of America,2006,103(20):7923-7928.
[32]Dong C H,Rivarola M,Resnick J S,et al. Subcellular co-localization of Arabidopsis RTE1 and ETR1 supports a regulatory role for RTE1 in ETR1 ethylene signaling[J]. The Plant Journal,2008,53(2):275-286.
[33]Shi J R,Drummond B J,Wang H Y,et al. Maize and Arabidopsis ARGOS proteins interact with ethylene receptor signaling complex,supporting a regulatory role for ARGOS in ethylene signal transduction[J]. Plant Physiology,2016,171(4):2783-2797.
[34]Shi J R,Habben J E,Archibald R L,et al. Overexpression of ARGOS genes modifies plant sensitivity to ethylene,leading to improved drought tolerance in both Arabidopsis and maize[J]. Plant Physiology,2015,169(1):266-282.

