蛇足石杉赖氨酸脱羧酶基因的克隆、原核表达及其功能分析
杜次,李菁,唐云涛,彭清忠
1 吉首大学生物资源与环境科学学院,湖南 吉首 416000
2 植物资源保护与利用湖南省高校重点实验室,湖南 吉首 416000
蛇足石杉赖氨酸脱羧酶基因的克隆、原核表达及其功能分析
杜次1,2,李菁1,2,唐云涛1,2,彭清忠1,2
1 吉首大学生物资源与环境科学学院,湖南 吉首 416000
2 植物资源保护与利用湖南省高校重点实验室,湖南 吉首 416000
赖氨酸脱羧酶 (Lysine decarboxylase, LDC) 是抗老年痴呆药——石杉碱甲生物合成的第一个酶。为了研究蛇足石杉中LDC的特性和功能,以其总RNA为模板,通过RT-PCR扩增得到2个赖氨酸脱羧酶基因LDC1和LDC2,克隆至pMD®19-T中测序发现,两基因同源性为95.3%,分别编码212和202个氨基酸。将两基因引入pET-32a(+)构建重组表达质粒pET-32a(+)/LDC1和pET-32a(+)/LDC2,分别转入BL21(ED3)中进行诱导表达,在30 ℃条件下获得可溶性表达产物Trx-LDC1和Trx-LDC2;采用Ni-NTA亲和层析法纯化目的蛋白,建立酶促反应体系分析其脱羧酶活性,薄层层析 (TLC) 检测表明重组融合蛋白Trx-LDC1和Trx-LDC2均能催化赖氨酸脱羧生成尸胺。利用生物信息学软件分析发现LDC1和LDC2理化性质存在差异,但预测的二级结构和三维结构基本一致。
蛇足石杉,赖氨酸脱羧酶,基因克隆,原核表达,酶活性,结构预测
蛇足石杉[Huperzia serrata (Thumb.) Trev.]为石杉科石杉属蕨类植物,别名千层塔、金不换、蛇足草等,系我国传统中草药,常用于治疗瘀血肿痛、跌打损伤、肌肉痉挛、精神分裂等疾病[1-2]。上世纪80年代我国科学家首次从蛇足石杉中分离出石杉碱甲 (Huperzine A),后经研究证明是一种高效、可逆、选择性强的乙酰胆碱酯酶抑制剂,治疗老年痴呆症 (Alzheimer's disease, AD) 疗效显著,而且具有毒性低、生物利用率高、易穿透血脑屏障、药效长等优点[2-4]。由于优于同类其他药物,石杉碱甲作为新一代治疗老年痴呆药物引起医药界的极度重视,而其药源植物蛇足石杉也受到国内外学者的广泛关注。
蛇足石杉生长缓慢、资源更新周期长,而石杉碱甲在全草中含量甚微,近些年由于提取石杉碱甲的高额利润,对野生蛇足石杉进行地毯式采收,使得该类植物资源日趋枯竭。一些学者尝试寻求新途径获取石杉碱甲,如化学合成法、组织培养法、微生物发酵法等[5-10],虽取得一些进展,但离生产实践有很远距离。解决资源短缺和实现新途径生产石杉碱甲,首先有必要从分子水平理解石杉碱甲的生物合成机制。目前一些研究表明,石杉碱甲生物合成是以赖氨酸脱羧酶 (Lysine decarboxylase, LDC)介导赖氨酸脱羧开始,接着由铜氨氧化酶(Copper amine oxidase) 催化尸胺生成四氢吡啶(2,3,4,5-Tetrahydropyridine),然后经过一系列的酶促反应生成石杉碱甲[3,11-12]。Sun等[13]曾对蛇足石杉中铜氨氧化酶基因进行克隆和功能鉴定,但是目前尚未见赖氨酸脱羧酶基因功能表征方面的研究报道,而关于石杉碱甲整个合成途径中的关键基因和调控机理也知之甚少。鉴于此,本研究拟采用RT-PCR方法从蛇足石杉中克隆赖氨酸脱羧基因,对其进行重组表达和功能分析,以及生物信息学分析,以期丰富赖氨酸脱羧酶家族分子信息,为阐明蛇足石杉中石杉碱甲生物合成机制提供参考数据。
1 材料与方法
1.1 材料
1.1.1菌株与质粒
大肠杆菌BL21(DE3)、大肠杆菌JM109,质粒pET-32a(+)为本实验室保藏;质粒pMD®19-T购自大连宝生物工程有限公司(TaKaRa)。
1.1.2酶和试剂
Ex Taq®DNA聚合酶、T4 DNA 连接酶、NcoⅠ和XhoⅠ限制性内切酶,DL2 000 DNA marker,Protein molecular weight marker (Low),Total RNA提取试剂 RNAiso Plus,PrimeScript®1st Strand cDNA Synthesis Kit,Agarose Gel DNA Fragment Recovery Kit Ver.2.0和MiniBEST DNA Fragment Purification Kit Ver.3.0均购自TaKaRa公司;Ni-NTA SefinoseTMResin Kit,IPTG和磷酸吡哆醛均购自上海生物工程有限公司;赖氨酸 (L-lysine) 和尸胺 (Cadaverine) 购自Sigma公司;其他试剂均为分析纯。
1.1.3材料
蛇足石杉样品采自湖南省古丈县高望界自然保护区,经吉首大学张代贵高级实验师鉴定为蛇足石杉。
1.2 方法
1.2.1总RNA提取及cDNA合成
野外采集蛇足石杉完整植株 (带土壤),运回实验室。从植株上剪取幼嫩叶片约100 mg,置于液氮预冷的研钵中迅速研磨成粉,立即使用RNAiso Plus试剂提取总RNA。采用1%的琼脂糖凝胶电泳检测RNA质量,并用分光光度计测定RNA浓度,提取的总RNA于–70 ℃保存备用。利用TaKaRa公司提供的PrimeScript反转录试剂盒,按其说明书操作流程,以蛇足石杉总RNA为模板反转录合成cDNA第一链。
1.2.2 LDC基因克隆与测序
从NCBI数据库中搜索赖氨酸脱羧酶相关基因进行同源比对,根据序列保守区域,采用vector NTI软件设计一对包含全长LDC基因的扩增引物LDC-F和LDC-R (表1),以cDNA第一链为模板扩增蛇足石杉的LDC基因片段。配制50 μL反应体系:10×Ex PCR缓冲液5.0 μL,dNTPs (2.5 mmol/L) 4.0 μL,引物 (LDC-F和LDC-R) 各1.0 μL,Ex Taq聚合酶0.25 μL,cDNA 4.0 μL,补加ddH2O至终体积50 μL;PCR反应程序:94 ℃预变性5 min;94 ℃变性30 s,52 ℃退火30 s,72 ℃延伸1 min,30个循环;72 ℃延伸10 min,4 ℃保存。1%琼脂糖凝胶电泳检测扩增结果。

表1 本研究所用引物序列Table 1 Primers used in this study
利用Agarose Gel DNA Fragment Recovery Kit Ver.2.0对PCR产物进行割胶纯化,电泳检测纯化结果,并用紫外分光光度计测定其浓度。按厂家说明书将纯化的DNA片段连接至载体pMD®19-T上,转化感受态细胞JM109,通过氨苄抗性和蓝白斑筛选,挑取阳性菌落进行PCR检测,从检测合格的重组菌株中提取质粒,送TaKaRa公司测序。
1.2.3 LDC基因的原核表达
根据LDC基因序列设计一对引物LDC-eF和LDC-eR (表1),分别在引物5'端引入NcoⅠ和XhoⅠ酶切位点。以1.2.2中的重组质粒为模板进行PCR扩增,纯化扩增产物,采用限制性内切酶NcoⅠ和XhoⅠ进行双酶切;同时对表达载体pET-32a(+)进行双酶切。采用MiniBEST DNA Fragment Purification Kit Ver.3.0对酶切产物进行纯化,分光光度计检测各酶切产物浓度。利用T4 DNA连接酶于4 ℃对两纯化产物进行连接,将连接产物转化BL21(DE3),在氨苄和氯霉素抗性平板上筛选转化子。从平板上挑取多个菌落进行全菌PCR检测,提取相应克隆子的重组质粒,并进行NcoⅠ和XhoⅠ双酶切鉴定。
将鉴定的阳性克隆子接种至LB液体培养基,于37 ℃摇床培养3−4 h (OD600约为1.0),添加IPTG (终浓度260 μg/mL),继续30 ℃恒温摇床培养 (150 r/min) 4 h。同时以空载体重组菌BL21(DE3)/pET-32a(+)作对照。取经过诱导表达的菌液于1.5 mL离心管中,离心收集菌体,用PBS洗涤和重悬菌体。重悬菌液分2份,其中一份加入2 μL溶菌酶 (100 mg/mL),室温静置10 min后反复冻融3次,12 000 r/min离心10 min,收集上清液和沉淀。分别在上清、沉淀、全菌 (另一份) 和对照组全菌中加入等体积的2×上样缓冲液,沸水浴3 min,短暂离心后于12%的SDS-PAGE中电泳检测。
1.2.4表达产物纯化及活性鉴定
将重组菌接种于1 L液体培养基中培养、诱导表达,收集菌体,PBS洗涤3次,使用Ni-NTA SefinoseTMResin蛋白纯化试剂盒中的结合缓冲液重悬菌体,溶菌酶酶解,冰浴超声破碎细胞,12 000 r/min离心收集上清液。按照试剂盒操作说明,采用Ni-NTA纯化柱进行重组蛋白纯化,12%的SDS-PAGE电泳检测纯化结果。
在1.5 mL离心管中配制500 µL酶反应体系,使各试剂终浓度分别为:磷酸钾缓冲液(pH 8.0) 100 mmol/L、磷酸吡哆醛 (PALP) 0.20 mmol/L、赖氨酸10.0 mmol/L、重组赖氨酸脱羧酶1.00 mg/mL。将反应混合液于37 ℃水浴锅中反应至出现浑浊,向反应液中加入1滴浓HCl混匀,然后加入500 µL氯仿剧烈振荡,10 000 r/min离心10 min弃去两相中间不溶物。氮吹仪40 ℃吹干,干燥后的两相残留物用1 mol/L的HCl溶解,取溶解后的上清用丹磺酰氯衍生剂进行衍生,同时对赖氨酸和尸胺标准品进行丹磺酰氯衍生,氯仿萃取浓缩衍生物进行薄层层析 (TLC) 检测,展开剂为氯仿、乙醚、三乙胺 (配比为8∶1∶2)。使用全自动点样仪(CAMAG ATS 4,瑞士) 点样,全自动展开仪(CAMAG ADC 2,瑞士) 进行展开,然后用TLC Visualizer进行检测成像。
1.2.5 LDC基因编码蛋白的生物信息学分析
LDC基因编码蛋白的理化性质预测采用ExPASy Bioinformatics Resource Portal 提供的在线工具Protparam (http://web.expasy.org/ protparam/);LDC蛋白质结构搜索分别采用在线工具PROSITE (http://prosite.expasy.org/) 和NCBI 在线工具 (http://www.ncbi.nlm.nih.gov/ structure/cdd/wrpsb.cgi/);蛋白质二级结构分析和三维建模采用SWISS-MODEL (http:// swissmodel.expasy.org/)。
2 结果与分析
2.1 LDC基因的克隆和序列分析
根据植物赖氨酸脱羧酶相关基因和EST同源序列设计保守引物,利用RT-PCR技术从蛇足石杉茎尖嫩叶总RNA中扩增获得2个基因片段,如图1所示,长度分别约为1 100 bp和650 bp。对扩增所得的2个基因片段进行克隆测序,采用Vector NTI 软件分析,发现两序列均包含有完整开放阅读框,长度分别为639 bp和609 bp,编码212个和202个氨基酸,两编码基因分别命名为LDC1和LDC2;同源比对分析发现,LDC1除在3'末端比LDC2多出30 bp小片段外,其余序列均完全一致,两者同源性为95.3%。将2个基因送GenBank注册,Accession number分别为JQ308624.1和JQ308625.1。
2.2 赖氨酸脱羧酶基因的原核表达
采用1.2.3中的引物进行PCR扩增时,分别在LDC基因起始密码部位引入NcoⅠ酶切位点,终止密码后引入XhoⅠ酶切位点。纯化的扩增产物经NcoⅠ和XhoⅠ双酶切、回收后,与经过同样双酶切的表达载体pET-32a(+)进行连接,即构建成重组质粒pET-32a(+)/LDC,构建过程如图2所示。
重组质粒pET-32a(+)/LDC转化BL21(DE3),通过PCR扩增和NcoⅠ/XhoⅠ双酶切筛选阳性克隆子。将鉴定正确的重组菌株接种于LB培养基中培养,经IPTG低温诱导表达发现,含pET-32a(+)/LDC1的重组菌株和含pET-32a(+)/LDC2的重组菌株分别产生Trx-LDC1和Trx-LDC2融合蛋白,如图3A所示。Trx-LDC1和Trx-LDC2融合蛋白大小约为40 kDa,在重组菌破碎后的上清液和沉淀部分均存在,其中沉淀部分含量较高,表明融合蛋白的可溶性比例较少。根据融合蛋白所带组氨酸标签,采用Ni-NTA柱对可溶性重组蛋白进行纯化,获得纯度较高的融合蛋白Trx-LDC1和Trx-LDC2,如图3B所示。
2.3 重组赖氨酸脱羧酶的活性鉴定
按照文献[14]和[15]方法分别建立Trx-LDC1和Trx-LDC2融合蛋白酶促反应体系,37 ℃进行催化反应后加入浓HCl终止反应,并通过氯仿萃取去除重组融合蛋白;两液相反应物干燥后与丹磺酰氯进行酰基化衍生反应,由于丹磺酰基具有强烈荧光,衍生物能利用薄层层析 (TLC) 进行检测,如图4所示,两融合蛋白反应体系中均有尸胺生成 (泳道2和3),而加热灭活的融合蛋白反应体系中未检测到尸胺(泳道4和5),据此,可以推测融合蛋白Trx-LDC1和Trx-LDC2具有赖氨酸脱羧酶活性,能催化赖氨酸脱羧生成尸胺。

图1 赖氨酸脱羧酶基因的RT-PCR扩增Fig. 1 RT-PCR amplification of LDC gene from Huperzia serrata. M: DNA marker; 1, 2: RT-PCR products of LDC gene; ct: control.

图2 重组质粒pET-32a(+)/LDC的构建Fig. 2 Construction of recombinant plasmid pET-32a(+)/LDC.

图3 重组蛋白Trx-LDC1和Trx-LDC2的SDS-PAGE电泳Fig. 3 SDS-PAGE analysis of recombinant Trx-LDC1 and Trx-LDC2. (A) Unpurified recombinant fusion proteins. (B) Purified recombinant fusion proteins. M: protein molecular weight markers; 1, 2 and 3: supernatant, precipitation and entire cell from BL21(DE3) transformants including pET32a(+)/LDC1 respectively; 4, 5 and 6: supernatant, precipitation and entire cell from BL21(DE3) transformants including pET32a(+)/LDC2 respectively; ct: recombinant cells only with pET-32a(+). LDC1: purified Trx-LDC1; LDC2: purified Trx-LDC2.
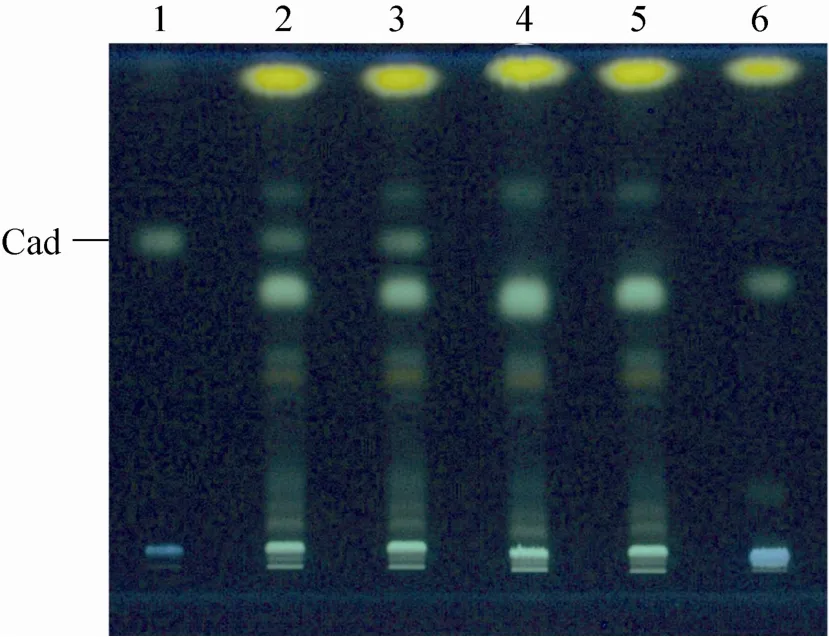
图4 重组蛋白Trx-LDC1和Trx-LDC2酶促反应的TLC检测Fig. 4 TLC detection of recombinant Trx-LDC1/ Trx-LDC2 enzyme reaction. 1: Cadaverine (Cad); 2, 3: products of Trx-LDC1/Trx-LDC2 enzyme reaction respectively; 4, 5: products of boiling-inactivated Trx-LDC1/Trx-LDC2 enzyme reaction respectively; 6: L-lysine.
2.4 LDC基因编码蛋白的理化性质和空间结构预测
2.4.1 LDC理化性质
LDC1基因预测编码212个氨基酸,LDC2编码202个氨基酸,根据ExPASy Bioinformatics Resource Portal 提供的在线工具Protparam对两基因编码蛋白进行分析,推测LDC1分子式为C1035H1650N274O302S10,分子量为23.08 kDa,等电点PI为5.59;带负电残基(Asp + Glu)为28,带正电残基(Arg + Lys)为23;LDC1的不稳定系数为31.96,脂肪系数为97.45,亲水系数为0.008。LDC2分子式为C985H1589N267O282S9,分子量为21.97 kDa,等电点PI为7.74;带负电残基(Asp + Glu)为24,带正电残基(Arg + Lys)为25;LDC2的不稳定系数为25.59,脂肪系数为100.35,亲水系数为0.013。结果表明两蛋白理化性质有一些差异。
2.4.2 LDC的二级结构和三维结构预测
通过在线软件对LDC1和LDC2二级结构进行预测,结果显示LDC1的二级结构主要由α-螺旋和β-折叠结构构成,分别占43.8%和34.4%,无规则卷曲占21.8%;同样LDC2的二级结构也主要由α-螺旋和β-折叠结构构成,分别占43.5%和34.2%,无规则卷曲占22.3%。由SWISS-MODEL在线分析软件对LDC1和LDC2进行三维结构模建,得到三维模型如图5所示。虽然LDC1比LDC2在C末端多出10个氨基酸,但从二级结构预测和三维结构模建可知,LDC1和LDC2的高级结构基本一致,从而表现出相同的生物活性。

图5 LDC1和LDC2的三维结构模建Fig. 5 Predicted three-dimensional structures of LDC1 and LDC2.
3 讨论
目前有关石杉碱甲生物合成途径、代谢调控机理及关键基因功能的研究资料非常匮乏。一些研究表明赖氨酸脱羧酶是参与石杉碱甲生物合成的第一个酶/入口酶[3,11-12],但是在蛇足石杉和其他石杉科植物中均未见编码该酶基因的研究报道。本研究根据其他植物赖氨酸脱羧酶基因和推测的EST序列设计保守引物,从蛇足石杉中成功克隆出2个赖氨酸脱酶基因 (LDC1和LDC2),同源性达95%,即LDC1除在3'末端比LDC2多30 bp外,其余序列均一致;由于两者的编码蛋白LDC1比LDC2多10个氨基酸,生物信息学分析表明LDC1和LDC2理化性质存在差异,但是其预测的二级结构和三维结构基本一致,这与其后分析的两重组表达蛋白均具有催化赖氨酸脱羧基功能相印证。根据这些结果我们可以推测LDC1在C末端多出的10个氨基酸并非其酶活性中心或底物结合区域序列,或也非其他必需氨基酸序列,不影响该酶的结构和功能;蛇足石杉中为什么存在如此形式的2个LDC基因尚不清楚,但在此可进一步推测LDC1和LDC2可能来源于同一祖先基因,在进化过程中LDC1出现冗余片段,或者一个基因是另一个基因整合到植物染色体中的拷贝形式,整合过程中发生了插入或缺失突变等。另外,通过氨基酸序列相似性比对还发现,LDC1和LDC2蛋白序列与野芭蕉Musa balbisiana (BAG70979.1)、玉米Zea mays (NP001148565.1)、蒺藜苜蓿Medicago truncatula (XP003588965.1) 和拟南芥Arabidopsis thaliana (CAB87668.1) 推测的LDC氨基酸序列具有较高的同源性 (数据未提供),该结果是否暗示低等蕨类蛇足石杉与上述高等植物在进化上有较近亲缘关系有待研究。
为了验证克隆获得的2个LDC基因功能,我们将其引入可溶性表达载体pET-32a(+),分别于37 ℃、30 ℃和25 ℃进行诱导表达,SDS-PAGE分析表明37 ℃条件下表达量最高,但重组蛋白均为包涵体,25 ℃表达量非常低,几乎检测不到重组蛋白 (数据未提供);30 ℃重组蛋白以包涵体和可溶性两种形式存在,但可溶性表达量比较低。如果要提高可溶性蛋白表达水平,还需对表达条件做进一步探讨。将30 ℃诱导的可溶性蛋白Trx-LDC1和Trx-LDC2进行分离和纯化后,我们曾多次尝试用肠激酶对融合蛋白进行酶切,遗憾的是酶切过程中蛋白变性产生沉淀析出,最后我们选用融合蛋白Trx-LDC1和Trx-LDC2进行酶活性分析,酶促反应结果表明它们均具有催化赖氨酸脱羧生成尸胺的能力。根据TLC检测结果中底物赖氨酸和产物尸胺的比例,可初步确定重组融合蛋白总体酶活性力比较低,其主要原因可能是酶反应条件不适宜所致,当然也不排除融合蛋白TrxA片段空间位阻效应妨碍LDC与底物结合等其他因素的影响。
虽然已有学者研究了大豆Glycine max、烟草Nicotiana glauca、多叶羽扇豆Lupinus polyphyllus和柳叶黄薇Heimia salicifolia等植物的赖氨酸脱羧酶活性和功能[16-20],但是至今有关植物LDC的分子信息仍然知之甚少。本研究首次从蛇足石杉中分离出2个LDC基因,通过生物信息学分析和原核重组表达研究了其编码蛋白的结构和酶活性,这些结果为进一步阐述LDC的生物学功能及其编码蛋白的酶学性质奠定基础。石杉碱甲作为新一代治疗老年痴呆的珍稀药物,随着对其生物合成入口酶LDC及代谢过程中其他关键酶 (基因) 的逐步认识,将能全面解析其生物合成路径和分子作用机制,为通过生物技术 (如组织/细胞培养、代谢工程等) 手段解决石杉碱甲药源问题提供科学依据。
REFERENCES
[1] Guo B, Xu LL, Wei YH, et al. Research advances of Huperzia serrata (Thunb.) Trev.. Chin J Chin Mater Med, 2009, 34(16): 2018–2023 (in Chinese).
郭斌, 徐玲玲, 尉亚辉, 等. 千层塔的研究进展.中国中药杂志, 2009, 34(16): 2018–2023.
[2] Ma XQ, Tan CH, Zhu DY, et al. A survey of potential huperzine A natural resources in China: Huperziaceae. J Ethnopharmacol, 2006, 104(1/2): 54–67.
[3] Ma XQ, Tan CH, Zhu DY, et al. Huperzine A from Huperzia species--an ethnopharmacological review. J Ethnopharmacol, 2007, 113(1): 15–34.
[4] Wang R, Yan H, Tang XC. Progress in studies of huperzine A, a natural cholinesterase inhibitor from Chinese herbal medicine. Acta Pharmacol Sin, 2006, 27(1): 1–26.
[5] Takahiro K, Satoshi Y, Tohru F. Total synthesis of (-)-huperzine A. Org Lett, 2009, 11(22): 5354–5356.
[6] Zeng FX, Jiang HL, Yang YS, et al. Progress in synthesis and structural modification of huperzine A. Prog Chem, 2000, 12(1): 63–76 (in Chinese).
曾繁星, 蒋华良, 杨玉社, 等. 石杉碱甲的合成及结构改造研究进展. 化学进展, 2000, 12(1): 63–76.
[7] Szypuła W, Pietrosiuk A, Suchocki P, et al. Somatic embryogenesis and in vitro culture of Huperzia selago shoots as a potential source of huperzine A. Plant Sci, 2005, 168(6): 1443–1452.
[8] Ma X, Gang DR. In vitro production of huperzine A, a promising drug candidate for Alzheimer's disease. Phytochemistry, 2008, 69(10): 2022–2028.
[9] Zhu D, Wang J, Zeng Q, et al. A novel endophytic huperzine A-producing fungus, Shiraia sp. Slf14, isolated from Huperzia serrata. J Appl Microbiol, 2010, 109(4): 1469–1478.
[10] Wang Y, Zeng QG, Zhang ZB, et al. Isolation and characterization of endophytic huperzine A-producing fungi from Huperzia serrata. J Ind Microbiol Biotechnol, 2011, 38(9): 1267–1278.
[11] Ma X, Gang DR. The Lycopodium alkaloids. Nat Prod Rep, 2004, 21(6): 752–772.
[12] Bunsupa S, Katayama K, Ikeura E, et al. Lysine decarboxylase catalyzes the first step of quinolizidine alkaloid biosynthesis and coevolved with alkaloid production in Leguminosae. Plant Cell, 2012, 24(3): 1202–1216.
[13] Sun JY, Morita H, Chen GS, et al. Molecular cloning and characterization of copper amine oxidase from Huperzia serrata. Bioorg Med Chem Lett, 2012, 22(18): 5784–5790.
[14] Kamio Y, Terawaki Y. Purification and properties of Selenomonas ruminantium lysine decarboxylase. J Bacteriol, 1983, 153(2): 658–664.
[15] Wang FQ, Liu F, Meng Y, et al. Biogenic amines in salted duck analyzed by TLC combined with HPLC. Food Sci, 2011, 32(14): 273–276 (in Chinese).
王凤芹, 刘芳, 孟勇. TLC和HPLC法相结合分析盐水鸭中的生物胺. 食品科学学报, 2011, 32(14): 273–276.
[16] Kim HS, Kim BH, Cho YD. Purification and characterization of monomeric lysine decarboxylase from soybean (Glycine max) axes. Arch Biochem Biophys, 1998, 354(1): 40–46.
[17] Ohe M, Scoccianti V, Bagni N, et al. Putative occurrence of lysine decarboxylase isoforms in soybean (Glycine max) seedlings. Amino Acids, 2009, 36(1): 65–70.
[18] Bagni N, Creus J, Pistocchi R. Distribution of cadaverine and lysine decarboxylase activity in Nicotiana glauca plants. J Plant Physiol, 1986, 125(1/2): 9–15.
[19] Schoofs G, Teichmann S, Hartmann T, et al. Lysine decarboxylase in plants and its integration in quinolizidine alkaloid biosynthesis. Phytochemistry, 1983, 22(1): 65–69.
[20] Pelosi LA, Rother A, Edwards JM. Lysine decarboxylase activity and alkaloid production in Heimia salicifolia cultures. Phytochemistry, 1986, 25(10): 2315–2319.
(本文责编 陈宏宇)
Cloning, prokaryotic expression and characterization of lysine decarboxylase gene from Huperzia serrata
Ci Du1,2, Jing Li1,2, Yuntao Tang1,2, and Qingzhong Peng1,2
1 College of Biology and Environmental Sciences Jishou University, Jishou 416000, Hunan, China
2 Key Laboratory of Plant Resource Conservation and Utilization, College of Hunan Province, Jishou 416000, Hunan, China
Huperzine A is a promising drug to treat Alzheimer's disease (AD). To date, its biosynthetic pathway is still unknown. Lysine decarboxylase (LDC) has been proposed to catalyze the first-step of the biosynthesis of huperzine A. To identify and characterize LDCs from Huperzia serrata, we isolated two LDC fragments (LDC1 and LDC2) from leaves of H. serrata by RT-PCR and then cloned them into pMD®19-T vector. Sequence analysis showed that LDC1 and LDC2 genes shared 95.3% identity and encoded the protein of 212 and 202 amino acid residues respectively. Thus, we ligated LDC genes into pET-32a(+) to obtain recombinant expressing vectors pET-32a(+)/LDC1 and pET-32a(+)/LDC2 respectively. We further introduced two expression vectors into Escherichia coli BL21(DE3) and cultured positive colonies of E. coli in liquid LB medium. After inducing for 4 hours with 260 μg/mL IPTG at 30 ℃, soluble recombinant Trx-LDC1 and Trx-LDC2 were obtained and isolated for purification using a Ni-NTA affinity chromatography. We incubated purified recombinant proteins with L-lysine in the enzyme reaction buffer at 37 ℃ and then derived the reaction products using dansyl chloride. It was found that both Trx-LDC1 and Trx-LDC2 had decarboxylase activity, could convert L-lysine into cadaverine by way of thin layer chromatography assay. Further, bioinformatics analysis indicated that deduced LDC1 and LDC2 had different physicochemical properties, but similar secondary and three-dimensional structures.
Huperzia serrata, lysine decarboxylase, gene cloning, prokaryotic expression, enzyme activity, structure prediction
October 15, 2013; Accepted: December 10, 2013
Qingzhong Peng. Tel: +86-743-8725365; E-mail: qzpengjsu@163.com
杜次, 李菁, 唐云涛, 等. 蛇足石杉赖氨酸脱羧酶基因的克隆、原核表达及其功能分析. 生物工程学报, 2014, 30(8):1299−1307.
Du C, Li J, Tang YT, et al. Cloning, prokaryotic expression and characterization of lysine decarboxylase gene from Huperziaserrata. Chin J Biotech, 2014, 30(8): 1299−1307.
Supported by: National Natural Science Foundation of China (No. 31260081), Scientific Research Fund of Hunan Provincial Education Department (No. 13A078).
国家自然科学基金 (No. 31260081),湖南省教育厅科学研究基金 (No. 13A078) 资助。

