采用DNA片段编辑技术反转CTCF结合位点改变基因组拓扑结构和增强子与启动子功能
郭亚,吴强上海交通大学系统生物医学研究院比较生物医学研究中心,系统生物医学教育部重点实验室,上海 200240
采用DNA片段编辑技术反转CTCF结合位点改变基因组拓扑结构和增强子与启动子功能
郭亚,吴强
上海交通大学系统生物医学研究院比较生物医学研究中心,系统生物医学教育部重点实验室,上海 200240

郭亚(博士研究生)
人类的祖先很早就观察到生物性状的遗传现象,但是直到19世纪60年代孟德尔发现遗传因子的分离定律和自由组合定律,遗传学才真正建立并快速发展起来。尽管人类很快有了基因的概念,并认识了核酸和蛋白质,但直到1953年沃森和克里克阐明了DNA的双螺旋结构之后,人类对基因才有了本质的认识,从而开启了分子遗传学时代。随着人类基因组计划测序工作的完成以及最近大规模测序技术的突破,越来越多的研究发现很多遗传疾病相关位点不仅位于启动子和基因编码区,而且也大量地存在于调控组织发育基因表达的增强子中。更多的遗传疾病相关位点则位于功能未知的基因组区域。近年研究发现这些遗传疾病相关位点可能与三维基因组(3D genome)息息相关并通过染色体高级拓扑架构 (Higher-order topological chromosome architecture)起作用。
在细胞分裂间期,人类大约两米长的30亿DNA碱基对在核酸和蛋白质相互作用下高度有机折叠在约5~10 μm的细胞核内,形成复杂的三维基因组。这种三维基因组随着时间的推移不断变化形成所谓的四维细胞核组 (4D nucleome),提供了细胞核发挥各种生物学功能的物质基础,包括在生命发育过程中实现精准的组织和细胞特异性基因表达调控。在细胞核中,不同染色体占据不同的区域,被称为“染色体领地” (Chromosome territory),染色体领地内又包含很多相对稳定的染色质拓扑结构域 (Topological chromatin domain)。在染色质拓扑结构域内存在着各种特异性的长距离DNA间相互作用,如增强子、沉默子、绝缘子和启动子等DNA功能元件之间的相互作用。近年研究发现哺乳动物基因组中增强子数量远大于启动子的数量,并且基因表达可能受到多个启动子的调控,这种特异性的增强子-启动子相互作用依赖于特定 DNA元件之间的三维高级空间构象。绝缘子元件在形成特异性DNA间相互作用中扮演着重要的角色,但是其分子机制还不清楚。
人类原钙粘蛋白 (Protocadherin, Pcdh) 基因座包括Pcdhα、Pcdhβ和Pcdhγ在内的3个基因簇,在脑发育和脑认知中起到重要作用,原钙粘蛋白的突变与自闭症和精神分裂症等复杂精神疾病相关。除原钙粘蛋白αc2、β1、γc4和γc5启动子外的每一个原钙粘蛋白基因启动子区都包括CBS位点。这些启动子 CBS位点都是正向的;此外增强子区也包括CBS位点,但增强子CBS位点却是反向的。CTCF蛋白能够特异性地介导原钙粘蛋白α增强子与启动子之间的长距离染色质环化,而且CBS位点位于染色质环的锚定点 (Anchor point) 并相互靠近且呈现相反的方向。原钙粘蛋白增强子与启动子特异性染色质环化可以实现在脑发育中单个细胞中原钙粘蛋白启动子的选择性激活和原钙粘蛋白基因的组合表达。这种独特的CBS位点的方向性可能与其介导的特异性增强子与启动子环化相关并在神经发育中具有重要的基因调控功能。该研究结果于 2012年 12月发表于Proc Natl Acad Sci USA (DOI:10.1073/pnas. 1219280110)。
本研究团队通过多种基于染色质构象捕获技术的方法进一步发现,原钙粘蛋白β和γ基因簇具有共同的调控区域,其调控区域与原钙粘蛋白 β和 γ基因的启动子形成染色质环化,这些相互重叠的染色质环具有特定的方向性,形成一个CTCF/Cohesin介导的原钙粘蛋白 β和 γ染色质拓扑结构域(CTCF/Cohesin-mediated chromatin domain: CCD);但是原钙粘蛋白α具有相对独立的调控区,其与原钙粘蛋白α基因形成染色质环化,这些染色质环同样具有方向性并形成原钙粘蛋白α染色质拓扑结构域。所以原钙粘蛋白基因簇包含两个相对独立的CTCF/Cohesin介导的染色质拓扑结构域 (CCD),而且原钙粘蛋白基因簇内CTCF介导的相互作用总是在正向-负向CBS位点之间建立。为阐明这种CBS位点的排列方向与长距离染色质相互作用的关系,本研究团队在CRISPR技术基础上利用Cas9核酸酶和一对sgRNA建立了一种高效的原位DNA片段编辑方法,该方法于2015年3月发表于J Mol Cell Biol (DOI: 10.1093/jmcb/ mjv016),详见本期“基因组编辑技术”专刊中综述文章《CRISPR/Cas9 系统在基因组DNA片段编辑中的应用》对该方法的介绍。
通过原位DNA片段编辑方法,本研究团队反转了包含CBS位点增强子并进一步研究发现增强子反转后其与上游Pcdhα启动子的相互作用显著降低,但产生了与下游Pcdhβ和Pcdhγ启动子的新的相互作用,也就是说反转引起染色质高级拓扑架构改变。因此CBS位点的方向性决定了染色质环化的方向性从而决定了染色质拓扑结构的形成。通过 RNA-seq实验发现,增强子反转造成多个 Pcdhα、Pcdhβ和Pcdhγ基因显著降低。这些结果表明反转增强子能够改变原钙粘蛋白范围内的增强子与启动子间的相互作用,并引起原钙粘蛋白基因表达的改变。利用计算生物学方法进一步验证了CBS位点排列方向与其相互作用形成染色质环化的方向密切相关,即大多数CTCF和Cohesin介导的染色质相互作用都是在正向-负向CBS位点之间建立的,少量CTCF和Cohesin介导的相互作用是在同向的CBS位点之间建立的。最后,通过 CRISPR技术在 β珠蛋白(β-globin) 基因簇区域敲除或反转CBS位点进一步验证了该规律的普适性。因此,基因组中至少一部分含有CBS的增强子是以方向依赖性的方式调控其靶向基因,而不是通常认为的增强子调控基因表达不具有方向性。该研究结果揭示了三维DNA结构是如何由一维的DNA序列所“编码”,因而阐明了DNA遗传信息在三维基因组结构建立与形成中的重要作用 (图 1),对于从三维基因组角度认识各种遗传性疾病的根本病因以及肿瘤的发生发展机理具有重要意义。该项研究结果于2015年8月发表于Cell(DOI: 10.1016/j.cell.2015. 07.038)。
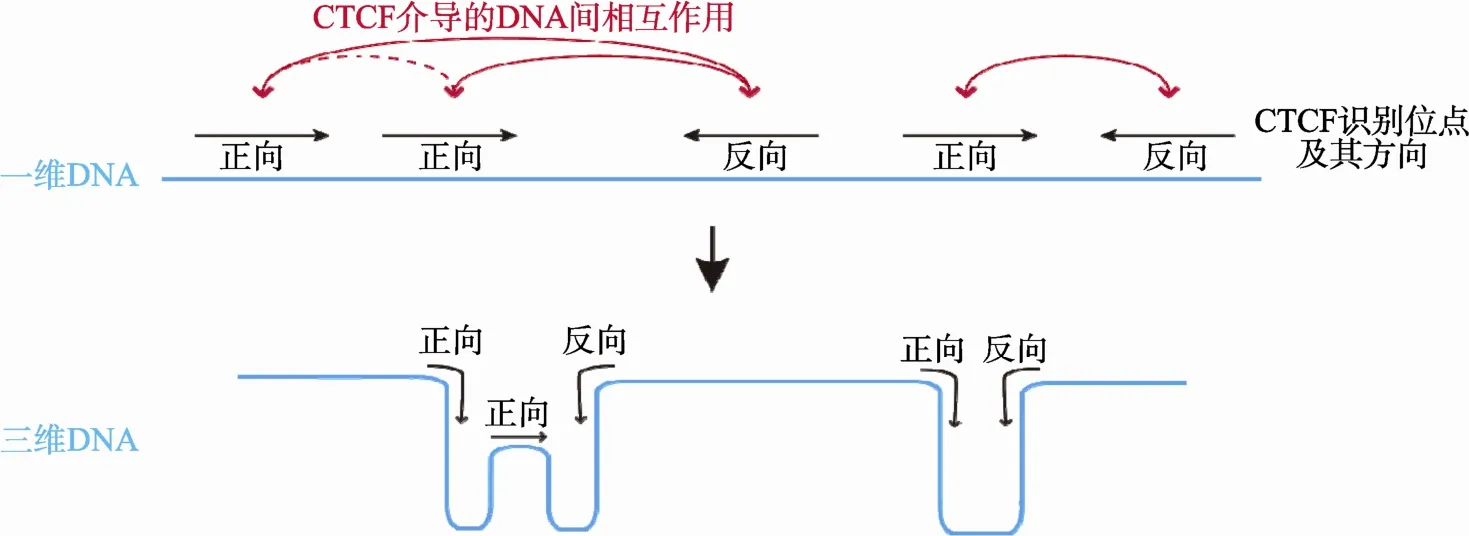
图1 基因组范围内CTCF结合位点(CBS)的一维DNA序列信息参与决定三维DNA结构

