贝类全基因组遗传育种评估与分析系统的开发❋
苏海林,王扬帆,胡晓丽,王 师,包振民
(中国海洋大学海洋生命学院,山东 青岛 266003)
贝类全基因组遗传育种评估与分析系统的开发❋
苏海林,王扬帆,胡晓丽,王师,包振民
(中国海洋大学海洋生命学院,山东 青岛 266003)
为在贝类育种工作中推广应用准确率高于传统系谱资料最佳线性无偏估计方法的全基因组选择育种评估技术,以实现全基因组选择算法的MixP软件为核心,结合国内贝类育种项目研究,本研究设计开发了一套以网站服务形式为基础的基因组选择网络育种平台。该系统在Ubuntu Linux环境下使用MySQL数据库和基于Java编程语言的服务器端动态页面(Java Server Pages, JSP)技术开发完成,主要包括系统公告、综合查询、信息维护、任务管理、育种评估、选种选配等6个功能模块,能够完成数据模拟,性状测量记录的储存和查询,遗传参数估计,分子标记效应估计从而计算基因组估计育种值,以及选种选配方案定制等主要工作。系统在可行性测试中分析了98个栉孔扇贝,包括2个亲本和96个子代的壳长数据和基因型信息,计算出分布于全基因组的单个苷酸多态性(Single Nucleotide Polymorphism, SNP)分子标记效应以及个体的估计育种值,并简单计算出估计育种值与表型值的相关系数为0.7039。这些结果表明系统运行正常。该系统在贝类育种工作中的推广和应用能够有效提升行业的整体技术水平,推动中国贝类育种工作的发展。
贝类育种;全基因组选择;网络育种系统
引用格式:苏海林, 王扬帆, 胡晓丽, 等. 贝类全基因组遗传育种评估与分析系统的开发[J]. 中国海洋大学学报(自然科学版), 2016, 46(10): 65-72.
SU Hai-Lin, WANG Yang-Fan, HU Xiao-Li, et al. Development of the genomic analysis and evaluation system for shellfish genetic breeding[J]. Periodical of Ocean University of China, 2016, 46(10): 65-72.
Meuwissen等于2001年提出全基因组选择(Whole-Genomic Selection, WGS)的概念[1]掀起了一阵育种评估技术的改革,科研工作者通过计算机模拟对其理论与实际应用前景开展了广泛的研究[2-5]。随着各物种全基因组高密度单核苷酸多态性(Single-nucleotide polymorphism, SNP)分子标记连锁图谱的开发与发布使得全基因组选择育种技术在实际选种工作上的应用成为可能。目前,全基因组选择育种技术在动物上的应用包括白鼠[6-7],鸡[8]以及奶牛[9]等,植物物种比如大麦[10],苹果[11]等。其中在奶牛上应用的准确性分析报道得最多,包括北美荷斯坦牛[12],澳大利亚荷斯坦牛[8]以及新西兰荷斯坦牛与娟姗牛[13]。Schaeffer[14]的研究报告表明,全基因组选择育种技术最大的优势在于其能有效的降低时间成本。与传统的使用系谱资料构建混合线性模型的最佳线性无偏估计(Best Linear Unbiased Prediction, BLUP)方法相比,全基因组选择育种技术需要同时具有表型记录和基因型信息的个体作为参考群体计算出分子标记的效应,并通过待评估个体的基因型信息计算出其育种值,因此在奶牛育种工作中采用全基因组选择技术可以有效减小世代间隔,从而使育种成本降低90%以上。而在水产动物上,于洋等[15]指出全基因组选择育种的策略在应用技术上是可行的。在扇贝育种工作中,随着高密度SNP分子标记的不断开发和积累,测序成本的不断下降,以及SNP分子标记基因型分型算法的改进[22],全基因组选择育种技术在扇贝育种工作中应用的基础也逐步完备。为了提高全基因组选择技术的易用性,有必要构建一个集数据管理、育种值与标记效应估算、育种策略制定等功能于一体的育种平台。在中国,李晓强[16]和公维嘉[17]分别开发并发表了使用BLUP法对猪和奶牛进行种用评估的软件和网站,而类似的育种评估工作在水产动物中应用的起步相对比较晚。近几年,科研工作者在水产领域也相继发表了水产动物的育种分析与管理系统:栾生等[18]针对鱼、虾等水产动物的育种工作发表了“水产动物育种分析与管理系统”;李艳等[19]构建并发布了使用BLUP方法的“贝类遗传育种分析与评估系统”。本研究设计开发了一套基于网站服务形式的网络育种平台,即“贝类全基因组选择育种分析评估系统”,探讨研究贝类基因组选择育种技术的应用与实现。
1 系统构建策略
1.1 系统开发技术及工具选择
为了降低开发成本,完成系统开发工作的所有软件、工具、技术和项目均为免费和开源的。在动态页面技术上,考虑到跨平台移植、分层管理、扩展功能及大负荷访问量等因素,系统采用了JSP技术,并使用Apache基金会的Tomcat项目的产品作为JSP容器。JSP技术与Java编程语言有良好的兼容性,作为网站后台技术能高效的解析和响应客户端请求,并且可以移植到任何支持Java虚拟机的操作系统平台上。同时,系统亦采用了同为Apache基金会项目的Struts 2.0框架技术,通过其实现了模型层、表现层和控制层(Modeling, Viewing, Controlling, MVC)的分离,令开发者得以更专注于对逻辑层M的编写,并使用JavaBeans实现框架中的跨层通信,极大地减轻工作量、提高开发效率。系统采用MySQL软件包作为数据库服务的实现,并采用MySQL为Java编程语言提供的JDBC数据库驱动连接器实现网站与数据库的数据传递,使得系统的所有功能和服务都能很容易的实现跨平台移植。系统采用Java编程语言实现动态网页、数据库、运算核心之间的数据交换及功能逻辑处理,在安装有网页工具平台(Web Tools Platform,WTP)扩展并配置好Tomcat服务的Eclipse集成开发环境软件上开发完成。网页的图形元素由GIMP软件设计和编辑。系统开发和调试过程所使用的个人电脑及服务器所运行的操作系统均为Ubuntu Linux,版本为12.10。
系统采用MixP软件包[20]作为实现全基因组选择算法的运算内核。该软件包可通过参考群体数据计算出所有分离的SNP分子标记的效应并能通过个体的基因型计算其基因组估计育种值,以此作为种用评估的标准,在选种选育工作中具有指导意义,而记录分子标记的效应可以为将来开展分子设计育种工作提供数量上的参考。
1.2 系统的数据流向
系统的数据来源于贝类育种场、育种中心的性状测定记录。工作人员或科研工作者将测定的数据按系统要求的格式,经互联网上传提交到系统平台上。系统后台模块将上传的资料存入数据库中,使得育种决策者或技术人员可以通过调用系统的运算内核对性状测量数据进行基因组选择分析,完成对养殖的贝类的遗传评估。系统将评估的结果应用于制定选种选配策略上,使得育种场与育种工作者可以通过互联网访问系统的选种选配页面获取策略信息,从而指导育种工作。系统的数据流程图如图1所示。

图1 系统数据流程示意图Fig.1 Data flow diagram of the system
1.3 数据库设计
数据库结构如图2所示。为了简化程序开发及系统将来的维护和升级,实现数据储存结构的合理性,完整性和安全性,系统在数据库结构设计中采用了十多个数据表,用于记录个体基本信息、个体生长记录、个体繁殖性能、家系抗病能力、家系养殖环境固定效应等数据记录。系统使用了多个自主开发的Java类,实现了与每个表相对应的数据读取和存储的过程,并通过SQL语言使用数据表索引、数据表关联以及数据表合并输出等数据库技术,充分利用了分布式关联数据库存储独立、关联数据查询结果共享性高等特点。系统还为遗传评估结果预留了一系列结果储存表,用于储存包括个体分子标记基因型、分子标记效应及个体估计育种值等信息。这些表未在图2中列出。
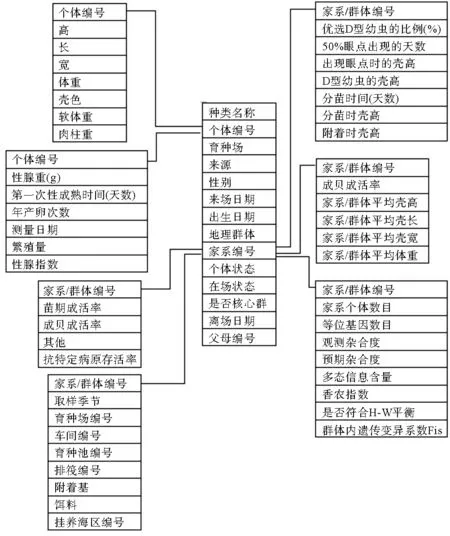
图2 系统数据库结构Fig.2 Database structure of the system
2 系统功能
系统采用模块化设计,主要由系统公告、综合查询、信息维护、任务管理、育种评估、选种选配6个功能独立的模块组成(见图3)。

图3 系统功能模块设计Fig.3 Functions and modular design of the system
2.1 系统公告
系统公告模块实现了系统的日志记录与公告信息展示的功能。用户每次录入或上传数据时,系统都会记录用户信息、提交时间、提交的数据量、数据存入什么表等信息。用户可在系统公告模块中查询这些信息。另外,一些系统动态也会不定期更新在公告中,例如系统升级、例行维护等可能会需要服务器重启的工作会提前做出公告,提醒用户避免在此期间使用系统。
2.2 综合查询
系统提供的综合查询模块使得用户可以对已经录入系统数据库中的数据进行查询和修改,或对存入数据库中的遗传参数计算结果进行查询。针对不同的数据表储存结构,系统提供了个体信息查询、家系信息查询、育种值查询等页面。在查询页面中,用户可以通过限定某些参数的取值范围对数据库中的信息进行条件查询,只有符合条件的数据记录才会在结果中显示。查询结果仅列出数个关键信息,比如个体编号、家系编号等,每条信息记录都有单独的删除该记录的链接,或转跳到详细信息页面的链接。详细信息页面与单笔数据录入页面功能相似,用户可以在详细页面中更改信息并保存,实现修正数据错误的功能。
2.3 信息维护
该模块将用户提交的个体、家系基本信息以及养殖环境信息导入到系统的数据库中,包括个体系谱信息、生长性能性状记录、繁殖性状记录、家系的抗逆性状、家系的苗期性状、家系遗传信息,以及养殖的设施和条件。在提交数据时用户可以选择两种方式:表单提交数据方式(见图4)和批量上传方式(见图5)。基因型信息数据规模较大,因此系统禁用了其表单提交页面并自动转跳至批量上传页面(见图6)。
2.4 任务管理
当用户提交的全基因组选择分析任务需要处理高密度标记及大规模数据时,后台程序的运算速度不足以在短时间内完成计算并产生结果,因此我们在系统开发的过程中引入了后台任务提交与管理模块。系统在默认设置中最多允许运行8个后台任务,如果当前运行的任务少于8个,则由用户提交的新任务将立克执行并显示在正在运行的任务列表中;如果正在运行的任务为8个,当用户提交新的运算任务时,新提交的任务信息将被储存到数据库中的待执行任务队列中,直到任一当前运行的任务计算完毕,系统将更新任务队列状态并从之前存入数据库中的待执行任务信息按照存入的先后顺序读取任务参数并自动提交运行。用户在新计算任务提交后,可以随时登出系统或关闭浏览器,无论新任务是否进入待执行队列(见图7)。

图4 系统的信息录入-单笔输入页面Fig.4 Web page for inputing single record

图5 系统的信息录入-批量上传页面Fig.5 Web page for uploading multiple records
图6 系统的基因型信息录入-批量上传页面Fig.6 Web page for uploading genotype info

图7 系统的任务管理-任务状态页面,显示核心使用情况Fig.7 Web page for monitoring jobs: idle
用户可在系统的后台任务管理页面中取消正在运行的任务或已提交到数据库中的待执行任务。系统的后台任务提交与管理模块为每个用户提交的任务分配所有权,用户可以看到所有已提交的任务的提交者、提交时间、已运行时间等(见图8),但是不能取消其他用户提交的任务。

图8 系统数据模拟新任务提交页面Fig.8 Web page for monitoring jobs: new submission
2.5 育种评估
该模块利用用户提交的有效的数据,通过调用后台运算核心程序估计SNP标记的效应,从而计算出已有基因型信息的个体的全基因组估计育种值。此外,该模块通过调用模拟算法实现了产生模拟基因组信息数据的功能。
2.5.1 SNP标记效应估计当扇贝个体在系统中同时具有表型性状记录与基因型数据时,用户便可通过点击系统页面左侧纵向导航菜单的“功能检索”菜单组下的“育种评估”主菜单项的“基因组估计育种值计算”菜单项打开相应页面,选取用于计算SNP标记效应的个体作为参考群体,同时勾选需要加入模型的固定效应,最后点击页面“计算”按钮向系统提交计算任务。提交任务后,系统将通过后台任务提交与管理模块分配运算资源,适时调用核心运算程序在服务器后台运行。当任务运行结束时,系统将计算结果中的SNP标记效应存入数据库中,用于估计已知基因型信息的待估群体的育种值。新提交的运算任务的结果将覆盖之前已存入数据库中的标记效应。
2.5.2 基因组估计育种值的计算与“SNP标记效应估计”类似,通过选取系统页面左侧纵向导航菜单的“功能检索”菜单组下的“育种评估”主菜单项的“基因组估计育种值计算”菜单项,用户可以在数据库中已存在SNP标记效应信息的情况下对选中的家系中的个体进行基因组估计育种值的计算(见图9)。在用户提交新的计算申请时,系统向数据库发出检索请求,判断是否已存有基因组估计育种值以及分子标记效应值。如果数据库中存在选中个体的估计育种值,系统将会提示用户并询问是否需要重新计算估计育种值:用户选择不需要重新计算,系统则直接调用并显示数据库中已经存在的结果;否则系统继续判断数据库中是否存在标记效应信息。如果数据库中不存在分子标记效应信息,系统将提交一个新的后台任务用于计算分子标记效应。此时用户可以登出或关闭网页浏览器,系统将在计算任务结束后向用户发送一条离线消息,用户下一次登陆时会在系统主页中看到提醒。如果数据库中有标记效应的信息,系统将从数据库中调用标记效应信息并根据所选个体的分子标记基因型计算出估计育种值,存入数据库中并返回计算结果页面。

图9 基因组估计育种值结果页面Fig.9 Web page showing GEBV estimations
2.5.3 基因组信息数据模拟系统的后台运算核心程序包含模拟功能,用户可通过在数据模拟页面上指定模拟时所需要的参数创建模拟任务并提交到系统后台任务队列(见图10)。模拟参数包括有效群体大小、染色体长度(摩尔根)、突变率(碱基突变个数每108碱基对)、QTL数目、遗传力、模拟自然交配世代数、最小等位基因频率等等。模拟功能产生的数据可存入数据库中,也可经系统压缩打包并提供下载链接返回给用户。
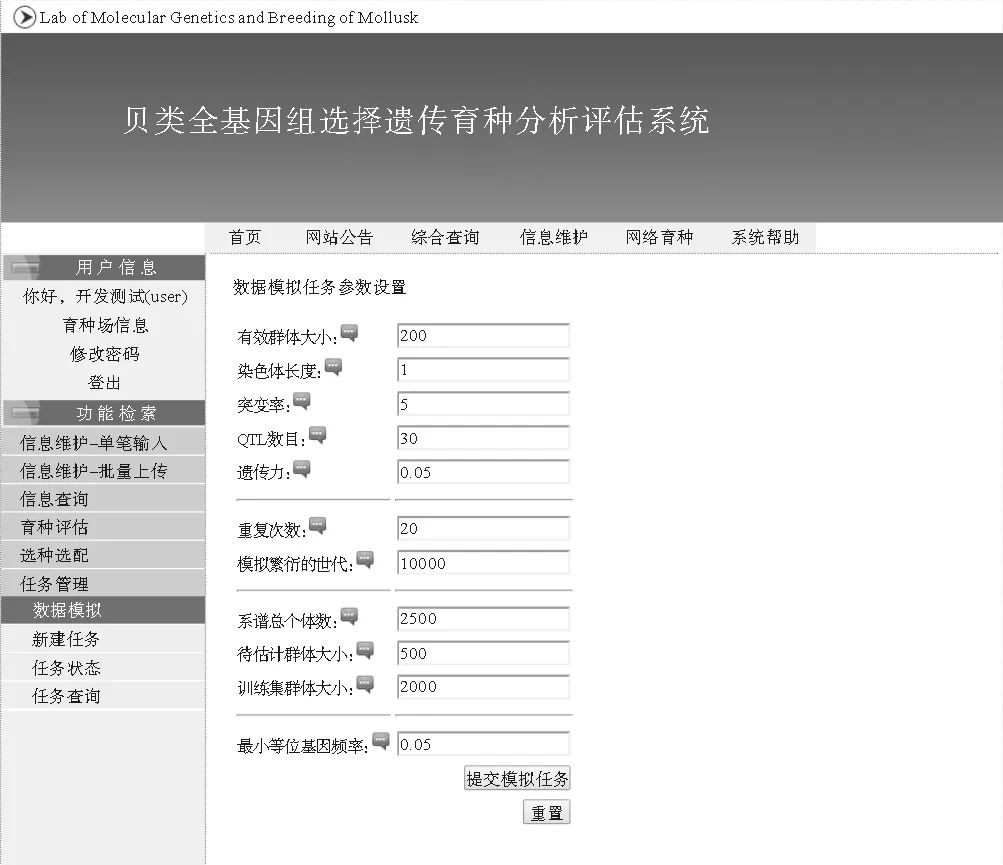
图10 系统的新建数据模拟任务页面Fig.10 Web page for submitting a simulation job
2.6 选种选配方案设计
在系统数据库中存入基因组估计育种值后,用户可通过选取系统页面左侧纵向导航菜单的“功能检索”菜单组下的“选种选配”主菜单项的“选种选配方案设计”菜单项打开相应页面,选择参加计算的家系,目标性状,以及可接受的拟后代的近交系数取值范围,系统将在页面提交后选出基因组估计育种值均数最优配置的雌雄个体组合,作为备选方案(见图11)。此功能需要系统数据库中存在参加计算的个体的基因组估计育种值,通过系谱资料计算出个体的近交系数以及个体之间的共亲系数,并以此为基础计算出备选个体作为亲本的拟后代的近交系数的期望值。系统将选出拟后代近交系数期望值符合用户所指定标准的亲本匹配结果按照亲本的平均基因组估计育种值大小降序显示(见图12)。

图11 系统的选种选配方案设计页面Fig.11 Web page for designing mating plans
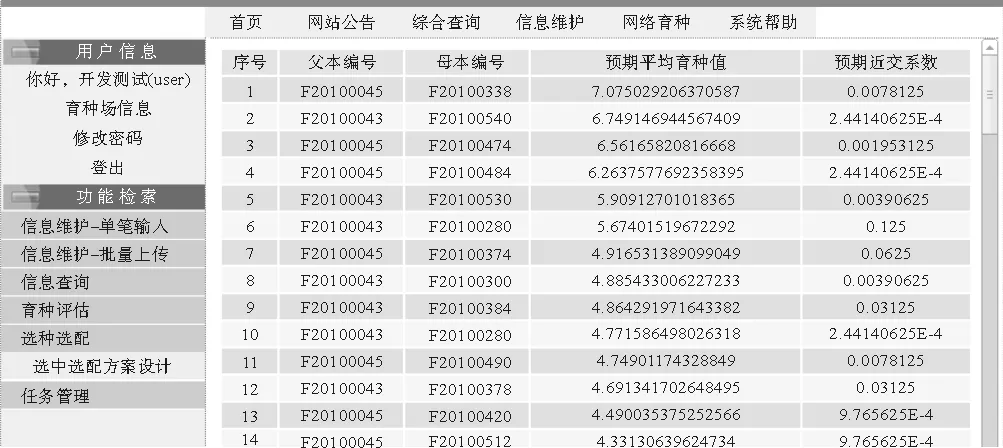
图12 系统的选种选配方案设计结果页面Fig.12 Web page showing results of mating plans
3 系统的应用
为测试系统的可用性,本研究采用了一个家系的栉孔扇贝壳长数据,包括2个亲本及其96个子代,利用2b-RAD技术[21]并通过Dou等[22]的SNP分型手段获得共有5369个可用标记的基因型数据。这些数据被导入系统并完成了一次基因组估计育种值的计算。

图13 估计育种值EBV与表型数据按照 个体序号排列的分布示意图Fig.13 Distributions of GEBV and phenotypes sorted by ID
计算结果显示,一半个体(48)的基因组估计育种值大于0,其中个体编号为67的估计育种值最高,为2.602276,而个体编号为47的估计育种值最低,为-1.704376。在存入数据库中的SNP标记估计效应的结果中,近一半(2626个,约50.82%)的标记效应大于0,其中编号为5167的SNP标记效应值最大,为0.005516943。图13展现了未经均值矫正的估计育种值(Estimated Breeding Value, EBV)与表型数据按照个体序号排列的分布示意图,其中估计育种值与表型值的简单相关系数达到0.7039。这些结果表明系统可以正常完成基因组估计育种值的计算。
4 讨论
4.1 系统安全性
在如今流行的模型层、表现层和控制层分离的MVC设计模式的众多实现中,本系统采用了Apache Struts 2.0框架。用户向系统提交访问请求时所有的访问页面链接都以Action的形式传递到系统的控制器,通过系统内部映射获取相应的资源和页面,从而隐藏系统资源的真实路径。当用户企图请求映射表之外的资源时,系统会返回请求无效的消息。这使得用户无法通过在浏览器地址栏构造恶意请求获得服务器限制访问权限的系统安全相关信息,从而提高了系统的安全性。
系统提供的服务是面向注册用户的:未注册或不能提供正确用户名、密码组合的用户无法登陆进入系统,因此无法使用系统提供的功能。另外,低权限的普通用户也无法访问其他用户所特有的数据,亦不能访问用户管理页面。Struts 2.0框架实现的拦截器(Interceptor)机制能高效处理系统所需要配置的用户权限:系统开发者将判断用户是否有效或是否具有访问某些页面的权限的Java代码写入一个继承了拦截器的类中,并在Struts2配置文件中相关页面的映射设置区域加入对该拦截器的调用,即可实现权限控制,在保证安全性的同时降低开发和维护的难度。
4.2 后台任务提交与管理
系统的后台任务提交与管理系统是通过建立2个Java类实现的。其中1个类的设计基于JavaBeans,用于产生代表1个后台任务的对象;另一个类封装了1个容量为8的类型为后台任务类的列表,用于管理任务调度。在系统服务运行的初始化过程中,系统向Tomcat容器申请应用程序级别的共享对象存储空间并声明由负责管理任务调度的类产生的对象,存进该共享对象存储空间。此后,系统通过与该空间内的负责任务调度的对象进行交互访问实现后台任务的统一管理,从而避免向Linux系统申请守护进程,极大减小了系统开销和开发难度,同时,用户登出系统、离开页面或关闭浏览器后,由于系统应用程序级别的对象在系统运行时不会被销毁,运行在其中的后台任务就不会停止。
4.3 贝类基因组选择网络育种平台
与“中国荷斯坦牛育种数据网络平台”、“全国种猪联合育种网络平台”、“贝类遗传育种分析评估系统”等网络育种平台相比,本研究开发的系统加入了对SNP分子标记信息的存储功能,使得系统能够进行全基因组选择育种分析。在育种工作中加入分子遗传标记信息可提高遗传选育的准确性,缩短世代间隔从而加速选择育种的进程[1]。系统采用的运算核心软件MixP运算速度与基因组最佳线性无偏估计(G-BLUP)法相当,却能获得与贝叶斯多元线性回归模型[1]无显著差异的准确率[20],因而更适合应用在实际育种工作中。
4.4 育种软件系统的发展方向
基于网络的遗传评估中心和数据处理中心是育种软件未来的发展趋势[15]。与桌面应用程序相比,网络应用程序对客户端没有硬性规定,通常耗费很少的用户硬盘空间,不需要检查、安装更新,在大多数情况下可以实现跨平台使用。育种分析软件在数据量较大的情况下对运算资源需求高,而基于网络的软件系统依靠服务器端配置的高性能硬件设备,降低对客户端使用的电脑的硬件规格的要求,因此基于网络的育种软件系统具有更强的普及性。运行在服务器端的程序不受客户端时间和空间的限制,因此科研工作者可以充分收集各地育种场提供的数据资料,提高数据的利用率和遗传育种评估与分析的准确性,从而节约人力资源和时间成本[19]。
5 结语
本研究设计、研发并实现了一套以网站服务形式为基础的基因组选择网络育种平台:贝类全基因组遗传育种评估与分析系统。开发者采用Apache Struts 2.0框架,提高了系统开发、维护和扩展的效率和易行性。贝类育种工作正进入基因组信息时代,新系统引入SNP标记效应估计、基因组估计育种值计算、基因组信息数据模拟、后台任务提交与管理等功能模块,为提供高质量、高精度、高通量的贝类全基因组选择育种网络应用服务打下了坚实的基础。
[1]Meuwissen T H, Hayes B J, Goddard M E. Prediction of total genetic value using genome-wide dense marker maps [J]. Genetics, 2001, 157(4): 1819-1829.
[2]Calus M P L, Veerkamp R F. Accuracy of breeding values when using and ignoring the polygenic effect in genomic breeding value estimation with a marker density of one SNP per cM [J]. J Anim Breed Genet, 2007, 124: 362-368.
[3]Habier D, Fernando RL, Dekkers J C M. The impact of genetic relationship information on genome-assisted breeding values [J]. Genetics, 2007, 177: 2389-2397.
[4]Kolbehdari D, Schaeffer L R, Robinson J A B. Estimation of genome-wide haplotype effects in half-sib designs [J]. J Anim Breed Genet, 2007, 124: 356-361.
[5]Solberg T R, Sonesson A K, Woolliams J A, et al. Genomic selection using different marker types and densities [J]. J Anim Sci, 2008, 86(10): 2447-2454.
[6]Lee S H, van der Werf J H J, Hayes B J, et al. Predicting unobserved phenotypes for complex traits from whole-genome SNP data [J]. PLoS Genet, 2008, 4: e1000231.
[7]Legarra A, Robert-Granie C, Manfredi E, et al. Performance of genomic selection in mice [J]. Genetics, 2008, 180: 611-618.
[8]Gonzalez-Recio O, Gianola D, Rosa G J M, et al. Genome-assisted prediction of a quantitative trait measured in parents and progeny: Application to food conversion rate in chickens [J]. Genetics Selection Evolution, 2009, 41: 3.
[9]Hayes B J, Bowman P J, Chamberlain A J, et al. Invited review: Genomic selection in dairy cattle: Progress and challenges [J]. J Dairy Sci, 2009, 92: 433-443.
[10]Zhong S, Dekkers J C M, Fernando R L, et al. Factors affecting accuracy from genomic selection in populations derived from multiple inbred lines: A barley case study [J]. Genetics, 2009, 182: 355-364.
[11]Kumar S, Chagne D, Bink, M C A M, et al. Genomic selection for fruit quality traits in apple (MalusxdomesticaBorkh. ) [J]. PLoS ONE, 2012, 7(5): e36674.
[12]VanRaden P M, Van Tassell, C P, Wiggans, G R, et al. Invited review: Reliability of genomic predictions for North American Holstein bulls [J]. J Dairy Sci, 2009, 92: 16-24.
[13]Harris B L, Johnson D L, Spelman R J. Genomic selection in New Zealand and the implications for national genetic evaluation [C]. Proceedings of the Interbull Meeting. Niagara Falls, 2008.
[14]Schaeffer L R. Strategy for applying genome-wide selection in dairy cattle [J]. J Anim Breed Genet, 2006, 123: 218-223.
[15]于洋, 张晓军, 李富花, 等. 全基因组选择育种策略及在水产动物育种中的应用前景[J]. 中国水产科学, 2011(4): 936-943.
Yu Y, Zhang X, Li F, et al. Strategy of whole genomic selection prospect in aquaculture [J]. Journal of Fishery Sciences of China, 2011(4): 936-943.
[16]李晓强. 猪遗传育种及种猪场管理计算机软件系统的研究[D]. 河南: 郑州大学, 2001.
Li X. Development of Computer Software for Swine Breeding and Genetics and Swine Farm Management System [D]. Henan: Zhengzhou University, 2001.
[17]公维嘉. 中国奶牛遗传评估系统的建立研究[D]. 北京: 中国农业大学, 2005.
Gong W. Genetic Evaluation System in China Dairy Cattle [D]. Beijing: China Agricultural University, 2005.
[18]栾生, 孔杰, 王清印, 等. 水产动物育种分析与管理系统的开发和应用[J]. 海洋水产研究, 2008(3): 92-100.
Luan S, Kong J, Wang Q, et al. Development and application of an aquatic animal breeding analysis and management system [J]. Marine Fisheries Research, 2008(3): 92-100.
[19]李艳. 贝类遗传评估体系的建立研究[D]. 青岛: 中国海洋大学, 2010.
Li Y. Research and Constitution of Genetic Evaluation System in Shellfish [D]. Shandong: Ocean University of China, 2010.
[20]Yu X, Meuwissen T H E. Using the Pareto principle in genome-wide breeding value estimation [J]. Genetics Selection Evolution, 2011, 43: 35.
[21]Wang S, Meyer E, McKay J K, et al. 2b-RAD: A simple and flexible method for genome-wide genotyping [J]. Nature Methods, 2012, 9(8): 808-810.
[22]Dou J, Zhao X, Fu X, et al. Reference-free SNP calling: Improved accuracy by preventing incorrect calls from repetitive genomic regions [J]. Biology Direct, 2012, 7: 17.
责任编辑高蓓
Development of the Genomic Analysis and Evaluation System for Shellfish Genetic Breeding
SU Hai-Lin, WANG Yang-Fan, HU Xiao-Li, WANG Shi, BAO Zhen-Min
(College of Marine Life Sciences, Ocean University of China, Qingdao 266003, China)
Working on shellfish breeding and in order to popularize genomic selection technology which is expected to be able to achieve higher prediction accuracy than the nowadays widely used Best Linear Unbiased Prediction (BLUP) breeding technology, we designed and developed an edge cutting web based breeding platform for the research and application of the genomic selection technology for domestic shellfish breeding programs. The MixP software, which utilized the Pareto principle in prior variance component assumptions and implemented a type of algorithm that performs genomic selection analysis that differs from Genomic Best Linear Unbiased Prediction (GPLUP) and Bayesian Regression based marker effect models, was adopted in this study and used as the computing kernel of the system. This system was built on the most widely used Linux operating system, i. e. the Ubuntu distribution, along with MySQL database system, Java programming language and the Java Server Pages technology, structured by the Apache Struts 2.0 frameworks. This system consists of 6 functioning modules consisting of the system announcement module, the comprehensive inquiry module, the information maintenance module, the task management module, the breeding evaluation module, and the selection and mating program design module. The system was competent for accomplishing jobs such as data simulation, storage and inquiry of phenotypic trait records and environmental information, genetic parameter estimation, Single Nucleotide Polymorphism (SNP) marker effect estimation and breeding value estimation, mating plan formulation, et al. In addition, a simple analysis was performed on this system using field data to test the system’s feasibility. The shell length measurement records and the 2b-RAD de novo genotyping technology enhanced genotypic information of 98ChlamysFarreriindividuals, including two parents and their 96 offspring, were inputted into the system and were analyzed using the breeding value estimation module of the system. Results have shown that this system is operating properly, and a moderate to high correlation of 0.703 9 between phenotype and estimated breeding value (EBV) was obtained, indicative of sound prediction accuracy. The application and extension of this system in the breeding programs of shellfish will effectively improve the overall technological level of the industry and promote the development of shellfish breeding in China, and in the near future a significant selection response is expected with data of large scale.
shellfish breeding; genome-wide select; breeding system based on internet
国家高技术研究发展计划项目(2012AA10A405);现代农业产业技术体系建设专项资金项目(CARS-48)资助
2016-05-11;
2016-06-24
苏海林(1983-),博士后。E-mail:cbkmephisto@gmail.com
Q38
A
1672-5174(2016)10-065-08
10.16441/j.cnki.hdxb.20150358
Supported by the National High-Tech R&D Program (2012AA10A405);the Earmarked Fund for Modern Agro-industry Technology Research System(CARS-48)

