基于质谱分析的代谢组学研究进展
任向楠,梁琼麟
(清华大学 化学系,清华大学分析中心,北京 100084)
基于质谱分析的代谢组学研究进展
任向楠,梁琼麟*
(清华大学 化学系,清华大学分析中心,北京 100084)
质谱分析技术是代谢组学研究的重要技术之一。该文通过近5年来的文献分析,对基于质谱分析的代谢组学研究方法的新进展,包括样品前处理方法、分析检测方法、数据处理方法等,以及近年来代谢组学在疾病诊断、药物研发、营养学、毒理学、运动医学等领域的应用进展,进行了较全面的综述,并对未来的发展趋势进行了展望。
质谱分析;代谢组学;研究进展;综述
代谢组学(Metabolomics,Metabonomics)是指生物体系因生物刺激、病理生理扰动或遗传信息改变等引起的总体、动态的代谢变化[1]。代谢组学作为系统生物学最下游的“组学”,是整体性研究生命体系功能变化的重要学科分支。代谢组学的研究对象为复杂的生物体系,种类非常多,常见的生物样本包括生物体液(尿液、血清、血浆、唾液、胆汁、脑脊液、羊水、眼泪、胰液、肠液等)、动物或人体组织(肿瘤、肝脏、脑、脂肪组织等)和细胞[2]。代谢组学常用的研究手段包括质谱和核磁两大类,本文重点综述了2010年以来基于质谱(MS)分析的代谢组学在医学领域的研究进展,主要包括疾病诊断、药物研发、营养学、毒理学、运动医学等方面。
1 代谢组学分析方法的新进展
代谢组学又分为靶向性代谢组学(Targeted metabolomics)和非靶向性代谢组学(Untargeted metabolomic)。靶向性代谢分析主要是定量特定的代谢物,典型的是关注一种或几种相关的代谢通路。常用于研究药物代谢的药代动力学,衡量某种疗法或基因修饰的效果。靶向性代谢分析的流程是代谢物标准品的液相色谱-质谱联用分析(LC-MS),选择性离子监测,分析方法优化和标准曲线制作,样品前处理,样品提取物LC-MS分析,通过对比样品和标准品进行数据分析,定量特定的代谢物[3-6]。非靶向性代谢组学范围宽泛,目标是从生物样本中同时分析尽可能多的代谢物。而非靶向性代谢组学的步骤是样品前处理,代谢物提取,LC-MS全扫描检测,数据预处理,统计分析及差异代谢物结构鉴定[3-4,7-9]。
1.1 样品前处理
样品前处理直接关系到信息获取的准确性,非靶向性代谢组学的样品前处理要求尽量不破坏代谢组分,以获得尽可能全的代谢信息。靶向性代谢组学则更关注提取的效率,用回收率、精密度、准确性等表征。本实验室用甲醇-乙腈(1∶1)去除血浆中的蛋白,建立了大鼠血浆的高效液相色谱-时间飞行质谱联用(HPLC-TOF-MS)的分析方法,同时定量了大鼠服用知母后的21种药物代谢物,用精确质量测定了代谢物的基本组成并确认其结构,其中包括同分异构体的代谢物[10]。成玉等[11]优化了去除血清蛋白的方法,发现甲醇-乙腈混合除蛋白检测的离子峰多于纯甲醇和纯乙腈,这与本实验室的研究结果相一致,另外一方面通过对比方法的重现性,发现甲醇-乙腈(1∶9)去除血清中的蛋白优于甲醇-乙腈(1∶1)。Want 等[12]和Sarafian 等[13]处理胆汁酸的样本时,均采用甲醇除蛋白法。非靶向分析实体组织代谢物研究方面,多篇文献均采用甲醇-水(1∶1)提取水溶性代谢物,二氯甲烷-甲醇(3∶1)提取有机类代谢物[14-18]。在液质联用(LC-MS)分析粪便代谢物方面,Su 等[19]处理粪便样本时,先用水提取,再用相同体积的乙腈提取,合并上清液、吹干,用同位素丹磺酰氯衍生化,数据结合HMDB数据库,鉴定出3位女性7 d粪便中的39种代谢物;Tian 等[20]则采用甲醇涡旋提取处理粪便样本,用液质联用分析出对照组和给药组的16个代谢标志物。气相色谱-质谱联用(GC-MS)能直接从标准谱库中比对化合物信息,易于定性,但其不适用于难挥发和高极性代谢物分析,前处理需衍生化,相对处理复杂,常用的衍生化方法有硅烷化、酰化、烷基化[21-22]。Sulek 等[23]用气质联用分析头发代谢物时,分别用氢氧化钠水解、酸中和、冷冻干燥、甲醇水混合物再提、蒸干、氯甲酸甲酯衍生、弃去水层的步骤处理头发样本。Tsutsui 等[24]用液质联用分析头发代谢物时,用甲醇盐酸混合物超声提取头发样本,前处理方面较气质联用简单。本实验室建立了从细胞中提取线粒体及其样本制备的方法,并将代谢组学的应用推广到亚细胞层面[25]。各类样本的靶向分析和非靶向分析的前处理方法详见表1。
1.2 质谱分析技术
质谱(MS)分析主要包括液相色谱-质谱联用(LC-MS)和气相色谱-质谱联用(GC-MS),液相色谱又分为超高效液相色谱(UPLC)和高效液相色谱(HPLC)。非靶向性代谢组学的分析,色谱可选择与飞行时间(TOF)、静电轨道阱(Orbitrap)、离子阱飞行时间(IT-TOF)、四极杆-飞行时间(Q-TOF)等质谱联用;靶向性代谢组学的分析,色谱可与三重四极杆(QQQ)或四极杆离子阱(Q-Trap)等质谱串联,用多反应监测器(MRM)进行检测。大气固体分析探针(Atmospheric solids analysis probe,ASAP)结合质谱技术(ASAP-MS)采用毛细管吸取的方式进样,操作简单,可用于较复杂物质的离子检测,例如鼠的尿液以及狗和鼠的胆汁,能有效地将样品来源归类,但其产生的数据不如液质和气质全面,可以用于样本筛选方面[26]。近年来逐渐发展了高集成和高通量的微流控芯片与质谱联用的方法,林金明等[27]在这方面做了很多工作,将微流控芯片与电喷雾质谱联用技术相结合用于细胞分析及药物动力学的研究,使细胞药物代谢研究和细胞间信号传递的研究深化到分子水平。

表1 生物样本的质谱代谢组学研究案例Table 1 The cases of metabolomics based on mass spectrometry of biological samples
(续表1)

代谢组学类型样本前处理方法分析方法数据处理方法参考文献非靶向分析(尿液代谢物)尿液稀释UPLC-MSPCA,ROCOPLS-DA相关性分析[2,14,33-34]非靶向分析(尿液代谢物)尿液衍生化处理GC-MSPCA[21]非靶向分析(尿液代谢物)尿液毛细管吸取ASAP-MSPCA[26]非靶向分析(尿液代谢物)尿液蛋白沉淀、稀释CE-ESI-MS-TOFPCAOPLS-DA[35]非靶向性分析肝脏组织甲醇-水(1∶1)水提,二氯甲烷-甲醇(3∶1)提取,甲醇-水(1∶1)复溶UPLC-MSHSST3柱PCA[16]非靶向性分析动脉斑块组织甲醇-水(1∶1)水提,二氯甲烷-甲醇(3∶1)提取有机物UPLC-MS水提物-HILIC柱有机物-反相柱PCAOPLS-DA变异系数文氏图代谢通路分析[17-18]非靶向性分析动物、人体组织甲醇-水(1∶1)水提,二氯甲烷-甲醇(3∶1)提取,甲醇-水(1∶1)复溶UPLC-MS水提物-HILIC柱有机物-C18柱HSST3柱PCAPLS-DA相关性分析变异系数文氏图代谢通路分析[14-15]非靶向分析(胆汁代谢物)胆汁毛细管吸取ASAP-MSPCA[26]非靶向分析(细胞代谢物)细胞细胞制备后用生物样品均质器提取代谢物干冰甲醇提取声波降解法UPLC-MSHILIC柱PCA相关性分析代谢通路分析比较代谢物分析[14,36-37]非靶向分析(脑脊液代谢物)脑脊液甲醇提取UPLC-MSHPLC-MS相关性分析PCA[14]非靶向分析(唾液代谢物)唾液除蛋白(气质需衍生化)UPLC-MSGC-MS相关性分析PCA[38-39]非靶向分析(羊水代谢物)羊水丙酮除蛋白并衍生化GC-MSPCAPLS-DAOPLS-DA代谢通路分析[22,40]非靶向分析(眼泪代谢物)眼泪80%冷甲醇提取HPLC-MSIDALC-MS/MSPCA代谢富集分析代谢通路分析[41-42]非靶向分析(粪便代谢物)粪便水提取,乙腈提取,合并上清液,同位素丹磺酰氯衍生化;甲醇提取UPLC-MSHPLC-QTOF-MSPCAPLS-DAOPLS-DA[19-20]非靶向分析(头发代谢物)头发氢氧化钠水解、酸中和、冷冻干燥、甲醇水混合物再提、蒸干、衍生化、弃去水层;甲醇盐酸混合物提取、超声、过滤GC-MSUPLC-ESI-TOFMSROC曲线T检验PCAOPLS-DA[23-24]
1.3 数据处理
代谢组学常用的数据处理软件有仪器公司的软件(例如Waters的MarkerLynx,Thermo Fisher的SIEVE,Agilent的MassHunter,Applied Biosystems的Marker View等)和一些公开免费的数据分析网站(例如XCMS[2,34,36,43-46],MzMATCH[36,47],MZmine[8,45-46],Metalign[45-46],Metaboanalyst[22,41,48],MeltDB[46,48],metaP-Server[48-49]等),还有专门的数据处理软件(SIMCA-P,Matlab,SAS等)。代谢组学常用的数据库主要有一般化学数据库(如PubChem)、代谢通路数据库(如BioCyc)、代谢物数据库(HMDB)、质谱数据库(如NIST)等[50]。Xia等[48]制定了Metaboanalyst软件处理代谢组学数据的标准,在格式化和上传数据、处理和标准化数据、鉴定有显著性的物质、利用代谢富集分析和代谢通路分析来阐明可能的生物机理方面提供了一系列流程,并且对比了Metaboanalyst,MeltDB,metaP-Server,SIMCA-P,SAS软件在处理代谢组学数据时的优缺点,发现Metaboanalyst网站在处理数据时应用较广,可以进行单变量分析、多变量分析、聚类分析、富集分析和通路分析。MeltDB网站则可以结合其它组学的数据。XCMS网站可以结合其它物质的数据库。各个代谢组学软件或网站的特点详见表2。
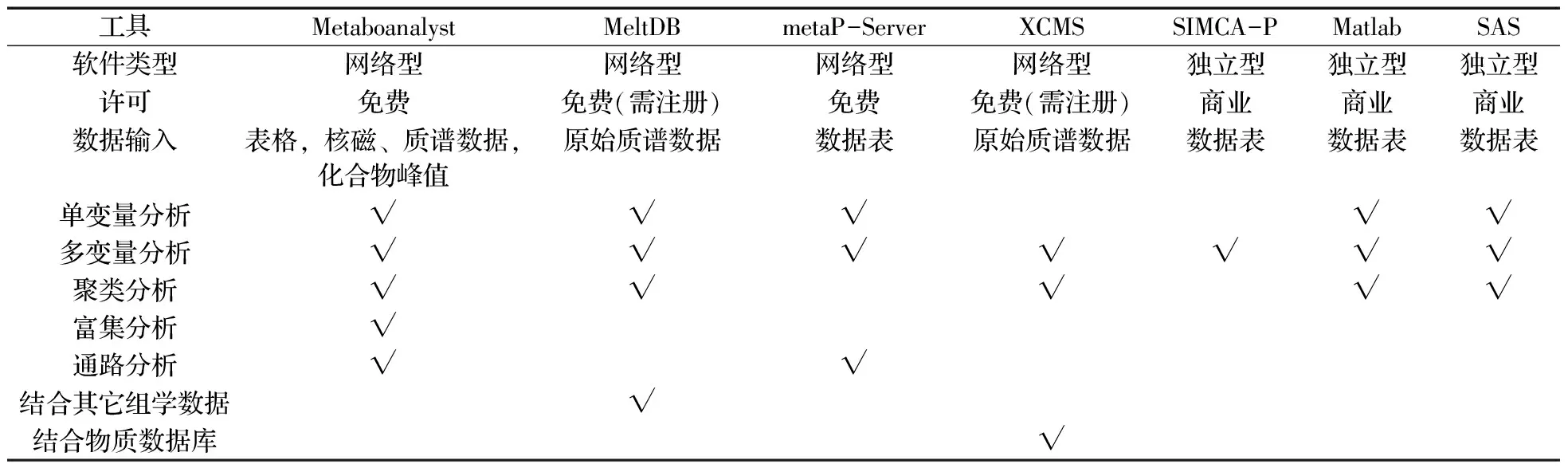
表2 代谢组学常用分析软件的特点Table 2 The characteristics of analysis software of metabolomics research
“√” means that the specific software can analyze the statistic analysis in column(“√”代表某种软件下可以进行纵列的统计分析)
2 基于质谱分析的代谢组学应用研究进展
基于质谱分析的代谢组学在医学领域的应用主要包括疾病诊断、药物研发、营养学、毒理学、运动医学等领域,表3总结了这几大领域主要关注的问题。
2.1 疾病诊断领域
传统的疾病诊断方法是检测单一的一个或几个指标,生物体的作用是一个协同的整体,评价单一的指标具有片面性。代谢组学是从整体上评价机体的代谢变化,较为全面。在实验设计上有横断面设计,比较疾病组和正常组,也有纵向设计,干预前后或不同时间点的代谢研究。Vorkas 等[17-18]利用UPLC-MS法对腹主动脉瘤、颈动脉狭窄性斑块、股动脉狭窄性斑块病人外科手术取的组织进行了非靶向性分析,发现这几类患者组织的代谢物均能得以区分,发现动脉粥样硬化相关的代谢包括胆固醇代谢、嘌呤代谢、嘧啶代谢、神经酰胺代谢。Roy 等[8]利用UPLC-MS分析了非靶向性的血浆代谢物和靶向性的代谢物,定量了8种氨基酸和8种酰基肉毒碱,被认为是发生心血管疾病风险的生物标志物。Fattuoni 等[22]研究了孕妇穿刺的羊水代谢物,有助于监测病毒性感染孕妇和胎儿的早期诊断。Musharraf 等[51]用GC-MS法研究了急性骨髓性白血病、急性淋巴细胞白血病、再生障碍性贫血和对照组病人的血清样本,发现了27个显著不同的代谢物,患者的脂肪酸代谢失调,这有助于早期诊断急性白血病。Ciavardelli 等[52]用代谢组学的方法研究了阿尔茨海默氏症患者的代谢物,与对照组及轻度认知功能的患者相比,阿尔茨海默氏患者的一部分血浆酰基肉碱含量较低,前额叶灰质体积较少,并且存在认知功能障碍。Yokoi 等[53]用GC-MS法研究了大鼠模型的糖尿病诊断,发现自发型糖尿病大鼠和正常大鼠相比,天冬酰胺、谷氨酰胺、甘油、犬尿氨酸、甘露糖、牛磺酸、苏氨酸和色氨酸有显著性差异,自发型糖尿病大鼠在12周龄以后色氨酸代谢途径的代谢产物(色氨酸和犬尿氨酸)明显降低,色氨酸及其代谢产物是糖尿病前期潜在的生物标志物,色氨酸的代谢可能是糖尿病治疗干预的一个潜在靶点。Zhu 等[54]利用代谢组学研究了二型糖尿病和糖尿病肾病患者的磷脂代谢物,发现了7大类磷脂中的18种显著的代谢标志物,其中3种是二型糖尿病的代谢标志物,8种是糖尿病肾病的代谢标志物,7种是二者共同的代谢标志物。有2个标志物,PI C18∶0/22∶6和SM dC18∶0/20∶2可用于区分健康人、二型糖尿病和糖尿病肾病人。Huang 等[55]利用代谢组学研究了早期二型糖尿病肾病的代谢标志物,发现了与其相关的6个临床指标、40种代谢物和5个基因。Cordeiro 等[72]利用电喷雾电离质谱(ESI-MS)研究了临床病人卵泡液中的脂质代谢,发现了对照组的代谢物中有磷脂酰甘油磷酸盐、磷脂酰胆碱、磷脂酰丝氨酸、磷脂酰肌醇二磷酸,子宫内膜异位症病人的代谢物中鞘脂类、磷脂酰胆碱含量高,子宫肌瘤病人的代谢物有较高丰度的鞘脂类、磷脂酰胆碱,有助于潜在生物标志物的发现。Zhao 等[73]利用代谢组学方法研究了多囊卵巢综合征患者的卵泡液代谢物变化,认为线粒体功能、氧化还原电势、卵丘细胞的氧化应激是促进卵母细胞受精前生长的必要因素,多囊卵巢综合征患者可能存在线粒体功能紊乱、氧化还原电位不平衡和卵丘细胞的氧化应激增加,从而为多囊卵巢综合征发病机理的研究提供了依据。
2.2 药物研究领域
代谢组学在药物领域的研究主要有药物品质评价、药物效果及作用机制、药物吸收代谢、药物生物转化代谢、药物活性成分研究等。本实验室前期在中药药效评价和药代动力学方面做了一些工作。Liang等[56]利用代谢组学评价了中药复方对大鼠心肌梗塞的作用,发现治疗效果优于人参和单参单方。由于中药复方在心肌能量新陈代谢方面的潜在药效,尿液中三羧酸循环及磷酸戊糖途径的改变表明其可能减少心肌梗塞中心脏的损伤。Xie等[10]研究了大鼠服用知母后的药代动力学,定量了21种代谢中间产物,涉及的代谢通路有水解作用、甲基化作用、去糖基化、糖脂化作用、糖苷结合、硫酸化、脱水作用和异构化反应,揭示了知母药片抗糖尿病的主要多酚组分。Huang等[57]用HPLC-ESI-MS方法研究了异丙嗪和咖啡因联合治疗运动病和抵抗异丙嗪的功效,发现了10多个药物代谢中间产物。Qin等[58]结合血浆代谢组学和脂质组学在大鼠模型上研究了中药清开灵的代谢,结果发现清开灵1 h有15种差异显著的代谢物(p<0.05),2 h有19种差异显著的代谢物(p<0.05),其中与退热功效有关的包括氨基酸、磷脂、溶血磷脂酰胆碱,涉及的代谢通路有氨基酸代谢和脂质代谢。Li等[59]用UPLC-MS代谢组学方法研究了人参皂苷Rg1和Rg2对阿尔茨海默氏症患者的记忆力下降的减缓效果,研究结果显示,在阿尔茨海默氏症大鼠模型发现了11个潜在的生物标志物,与溶血磷脂酰胆碱、次黄嘌呤和鞘脂代谢有关,在给予人参皂苷Rg1和Rg2后这些代谢物水平有恢复,通过脑代谢通路平衡而改变认知功能和形态学变化。Tian等[20]利用大鼠粪便代谢物研究了太子参、丹参、沙参、苦参4味药材对于动脉粥样硬化的治疗效果,在大鼠粪便中发现了16个潜在的生物标志物,其中5个在服用混合中药后发生改变,涉及相关的代谢通路有脂代谢、胆固醇代谢、能量周期和炎症反应。刘琦等[60]利用中医方证代谢组学研究了男仕胶囊对肾阳虚证大鼠的治疗效果,研究确定了11个与肾阳虚证大鼠模型相关的代谢标记物,涉及类固醇激素的生物合成、色氨酸代谢和酪氨酸代谢等途径,并发现了男仕胶囊治疗肾阳虚证的8种药效物质基础。秦昆明等[74]综述了常见中药材的药效作用机制和常见中药复方的配伍规律及作用机制。吴昱铮等[75]综述了典型中药材对于疾病的代谢组学研究。张爱华等[76]综述了代谢组学在中药药理、中医证候、中医方证、中药安全性评价几个方面的应用。
2.3 营养学领域
代谢组学在营养学领域的研究主要包括活性成分的功能研究、膳食模式的评价、营养素需要量的研究、营养代谢的研究、营养流行病学研究等。Liu等[61]利用代谢组学的手段研究了不同季节绿茶的代谢表型;Lee等[62]研究了绿茶对大鼠高脂血症的预防效果,分析了大鼠血清和肝脏代谢组学,发现绿茶能减缓由于高脂饲料诱导的线粒体β氧化异常;Perez-Cornago等[63]利用代谢组学手段评价了对超重、肥胖人群能量控制饮食干预后代谢的变化,发现总饱和脂肪酸、棕榈酸、硬脂酸、单不饱和脂肪酸降低;Martin等[64]研究了不同喂养模式婴儿的代谢特点,发现用配方奶粉喂养婴儿,在其粪便中会发现由蛋白衍生成的短链脂肪酸和氨基酸含量较高,尿液代谢组学揭示了配方奶粉中膳食蛋白质的细菌繁殖过程和蛋白含量增加刺激宿主蛋白质代谢之间的关系。Zhao等[65]利用代谢组学和脂质组学研究了中药复方对糖尿病肾病的疗效,发现中药复方可以保护糖尿病肾病患者的肾脏,发现服用中药复方的糖尿病肾病患者能显著抑制有机毒物的升高及磷脂的减少。
2.4 毒理学领域
由于中毒或代谢失常而引起机体功能紊乱会反映在血液、尿液或组织代谢物中,代谢组学在毒理学方面可以应用在毒性生物标志物筛选、食品毒品毒理和食品安全研究等。Shi等[66]利用代谢组学手段研究了丙烯酰胺的慢性暴露毒性,发现了3种新的生物标志物,认为丙烯酰胺会使脂类和氨基酸代谢紊乱、诱发氧化应激。Yu等[67]用代谢组学研究了全氟辛酸的暴露会引起个体代谢通路的改变,主要影响氨基酸、脂类、碳水化合物和能量代谢,在脑中和肝脏中分别发现有10和18种生物标志物。在大脑中,全氟辛酸会影响神经递质的浓度,包括5-羟色胺、多巴胺、去甲肾上腺素、谷氨酸盐,在肝脏中,因全氟辛酸引发肝中毒,脂类代谢方面出现β脂质氧化、饱和及不饱和脂肪酸的生物合成,花生四烯酸代谢的改变提示了全氟辛酸可能引起肝脏的炎症反应。Qi等[68]在大鼠模型上利用代谢组学方法评价了转基因大米的安全性,结合肠道菌群、血液指标和尿液代谢物综合分析,发现与食用非转基因大米的大鼠相比,服用转基因大米的大鼠的代谢物存在显著性差异,但与对照组相比差异不显著。
2.5 运动医学领域
代谢组学在运动领域常用的研究方向有竞技运动训练方面的问题、运动营养问题、急性慢性运动对代谢的影响、兴奋剂问题、运动对慢性疾病的影响等。Wang等[69]研究了不同运动强度下的尿液代谢变化,结果发现随着运动强度和运动量的增加,乳酸、丙氨酸、三甲胺、丙二酸、牛磺酸、甘氨酸水平下降,随后随着运动量下降,代谢物无显著变化。通过全面评估不同运动形式、不同运动强度、不同运动时间以及不同运动频率对机体所产生的代谢物变化来指导科学合理的运动训练,可以对运动训练方案的设计、运动训练过程的监控和运动效果评价等提供更全面的证据。Mukherjee等[70]用DNA芯片技术和代谢组学的方法研究了老龄化的代谢紊乱问题,选取运动员和非运动员两组,给予不同的运动强度,发现每周连续5 h以上高强度运动员组的基因、能量代谢、脂类代谢、胰岛素信号、心血管功能与非运动员组相比均有显著差异。Yan等[71]用代谢组学手段监测了力量型训练下运动员生理状态的变化,发现不同训练年限的运动员、不同训练时间的代谢物能被显著区分,显著不同的代谢物有丙氨酸、乳酸、焦谷氨酸、半胱氨酸、谷氨酸、柠檬酸、游离脂肪酸、缬氨酸、苯丙氨酸、酪氨酸、谷氨酸盐、苯基丙氨酸、酪氨酸等,涉及到的代谢有糖代谢、氧化应激、能量代谢、脂代谢、氨基酸代谢。
3 展 望
靶标代谢组学和非靶向代谢组学(发现代谢组学)已成为代谢组学应用的两种主要模式。靶标分析具有定量准确、刻画精细、先验知识充分利用的特点,但往往只关注于其中的一点而漏掉其它重要的信息,不具有整体性,过于依赖基于先验知识所预设的目标分析物的合理性。代谢指纹谱属于无歧视的整体表征方法,不要求预先设定目标分析物,可以发现未知生物标志物,但先验知识利用率低,分析方法未针对特定的目标分析物进行优化,难以做到准确的定量分析,且生物样本中常见的高丰度以及质谱离子化效率高的代谢物往往容易检查并可能对某些低丰度、质谱信号响应性差的代谢物检测造成干扰,因此代谢指纹谱的无歧视分析在实际上难以做到,某些重要的低丰度特征信息可能丢失,所发现潜在代谢标志物的针对性较差。因此,对于生物复杂系统的表征需要将代谢指纹谱分析(非靶向代谢组学)与多靶标代谢物定量分析(靶向代谢组学)相结合,整体表征与局部表征相结合,定性与定量分析相结合,代谢分析与临床生化分析相结合,构建多学科参与、多种分析方法整合、大数据分析的临床定量代谢组学平台[77]。定量代谢组学平台既具有全局的视野和对代谢轮廓谱的整体把握,又能实现对特定的代谢途径和生物标志物的精准分析,在多因素复杂疾病和生物复杂系统的研究中具有广阔的应用前景。在分析方法的发展方面,如何使得代谢指纹谱(轮廓谱)测定的代谢物更加全面,如何使得靶标代谢物分析更加精准,特别是低丰度、强极性等难检出化合物的分离分析等,仍然值得重点关注。在代谢组学分析方法的标准化方面,目前Nature Protocol已陆续发表了尿液、脑脊液以及大部分实体组织和细胞样本代谢组学分析的操作规程[14-15,21,33,37],但一些特殊的样本,如精液、卵泡液、羊水、眼泪、粪便、头发等样本,目前还缺乏公认度较高的标准操作规程。对于细胞代谢物的亚细胞代谢组学是将宏观与微观相结合的一个新视角。在数据分析方面,包括非监督分析和有监督分析方法,近年来人工神经网络等一些非线性分析方法在代谢组学中的应用值得关注。如何将代谢组学数据与其它组学技术相结合是一大难点,大数据的挖掘将有助于发现新的有价值信息。随着科学技术的发展,大数据时代的到来,将各种组学技术相整合,必将促进分析测试技术的大发展。
[1] Nicholson J K,Lindon J C.Nature,2008,455:1054.
[2] Gika H G,Zisi C,Theodoridis G,Wilson I D.J.Chromatogr.B,2016,1:15-25.
[3] Klepacki J,Klawitter J,Karimpour-Fard A,Thurman J,Ingle G,Patel D,Christians U.Clin.Biochem.,2016,49(13/14):955-961.
[4] Patti G J,Yanes O,Siuzdak G.Nat.Rev.Mol.CellBiol.,2012,13(4):263-269.
[5] Millan L,Sampedro M C,Sanchez A,Delporte C,Van Antwerpen P,Goicolea M A,Barrio R J.J.Chromatogr.A,2016,8:67-77.
[6] Zhao L J,Zhao A,Chen T L,Chen W L,Liu J W,Wei R M,Su J,Tang X L,Liu K,Zhang R,Guo X X,Panee J,Qiu M F,Jia W.J.ProteomeRes.,2016,15(7):2327-2336.
[7] Ranninger C,Schmidt L E,Rurik M,Limonciel A,Jennings P,Kohlbacher O,Huber C G.Anal.Chim.Acta,2016,930:13-22.
[8] Roy C,Tremblay P Y,Bienvenu J F,Ayotte P.J.Chromatogr.B,2016,14:40-49.
[9] Zhao C X,Xu G W.J.Anal.Sci.(赵春霞,许国旺.分析科学学报),2014,30(5):761-766.
[10] Xie Y Y,Wang X M,Wang S H,Wang Y M,Tian H F,Yuan Y S,Li H Y,Liang Q L,Luo G A.J.Chromatogr.B,2015,1026:134-144.
[11] Cheng Y,Liu Y M,Huang F J,Chen T L,Zheng X J,Zhao A H,He P G,Jia W.Chem.J.Chin.Univ.(成玉,刘玉敏,黄凤杰,陈天璐,郑晓皎,赵爱华,何品刚,贾伟.高等学校化学学报),2013,34(1):77-83.
[12] Want E J,Coen M,Masson P,Keun H C,Pearce J T,Reily M D,Robertson D G,Rohde C M,Holmes E,Lindon J C,Plum R S,Nicholson J K.Anal.Chem.,2010,82(12):5282-5289.
[13] Sarafian M H,Lewis M R,Pechlivanis A,Ralphs S,McPhail M J,Patel V C,Dumas M E,Holmes E,Nicholson J K.Anal.Chem.,2015,87(19):9662-9670.
[14] Yuan M,Breitkopf S B,Yang X,Asara J M.Nat.Protoc.,2012,7(5):872-881.
[15] Want E J,Masson P,Michopoulos F,Wilson I D,Theodoridis G,Plumb R S,Shockcor J,Loftus N,Holmes E,Nicholson J K.Nat.Protoc.,2013,8(1):17-32.
[16] Masson P,Alves A C,Ebbels T M,Nicholson J K,Want E J.Anal.Chem.,2010,82(18):7779-7786.
[17] Vorkas P A,Isaac G,Anwar M A,Davies A H,Want E J,Nicholson J K,Holmes E.Anal.Chem.,2015,87(8):4184-4193.
[18] Vorkas P A,Shalhoub J,Isaac G,Want E J,Nicholson J K,Holmes E,Davies A H.J.ProteomeRes.,2015,14(3):1389-1399.
[19] Su X L,Wang N,Chen D Y,Li Y N,Lu Y F,Huan T,Xu W,Li L J.Anal.Chim.Acta,2016,903:100-109.
[20] Tian F,Gu L,Si A Y,Yao Q B,Zhang X W,Zhao J H,Hu D D.J.Chromatogr.B,2015,15:140-148.
[21] Chan E C,Pasikanti K K,Nicholson J K.Nat.Protoc.,2011,6(10):1483-1499.
[22] Fattuoni C,Palmas F,Noto A,Barberini L,Mussap M,Grapov D,Dessi A,Casu M,Casanova A,Furione M,Arossa A,Spinillo A,Baldanti F,Fanos V,Zavattoni M.Clin.Chim.Acta,2016,460:23-32.
[23] Sulek K,Han T L,Villas-Boas S G,Wishart D S,Soh S E,Kwek K,Gluckman P D,Chong Y S,Kenny L C,Baker P N.Theranostics,2014,4(9):953-959.
[24] Tsutsui H,Maeda T,Min J Z,Inagaki S,Higashi T,Kagawa Y,Toyo’oka T.Clin.Chim.Acta,2011,412(11/12):861-872.
[25] Wang H,Liang X P,Luo G A,Ding M Y,Liang Q L.Mol.Biosyst.,2016,12(7):2257-2264.
[26] Twohig M,Shockcor J P,Wilson I D,Nicholson J K,Plumb R S.J.ProteomeRes.,2010,9(7):3590-3597.
[27] Li H F,Zhang Y,Lin J M.Sci.Sin.:Chim.(李海芳,张英,林金明.中国科学),2014,44(5):777-783.
[28] Kadar H,Dubus J,Dutot J,Hedjazi L,Srinivasa S,Fitch K V,Grinspoon S K,Nicholson J K,Dumas M E,Gauguier D.ArchBiochem.Biophys.,2016,597:12-20.
[29] Sen A,Knappy C,Lewis M R,Plumb R S,Wilson I D,Nicholson J K,Smith N W.J.Chromatogr.A,2016,3:141-155.
[30] Benton H P,Want E,Keun H C,Amberg A,Plumb R S,Goldfain-Blanc F,Walther B,Reily M D,Lindon J C,Holmes E,Nicholson J K,Ebbels T M.Anal.Chem.,2012,84(5):2424-2432.
[31] Hou W E,Zhong D M,Zhang P T,Li Y M,Lin M N,Liu G H,Yao M C,Liao Q F,Xie Z Y.J.Chromatogr.A,2016,15:207-217.
[32] Dunn W B,Broadhurst D,Begley P,Zelena E,Francis-McIntyre S,Anderson N,Brown M,Knowles J D,Halsall A,Haselden J N,Nicholls A W,Wilson I D,Kell D B,Goodacre R.Nat.Protoc.,2011,6(7):1060-1083.
[33] Want E J,Wilson I D,Gika H,Theodoridis G,Plumb R S,Shockcor J,Holmes E,Nicholson J K.Nat.Protoc.,2010,5(6):1005-1018.
[34] Wang Y,Caldwell R,Cowan D A,Legido-Quigley C.Anal.Chem.,2016,88(4):2243-2249.
[35] Garcia-Perez I,Lindon J C,Minet E.Bioanalysis,2014,6(20):2733-2749.
[36] Stipetic L H,Dalby M J,Davies R L,Morton F R,Ramage G,Burgess K E.Metabolomics,2016,12:75.
[37] Martano G,Delmotte N,Kiefer P,Christen P,Kentner D,Bumann D,Vorholt J A.Nat.Protoc.,2015,10(1):1-11.[38] Halama A,Kulinski M,Kader S A,Satheesh N J,Abou-Samra A B,Suhre K,Mohammad R M.J.Transl.Med.,2016,14(1):140.
[39] Martel J,Wu C Y,Hung C Y,Wong T Y,Cheng A J,Cheng M L,Shiao M S,Young J D.Nanoscale,2016,8(10):5537-5545.
[40] Dudzik D,Revello R,Barbas C,Bartha J L.J.ProteomeRes.,2015,14(3):1432-1444.
[41] Karamichos D,Zieske J D,Sejersen H,Sarker-Nag A,Asara J M,Hjortdal J.Exp.EyeRes.,2015,132:1-8.
[42] Chen L Y,Zhou L,Chan E C,Neo J,Beuerman R W.J.ProteomeRes.,2011,10(10):4876-4882.
[43] Telu K H,Yan X,Wallace W E,Stein S E,Simon-Manso Y.RapidCommun.MassSpectrom.,2016,30(5):581-593.[44] Mahieu N G,Genenbacher J L,Patti G J.Curr.Opin.Chem.Biol.,2016,30:87-93.
[45] Chang H Y,Chen C T,Lih T M,Lynn K S,Juo C G,Hsu W L,Sung T Y.PLoSOne,2016,11(1):e0146112.
[46] Niu W,Knight E,Xia Q,McGarvey B D.J.Chromatogr.A,2014,29:199-206.
[47] Daly R,Rogers S,Wandy J,Jankevics A,Burgess K E,Breitling R.Bioinformatics,2014,30(19):2764-2771.
[48] Xia J,Wishart D S.Nat.Protoc.,2011,6(6):743-760.
[49] Kastenmuller G,Romisch-Margl W,Wagele B,Altmaier E,Suhre K.J.Biomed.Biotechnol.,2011,1-7.
[50] Kong H W,Dai W D,Xu G W.Chin.J.Chromatogr.(孔宏伟,戴伟东,许国旺.色谱),2014,32(10):1052-1057.
[51] Musharraf S G,Siddiqui A J,Shamsi T,Naz A.Hematol.Oncol.,2016,1-7.
[52] Ciavardelli D,Piras F,Consalvo A,Rossi C,Zucchelli M,Di Ilio C,Frazzini V,Caltagirone C,Spalletta G,Sensi S L.Neurobiol.Aging,2016,43:1-12.
[53] Yokoi N,Beppu M,Yoshida E,Hoshikawa R,Hidaka S,Matsubara T,Shinohara M,Irino Y,Hatano N,Seino S.Metabolomics,2015,11(5):1277-1286.
[54] Zhu C,Liang Q L,Hu P,Wang Y M,Luo G A.Talanta,2011,85(4):1711-1720.
[55] Huang M,Liang Q L,Li P,Xia J F,Wang Y,Hu P,Jiang Z T,He Y X,Pang L Q,Han L D,Wang Y M,Luo G A.Mol.Biosyst.,2013,9(8):2134-2141.
[56] Liang X P,Chen X,Liang Q L,Zhang H Y,Hu P,Wang Y M,Luo G A.J.ProteomeRes.,2011,10(2):790-799.
[57] Huang M,Gao J Y,Zhai Z G,Liang Q L,Wang Y M,Bai Y Q,Luo G A.J.Pharm.Biomed.Anal.,2012,62:119-128.
[58] Qin L M,Zhang Z X,Guo M X,Zhang Q Q,Wang Q,Lu Z W,Zhao H Z,Liu Y H,Fu S,Wang M L,Gao X Y.Chem.Biol.Interact,2016,254:24-33.
[59] Li N J,Liu Y,Li W,Zhou L,Li Q,Wang X Q,He P.J.GinsengRes.,2016,40(1):9-17.
[60] Liu Q,Zhao H W,Zhang A H,Sun H,Zhou X H,Nan Y,Kong L,Guan Y,Zou S Y,Ma Z H,Wang X J.J.Chin.Mater.Med.(刘琦,赵宏伟,张爱华,孙晖,周晓航,南洋,孔玲,关瑜,邹士玉,马忠华,王喜军.中国中药杂志),2016,41(15):2901-2914.
[61] Liu J W,Zhang Q F,Liu M Y,Ma L F,Shi Y Z,Ruan J Y.J.Agric.FoodChem.,2016,64(16):3302-3309.
[62] Lee L S,Choi J H,Sung M J,Hur J Y,Hur H J,Park J D,Kim Y C,Gu E J,Min B,Kim H J.Mol.Nutr.FoodRes.,2015,59(4):784-794.
[63] Perez-Cornago A,Brennan L,Ibero-Baraibar I,Hermsdorff H H,O’Gorman A,Zulet M A,Martinez J A.J.Physiol.Biochem.,2014,70(2):593-602.
[64] Martin F P,Moco S,Montoliu I,Collino S,Da Silva L,Rezzi S,Prieto R,Kussmann M,Inostroza J,Steenhout P.Pediatr.Res.,2014,75(4):535-543.
[65] Zhao T,Zhang H J,Zhang X L,Zhao T T,Lan H Y,Liang Q L,Luo G A,Li P.J.Ethnopharmacol.,2015,166:31-41.
[66] Shi H D,Hu L Y,Chen S,Bao W,Yang S,Zhao X J,Sun C H.Xenobiotica,2016,27:1-11.
[67] Yu N Y,Wei S,Li M Y,Yang J P,Li K,Jin L,Xie Y W,Giesy J P,Zhang X W,Yu H X.Sci.Rep.,2016,6:1-10.
[68] Qi X Z,Chen S Y,Sheng Y,Guo M Z,Liu Y F,He X Y,Huang K L,Xu W T.FoodChem.Toxicol.,2015,76:1-10.
[69] Wang F Q,Han J,He Q,Geng Z F,Deng Z W,Qiao D C.J.Anal.MethodsChem.,2015,1-9.
[70] Mukherjee K,Edgett B A,Burrows H W,Castro C,Griffin J L,Schwertani A G,Gurd B J,Funk C D.PLoSOne,2014,9(3):e92031.
[71] Yan B,A J,Wang G J,Lu H L,Huang X P,Liu Y,Zha W B,Hao H P,Zhang Y,Liu L S,Gu S H,Huang Q,Zheng Y T,Sun J G.J.Appl.Physiol.,2009,106(2):531-538.
[72] Cordeiro F B,Cataldi T R,Perkel K J,do Vale Teixeira da Costa L,Rochetti R C,Stevanato J,Eberlin M N,Zylbersztejn D S,Cedenho A P,Turco E G.J.Assist.Reprod.Genet.,2015,32(12):1817-1825.
[73] Zhao H C,Zhao Y,Li T J,Li M,Li J S,Li R,Liu P,Yu Y,Qiao J.FreeRadic.Biol.Med.,2015,86:295-307.
[74] Qin K M,Wang B,Chen L W,Zhang M S,Yang G M,Su Y C,Cai B C.Chin.Mater.Med.(秦昆明,王彬,陈林伟,章茂森,杨光明,束雅春,蔡宝昌.中国中药杂志),2014,39(16):3010-3017.
[75] Wu Y Z,Wang G J,Hao H P.Chin.Pharm.Univ.(吴昱铮,王广基,郝海平.中国药科大学学报),2014,45(2):129-135.
[76] Zhang A H,Wang X J.Mod.Tradit.Chin.Med.Mater.Med.(张爱华,王喜军.世界科学技术_中医药现代化),2013,15(4):643-647.
[77] Luo G A,Wang Y M,Liang Q L,Liu Q F.SystemsBiologyforTraditionalChineseMedicine.New York:A John Wiley & Sons,2012:191-194.
Advance in Metabolomics Based on Mass Spectrometry
REN Xiang-nan,LIANG Qiong-lin*
(Department of Chemistry,Analysis Center,Tsinghua University,Beijing 100084,China)
Mass spectrometry(MS) is one of the most important analytical technologies for metabolomics.In recent years,metabolomics based on MS has been widely used in disease diagnosis,drug effect research,nutrition,toxicology,sports medicine,etc.This paper reviews the sample pretreatment,analytical tools,data processing method and main application fields in the past five years.The development tendency of metabolomics in future is also proposed.
mass spectrometry(MS);metabolomics;advance;review
2016-09-30;
2016-11-28
国家自然科学基金项目(U1333132,81230079);科技部国际科技合作项目(2011DFA31860);国家科技重大专项(2014ZX09304307001,2014ZX09201022-004,2014ZX09201022-005)
10.3969/j.issn.1004-4957.2017.02.002
O657.63;O629.73
A
1004-4957(2017)02-0161-09
*通讯作者:梁琼麟,研究方向:生命分析化学,Tel:010-62772263,E-mail:liangql@tsinghua.edu.cn

