基于Docker Swarm集群的药物基因组解读及用药指导软件设计*
孙涛,徐秀林,丁瑞丰,胡秀枋
(上海理工大学医疗器械与食品学院,上海 200093)
1 引 言
药物基因组学(pharmacogenomics,PGx)是研究药物反应与遗传基因之间关系的一门学科。临床上PGx通过药物基因变异信息来指导用药剂量,以实现个体化合理用药,减少或者避免药物的不良反应,改善患者预后,实现精准治疗[1]。
近年来,随着人类基因组精准医学的发展,国内外均有提出精准医学计划和实践应用。2015年1月20日,美国国情咨文中宣布了“精准医学计划”,旨在借助精准医学为临床提供科学的治疗依据[2]。同时依托先进的计算机技术,欧美国家率先建立了一系列重要的PGx数据库和临床信息系统,如美国St.Jude儿童研究院建立了药物基因警报系统,该系统能及时添加患者高危基因分型及潜在的药物不良反应,制定相应药物治疗方案,辅助临床决策[3]。2016年,我国颁布了“精准医学计划”。之后,国家卫生计生委推出了两套药物基因组学试行指南,以规范临床用药规则和精准医学行业市场[4]。当前,通过利用先进的计算机技术来整合PGx的相关指南,建立实时更新的临床信息系统,是广大医学工作者迫切关注的问题。
本研究开发了一种基于Docker Swarm集群的药物基因组解读及用药指导软件。该软件采用国际权威的遗传药理学与药物基因组学数据库(PharmGKB)及美国食品药品监督管理局(FDA)药物标签数据库的数据作为PGx知识库内容[5];采用Docker Swarm集群负载均衡算法,实现药物基因检测位点的快速解读,为临床用药提供快捷、有效的指导。
2 软件总体设计
2.1 开发平台构建
本软件开发平台为Ubuntu 16.04 LTS系统,前端Web页面视图使用超文本标记语言5(HTML 5)[6]、层叠样式表3(CSS 3)[7]、JavaScript脚本等实现,使用MySQL5.7作为数据库管理系统,使用超文本预处理器5.6(PHP 5.6)和RScript作为服务器端脚本,使用Docker Swarm集群搭建集群环境。
2.2 软件架构
通过创建三个Docker Engine实例节点(Node):Manager Node、Worker1 Node和Worker2 Node来构建Swarm集群。软件设计框架见图1,客户端登录浏览器,输入Web域名向服务器端发送HTTP请求,域名指向服务器集群中的Manager Node。Manager Node将HTTP请求根据负载均衡算法下发至Worker Node,HTTP 代理服务器Nginx一旦接收到Manager Node下发的HTTP请求,立即处理静、动态文件请求。其中PHP动态请求包括通过应用程序函数接口(API)调用后台MySQL数据库中的药物基因组数据库,以及异步调用RScript基因组解读脚本。静、动态请求响应结束后,Nginx再将数据返回到客户端浏览器页面。

图1 软件设计框架图Fig.1 Software design framework
3 关键技术
3.1 基于LNMP的服务器端设计与实现
服务器端是通过Linux+Nginx+MySQL+PHP的组合方式实现的,设计与实现见图2。前端表单数据提交至服务器端,由PHP通过MySQLi连接数据库来处理表单,同时PHP调用RScript位点解读脚本进行基因位点与数据库进行比对计算,计算流程见图3,首先计算测序深度(Depth)和变异质量值(QUAL),将位点测序深度(Depth)小于5或大于100以及位点变异质量值小于20的SNP位点过滤,得到可靠性较高的SNP位点,然后与药物基因组数据库比对计算,得到药物作用的基因突变位点信息,再将此突变位点与药物数据库比对计算,得到用药指导方案,实现药物基因位点的解读。其中位点解读结果及用药指导方案等信息可在患者报告中获得并提供PDF格式文件的下载,患者报告见图4。

图2 服务器端设计与实现图Fig.2 Server-side design and implementation diagram
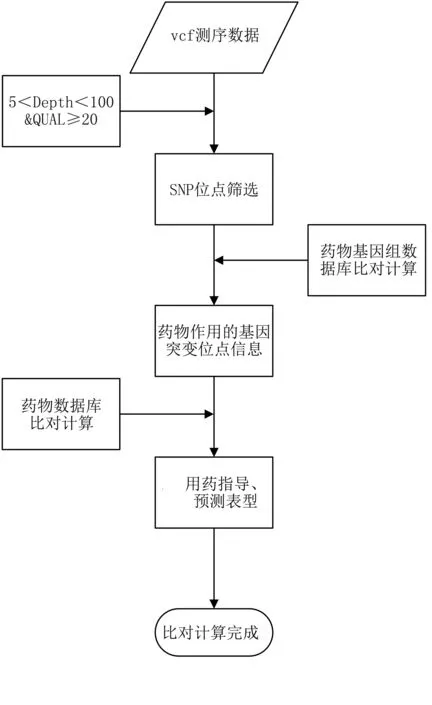
图3 药物基因位点解读流程图Fig.3 Drug gene locus interpretation flow chart

图4 患者报告图Fig.4 Patient report diagram of software system
3.2 基于Docker Swarm的服务器集群设计与实现
由于药物基因库内数据种类繁多、数量庞大,利用单主机架构的软件系统存在响应时间长、请求队列排队时序久,导致位点解读效率较低。本软件使用Docker Swarm搭建服务器集群,实现了软件系统负载均衡,提高了位点解读效率。
Docker是一种轻量级的虚拟容器技术[8],具有启动快,资源占用少,扩展性强,管理方便等优点。Swarm是一款Docker集群管理工具[9],它将多台部署有Docker服务的主机抽象成一个虚拟集群,提高了Docker的跨主机能力,减少了Docker与主机的耦合。
3.2.1基于IPVS的动态负载均衡算法 IPVS(IP virtual server,IPVS)是Linux虚拟服务器实现负载均衡的关键技术[10]。本软件使用的负载均衡算法是IPVS的加权最小链接动态负载均衡算法,通过计算服务器节点连接数与服务器节点权重的比值,将新请求分配给比值最小的服务器节点来实现动态负载均衡。服务器节点连接数计算公式,见式(1):
AC=ac×K+Iac
(1)
其中,AC表示服务器节点连接数,ac表示Swarm负载均衡器上记录的活动连接数,Iac表示Swarm负载均衡器上记录的非活动连接数,K为连接系数,一般取256。
最小比值计算公式见式(2):
(2)
其中,AC(Ni)表示当前服务器节点连接数,W(Ni)表示当前服务器节点权重,Pi表示分配请求的服务器节点。在本软件的集群设计中,由于两个Work Node均为性能相同的Docker Machine,故设置两个Work Node的节点权重值相等。加权最小链接动态负载均衡算法实现过程见图5,Swarm集群共有两个工作节点,分别为Worker1 Node和Worker2 Node,在图5中分别表示为S0和S1,Wm表示第m个服务器节点的权重值,Cm表示第m个服务器节点的连接数。
3.2.2Swarm集群设计与实现 利用Docker Machine创建一个Manager Node以及Worker1 Node、Worker2 Node构建Swarm集群,见图6。 其中Manager Node负责管理整个Swarm集群的容器资源,Worker1 Node和Worker2 Node负责接收并执行Manager Node下发的任务。集群使用docker compose.yml配置文件创建了两个Stack实例:My_web和Monitor。My_web包含三个服务项,分别为PHP-R、MySQL、Nginx服务,是软件的Web实例服务。Monitor包含三个服务项,分别为Cadvisor、Grafana、Influx服务,是软件的监控实例服务。集群内部还创建了Overlay网络(Overlay Network),通过Overlay Network,不同的Node之间以及容器之间可以实现通信交互。
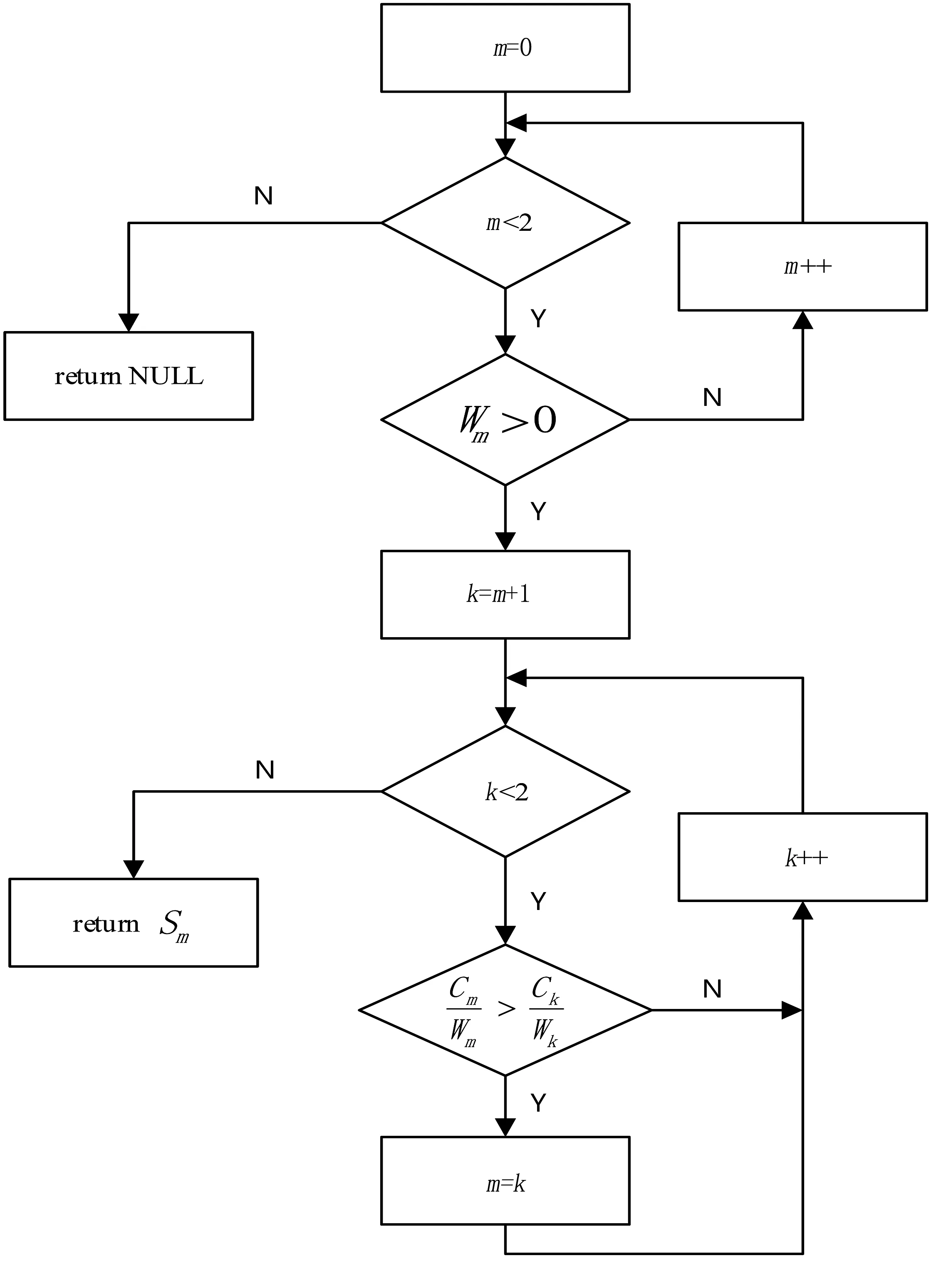
图5 IPVS加权最小链接动态负载均衡算法实现图Fig.5 Implementation diagram of IPVS weighted minimum link dynamic load balancing algorithm
4 软件有效性分析与测试
4.1 有效性分析
甲氨蝶呤(methotrexate,MTX)是一种抗叶酸类抗肿瘤化疗药物,它在多聚谷氨酸合成酶(FPGS)的作用下转化为甲氨蝶呤多聚谷氨酸盐(MTXPGs),可使DNA合成受阻,促进细胞凋亡[11]。较大剂量的MTX在抗肿瘤治疗中虽有较好的疗效,但不同基因位点的基因多态性会导致不同个体产生不同程度的药物不良反应,例如亚甲基四氢叶酸还原酶(MTHFR)基因型已被证实与MTX不良反应中的白细胞减少、骨髓抑制等密切相关[12]。
王轶睿等[12]在未通过药物基因解读用药的基础上,利用大剂量甲氨蝶呤对112名恶性肿瘤患者进行化疗,并按照2009年公布的常见不良反应事件评价标准4.0版,统计了患者在化疗后7 d内出现的各类不良反应在5种不同等级水平上的不良反应率,其中骨髓抑制在2级及2级以上的不良反应率为52.7%,白细胞减少的不良反应率为74.1%。

图6 Swarm集群设计与实现图Fig.6 Swarm cluster design and implementation diagram
经过华山医院伦理委员会(编号为KY-2015-256)批准,采用本软件对9例临床肿瘤患者进行研究。针对甲氨蝶呤药物,对比经过本软件药物基因解读用药与文献[12]未通过药物基因解读用药(以下简称文献)的不良反应统计结果,进行有效性验证。患者信息如下:男性5名,女性4名;年龄均大于18周岁;已确诊为左腺肺癌1例,乙状结肠癌1例,肝癌1例,甲状腺癌1例,肺癌1例,胃肠道间质瘤2例,胃癌2例;均无甲氨蝶呤严重过敏史、骨髓抑制等MTX不良反应病史,白细胞数正常;未因其他恶性肿瘤接受过较大剂量甲氨蝶呤化疗。将患者基因测序文件上传至本软件得到解读结果,见表1。9例患者的MTHFR基因在rs1476413、rs4846051、rs1801131、rs1801133四个位点上均存在基因型突变。对不同突变,采用解读结果提供的用药指导方案为患者提供化疗。化疗结束后,将9例患者7 d内的不良反应随访信息统计结果与文献结果进行对比,见表2。结果表明,经过本软件药物基因解读进行用药指导后,患者均未发生骨髓抑制、白细胞数减少等不良反应,与未经药物基因解读用药的结果比较,两种不良反应率差异均具有统计学意义(P<0.05),证明本软件具有良好的有效性。

表1 甲氨蝶呤药物基因位点解读结果Table 1 Interpretation results of MTIX drug gene loci

表2 甲氨蝶呤化疗不良反应率对比Table 2 Comparison of adverse reaction rate of methotrexate in chemotherapy
4.2 软件测试
为了验证本软件的高并发处理能力,使用Apache Jmeter压力测试工具模拟并发情况,以95%用户响应时间(ms)、吞吐量(请求数/s)两种性能指标比较本研究开发的基于Swarm集群架构的系统与基于单主机架构的系统在处理高并发时的性能差异,其中Swarm集群系统与单主机系统的CPU能力资源均为3 GHz,内存能力资源均为3 GB。设定用户在线并发数为100,药物基因大数据分析解读任务的并发数以100、150、200、250、300的序列递增,每个过程执行50次迭代,测试结果见表3、表4。
由表3、表4可知,在相同并发压力下,Swarm架构的软件响应时间较单主机架构的软件缩短了一半左右,吞吐量明显高于单主机架构的软件,说明对于单主机架构,由于多个并发项处于相同进程,任务并发请求和用户并发请求争抢主机资源,出现请求进入队列等待,响应时间过长,并发请求数量受限。而Swarm架构通过内部IPVS动态负载均衡算法由多个节点服务器并行响应,响应时间较单主机架构明显缩短,显著提高了软件解读基因位点任务的吞吐量。
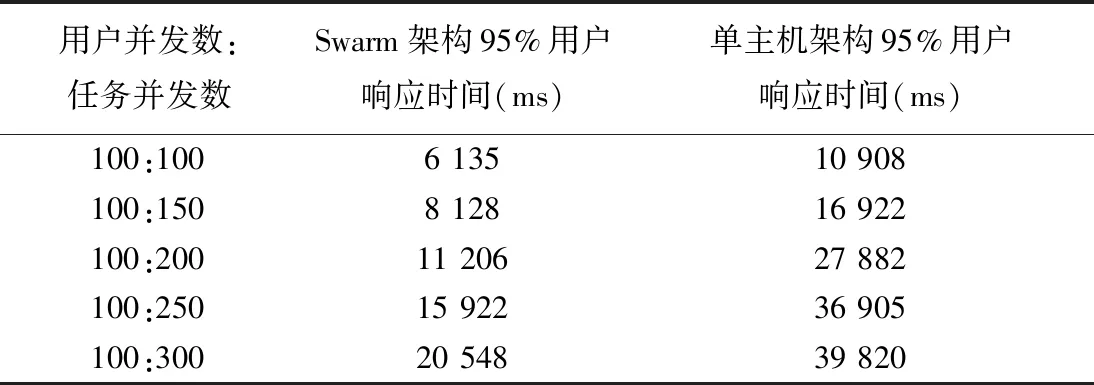
表3 相同并发情况下的Swarm集群与单主机软件系统95%用户响应时间对比Table 3 The 95% user′s response time comparison of Swarm cluster and single host system under the same concurrency

表4 相同并发情况下的Swarm集群与单主机吞吐量对比Table 4 Throughput comparison of Swarm with single host architectures under the same concurrency situation
5 总结与讨论
目前,由于PGx数据庞大且更新速度快,采用先进的计算机技术整合PGx数据库,建立具有快速数据处理能力的临床信息系统是十分迫切的。本研究提出的基于Docker Swarm集群的药物基因组解读及用药指导软件以多主机网络形式快速部署Web服务,在应对高并发压力时具有良好的负载均衡能力。同时,临床测试研究证明本软件具有良好的有效性。
然而,本研究设计的系统仍然存在一些不足。受硬件设备条件限制,集群服务器节点未采用实体物理机来搭建集群。另外,在软件系统微服务方面还需要分析优化,尽量减少Manger Node参与实例服务,充分发挥Manger Node管理容器资源的优势,这些均有待于下一步研究。

