1株携带质粒介导喹诺酮耐药基因qnrS1和qepA1的多耐药沙门菌的基因特点分析
邢 燕,钱玉春,张文艳,胡忠旺,孙 永
沙门菌是一类重要的人兽共患病原体,血清型多达2 000多种,其中绝大多数血清型既可存在于动物、环境中,也可传播至人。沙门菌感染会引起腹泻、发热等症状[1],属于自限性疾病,但对于细菌入血的少数重症患者,必须使用抗生素治疗。
喹诺酮类抗生素是成人治疗沙门菌感染的一线药物之一。沙门菌对喹诺酮类药物产生耐药对于治疗非常不利。目前沙门菌对一代药物萘啶酸已表现出明显耐药,对第三代药物环丙沙星的敏感性仍较高,耐药率大约在2%~3%[2-3]。
对喹诺酮类药物耐药机制的研究表明,喹诺酮耐药决定区的基因突变,即位于细菌染色体上的gyrA、gyrB基因的突变,是导致喹诺酮耐药的主要原因[4]。随着研究的深入,由质粒介导的耐药相关基因,如qnr、aac(6′)-Ib-cr、qepA等都被发现与喹诺酮耐药有关[5-7]。研究发现质粒介导的耐药通常引起喹诺酮类药物的MIC浓度降低或药物敏感性下降,不能达到临床耐药水平[8-10]。但Toyotakasato[11]等人于2011年报道了一例临床分离的大肠杆菌HUE1,它不含染色体编码基因的耐药突变,仅有质粒上携带的基因qnrS1和oqxAB并表现出环丙沙星耐药。
本实验室在对临床分离沙门菌菌株的抗生素耐药筛查中,发现一株多耐药菌株SM846,它对喹诺酮类药物萘啶酸和环丙沙星都耐药,在PCR研究耐药机制时,没有检测到gyrA和gyrB基因的突变。随后对它进行了全基因组测序及序列比较分析,来研究它的耐药机制。
1 材料与方法
1.1菌株来源及分离鉴定 沙门菌SM846于2014年从1名11个月的婴儿腹泻粪便标本中分离。沙门菌从沙门显色培养基(购于上海科马嘉)中分离出单个菌落,纯菌落使用梅里埃的微生物鉴定系统(购于梅里埃VITEK2)鉴定,经血清学(购于宁波天润)鉴定为汤普逊沙门菌(S.thompson)。
1.2药敏试验 使用thermo公司的SensititreAIM仪器和基于微量肉汤法定制的药敏板(购于上海星佰),测定沙门菌SM846对14种抗生素的最低抑菌浓度(MIC)。试验菌株培养至对数期,用无菌生理水调整菌悬液浓度至0.5McF,取50 μL菌悬液加入到11 mL CAMHT肉汤中,将稀释后的菌液(细菌数1×105~1×106)接种在药敏板上。培养24 h,测定结果。使用大肠杆菌ATCC25922为试验的质控菌株。14种抗生素分别为:氨苄西林(AMP)、氨苄西林/舒巴坦(AMS)、四环素(TET)、氯霉素(CHL)、头孢唑林(CFZ)、环丙沙星(CIP)、甲氧苄啶/磺胺甲恶唑(SXT)、头孢他啶(CAZ)、亚胺培南(IPM)、萘啶酸(NAL)、头孢西丁(CFX)、头孢噻肟(CTX)、庆大霉素(GEN)、阿奇霉素(AZM)。根据CLSI的判读标准确定MIC值。
1.3全基因组测序 对于组装基因组的菌株,提供8~10 μg高质量的基因组DNA,用Covaris g-TUBE随机打断成10 kb左右的片段,随后用DNA Template Prep试剂盒构建SMRTbell文库。构建好的SMRTbell文库结合V3测序引物和聚合酶采用自由扩散的方式加入到PacBio Sequel平台的测序芯片中。测序过程由金唯智公司处理和分析。
1.4测序后数据分析 使用Prodigal软件(version 3.02)进行染色体编码基因预测[12];与Rfam(version 12.0)数据库进行序列比对获得非编码RNA结果[13]。使用在线网站RAST对质粒进行注释[14]。使用resfinder[15]与CARD[16]进行耐药基因的预测分析。使用SISTR[17]预测血清型,PlasmidFinder预测质粒类型[18]。使用SnapGene(from GSL Biotech; available at snapgene.com)对质粒的编码基因进行展示。基因组线图使用CGView1.3.4[19]生成。质粒上耐药基因图由ApE软件制作(available at https://jorgensen.biology.utah.edu/wayned/ape/)。质粒序列使用clustalw进行比对(available at https://www.ebi.ac.uk/Tools/msa/clustalo/),进化树由软件mega-X根据NJ法生成[20]。
2 结 果
2.1测序菌株信息 SM846的全基因组包含1条染色体和1条质粒。染色体全长4 754 945 bp,GC含量52.3%,预测包含编码序列4 425个,含64个rRNA,89个tRNA,其它RNA178个。质粒全长152 940 bp,GC含量53.4%,预测包含编码序列193个,不含RNA。染色体和质粒基因组已提交到NCBI,序列号分别为:CP028729、CP029249。基于SISTR数据库预测的血清型为汤普逊,与血清学鉴定结果一致。基于PlasmidFinder质粒基因组数据库预测的质粒类型为IncA/C2。SM846测序原始数据见表1。

表1 SM846测序原始数据统计信息
2.2 药敏试验结果与耐药基因分布
2.2.1药敏试验 SM846对IPM、GEN敏感,对其它12种抗生素都高度耐药,包括喹诺酮药物萘啶酸和环丙沙星。虽然SM846对喹诺酮耐药,PCR检测gyrA和gyrB基因的耐药突变,没有获得任何突变位点的结果。对SM846进行了基因组测序以研究它的喹诺酮耐药机制(详见表2)。

表2 SM846药敏试验结果及相关耐药基因
2.2.2耐药基因分布 将SM846序列与ResFinder数据库比对,注释结果显示,SM846的染色体不含有任何和已知耐药有关的基因和突变,它有3个parC蛋白的未知突变,分别是p.T57S、p.T255S、p.V657I。它的质粒p846携带13个耐药相关基因,分别是耐氨基糖苷类的aadA2、APH(6)-1d、APH(3″)-1b;耐氯霉素的floR;耐β-内酰胺类的blaTEM-1、blaCMY-2;耐喹诺酮类的qnrS1和qepA1;耐大环内酯类mph(A);耐磺胺类的sul1、sul2;耐四环素类tet(A);以及耐甲氧嘧啶的dfrA12(见表1)。由于ResFinder和CARD对耐药基因的筛选标准有差别,ResFinder比对的是明确并且特异地与耐药相关的耐药基因,而CARD数据库的比对结果中还包括外排泵系统和基因表达调控系统等非特异的基因。CARD数据库的预测结果,除了包含ResFinder中的耐药基因,还有30个位于染色体上的基因,分别属于RND抗生素外排泵、ABC抗生素外排泵、MFS抗生素外排泵、以及上述外排泵基因上下游的正负调控基因。
2.3 耐多药质粒信息
2.3.1由于SM846的耐药基因,包括与喹诺酮耐药相关的基因,都在质粒上,我们对质粒p846进行了进一步的分析。p846共编码193个基因,去除小于100 bp和没有明确功能的,还有159个。其中13个基因与耐药相关。
将p846的序列与NCBI上其它公开数据比对,可以得到很多相似度大于99%的质粒。表3中列出了代表性的13条质粒,分别是4条相似度最高的和9条覆盖度最高的序列。用ResFinder注释上述质粒的耐药基因,获取它们的耐药基因的携带情况(表3)。
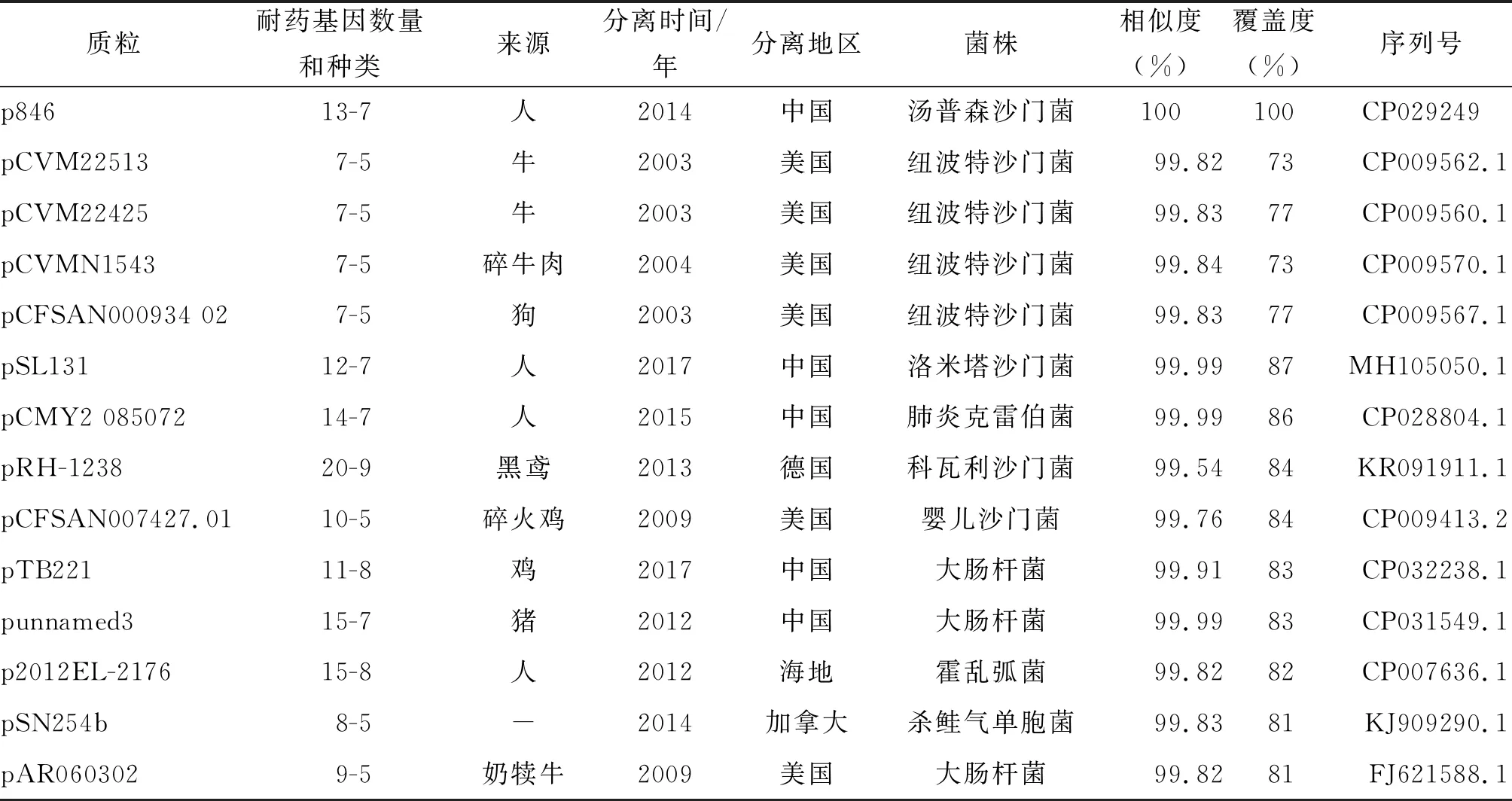
表3 p846与相似质粒比较
上述质粒中,分离时间早的,如分离自2003年和2004年的质粒。耐药性都相对较弱,2013年及以后分离出的则表现出很强的耐药性;质粒菌株来自于人的,耐药性也强,由于中国分离出的质粒都是2010年以后且自人体分离出,因此中国分离的质粒都显示出了很强的耐药性。以质粒的序列进行进化分析,可以得到2个进化簇(见图1)。与p846亲缘关系近的质粒有3个,分别是pCVMN1543、pCFSAN00093402和pRH-1238。其中p846和pRH-1238耐药性极强,pCVMN1543、pCFSAN000934-02耐药性一般;p846来自于人分离的菌株,其它3个来自于动物分离的菌株;p846来自中国,pRH-1238来自德国,pCVMN1543和pCFSAN000934-02来自美国。

进化分析序列基于以下质粒序列:p846,pSN254b,p2012EL-2176,pSL131,punnamed3,pTB221,pCVM22425,pCMY2 085072,pCFSAN007427 01,pCVM22513,pCVMN1543,pCFSAN000934 02,pRH-1238。
为了更深入研究p846的遗传情况,将p846与上面的质粒进行序列对比分析。从对比的圈图,可以看出p846含有2个较为特殊的区域,P1和P2。pCFSAN007427.01也含有P1区域,它是一条分离自2009年的美国的沙门菌的质粒(见图2)。在NCBI搜索P1序列,发现很多宿主细菌的质粒中都含有这段序列,如沙门菌、志贺菌、肺炎链球菌等。与P1序列的广泛存在不同,P2序列特异性较强,它与NCBI中的序列没有很好的匹配。QepA1和qnrS1基因位于P2序列中,它们正是质粒中与喹诺酮耐药相关的2个基因。QnrS1在沙门菌质粒中分布较为普遍[21],而qepA1未在耐喹诺酮的沙门菌中发现过。

由内而外分别是GC content、pAR060302、pSN254b、p2012EL-2176、 punnamed3、 pTB221、pCFSAN007427.01、pRH-1238、pCMY2 085072、pSL131、pCFSAN000934_02、 pCVMN1543、pCVM22425、 pCVM22513、 p846。P1和P2是p846中未比对上的序列。
P2序列全长32 048 bp,GC含量57%。共编码42个基因,其中13个是和基因移动相关的,如解离酶、转化酶、内含子等。携带6个耐药基因——qnrS1、qepA1、dfrA12、sul1、aadA12和mph(a),分别编码对喹诺酮类、氨基糖苷类、甲氧嘧啶类、磺胺类和大环内酯类抗生素的耐药(见图3)。P2主要由I类整合子构成,包括它的5′端的intI整合酶及3′端的sul1和溴乙锭的耐药基因以及两者之间的可变区。研究发现,I类整合子的可变区包含的几乎都是与耐药相关的基因[22]。在P2的I类整合子序列里,可变区包含耐药基因qepA1、dfrA12和aadA2。P2序列的两端均有IS6和反向重复序列,提示3′端的qnrS1、5′端的mph(a)基因可能与此相关。这些插入或整合到I类整合子的耐药基因的存在,使细菌获得在抗生素压力下的生存优势。

红色为可移动蛋白;灰色为IR、IS及Tn等可移动元件箭头方向为基因编码方向
3 讨 论
根据Resfinder的注释,parC基因含有3个未知突变p.T57S、p.T255S、p.V657I。经过分析,认为它们对于蛋白结构的变化影响不大,与喹诺酮耐药无关。有以下原因:首先从氨基酸化学性质分析,T和S、V和I都是化学性质相似的氨基酸,不会引起蛋白质局部电荷以及疏水性质的变化;其次,在Uniport网站搜索到蛋白ParC,已经报导的影响功能的氨基酸突变都有标注,可以参考,而它不含以上3个突变;第三是从蛋白质结构上,虽然沙门氏菌的parC结构未被解析,但和它高度同源的大肠杆菌ParC结构已经解析,这些残基并不位于关键的结合界面,不会影响蛋白质二级结构;最后,这几个突变位点与同期测序的实验室其它对喹诺酮敏感菌株的突变位点相同(数据未列出),可以说明这些突变对于喹诺酮耐药是不起作用的,并且p.T57S位点的突变在文献中已经有多次报道,证实其对喹诺酮耐药是无效的[23]。
综合染色体和质粒序列耐药基因的注释结果,可以确定SM846基因组中和喹诺酮耐药有关基因有acrAB-TolC、sdiA、marR,、marA、qnrS1、qepA1。SdiA位于染色体上,它的作用是无效的[24]。MarR、marA都是编码acrAB的调控蛋白[25]。QepA1曾在敏感的临床鼠伤寒沙门菌中检测出,说明qepA1单独存在不能介导对喹诺酮耐药,这与大肠中qepA的研究结果一致[21-23]。SM846的喹诺酮耐药可能是由acrAB外排泵,QnrS1靶位结合蛋白和qepA1外排泵共同作用来实现。QnrS1或者qepA1或者acrAB单独存在于沙门菌中,均没有对喹诺酮药物产生耐药性的能力。它们组合在一起的具体作用方式,是否与Sato[11]报道的大肠杆菌HUE1的耐药机制类似,还需要更多的实验证实。
对IncA/C2质粒的分析表明,不同来源的质粒,都能在寄生于人和动物的病原体中传播。当同一型的质粒位于不同地区时,它们所携带的耐药基因的数量和种类会发生改变,使宿主获得不同的耐药能力,说明同源性强的质粒可通过灵活获取耐药基因更好的实现本地化生存[26]。Han等[26]分析了4条质粒并与之前发表的质粒进行比对,发现相似质粒之间的独特性是由可移动部分造成的。在SM846中也是如此。p846及相似的质粒,质粒骨架(backbone)区域在时间跨度十几年间,相似度达99%,相对保守。IncA/C2型质粒骨架不含qnrS1和qepA1基因,推测抗生素或其它压力可能促使细菌在可移动元件的帮助下快速获得相应的耐药基因,提高细菌的生存能力。
通过质粒信息的比对我们发现来自于临床样本的质粒,它们的宿主细菌种类比动物和环境来源的样本要更多样,也就意味着多耐药的质粒能通过多种病原菌感染人类。应当注意到,共同的问题包含着不同的细节,不同地区的生态因素和选择压力造成了某一地区独特的耐药模式[27-28]。一项根据基因组序列分析多耐药沙门菌的研究表明,不同地区的耐药相关的特征在基因型和表现型上都表现出了地区差异[27]。因此需要建立地区性的耐药监测和耐药控制策略,我们的质粒比对结果也支持这个观点。在p846相似的质粒中,耐药相关性最强的质粒来自中国和海地,这2个国家都是经济不够发达,在抗生素使用管理上可能不够完善,这两个地区的细菌耐药性更强,不适宜直接套用发达国家对于抗生素管理和控制的策略,因为它们的策略是建立在自己的细菌耐药监测的结果上的。值得一提的是分离自德国的质粒pRH-1238,它携带的耐药基因数量最多,达到20个,所属的抗生素类型也是最多,可耐9种类型的抗生素。对这条质粒上耐药基因fosA3的分析表明,它很可能来自中国[29]。我们根据基因组序列所做的进化分析显示,德国分离的pRH-1238与中国分离的p846在进化上确实属于一个分支,支持了这个质粒可能与中国有关的结论。因此,不同地区的细菌耐药监测是必要的,各地区自己的耐药监测结果才是制定其耐药策略的最合理的依据,也只有根据合理的依据,才能得到有效的控制策略,切实减轻疾病负担。SM846携带qnrS1和qepA1来介导对喹诺酮的耐药,这个发现正是我们监测的成果之一。
利益冲突:无
——一道江苏高考题的奥秘解读和拓展

