用于CD56图像分割的细胞标注精细化与自适应加权损失*
刘 榕,伍 欣,敖 斌,文 青,李 宽
(东莞理工学院网络空间安全学院,广东 东莞 523808)
1 引言
CD56是一种神经细胞黏附分子,也是自然杀伤细胞的标志物。神经内分泌组织及其肿瘤和少数淋巴瘤呈强阳性反应,也是食管小细胞癌的阳性标志物。计算机医学图像处理领域对CD56图像的研究刚刚起步。CD56是一种褐色的物质,它可使相关肿瘤的绝大部分癌细胞的细胞膜表现为阳性,医生通过判断某个细胞是阴性细胞还是阳性细胞,进而推算出阴性细胞和阳性细胞的比例,以确定对癌症患者用药的剂量。图1为CD56图像,左下角为阳性细胞,右下角为阴性细胞。传统的计算阴性细胞和阳性细胞比例的方法主要有:(1)医生分别统计出图像中阴性细胞和阳性细胞的具体数量,再计算出阴性细胞和阳性细胞的比例;(2)医生根据自己的经验,通过观察图像大致估算出阴性细胞和阳性细胞的比例。方法(1)会消耗医生大量的时间和精力,这种重复的工作完全可以交给计算机来完成。同时,细胞分割的精度很大程度上取决于医生的技巧和经验[1],对于同一个细胞,不同的医生可能把它划分为不同的类别。方法(2)容易受到医生的主观性、医生的认知差异和疲劳与分心等因素的影响。面对相同的图像,不同医生估算出的阴性细胞和阳性细胞的比例可能差别很大。如果用药的剂量过大会对癌症患者的身体造成很大的副作用,如果用药的剂量不足会导致无法抑制患者病情的发展,所以准确地计算出病人体内阴性细胞和阳性细胞的比例对疾病治疗是至关重要的。
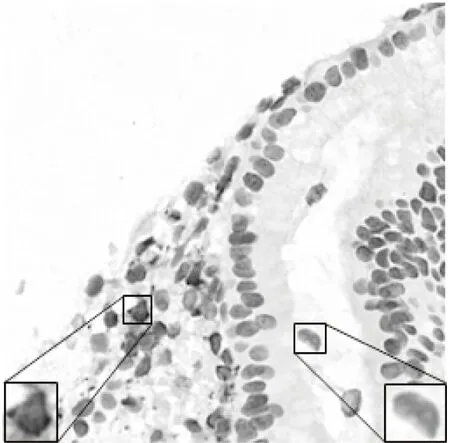
Figure 1 CD56 image图1 CD56图像
近年来,诸如语义分割的深度学习技术在医学图像处理等领域获得了很好的应用,能够对图像中的每个像素点进行分类。将语义分割技术应用于CD56图像,可将统计阴阳细胞数量的比例转化为统计阴阳细胞像素点个数的比例,从而实现从人工统计到计算机统计的转变。但是,在CD56图像中背景、阴性细胞和阳性细胞的像素点个数比例失衡,这会影响语义分割模型对CD56图像的分割效果。
本文在语义分割模型的损失函数中为不同类别的像素点添加不同的损失权重,并且对每个像素点添加自适应权重,缓解了类别不平衡的问题。此外,由于细胞数量很多,精细标注费时费力,所以通常使用一个矩形框来标注细胞。一个矩形框的标注不仅包含一个细胞,而且还可能包含部分背景,这些标注框中的背景像素会对语义分割模型的训练造成干扰。本文使用聚类的方法,将每个标注框中的像素聚类成2个簇,再将属于背景的那一簇像素点从标注中去除,从而实现标注的精细化。
2 相关工作
CD56图像中细胞数量很多,如果沿着细胞的边界精细地标注出细胞会消耗大量的时间和精力,所以研究人员通常使用一个矩形框来标注细胞。图2为标注好的CD56图像,其中,“-”代表阴性细胞,“+”代表阳性细胞。

Figure 2 Annotated CD56 image图2 已标注的CD56图像
通过训练集矩形框的标注来预测测试集,这就是目标检测的任务。关于目标检测的研究,目前有双阶段的区域卷积神经网络R-CNN(Region-Convolutional Neural Network)系列[2 -4]模型,该系列模型先产生一些待检测框,再对检测框进行分类;有单阶段的YOLO(You Only Look Once)系列[5 -7]模型,该系列模型将全图划分为格子,如果一个物体的中心正好位于某个方格之内,那么该方格就负责预测这个物体,并使用边界框回归得到检测框的准确位置和大小;有不需要锚框的CornerNet模型[8],该模型预测目标的左上角和右下角2个点,通过这2个点形成检测框,从而检测出物体。但是,在CD56图像中,细胞面积较小,而目标检测模型对小目标的检测效果并不理想;并且细胞密集地贴在一起,甚至边界都连在一起,这种情况下用目标检测无法准确地检测出细胞的个数。表1为Faster-RCNN模型、YOLOv3模型和CornerNet模型在CD56图像数据集上的性能评估结果(表中Negative表示阴性细胞,Positive表示阳性细胞,AP50表示当检测框和标注框的交并比的阈值设为0.5时,模型对目标进行检测的平均精度)。图3为经过CD56图像数据训练的YOLOv3模型对2幅CD56图像预测的效果。从表1可以看出,目标检测模型对CD56图像的检测效果并不理想,从图3可以看出,YOLOv3模型对CD56图像检测时会遗漏许多细胞。

Table 1 Performance of three object detection models on CD56 image dataset表1 3个目标检测模型在CD56图像数据集上的性能

Figure 3 CD56 image detection results using YOLOv3 model图3 YOLOv3模型对CD56图像的检测结果
语义分割能对图像中的每个像素点进行分类,将语义分割模型应用于CD56图像,能将统计阴阳细胞数量的比例转化为统计阴阳细胞像素点个数的比例。2015年,Long等[9]提出了全卷积网络FCN(Fully Convolutional Network)模型,该模型是一种编码器-解码器结构,这是后来的语义分割模型主要采用的结构之一。这种结构先在编码过程中通过卷积池化操作,获取图像的高维信息;再在解码过程中通过上采样操作,将编码过程得到的特征图逐步恢复到和输入图像相同的尺寸,从而对图像的每个像素点进行分类。为了保留图像中的上下文空间信息,FCN在解码过程中将浅层的特征图和高层的特征图进行融合。同年,Ronneberger等[10]提出了U-Net模型,该模型应用跳跃连接将编码过程产生的特征图和解码过程产生的特征图进行拼接,使最后得到的特征图既保留了图像低维高分辨率的信息,又有图像高维低分辨率的信息。U-Net被广泛应用于医学图像分割。2018年,Chen等[11]提出了DeepLabv3+模型,该模型通过条件随机场来提高模型捕获精细细节的能力;在最后几个最大池化层用空洞卷积(Atrous Convolution)替代下采样,以更高的采样密度来计算特征图;提出了空洞空间金字塔池化(Atrous Spatial Pyramid Pooling)结构,对给定输入以不同采样率的空洞卷积进行采样,从而能够抽取丰富的多尺度上下文信息。2020年,Wu等[12]提出了一种轻量化语义分割模型——上下文引导网络CGNet(Context Guided Network),CGNet主要由CG(Context Guided)块构建而成,CG块可以学习局部特征和周围环境上下文的联合特征,最后通过引入全局上下文特征进一步改善联合特征的学习。
CD56图像上的标注是矩形框,每个矩形框不仅包含一个细胞,而且还可能包含部分背景像素点。如果将图像和原标注放入语义分割模型中进行训练,可能会影响模型对细胞像素点的判断,比如会将细胞周围的背景像素点也预测为细胞像素点。图4为经过CD56图像和原标注训练的U-Net模型对CD56图像的预测结果。从图4中可以看出,U-Net对每个细胞的预测结果十分接近矩形,不但包含细胞,而且还包含大量的背景像素点,这使得计算阴阳细胞像素点的比例可能产生误差,因此需要将一个标注框内的细胞像素和背景像素区分开来,即精细化细胞标注。
Li等[13]使用设置颜色阈值的方法,将细胞图像中处于阈值内的像素点作为感兴趣区域ROI(Region Of Interest)提取出来,将处于阈值外的像素点作为干扰因素过滤掉。这种方法对CD56图像不可行,因为在CD56图像中,判断一个细胞是不是阳性细胞,要看蓝色的细胞周围是否有褐色的CD56黏附着,设置颜色阈值的方法无法将阳性细胞和背景区分开来。在CD56图像中,一个标注框主要包含一个细胞的像素点和背景像素点,细胞的像素点很接近,同时背景像素大多是简单的白色,因此可以考虑用聚类的方法将每个标注框中的细胞像素点和背景像素点聚成2个簇,从而将细胞像素点和背景像素点区分开来。
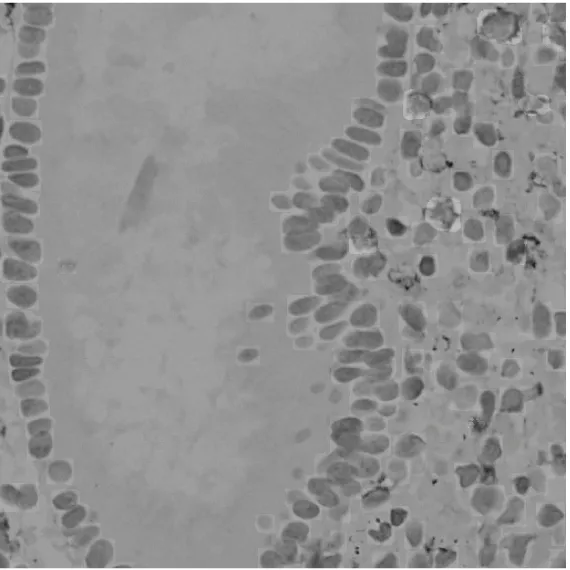
Figure 4 Prediction results of CD56 image using the U-Net model trained by original annotations图4 使用原标注训练的U-Net模型对CD56图像的预测结果
关于不平衡样本的研究,Lin等[14]于2017年提出了焦点损失(Focal Loss),使得模型更关注难以正确分类的样本,减少对数量众多且容易分类的样本的关注。Law等[8]和Zhou等[15]将Focal Loss的变体用于物体关键点的预测。本文改进了Focal Loss,使其更适用于CD56图像的语义分割。
3 方法描述
3.1 细胞标注精细化
K-Means是一种实现简单、效果不错的聚类算法,因此本文使用K-Means算法对CD56图像数据集的标注框中的像素点进行聚类。K-Means算法的步骤如下:先将数据分为K组,随机选取K个对象作为初始的聚类中心,然后计算每个对象与各个聚类中心之间的距离,把每个对象分配给距离它最近的聚类中心。聚类中心以及分配给它们的对象就代表一个簇,这样就能得到的K个簇。更新得到K个簇的聚类中心,再根据K个新的聚类中心得到K个新的簇。重复以上步骤,直到算法收敛。
本文将CD56图像训练集中所有图像的所有阴性框中的像素点和所有阳性框中的像素点分别进行聚类。在这2次聚类中,一个对象为一个像素点的RGB值构成的数组,每次聚类将本次所有的对象分成2个簇。第1次聚类得到的2个簇为阴性细胞像素点和背景像素点,第2次聚类得到的2个簇为阳性细胞像素点和背景像素点,这样就实现了细胞像素点和背景像素点的区分,从而实现细胞标注的精细化。图5展示了通过聚类对标注精细化的结果。图5a为CD56图像原图;图5b为原标注;图5c为分别对所有阴性标注框中的像素点和所有阳性标注框中的像素点聚类的结果,对所有阴性标注框中的像素点聚类得到的2个簇的像素点分别用灰度值60和120表示,对所有阳性标注框中的像素点聚类得到的2个簇的像素点分别用灰度值180和240表示(即较暗的标注框为阴性细胞的标注框,较亮的标注框为阳性细胞的标注框);图5c中灰度值为120和240的像素点是背景像素点,灰度值为60的像素点是阴性细胞像素点,灰度值为180的像素点是阳性细胞像素点,将背景像素点、阴性细胞像素点和阳性细胞像素点的值分别设为0,120和240(即黑色区域为背景,较暗的区域为阴性细胞,较亮的区域为阳性细胞),得到图5d,即为对细胞标注精细化后得到的新标注。对比图5b和图5d可知,经过聚类精细化后的标注相比原标注少了许多背景像素,甚至能将细胞精确地标注出来。

Figure 5 Results of annotations refined by clustering图5 通过聚类对标注精细化的结果
3.2 类别权重计算
Eigen等[16]针对语义分割中不同类别的像素点的不平衡提出了一种用类别像素点的频率计算类别权重的方法,再将得到的类别权重加入交叉熵损失函数中。具体的权重计算公式如式(1)和式(2)所示:
(1)
(2)
其中,median_freq表示所有类别像素点频率的中值,freq(c)表示为类别c像素点的频率,sum(c)表示训练集中类别c像素点的总数,sum(Ic)表示训练集中所有含有类别c像素点的图像的像素点的总数。在CD56图像数据集中,背景和阳性细胞像素点的总数相差很大,用式(1)计算背景像素点的权重和阳性细胞像素点的权重也会相差很大,使得一些原本被正确预测为背景的像素点反而被错误地预测为阳性细胞像素点。因此,本文用式(1)计算出类别权重之后,再对得到的类别权重开n次方根,得到最后的类别权重,如式(3)所示:
(3)
3.3 自适应加权
本文基于Focal Loss在损失函数中对每个像素点添加了一个自适应权重。在二分类任务中,每个样本的交叉熵损失的计算公式如式(4)所示:
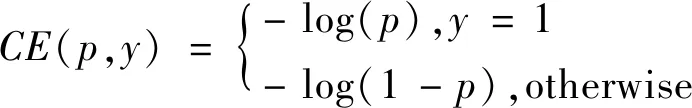
(4)
其中,y∈{±1}表示样本的标签值,p∈[0,1]表示模型预测样本属于类别1的概率。为了方便起见,定义py如式(5)所示:
(5)
则CE(p,y)=CE(py)=-log(py)。
为了解决类不平衡问题,一种方法是在交叉熵损失中给类别1添加权重α∈[0,1],给类别-1添加权重1-α,类似定义py一样定义αy,则用α平衡的交叉熵损失如式(6)所示:
CE(py)=-αylog(py)
(6)
Focal Loss在交叉熵损失中加入了调制因子(1-py)γ:
FL(py)=-(1-py)γlog(py)
(7)
其中γ≥0是称为聚焦参数(Focusing Parameter)的超参数。
从式(7)可以看出,模型对一个样本预测的py越大,模型对其越容易正确预测,这时调制因子会给它赋予一个小权重,以减少模型对它的关注;反之,py越小,模型对其越难正确预测,这时调制因子会给它赋予一个大权重,以增加模型对它的关注,这样调制因子就给样本添加了一个自适应的权重。本文实验中采用的是带α平衡的Focal Loss的变体,如式(8)所示:
FL(py)=-αy(1-py)γlog(py)
(8)
在多分类任务中,每个样本的交叉熵损失的计算公式如式(9)所示:
CE(py,y)=-log(py)
(9)
其中,y表示样本的标签值,py∈[0,1]表示模型预测样本属于类别y的概率。类似式(8)可为式(9)添加自适应权重,本文将αy替换成β,将γ的值设为1。其中,β>0不再是为了平衡调制因子(1-py)γ,而是为了控制调制因子的范围;将γ的值设为1是因为在实验中其效果更好。故本文对CD56图像像素点添加自适应权重的公式如式(10)所示:
AWCE(py,y)=-β(1-py)log(py)
(10)
4 实验与结果分析
本文分别使用U-Net、DeepLabv3+和CGNet模型进行了实验,以验证细胞标注精细化,对不同类别的像素点加权和对像素点自适应加权在CD56图像分割中的有效性。
4.1 数据集
本文使用的所有CD56图像均来自某医院,共有598幅图像,原图像的分辨率为1024×1024。将每幅图像裁剪为4幅512×512的图像,除去不含细胞的无效图像,最终得到2 198幅图像。本文实验采用5-折交叉验证,将所有图像分成5组,前4组图像每组包含440幅,最后一组图像包含438幅。
4.2 训练参数
训练时采用Momentum[17]梯度下降优化算法,初始学习率设为0.01,迭代次数设置为40 000。训练平台为NVIDIA GeForce RTX 2080×1和NVIDIA GeForce RTX 3090×1,没有经过聚类处理的数据集在NVIDIA GeForce RTX 2080上进行训练,经过聚类处理的数据集在NVIDIA GeForce RTX 3090上进行训练。
4.3 测试结果评估
4.3.1 细胞标注精细化
这组实验是为了验证细胞标注精细化对CD56图像分割的有效性。将训练集和原标注与训练集和精细化后的标注分别使用3个模型进行训练,得到的性能评估结果如表2和表3所示。可以看出,采用精细化后的标注进行训练能让U-Net对阴阳细胞的测试Dice(Dice=(2*|X∩Y|)/(|X|+|Y|),其中,|X|表示模型对某类物体预测的像素点数,|Y|表示该类物体实际标注的像素点数,|X∩Y|表示模型对该类物体预测正确的像素点数)分别提升1.62%和3.92%,DeepLabv3+对阴阳细胞的测试Dice分别下降0.13%和提升2.79%,CGNet对阴阳细胞的测试Dice分别下降3.28%和上升2.89%。总体来说,细胞标注精细化对CD56图像分割是有效的。
图6是用原细胞标注和用精细化后的标注分别进行训练得到的模型对CD56图像的预测结果。图6a是用原细胞标注进行训练得到的模型对CD56图像的预测结果,图6b是用精细化后的标注进行训练得到的模型对CD56图像的预测结果。可以看出,用原细胞标注进行训练得到的模型对细胞的预测十分接近矩形,包含大量的背景像素,而用精细化后的标注进行训练得到的模型对细胞的预测很吻合细胞的真实区域。

Table 2 Performance evaluation results of models trained with original cell annotations表 2 用原细胞标注进行训练的模型性能评估结果

Table 3 Performance evaluation results of models trained with refined annotations表3 用精细化后的标注进行训练的模型性能评估结果
4.3.2 对类别添加权重
这组实验是为了分别找到原标注数据集和标注精细化后的数据集在3个模型上合适的权重,方法为根据式(3)计算获得。对于原标注数据集,当式(3)中的n=1时得到类别权重比为背景∶阴性细胞∶阳性细胞=0.3405∶1∶12.9120(后面提到的类别权重比均为背景∶阴性细胞∶阳性细胞)。当用于标注精细化后的数据集,式(3)中的n=1时得到类别权重比为背景∶阴性细胞∶阳性细胞=0.1567∶1∶10.5778。可以看出,当式(3)中的n=1时,计算得出的背景和阳性细胞的权重相差太大,这会使得一些原本被正确预测为背景的像素点反而被错误地预测为阳性细胞,所以本文从n=2开始计算类别权重比,n逐次加1,直到模型在数据集上的性能评估指标下降。表4为类别权重不同时3个模型在原标注数据集上的性能评估结果。表5为类别权重不同时3个模型在标注精细化后的数据集上的性能评估结果。

Table 4 The performance of the three models on the original annotated dataset with different class weights表 4 类别权重不同时3个模型在原标注数据集上的性能
从表4可知,在获得的权重比中,原标注数据集在3个模型上的最佳类别权重比分别为式(3)中n=3时计算的值(0.6983∶1∶2.3460),n=4时计算的值(0.7639∶1∶1.8956)和n=5时计算的值(0.8161∶1∶1.6680)。从表5可知,在获得的权重比中,经过标注精细化后的数据集在3个模型上的最佳类别权重比分别为式(3)中n=4时计算的值(0.6292∶1∶1.8034),n=5时计算的值(0.6903∶1∶1.6028)和n=4时计算的值(0.6292∶1∶1.8034)。本文分别选取这些类别权重比进行下一组实验。
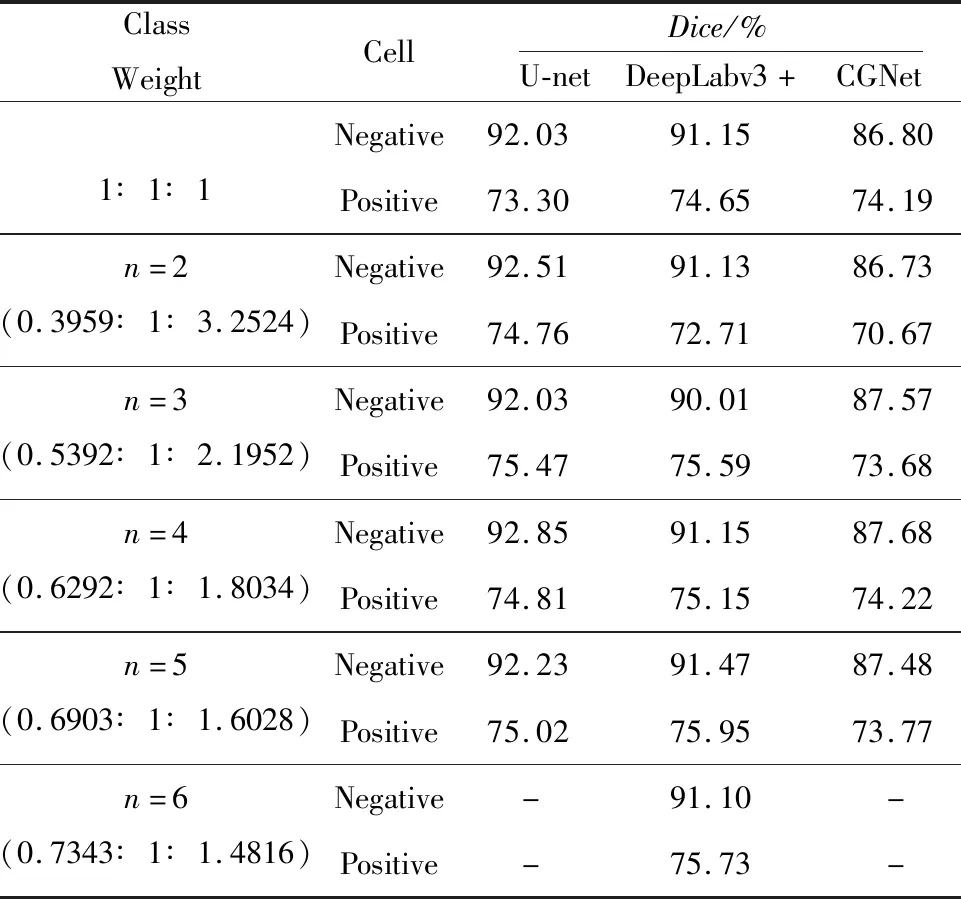
Table 5 The performance of the three models on the refined annotated dataset with different class weights表5 类别权重不同时3个模型在标注精细化后的数据集上的性能
4.3.3 对像素点添加自适应权重
这组实验是为了验证对像素点添加自适应权重对CD56图像分割的有效性。实验采用原标注数据集和经过标注精细化后的数据集分别在3个模型上进行训练,在训练之前给类别添加上组实验中对应的权重,对比不对像素点添加自适应权重和对像素点添加自适应权重的性能。对像素点添加的自适应权重由式(10)中的β取不同值获得,结果如表6和表7所示,表中的β是在实验中得到的对应类别权重的最佳值。
从表6可以看出,在原标注数据集上,对每个像素点添加自适应权重相比不对每个像素点添加自适应权重进行训练,能让U-Net对阴阳细胞的测试Dice分别提升0.07%和0.75%,DeepLabv3+对阴阳细胞的测试Dice分别提升0.27%和0.01%,CGNet对阴阳细胞的测试Dice分别下降0.18%和上升0.34%。从表7可以看出,在标注精细化后的数据集上,对每个像素点添加自适应权重比不对每个像素点添加自适应权重进行训练,能让U-Net对阴阳细胞的测试Dice分别提升0.2%和1.91%,DeepLabv3+对阴阳细胞的测试Dice分别提升0.35%和0.22%,CGNet对阴阳细胞的测试Dice分别下降0.14%和上升0.58%。CGNet的CG块不仅学习局部特征,还学习周围环境的上下文特征;而对每个像素点添加自适应权重强调的是局部特征,没有涉及到上下文特征,由此可能造成CGNet对阴性细胞的测试Dice的下降。但是总体来说,给每个像素点添加自适应权重对CD56图像分割是有效的。
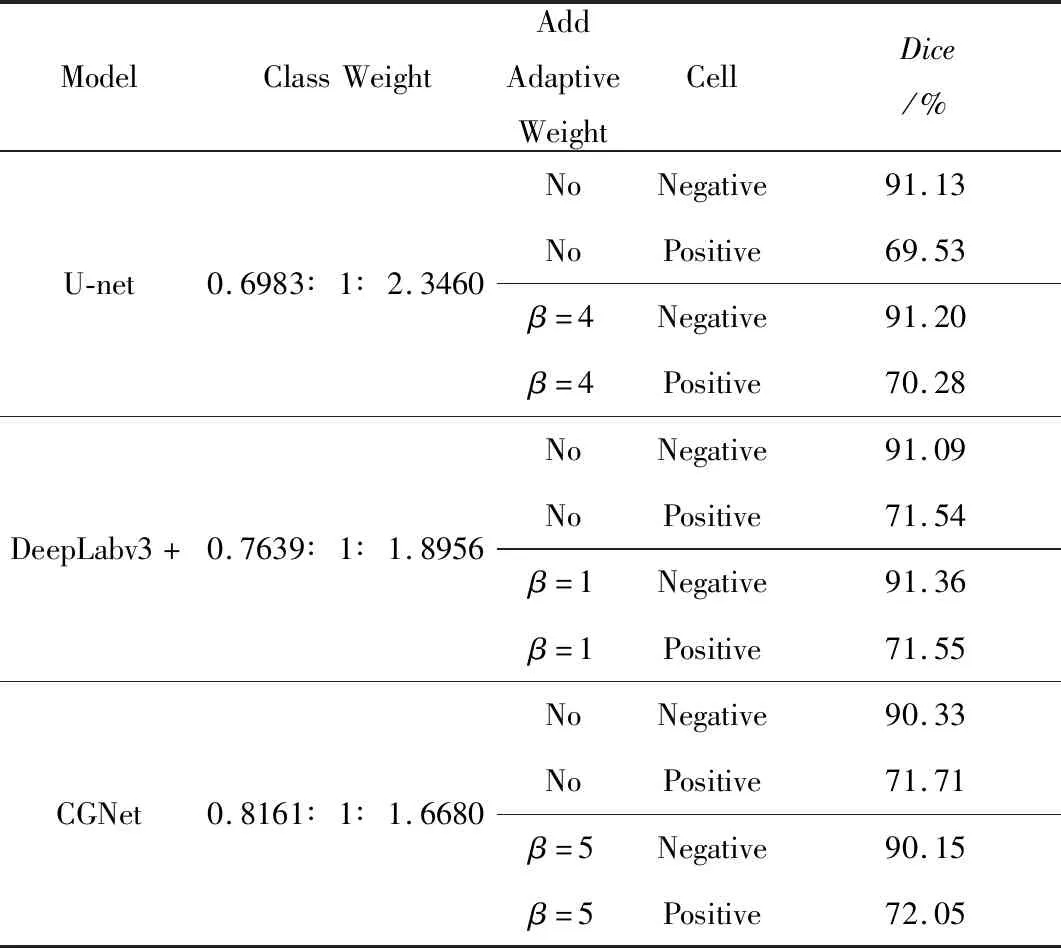
Table 6 Performance without or with adding adaptive weights to the pixels on the original annotated dataset表6 不对像素点添加自适应权重和对像素点添加自适应权重在原标注数据集上的性能
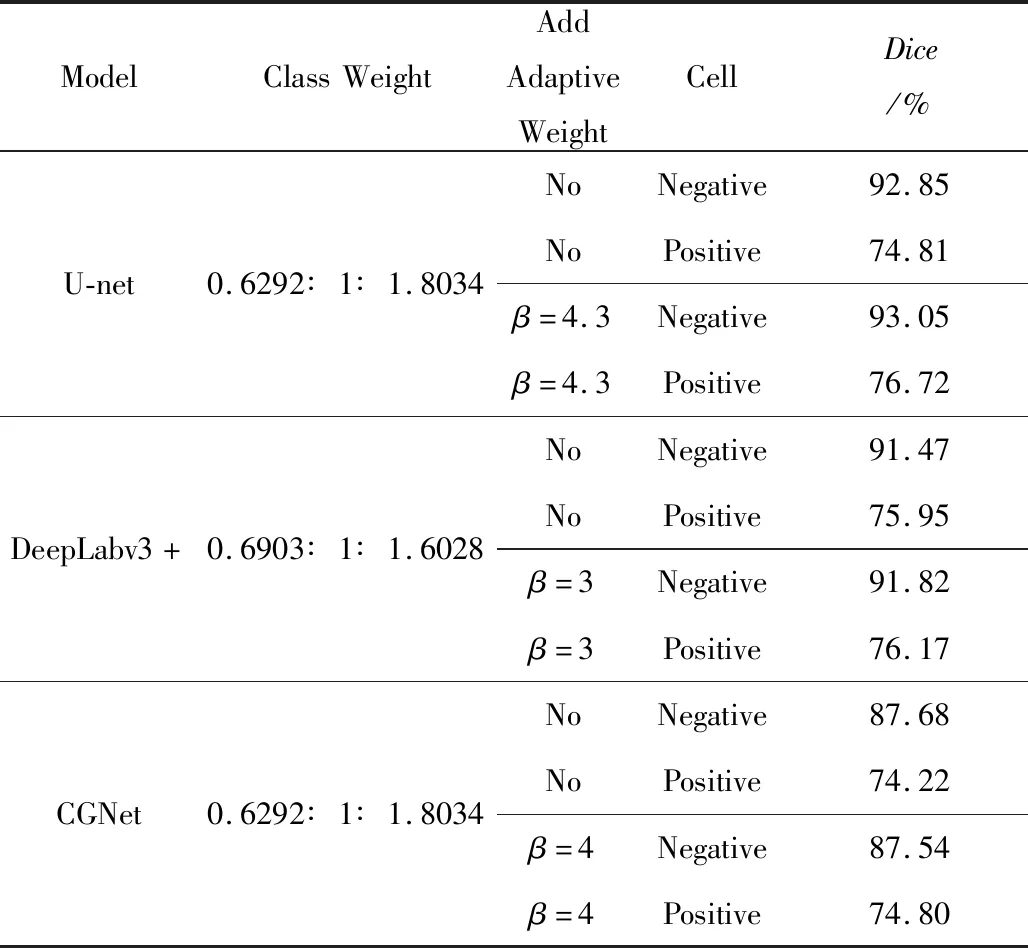
Table 7 Performance without or with adding adaptive weights to the pixels on the refined annotated dataset表7 不对像素点添加自适应权重和对像素点添加自适应权重在标注精细化后的数据集上的性能
5 结束语
本文以CD56图像为主要研究对象,基于深度学习中的语义分割技术实现对CD56图像像素级别的分割,以辅助医疗诊断。本文使用聚类的方法对原CD56图像数据集的标注进行精细化处理,对数据集中不同类别的像素点进行加权并对数据集中所有像素点进行自适应加权,在U-Net、DeepLabv3+和CGNet 3个模型上进行了实验,验证了方法的有效性。实验结果显示,在3个模型上,使用聚类的方法对原CD56图像数据集的标注进行细化、对数据集中不同类别的像素点进行加权并对数据集中所有像素点进行自适应加权比原始数据集、原始模型的阴阳细胞的Dice指标分别提升了1.94%和7.34%,0.54%和4.31%,-2.54%和3.5%。在以后的工作中,我们将获取更多的CD56图像,并应用数据增强等技术以解决数据集规模小、数据不平衡的问题,以进一步提高对CD56图像的分割精度。此外,本文应用语义分割技术实现对CD56图像像素级别的分割,统计的是阴性细胞和阳性细胞像素点个数的比例,并没有直接对细胞的个数进行统计,这也是今后工作的一个方向。

