链霉菌TRM SA0054 Tubercidin合成基因簇挖掘
张 萍,罗晓霞,万传星,张利莉
(塔里木盆地生物资源保护利用兵团重点实验室/塔里木大学生命科学与技术学院,新疆阿拉尔 843300)
0 引 言
【研究意义】近年来,随着高通量测序技术的不断发展,微生物基因组测序的成本不断降低,在基因水平上预测其具有怎样的生物学功能和可能产生的化合物的结构,可以作为目标天然产物分离及鉴定的依据[1,2],也是探索新的生物合成基因簇的有效途径,并有希望发现新的化合物[3,4]。Tubercidin(杀结核菌素)是从Streptomycestubercidicus中提取的一种吡咯嘧啶核苷类似物,能够与ATP在微管蛋白的结合位点进行结合来稳定微管,具有潜在的研究价值[5]。【前人研究进展】王珊珊等[6]在基因组信息指导下从茂源链霉菌StreptomycesmobaraensisUS-43中分离鉴定了一个α-吡啶酮类化合物杀粉蝶菌素A1。植物根际分离的Streptomycessp.BA2新物种由E Kum等[7]通过基因组分析从菌株次级代谢物中鉴定了actinomycin和desferrioxamine,验证了基因组挖掘结果。T Hashimoto等[8]利用基因组分析挖掘Streptomycssp.4083-SVS6中的一个新的合成基因簇AT-反式PKS基因,在另一株链霉菌中进行了表达,获得了一种新的多烯化合物JBIR-159。【本研究切入点】胭脂红链霉菌(StreptomycescarminiusTRM SA0054)是分离自新疆阿拉尔药用植物苦豆子根际土壤的新物种,对粪肠球菌和金黄色葡萄球菌有明显的抑菌活性,确定其为链霉菌新物种,并被命名为胭脂红链霉菌(StreptomycescarminiusTRM SA0054)[9]。【拟解决的关键问题】对StreptomycescarminiusTRM SA0054进行全基因组测序,对基因组进行功能注释,并预测比对次级代谢生物合成基因簇,为挖掘结构独特、生物活性较好且具有特殊作用机制的目标天然产物提供理论依据。
1 材料与方法
1.1 材 料
1.1.1 菌 株
菌株TRM SA0054从中国西北部新疆阿拉尔(E 81°17′,N 40° 32′)的苦豆子的根际土壤中分离得到。保藏于塔里木盆地生物资源保护利用兵团重点实验室,中国典型培养物保藏中心和韩国典型菌种保藏中心(StreptomycescarminiusTRM SA0054T= CCTCC AA2016041T= KCTC39903T)[9]。
革兰氏阳性代表病原细菌:金黄色葡萄球菌(StaphylococcusaureusATCC25923),粪肠球菌(EnterococcusfaecalisATCC29212);革兰氏阴性代表病原细菌:大肠杆菌(EscherichiacoliATCC25922);真菌代表病原菌:白色念珠菌(CandidaalbicansATCC64550),棉花黄萎病(VerticilliumdahliaeACCC 36211),棉花枯萎病(FusariumvasinfectumACCC31038)。
1.1.2 培养基
玉米-豆粕发酵培养基:20.0 g 葡萄糖,30.0 g 可溶性淀粉,10.0 g 玉米浆,10.0 g 豆粕,5.0 g 蛋白胨,2.0 g NaCl,5.0 g 碳酸钙,水1 L,pH 7.0。
MHA培养基:牛肉提取物 2.0 g,酪蛋白水解物17.5 g,淀粉2.0 g,琼脂17.0 g,水1 L,pH 7.3。
SDA 培养基:葡萄糖20.0 g,蛋白胨10.0 g,琼脂17.0 g,水1 L,pH 7.3 。
LB培养基:酵母浸粉5 g,蛋白胨10 g,NaCl 10 g,水1 L, pH 7.0~7.4。
PDA培养基:土豆200.0 g,葡萄糖 20.0 g,琼脂17.0 g,水1 L,pH 7.0。
1.1.3 主要仪器设备
上海博迅YXQ-70A 高压蒸汽灭菌锅,上海博迅SW-CJ-ZF超净工作台,上海博迅BMJ-250C电热恒温培养箱,河北国辉FC-27AS-E真空冷冻干燥机,上海安科AnkeyqN-1100旋转蒸发仪,日本岛津LC-20AT高效液相色谱仪,美国Agilent高效液质联用仪。
1.2 方 法
1.2.1 菌株TRM SA0054抑菌活性检测
小量发酵粗提物制备,在无菌超净工作台中,选取3个长势良好的菌株TRM SA0054平板,用灭过菌的直径为1 cm的蓝色枪头打5个菌饼于121℃,30 min灭菌的玉米-豆粕液体发酵培养基250 mL锥形瓶中,30℃,150 r/min摇床培养7 d,3个平行。发酵液经8 000 r/min,离心10 min后,经真空冷冻干燥研磨,100 mL乙酸乙酯和100 mL甲醇分别萃取。
采用滤纸片法检测菌株TRM SA0054不同极性段萃取的发酵液的粗提物对病原菌的抑制作用,各吸取100 μL,发酵液滴加在直径6 mm的滤纸片上,乙酸乙酯和甲醇空白对照,每组3个平行,将晾干后滤纸片放在病原菌平板上,病原真菌放置 28℃培养箱培养3~5 d,病原细菌放置37℃培养箱中培养24 h后,测量抑菌圈直径大小。
1.2.2 全基因组测序及基因组组装
菌株TRM SA0054基于Illumina Hiseq 2000 测序平台进行全基因组测序,对基因组数据采用SOAP denovo短序列组装软件[10]拼接,AbySS 2.0软件注释分析。
1.2.3 菌株TRM SA0054基因组
1.2.3.1 编码基因
对基因组组装结果采用 GeneMarkS[11]和Glimmer3.0[12]软件预测菌株TRM SA0054基因组中的基因组ORFs,利用RNAmmer-1.2[13],tRNAscan-SE 2.0.4[14]和Infernal v1.1.14 软件对基因组中包含的rRNA,tRNA,其他ncRNA进行预测。
1.2.3.2 基因功能注释
采用的基因功能注释数据库有COG[15]是Clusters of Orthologous Groups of proteins的缩写(http://www.ncbi.nlm.nih.gov/COG/),进行COG数据库比对可以对预测蛋白进行功能注释、归类类以及蛋白进化分析。GO是基因本体论Gene Ontology的缩写(http://www.geneontology.org/),GO注释更加便于理解基因背后所代表的生物学意义[16]。KEGG(Kyoto Encyclopedia of Genes and Genomes,京都基因和基因组百科全书)( http://www.genome.jp/kegg/)是系统分析基因功能,联系基因组信息和功能信息的大型知识库[17]。
1.2.3.3 次级代谢产物生物合成基因簇预测
AntiSMASH(version 6.0)(https://antismash.secondarymetabolites.org/)是一种对次级代谢产物合成基因簇分析预测的软件,运用软件对菌株基因组数据进行注释、预测和分析[18]。
1.2.4 菌株TRM SA0054发酵产物检测
采用 HPLC-MS对产物进行检测。液相分离柱选用 C18(2.1×50 mm,3.5 μm);检测波长269 nm;流动相 A:超纯水加 0.5%甲酸,流动相 B:甲醇;流速为 0.5 mL /min。液相洗脱程序:2~11 min,流动相 B 从5% 到 35% ;11~20 min,流动相 B从 35% 到 100% ;流动相 B 保持100%,然后停止洗脱。质谱采用电喷雾电离(ESI),正离子模式,初始条件气化室温度 350℃, 11 L/ min,离子化温度 300℃, 3 L/min;电压4 000 V,扫描范围 m /z 为 100~1 000。
2 结果与分析
2.1 菌株TRM SA0054的抑菌作用检测
研究表明,3个平行得出抑菌圈平均值结果菌株TRM SA0054小量摇瓶发酵,甲醇萃取物对革兰氏阳性代表病原细菌粪肠球菌(E.faecalisATCC29212)具有显著的抑菌作用抑菌圈直径达到(25.45±0.3) mm,对金黄色葡萄球菌(S.aureusATCC25923)抑菌圈直径达到(10.03±0.2) mm。菌株TRM SA0054对革兰氏阴性代表病原细菌大肠杆菌(E.coliATCC25922)和真菌代表病原菌白色念珠菌(C.albicansATCC64550 ),棉花黄萎病(V.dahliaeACCC 36211),棉花枯萎病(F.vasinfectumACCC31038)无抑菌活性。图1

注:a.粪肠球菌(E.faecalis)G+;b.金黄色葡萄球菌(S. aureus)G+;1.甲醇萃取粗提物;2.甲醇空白对照;3.水相粗提物;4.菌体水相;5.菌体甲醇萃取相;6.水空白对照;7.Tubercidin标品(1 mg/mL);8.乙酸乙酯空白对照;9.菌体乙酸乙酯萃取相;10.乙酸乙酯萃取粗提物
2.2 基因组组装
研究表明,测得其全长7 200 902 bp,GC含量为73.16%,菌株TRM SA0054的基因组序列由85个contigs组成,N50大小为 300 464 bp ,最长contig为 866 627 bp 。基因组预测到共编码6 410个基因,总长度为 6 118 173 bp,平均长度为 954 bp,占基因组全长的85%。预测到菌株TRM SA0054基因中存在非编码RNA中含有7个5S rRNA,6个16S rRNA,2个23S rRNA,65个tRNA。表1

表1 S.carminius TRM SA0054 的基因组信息
2.3 菌株TRM SA0054基因组功能注释
2.3.1 COG 功能注释分类
研究表明,菌株TRM SA0054共有5 013个基因在COG数据库得到注释。菌株TRM SA0054的COG 聚类分析发现其蛋白质功能主要有:746 个基因参与转录(Transcription,K)、441个基因参与氨基酸代谢及转运(Amino acid transport and metabolism,E)、416个基因参与碳水化合物转运和代谢(Carbohydrate transport and metabolism,G)、416个基因参与信号转导机制(signal transductionmechanisms,T)、348个基因参与复制、重组和修复(replication, recombination and repair,L)、320个基因参与能量生产和转换(energy production and conversion,C)、280个基因参与细胞壁/膜/包膜生物发生(Cell wall/membrane /envelope biogenesis,M)、267个基因参与脂质转运与代谢(Lipid transport and metabolism,I)、250个基因参与无机离子转运和代谢(inorganic ion transport and metabolism,P),各功能注释的基因占 COG 功能注释总基因数的百分比依次为14.9%、8.8%、8.3%、8.3%、6.9%、6.4%、5.6%、5.3%和5.0%。图2
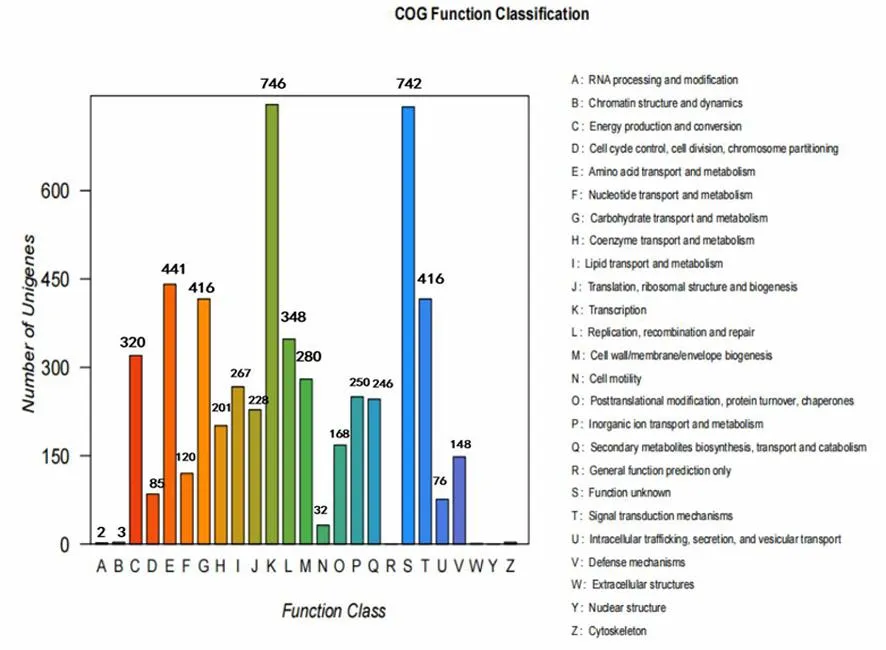
图2 S.carminius TRM SA0054的COG功能注释分类
2.3.2 GO 功能分类
研究表明,S.carminiusTRM SA0054基因组中有916个基因能够在GO数据库提取到注释信息见图3,按各基因功能分为三大亚类,其中,生物学过程(Biological process)类别中涉及基因最多的是细胞过程 (cellular process)和代谢过程(metabolic process);细胞组分(Cellular component)类别中涉及基因最多的是细胞内的和细胞实体(cellular anatomical entity),分子功能(Molecular function)类别中涉及基因最多的是结合(binding)和催化活性(catalytic activity)。图3
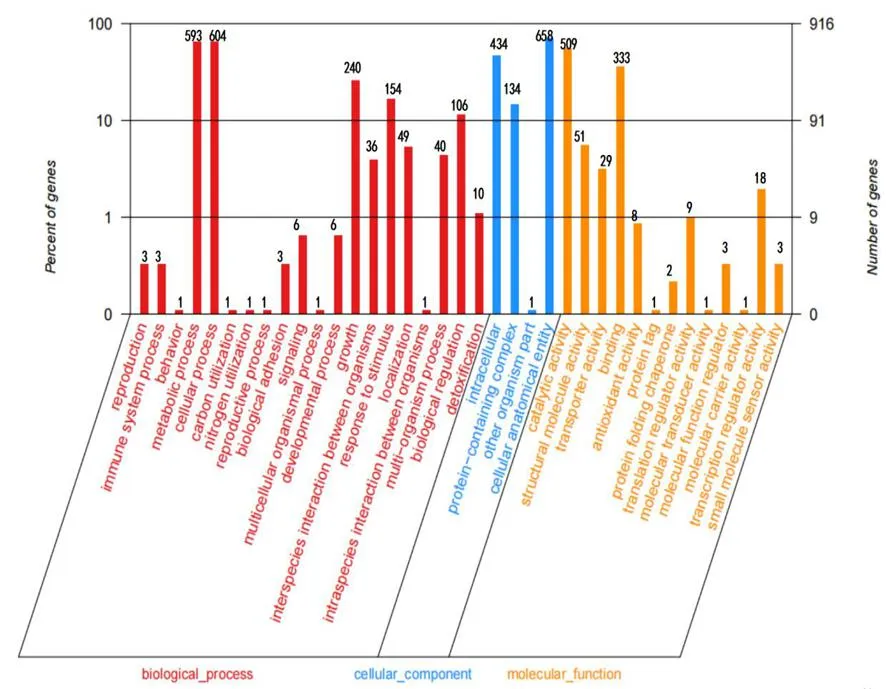
图3 S.carminius TRM SA0054的GO功能分类
2.3.3 KEGG代谢途径
研究表明,TRM SA0054共有2 465个基因在KEGG数据库中得到功能注释,占总蛋白质序列的38.46%。其中涉及基因最多的通路主要有:309个基因富集在氨基酸代谢(Amino acid metabolism),399个基因富集在碳水化合物代谢(Carbohydrate metabolism),172个基因富集在能量代谢(Energy metabolism),60个基因富集在聚糖生物合成和代谢(Glycan biosynthesis and metabolism),105个基因富集在脂类代谢(Lipid metabolism ),185个基因富集在辅助因子和维生素的代谢(Metabolism of cofactors and vitamins),67个基因富集在萜类和聚酮类物质的代谢(Metabolism of terpenoids and polyketides),79个基因富集在核苷酸代谢(Nucleotide metabolism),62个基因富集在外源生物的生物降解和代谢(Xenobiotics biodegradation and metabolism)。KEGG数据库注释的结果主要与遗传信息处理、信号和细胞过程、碳水化合物代谢及氨基酸代谢有关。图4

注:纵坐标为KEGG代谢通路的名称,横坐标为注释到该通路下的基因个数及其个数占被注释上的基因总数的比例
2.4 次级代谢产物合成基因簇预测
研究表明,S.carminiusTRM SA0054 的 全 基 因 组 经 过 antiSMASH预测,发现其潜在的天然产物生物合成基因簇共有26个,包括1个Nucleoside核苷类基因簇,3个NRPs (Non- ribosomal peptide synthetase,非核糖体肽合成酶)类基因簇,4个PKS (Polyketide synthase , 聚 酮 合 酶 ) 类 基 因 簇 ,5 个 NRP+Polyketide 杂合基因簇 , 3个 RiPP (Ribosomally synthesized and posttranslational modified peptides,核糖体合成和翻译后修饰 肽)基因簇,4个Terpene(萜烯)类,1个Ectoine(四氢嘧啶)类,1个Polyketide:Iterative typeI + Saccharide:Hybrid/tailoring,1个lanthipeptide-class-i,3个other功能的基因簇。表2
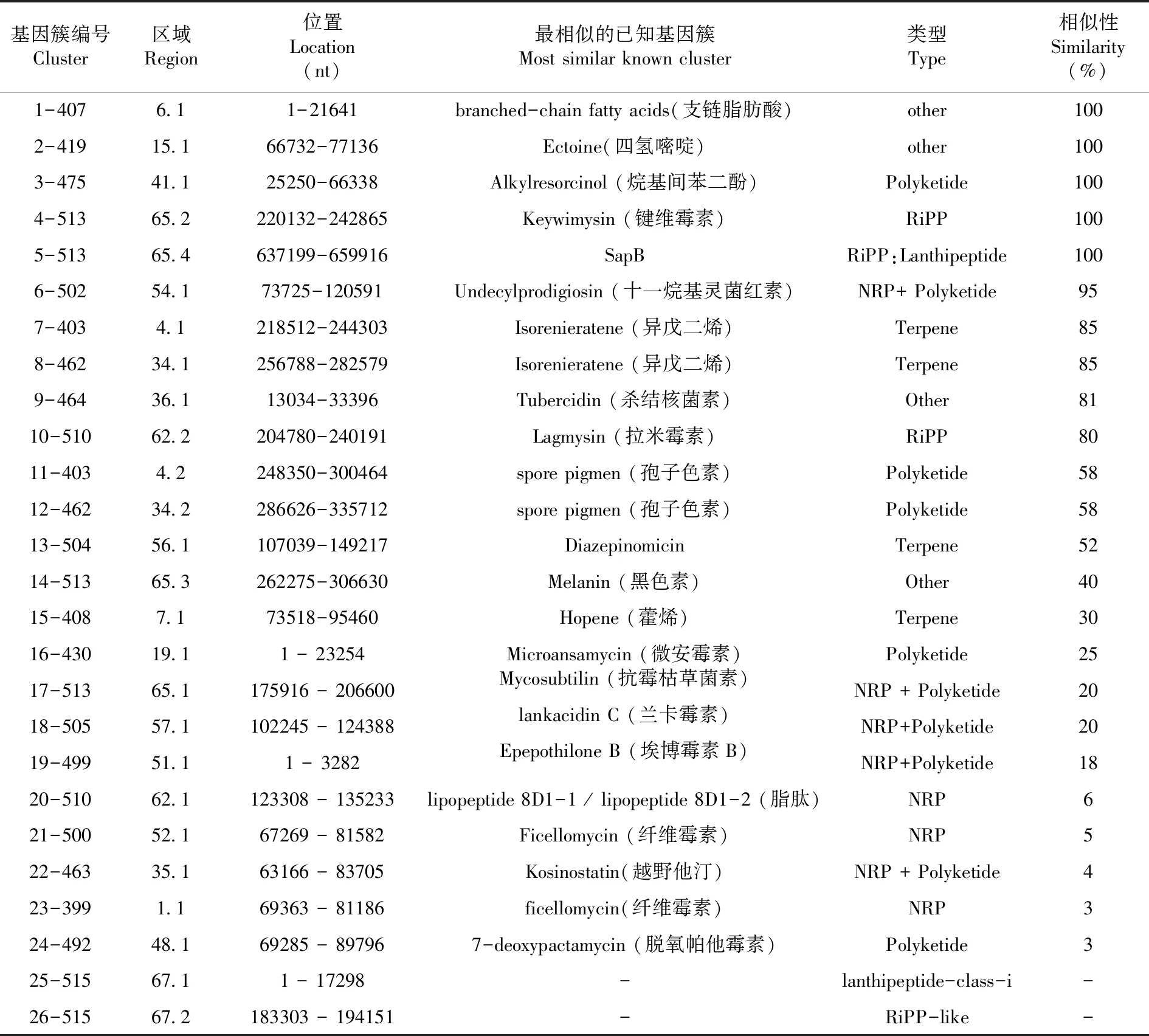
表2 antiSMASH预测S.carminius TRM SA0054的相似度由大到小的基因簇
菌株TRM SA0054 的 全 基 因 组 经 过 antiSMASH预测,其潜在的天然产物生物合成基因簇共有26个,存在8种潜在抗生素生物合成基因簇,具有产生Undecylprodigiosin (十一烷基灵菌红素)、Tubercidin (杀结核菌素) 、Diazepinomicin 、Microansamycin (微安霉素) 、 Mycosubtilin (抗霉枯草菌素)、 Lankacidin C (兰卡霉素)、 Epothilone B (埃博霉素B) 和 Kosinostatin(越野他汀) 8种抗生素及其类似物的潜力。其中,菌株TRM SA0054的Region 36.1基因簇经BlastP比对,得出从基因标号8-ctg36_24至16-ctg36_32号的基因片段分别与文献中报道负责合成StreptomycestubercidicusNBRC 13090的Tubercidin的生物合成基因簇中的主要9个基因tubA~tubI对应,这些基因片段对应的功能蛋白酶可能对生物合成Tubercidin及其类似物有着至关重要的作用,有较大几率产生Tubercidin及类似化合物。
菌株TRM SA0054的Region 36.1基因簇中基因标号8-ctg36_24与基因tubA相似性为74.17%,9-ctg36_25与tubB相似性为72.66%,10-ctg36_26与tubC相似性为78.28%,11-ctg36_27与tubD相似性为78.59%,12-ctg36_28与tubE相似性为68.51%,13-ctg36_29与tubF相似性为78.01%,14-ctg36_30与tubG相似性为79.76%,15-ctg36_31与tubH相似性为51.95%,16-ctg36_32与tubI相似性为68.39%。表3

表3 S.carminiusTRM SA0054基因组中潜在的Tubercidin生物合成基因簇中的功能预测
Tubercidin的生物合成途径中,鸟嘌呤三核苷磷酸(Guanosine triphosphate, GTP)转化为H2NTP(7, 8-dihydroneopterin-3'-triphosphate,7, 8-二氢新蝶呤-3'-三磷酸),tubA编码的TubA是6-carboxy-5,6,7,8-tetrahydropterin (CPH4) synthase(6-羧基-5、6、7、8-四氢蝶呤合成酶),H2NTP经TubA转化为CPH4;tubB编码的TubB是7-carboxy-7-deazaguanine (CDG) synthase (7-羧基-7-脱氮鸟嘌呤(CDG)合成酶)一个自由基SAM酶,CDG生物合成过程中自由基介导的环收缩步骤;tubC编码的TubC是GTP cyclohydrolase I(三磷酸鸟苷环化水解酶 I 型);tubD编码的TubD是一种NADPH依赖的鸟嘌呤核苷酸(GMP,Guanosine monophosphate );还原酶/肌苷一磷酸(IMP,Inosine monophosphate)脱氢酶在生物合成中催化物质 2 (GMP类似物)转化为物质3 (IMP类似物)不可逆的还原脱氨;TubE是Phosphoribosylpyrophosphate transferase (PRPP转移酶, 磷酸核糖转移酶);TubF是UbiD家族脱羧酶;物质3 在结构上与IMP相似,推测物质3 到5 转化的两种酶PurA是(adenylosuccinate synthetase ,腺苷酸琥珀酸合成酶)和PurB(adenylosuccinatelyase,腺苷酸琥珀酸裂解酶),是从初级嘌呤代谢途径中“借用”的。TubG作为一种Nudix水解酶,对于物质5,可以用TubG水解并去除磷酸盐以完成Tubercidin (TBN)的生物合成。图5

注:a.Streptomyces tubercidicus NBRC13090的Tubercidin的基因簇结构图;b.Streptomyces carminius TRM SA0054的Tubercidin的基因簇结构图;c.Tubercidin的生物合成途径[20]
2.5 菌株TRM SA0054发酵粗提物检测
研究表明,标品Tubercidin的分子量为266,Tubercidin标品(300 μg/mL)所示MS检测的分子离子峰MS(ESI): m/z 267.1[M+H]+。优化标品Tubercidin条件后进行LC-MC全扫,寻找到菌株TRM SA0054发酵粗提物样品TB4(分离自硅胶柱层析洗脱的甲醇相30:1段,再过凝胶柱层析获得的样品)以及样品TB7(分离自硅胶柱层析洗脱的甲醇相10:1段,又过凝胶柱层析后得到的馏分)中存在Tubercidin 的分子离子峰MS(ESI): m/z 267.2[M+H]+,以上可确认菌株TRM SA0054发酵可产生Tubercidin。图6
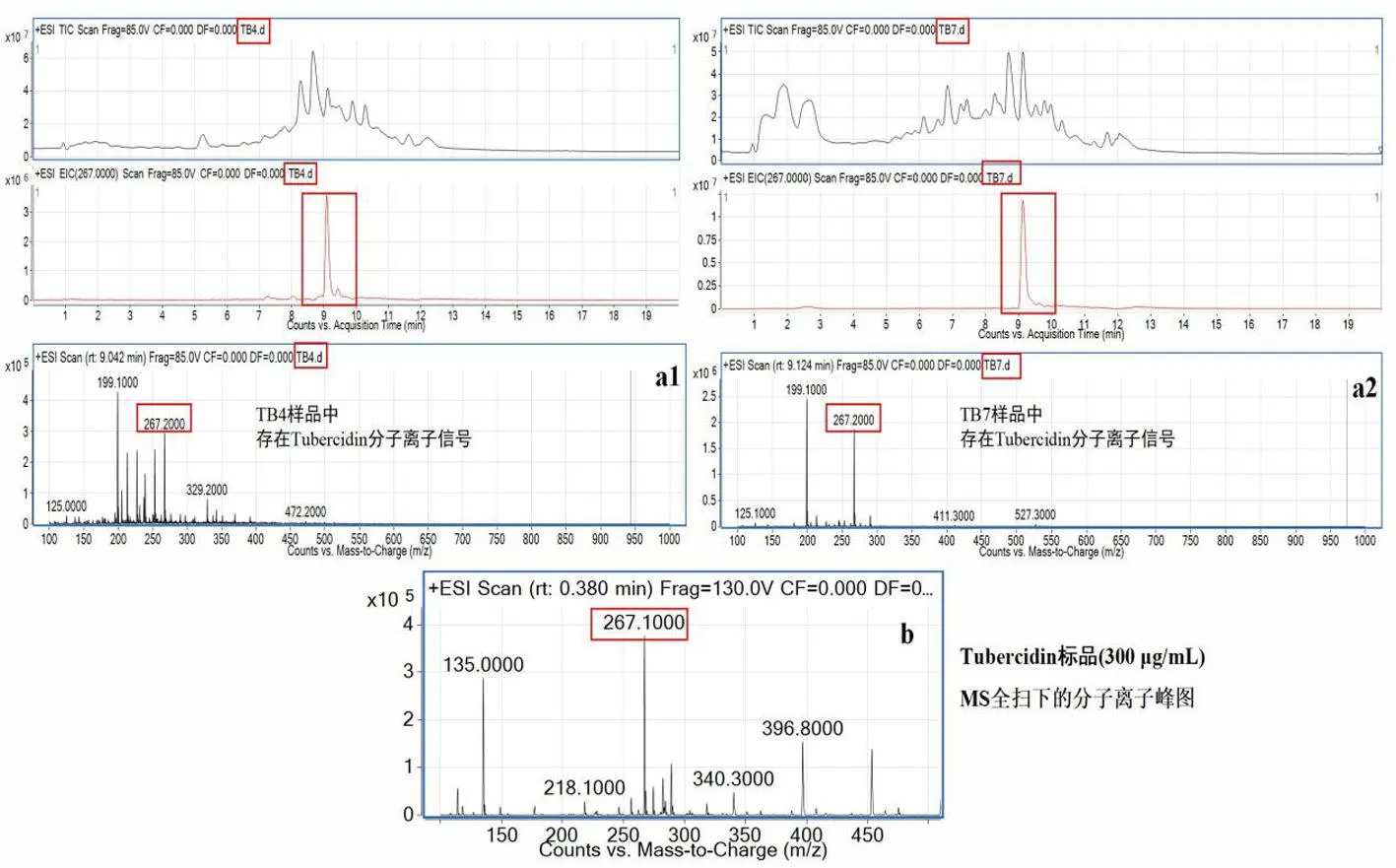
图6 TB4、TB7以及标品Tubercidin(300 μg/mL)的LC-MS全扫检测
3 讨 论
3.1该菌株进行全基因组测序,并且组装后将菌株TRM SA0054基因组进行GO、COG、KEGG功能注释发现大量基因都涉及到了调控、合成、转运、各种次级代谢活动和环境适应。菌株TRM SA0054 的 全 基 因 组 经 过 antiSMASH预测,发现其潜在的天然产物生物合成基因簇共有26个,存在8种潜在抗生素生物合成基因簇,Region 54.1、Region 36.1、Region 56.1三个基因簇预测的化合物分别为Undecylprodigiosin具有产生抗菌、抗肿瘤以及免疫抑制等多种重要的生物活性[19];Tubercidin对血吸虫感染具有显著的活性,同时是潜在的肿瘤化疗药物,Tubercidin结构改造后,还具有腺苷酸激酶的活性[20-22];Diazepinomicin作为抗肿瘤药物具有双重活性,其广谱抗肿瘤作用与多靶向抗肿瘤机制密切相关[23,24],这三个基因簇与己知基因簇具有较高相似度>50%,预示着菌株TRM SA0054有合成这些化合物以及类似物的潜力。
3.2研究基于基因组信息对链霉菌TRM SA0054进行了代谢次级产物的挖掘并在基因组序列中预测分析了相应代谢产物的生物合成基因簇,使得该菌的次级代谢产物背景更为清晰,同时为该菌株的次级代谢产物挖掘建立了生物信息学分析、发酵及产物检测和化合物分离鉴定等系统的研究基础。
3.3在基因水平上预测产物结构,对于微生物天然产物的发现具有重要的指导意义。基因组挖掘更善于合理利用生物信息学分析工具对次级代谢基因族进行预测从而实现产物的定向挖掘,目前是药物发现工作的重要组成部分[25]。
4 结 论
利用全基因组测序获得菌株基因组信息。对S.carminiusTRM SA0054基因组进行分析,挖掘到该基因组中有Tubercidin合成基因簇。该基因簇具有完整的与Tubercidin合成相关的基因。基于基因组挖掘以Tubercidin作为目标化合物,通过 LC-MS检测菌株TRM SA0054发酵产物,验证其具有合成 Tubercidin的能力,预测到的Tubercidin基因簇是表达的。

