基于深层多尺度聚合3D U-Net 的肾脏与肾肿瘤分割方法
张 芳,郝思敏,耿 磊
(1.天津工业大学生命科学学院,天津 300387;2.天津工业大学电子与信息工程学院,天津 300387;3.天津工业大学天津市光电检测技术与系统重点实验室,天津 300387)
肾脏是泌尿系统中至关重要的器官之一,肾肿瘤是一种常见的癌症,是泌尿生殖道癌症中死亡率最高的癌症之一。CT 图像能够准确地呈现肾脏组织和病变组织位置及形态的详细特征,常作为医生初步诊断以及后续决策的重要依据。肾肿瘤的内部组织情况复杂、纹理以及位置变化多端,导致临床诊断难度大,肾肿瘤切除手术难度高,操作过程复杂。医生要制定最佳手术方案,需要清楚地掌握肾脏以及肾肿瘤的位置信息、病变组织的形态大小、边缘信息等。目前临床中主要依靠医生人工标定肾脏与肾肿瘤的位置并测量肿瘤大小,由于医生临床经验不同,存在一定的主观性,常导致诊断结果不唯一。除此之外,肾脏和病灶的识别也是影像科医生的日常工作,尤其是在体检和筛查中,医生的工作量大,耗时费力。因此实现肾脏和肾肿瘤的自动化准确分割至关重要。
近几年,国内外学者对于CT 图像中肾脏和肾肿瘤分割进行了大量探索研究,主要分为两类:一类是基于传统图像处理的分割方法[1-2];另一类是基于深度学习的分割方法[3-6]。传统方法是指利用先验知识以及图像信息进行图像分割。基于传统图像处理的肾脏和肾肿瘤分割方法主要包括3 类:基于区域的分割方法[7]、基于先验知识模型的分割方法[8]和基于可变形模型的分割方法[9]。传统图像处理方法分割肾脏时大多需要人工干预,难以满足实际的医疗需要。深度卷积神经网络是一种具有表征学习能力的运算方法,能够在不需要人工干预的情况下进行自主学习并提取目标特征。深度卷积神经网络通过学习肾脏和肾肿瘤的深层特征,更好地自动分割出肾脏和肾肿瘤。许多研究人员构建了新的解决方案,Zhao 等[10]提出PSPNet 架构,利用金字塔池化模块(pyramid pooling module,PPM)将多尺度特征与不同区域的上下文关联信息相结合,对肾脏和肾肿瘤的分割取得了良好的效果。Yang 等[11]将基本的3D 多模态完全卷积网络(FCN)和金字塔池化模块相结合,实现了肾脏和肾肿瘤的同步分割。Cicek 等[12]提出3D U-Net 架构,将U-Net 网络中的2D 卷积全部替换为3D 卷积,输入网络的数据也从二维数据变为三维数据,CT 切片之间的空间信息得以保留。在此之后,Yu 等[13]又进一步提出了3D Res-U-Net 模型,采用在3D U-Net 收缩路径和扩展路径中使用残差模块的方法提高特征提取能力。Isensee 等[14]提出了基于2D UNet 和3D U-Net 的自适应框架nnU-Net,把重心放在预处理、训练和后处理等工作上,在多个数据集上取得了不错的分割效果。Zhao 等[15]提出的MSS U-Net 在基于nnU-Net 的每个解码层中引入了多尺度监督方案。
以上方法虽然获得了分割结果,但仍然存在以下问题:①肾脏和肾肿瘤与相邻的器官和组织之间边界模糊且CT 值接近,难以区分,造成部分边界分割不准确;②个体差异导致每个患者的肾脏形状、大小、位置情况都不尽相同,分割结果不精确;③肾脏和肾肿瘤的尺度相差悬殊,对部分小尺度肾肿瘤分割效果欠佳。此外,深度学习方法非常依赖于训练数据,如何在数据量较少或数据量不平衡时提高模型的分割效果,也具有重要的研究意义。针对这些问题,本文提出了深层多尺度聚合(DMSA,deep multi-scale aggregation)3D U-Net 网络分割模型。
1 研究方法
1.1 肾脏和肾肿瘤自动分割方法流程
针对肾脏和肾肿瘤的分割难点,本文提出了一种肾脏和肾肿瘤自动分割方法,整体框图如图1 所示。
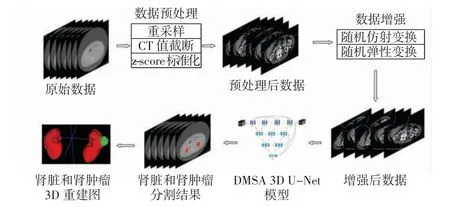
图1 肾脏和肾肿瘤CT 图像分割整体框图Fig.1 Overall block diagram of CT image segmentation of kidney and renal tumor
首先,针对目标与周围组织难以区分、分割结果容易受周围器官组织影响的问题,本文对数据集进行了重采样、CT 值截断和z-score 标准化等一系列数据预处理操作,增强CT 图像中肾脏和肾肿瘤的特征,削弱周围组织器官的特征,降低其对肾脏和肾肿瘤分割结果的影响。其次,针对数据量少的问题,本文对训练集进行随机仿射变换和随机弹性变换增强数据,使得训练集扩增为原来的近10 倍,为网络学习提供了更加多样化的训练集,提高模型的泛化能力,增强模型的分割精度。最后,针对部分目标尺度较小、边缘复杂的分割难点,本文采用了基于3D U-Net、U-Net++[16]和U-Net3+[17]改进的深层多尺度聚合(DMSA)3D U-Net网络模型,利用嵌套不同尺度的3D U-Net 和相邻节点之间的短连接来共享多尺度特征,还在解码器层的每个节点上融合了同样来自解码器层的较大尺度的特征图,增强了网络模型对于较小目标的细节特征提取能力,使得小尺度肾肿瘤边缘得到良好分割。
1.2 数据预处理和数据增强
数据集预处理包括以下3 个步骤:①重采样,即在数据预处理中将所有样本中z方向的层厚距和x、y方向的层间距全部调整为(3.22 mm、1.62 mm、1.62 mm),在分割网络模型中所有样本的空间分辨率保持不变。②CT 值截断[18],即为了增强肾脏和肾肿瘤的特征,减小周围组织器官对分割任务的干扰,经反复实验确定截断阈值,将每个样例序列图像的CT 值限制在[-200,400]的范围内。③z-score 标准化,即用图像中每个像素的灰度值减去肾脏区域的灰度均值再除以肾脏区域的灰度方差,数据样本的CT 值都进行规范化方便后续处理。
将数据集划分为训练集、验证集和测试集,为了避免实验过程中数据量过小导致的模型泛化能力较低,采用随机仿射变换和随机弹性变换的方法,对训练集进行扩充,提高训练样本的多样性。验证集和测试集是检验模型性能的数据集,不能改变其原始的数据分布特征,因此不对验证集和测试集进行数据增强。
1.3 基于DMSA 3D U-Net 的肾脏与肾肿瘤分割网络
现有的网络对于部分形态差异较小、肾脏及肾肿瘤边缘清晰的CT 图像分割效果较好,但是对于特征分布复杂且尺度较小的肾肿瘤以及肿瘤边缘分割效果不佳,这是由于网络在训练过程中学习到的肾脏和肾肿瘤的细节特征不够丰富,导致错过小尺度肾肿瘤和精细的边缘信息。U-Net++网络采用大量的密集跳跃连接将不同层级中尺度相同的特征图进行特征共享,相邻节点之间的语义信息相差较小,易于融合,但编码器层与解码器层之间的特征信息随着中间节点数的增加,语义信息差异逐渐累加,无法实现充分融合。U-Net3+网络通过全尺度的跳跃连接将解码器层各节点与来自编码器的小尺度和等尺度特征映射以及来自解码器的大尺度特征映射直接融合,尽管在一定程度上探索了全尺度的特征信息,但解码器层的特征图与来自编码器的不同尺度的特征图仍然存在差异,无法充分融合,全尺度信息没有得到充分利用。针对上述情况,本文基于U-Net++和U-Net3+提出了深层多尺度聚合(DMSA)3D U-Net 网络,结构示意图如图2 所示。
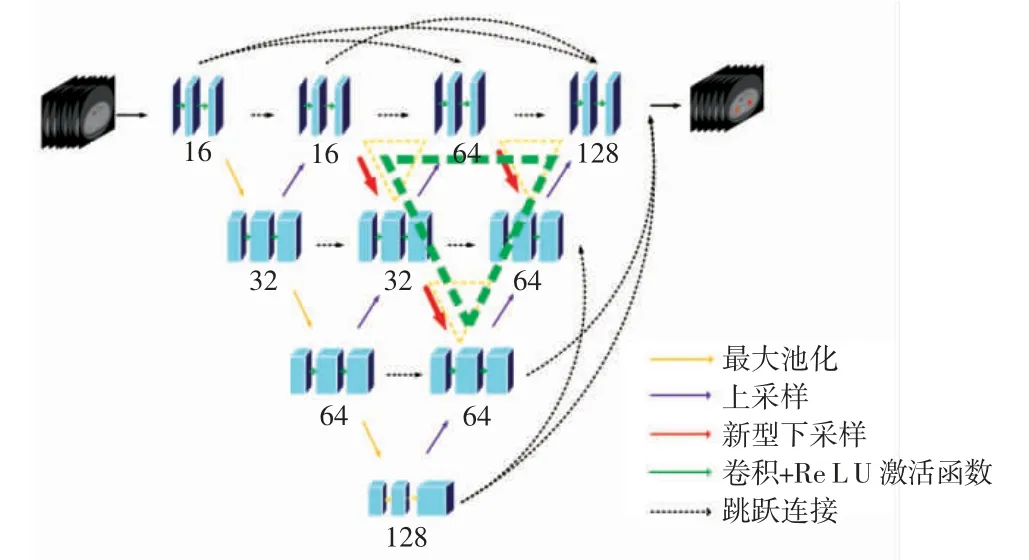
图2 DMSA 3D U-Net 网络结构示意图Fig.2 Schematic diagram of DMSA 3D U-Net network structure
图2 中,DMSA 3D U-Net 网络结构中所有节点之间的黑色虚线短箭头为跳跃连接,该连接为数据前馈过程,仅进行特征共享;黄色箭头为池化下采样,其步长为2;绿色剪头表示对输入图像先进行大小为3×3、步长为1 的卷积操作,然后经过ReLU 激活函数处理的过程;红色箭头代表DMSA 3D U-Net 网络相比于U-Net++网络新增的步长为2 的下采样操作;紫色箭头表示步长为2 的上采样操作;黑色长虚线箭头表示解码器之间和不同深度U-Net 输出之间的全尺度跳跃连接。该网络基于U-Net++和U-Net3+在3 个方面做出改进:①增加了4 个3D U-Net 子网络的嵌套,如图2 中黄色三角和绿色三角部分所示,利用嵌套不同层级的3D U-Net 确定分割任务的最佳深度,促进各3D U-Net 子网络之间的特征共享,对提取到的不同尺度的特征进行压缩和还原,进一步获取多尺度特征;②利用短连接替换传统U-Net 中的跳跃连接,消除网络对于跳跃连接限制的同时,减小参数量;③利用跳跃连接使每个解码器层获得来自解码器层更大尺度的特征图,充分融合解码器层的多尺度特征信息。
本文设计的网络结构具有以下特点:
(1)保留U-Net++的优势,针对不同的数据集或者分割任务进行最佳深度匹配,提升网络运行效率;改进的跳跃连接在解码器上实现了灵活的特征融合,缓解了对跳跃连接的限制;共享部分编码器,简化了网络结构,提高了网络运行效率。
(2)保留U-Net3+优势,将每个解码器层融合了来自解码器的较大尺度的特征图,促进特征共享,获得多尺度特征信息。
(3)在U-Net++基础上增加的下采样操作,进一步提取了目标的抽象特征,多个3D U-Net 子网络的嵌入促进了编码器层和解码器层的特征共享,有利于网络掌握目标更细节的特征信息。
(4)将U-Net3+内部编码器和解码器之间联系各个尺度特征图的跳跃连接,替换为密集短连接,相邻节点之间的特征共享易于不同尺度且携带不同语义信息的特征图的特征共享,能够充分利用多尺度特征信息,进一步促进特征融合。
上述优势可以充分利用多尺度信息,提升网络对于尺度较小、边缘复杂的肾肿瘤的分割力。
2 实验结果分析
2.1 实验数据
本文使用的数据集是KiTS19 公开数据集[19],该数据集共有210 例样本以及所对应的样本金标准,随机抽取168 例样本作为训练集、21 例样本作为验证集,另有21 例样本构成测试集。在经过上文所提的数据增强后,训练集样本数量扩充至1 180 例,验证集和测试集保持不变。数据集裁剪成大小为(32,128,128)的格式后输入网络。以下实验所用数据都源于该数据集。
2.2 评价指标
本文的任务是在三维CT 扫描图像中分割肾脏和肾肿瘤,采用医学图像分割中最常用的评价指标Dice相似性系数(Dice similarity coefficient)[20]对本文方法的分割性能进行评价。
Dice 系数是集合相似度度量函数,常用于计算2个样本的相似度,其定义如下:
式中:P为网络模型预测输出的肾脏和肾肿瘤分割结果;G为真实的肾脏和肾肿瘤区域;P和G的交集为正确分割区域。该评价指标越高表示模型的分割效果越好。
2.3 实验环境及超参数配置
本文实验中硬件配置包括:①CPU 为Intel i7-10700k@3.8GHz;②GPU 为NVIDIAGeForceGTX3060-Ti;③操作系统为Windows10 操作系统;④深度学习框架为Pytorch1.8.1+cuda11.1;⑤图像预处理库为MONAI。网络在训练过程中采用Adam 优化算法,初始学习率设置为0.001,并采用ReduceLROnPlateau 学习率自动衰减,Patience 设为50,表示当Loss 在连续50个Epoch 中均未下降时,则学习率自动减小为原来的1/10。batch size 设为4。
2.4 实验结果分析
图3 为肾脏和肾肿瘤分割效果图。

图3 DMSA 3D U-Net 网络分割肾脏和肾肿瘤Fig.3 Segmentation of kidney and renal tumors by DMSA 3D U-Net network
由图3 可见,DMSA 3D U-Net 网络能够准确地分割出肾脏和肾肿瘤的形状特征。图4 为DMSA 3D U-Net网络训练过程中损失函数随迭代次数的曲线变化图。

图4 DMSA 3D U-Net 的Dice、Loss 曲线Fig.4 Dice and Loss curves of DMSA 3D U-Net
从图4 中的准确率曲线可以看出,网络对肾肿瘤分割的准确率随着Epoch 数量增加而逐步提升,最终趋于稳定。从损失曲线可以看出,网络的损失下降较快,并且最终趋于稳定状态。
2.4.1 与经典网络实验结果对比
为进一步评估本文网络的性能,将本文网络与U-Net、2D PSPNet、3D U-Net 、3D FCN_PPM[21]、3D UNet++和3D U-Net3+网络进行比较。表1 展示了不同网络模型在测试集上的Dice 系数,由表1 可以看出,本文提出的网络分割性能最佳。

表1 经典网络的分割评价指标Tab.1 Segmentation evaluation indexes of classical network
图5 为上述几种网络和本文提出的DMSA 3D UNet 网络的肾脏和肾肿瘤分割结果对比图。

图5 肾脏和肾肿瘤分割结果图Fig.5 Segmentation results of kidney and renal tumor
图5(a)为肾脏和肾肿瘤的3D 重建图,图5(b)为矢状面,图5(c)为横断面,图5(d)为冠状面。由图5可以看出,在3D U-Net 网络和3D FCN_PPM 网络的分割结果中,肾脏外围出现了极小一部分误分割的红色区域,并且分割得到的肾脏表面有明显凸起,肾肿瘤分割结果偏大。3D U-Net++网络的分割结果在右边肾脏顶部出现明显的缺口,肾肿瘤分割结果偏大。3D U-Net3+网络的分割结果在右边肾脏顶部有较小的缺口且肾脏表面凸起,肾肿瘤分割结果偏大且肿瘤表面存在不同于金标准的凸起部分。本文提出的DMSA 3D U-Net 网络对特征分布复杂的肾肿瘤目标分割效果最好,分割结果最接近图5 中的金标准。
实验结果表明,由于3D 网络进一步利用了空间信息,所以3D 网络模型的准确率高于2D 网络模型,分割精度得到提升。在所有的3D 网络分割模型中,对肾肿瘤分割精度差异较大,相比之下本文设计DMSA 3D U-Net 的网络对肾脏以及肾肿瘤的分割结果都最接近于金标准,平均准确率较高,尤其是对肾肿瘤分割精度提升最为明显,说明该网络可以充分优化参数,精确分割出肾脏和肾肿瘤。
2.4.2 将U-Net 与不同模块结合的实验结果对比
最后,对比了在2D 或3D U-Net 网络上加入不同模块的分割效果。将本文提出的网络结构与Attention-U-Net[22]、3D ResU-Net 网络进行比较,实验结果如表2 所示。
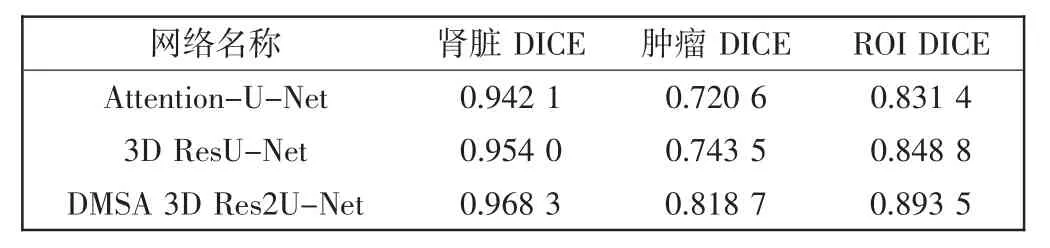
表2 U-Net 与不同模块结合网络分割评价指标表Tab.2 Segmentation evaluation index table of U-Net combined with different modules
由表2 可以看出,本文提出的DMSA 3D U-Net 网络对肾脏和肾肿瘤的分割准确率最高。
肾脏和肾肿瘤尺度相差悬殊,通过对数据集中肾脏和肾肿瘤的分析发现,肾脏区域的平均像素数在5 200 左右,部分样本中肾肿瘤要比肾脏小得多,再加上肾肿瘤数量和位置不确定,模型训练难度较高。为了进一步展示本文方法对边缘复杂以及小尺度的肾肿瘤的分割性能。图6 和图7 分别展示对边缘复杂的肾肿瘤和小尺度肾肿瘤的处理效果。为更清晰地比较分割结果的细节,每个分割结果包含3D 建模正面和俯视2 个视角。

图6 肾脏和边缘复杂肾肿瘤分割结果3D 建模Fig.6 3D modeling of the segmentation results of kidney and marginal complex renal tumor

图7 肾脏和小尺度肾肿瘤分割结果3D 建模Fig.7 3D modeling of the segmentation results of kidney and small-scale renal tumor
由图6 可见,3 种模型均可以初步定位出肾脏和肾肿瘤的位置及边缘信息,但Attention-U-Net 和3D ResU-Net 网络分割的肾肿瘤不完整且伴有缺口。例如Attention-U-Net 中左肾出现了肾肿瘤错误预测,3D ResU-Net 网络分割中右肾的左上方出现与其他区域不连通的肾肿瘤部分,误分割明显。而本文提出的DMSA 3D U-Net 网络的分割结果与金标准最接近,说明DMSA 3D U-Net 网络对于边缘复杂的肾肿瘤分割效果最好。
图7 中,因肾脏尺度较大,所以分割难度较小,3种网络均得到了较好的肾脏分割效果。而对于小尺度的肾肿瘤部分,Attention-U-Net 和3D ResU-Net 网络分割出的肾肿瘤明显大于图7 中的肾肿瘤金标准,还出现了肾肿瘤与肾脏粘连现象,分割效果不佳,如Attention-U-Net 网络的分割结果和3D ResU-Net 网络分割效果所示。相比而言,DMSA 3D U-Net 网络对肾肿瘤的大小、边缘分割较为精确,分割效果良好,如3D ResU-Net 网络分割效果所示。实验表明,与其他网络相比,DMSA 3D U-Net 网络对于小尺度肾肿瘤的分割效果较好。
以上实验结果充分体现了本文提出的DMSA 3D U-Net 网络的有效性,通过提取多尺度的特征信息并进行融合,可以更好地识别目标的细节特征,说明本文所提出方法适用于三维CT 图像的肾脏和肾肿瘤分割任务。
3 结 论
提出了一种基于深层多尺度聚合3D U-Net 网络的肾脏与肾肿瘤自动分割方法。通过嵌套不同深度的3D U-Net,通过相邻节点短连接的方式进行特征融合。各层级3D U-Net 子网络的输出以及解码器层均采用跳跃连接,将不同尺度的特征图拼接融合,进一步增强模型对于细节特征的提取能力。实验结果表明,DMSA 3D U-Net 网络能够有效解决小肿瘤漏诊和大肿瘤欠分割的问题,在边缘复杂、小尺度肾肿瘤等多种困难样本中分割效果良好。在KiTS19 公开数据集上进行评估,DMSA 3D U-Net 网络对肾脏分割的Dice系数为0.968 2,对肿瘤分割的Dice 系数为0.790 8。

