融合跨物种科学数据的性状调控基因本体模型构建及应用
张丹丹 赵瑞雪,2 鲜国建,3 熊赫
(1.中国农业科学院农业信息研究所,北京 100081;2.国家新闻出版署农业融合出版知识挖掘与知识服务重点实验室,北京 100081;3.农业农村部农业大数据重点实验室,北京 100081)
在作物育种科学研究中,挖掘同时调控抗旱、抗病、抗虫等多个优异性状的多效基因,是获得高产、优质作物新品种的关键。然而,基因和性状之间关联假设的产生,往往需要组织关联分析多维度的科学数据。2018 年初,美国康奈尔大学玉米作物育种学家、美国科学院院士Edwards Buckler 教授提出了“育种4.0”的理念,强调生命科学、信息科学与育种科学的深度融合[1]。这一理念的提出,推动着作物育种科研范式,从以假设驱动的被动探索,向数据驱动的主动知识发现转变。因此,基于多维度科学数据融合的领域知识表示模型,进而提出科学假设已经成为重要的研究方向。然而,领域科学数据的复杂性和异构性使得数据间的互操作性和数据集成极其困难,阻碍了学科新知识的发现。有研究表明关联融合不同的科学数据集,可以有效提高知识的获取效率和实现新学科知识的发现[2-3]。本体模型作为多源科学数据集关联组织的关键框架体系,为后续多维度科学数据的融合和深层次学科知识挖掘奠定了重要的基础。
近年来,国内外学者在生物科学领域开展了大量的本体模型构建与应用方面的研究。在本体模型数据组织方面,已有的本体模型多以某类科学数据为中心实体构建语义表示模型,用于语义搜索和知识问答。例如,基因本体(gene ontology,GO)是一个适用于多物种、对基因和蛋白质功能进行限定和描述的标准词汇体系,对基因产物从功能、参与的生物途径、细胞中的定位进行注释,包括了基因的细胞组分、分子功能和生物学过程[4]。基因组和蛋白组注释本体GoMiner(gene ontology miner),以有向无环图和结构树的形式,展示了基因本体结构框架中的基因注释信息[5]。细胞周期本体(cell cycle ontology,CCO),是一个用于表示和集成分析细胞周期过程的应用本体,集成了与细胞周期调控相关的概念以应用于细胞周期研究中[6]。作物本体(crop nntology,CO)包含了作物的形态、生理、品质以及生物和非生物胁迫等性状的相关信息,可以为任何给定的性状提供结合地理和环境数据的表型数据[7-8]。植物本体(plant ontology,PO),采用RDF(resource description framework)数据模型来描述植物不同发育阶段的器官、细胞等形态解剖结构[9]。在本体模型应用方面,英国洛桑研究所提出一种开放关联的领域本体模型,最终形成了面向作物领域的知识图谱KnetMiner(knowledge network miner),以支持基因调控网络的知识查询[10]。法国农业国际合作研究发展中心采用语义网技术,对来自多个专业数据库的数百种数据集和相关领域本体进行关联整合,以应用于基因组学、蛋白组学和表型组学相关知识的查询[11]。Linked Life Data 关联整合了含有基因、蛋白质、药物、靶点多个类型的生物医学实体,支持医学领域知识更为全面地检索[12]。隗玲等[13]提出基于“主语-谓语-宾语(subject -predicationobject,SPO)”三元组的生物医学领域知识发现本体模型,可直观地支持旧药新用的知识发现。Luciano等[14]开发了转化医学本体(translational medicine nntology,TMO),利用语义Web 技术将化学、基因组和蛋白质组数据与疾病、治疗和电子健康记录集成在一起,用于探索致病机理,以更好地了解治疗方案、疗效和作用机制。Lam 等[15]提出了一种语义Web方法,通过使用资源描述框架来构建本体模型以整合各种神经科学数据,并基于该本体构建了知识图谱,为神经科学家提供一个发现新知识的综合平台。
调研结果表明,现有的领域本体模型还存在以下的局限。一是,较少涉及多维度科学数据的融合,知识获取效率低;二是,多应用于语义检索和知识问答,缺少知识发现的语义关联应用。本研究针对现有作物领域本体模型应用中,知识获取效率低和优异多效基因发现困难的问题,提出一个性状调控基因本体模型的构建方法;并基于功能基因挖掘的知识服务需求,详细阐述性状调控基因本体模型的应用功能,旨在为实现跨物种间科学数据的融合和多维度科学数据集的组织关联奠定良好的基础。
1 材料与方法
1.1 数据来源
本研究以PubMed 数据库为文献数据来源,同时还选取了其他8 个领域知识库作为数据来源,包括Phytozome(4 个物种的基因组信息)、Ensemble(European molecular niology laboratory's European bioinformatics institute)(4 个物种的基因组信息)、RGAP(rice genome annotation project)(水稻基因组的注释信息)、UniProt(universal protein resource)(4 个物种的蛋白注释信息)、STRING(search tool for recurring instances of neighboring genes)(4 个物种的蛋白互作信息)、Pfam(protein family)(4 个物种的蛋白质家族信息)、KEGG(Kyoto encyclopedia of genes and genomes)(4 个物种的通路注释信息)和GO(gene ontology)(4 个物种的通路注释信息)。在此数据来源的基础上,选取模式植物拟南芥和主粮作物水稻、玉米、小麦为所有实体及其相关属性的科学数据采集对象。从生长发育指标、抗逆指标、抗病虫指标和经济指标等多个不同育种目标层面综合考量后,选取了6 个关键词作为“性状”实体,包括株高(plant height)、抗旱(drought resistance)、抗盐(salt resistance)、抗病(disease resistance)、抗虫(insect resistance)和粒重(grain weight)。
1.2 方法
1.2.1 构建本体模型 本体模型描述了抽象层次上由实体及其他之间的关系建立而来的逻辑模型。本研究构建的性状调控基因本体模型框架如图1 所示,以性状、基因和蛋白为中心实体,关联融合基因水平、蛋白水平、富集通路水平与性状水平4 种数据类型维度的科学数据,通过14 个对象属性组织关联了13 种实体,并且赋予了每种实体相关的数据属性。最终,构建了涵盖13 种实体、16 种数据属性和14个对象属性的性状调控基因本体模型。
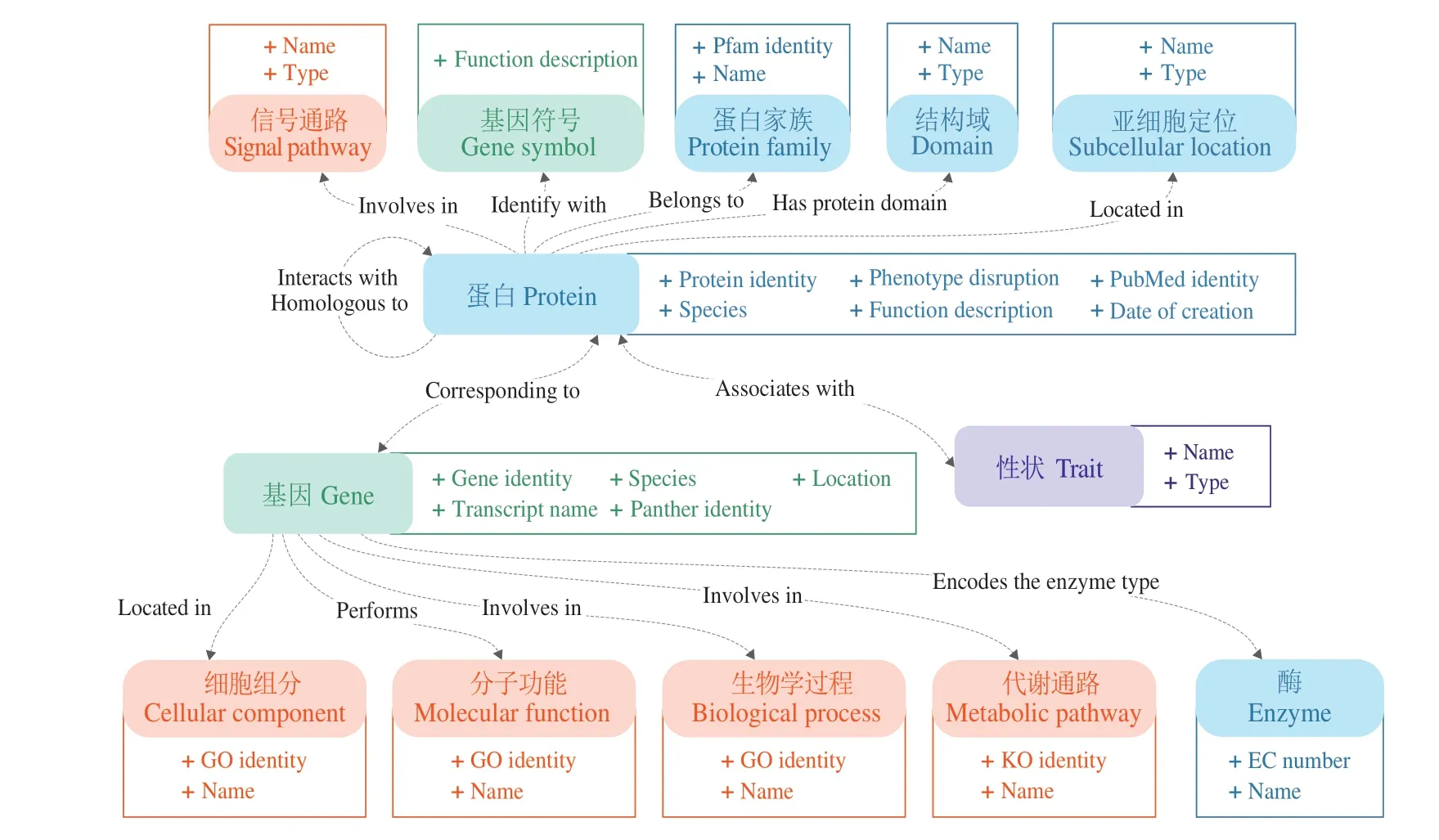
图1 性状调控基因本体模型框架Fig.1 Trait-regulated-genes ontology model framework
(1)定义实体的层次结构。实体用于描述具有相同属性的一类概念的集合。以“性状(trait)” “基因(gene)” “蛋白(protein)”为中心实体,以此延伸定义了另外10 种相关实体,包括根据蛋白功能描述缩写定义的“基因符号(gene symbol)”实体,根据蛋白质结构定义的“蛋白家族(protein family)”和“结构域(domain)”实体,根据蛋白功能定义的“酶(enzyme)”和“亚细胞定位(subcellular localization)”实体,根据基因参与的代谢富集通路定义了“生物学过程(biological process)” “细胞组分(cellular component)” “分 子功能(molecular function)” “KEGG 通路(KEGG pathway)”和“信号通路(signal pathway)”实体。依据不同的科学数据类型维度,将实体分为基因水平、蛋白水平、富集通路水平和性状水平4 个层次维度(表1),并对每类实体进行了科学的描述释义和数据属性的确定。
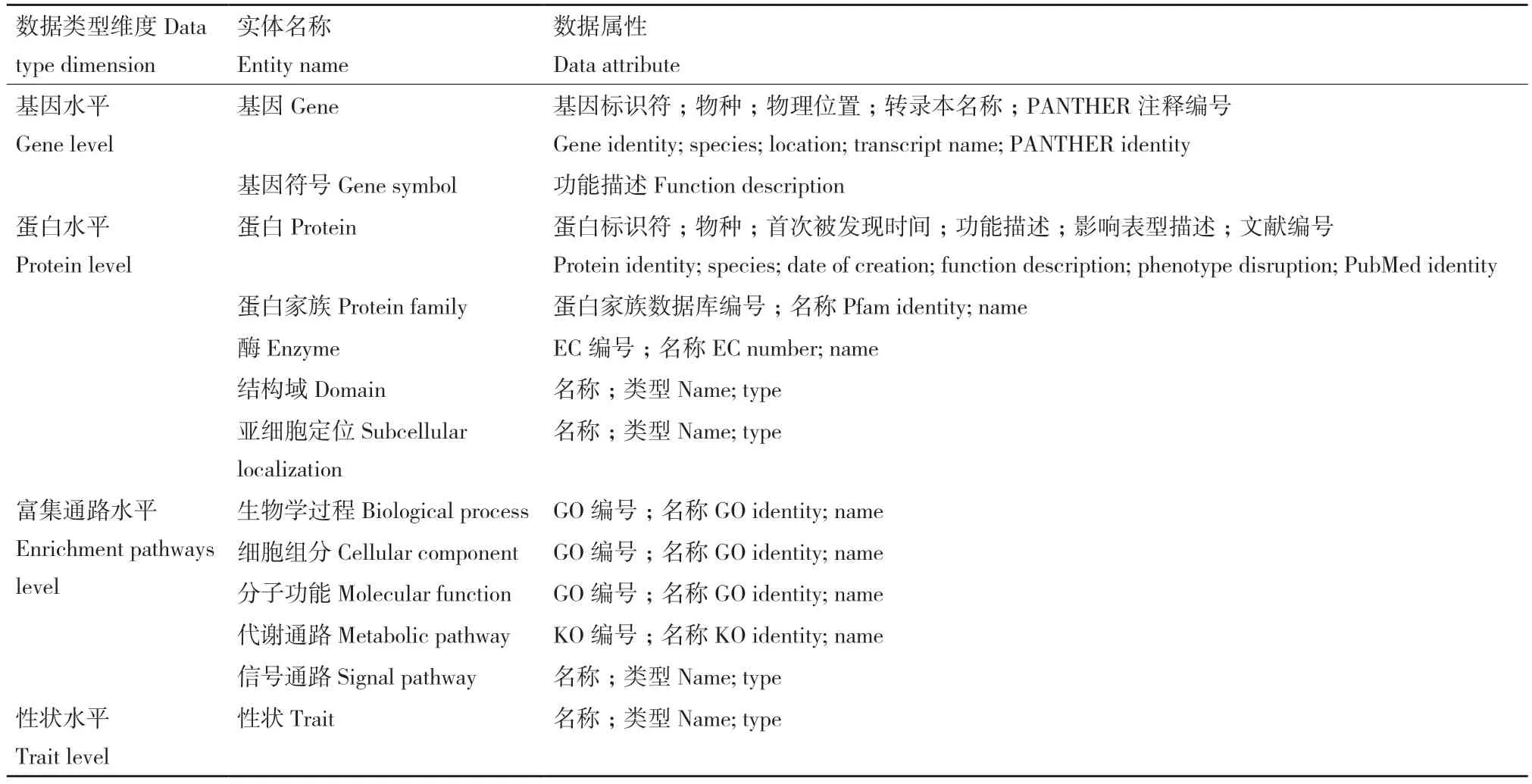
表1 性状调控基因本体模型中实体的释义Table 1 Description of entity classes in trait-regulated-genes ontology model
(2)定义实体的数据属性。实体的数据属性即实体自身所具有的特征,即如果一个实体具有某一属性,则这个实体类型中的所有实体均有此种属性。针对本体模型框架中的13 种实体,结合本体模型的应用需求,优先选择了与其他实体类型关联的数据属性保留。基于以上的数据属性筛选原则,共定义了16 种数据属性(表2)。例如,定义蛋白实体的数据属性有“蛋白标识符(protein identity)”,作为蛋白实体中节点名称的唯一标识符,便于用户的关联检索;“首次被发现时间(date of creation)”,作为性状调控基因发现结果验证的文献回溯依据;“功能描述(function description)”,用于描述蛋白的主要分子功能;此外,还为蛋白实体添加了相关的“物种(species)” “文献编号(PubMed identity)” “影响表型描述(phenotype disruption)”数据属性信息。

表2 性状调控基因本体模型中实体数据属性释义Table 2 Description of data attributes in trait-regulated-genes ontology model
(3)定义实体的对象属性。实体的对象属性能够揭示两个实体之间的语义关系,是本体模型中进行逻辑推理的关键,决定领域知识图谱的丰富程度和应用效果。根据本体模型应用场景的需求,最终定义了14 个对象属性(表3)。下面仅以蛋白实体的对象属性为例,阐述具体的定义过程。以基因、蛋白和性状为中心实体,通过“与……有关(associates with)”对象属性将性状实体与蛋白实体进行链接,实现蛋白与性状关联关系的建立。通过“与……同源(homologous to)”对象属性建立起两个蛋白之间的关联,作为本体模型中的关键对象属性,也是实现跨物种间多维度科学数据融合的重要基础。同时,通过“与……互作(interacts with)”建立起互作蛋白之间的关系。在此基础上,通过“与……相对应(corresponding to)”构建起蛋白和基因间的关联关系。通过“与……一致(identify with)”对象属性建立蛋白和基因符号间的关联关系,作为跨物种间基因功能知识发现的关键。

表3 性状调控基因本体模型中对象属性释义和数据来源Table 3 Description of object attributes in trait-regulated-genes ontology model and data sources
1.2.2 构建知识图谱 首先,以性状(traits)描述关键词分别为检索词,在Uniprot 数据库中获得蛋白ID 和所对应的相关文献(源于PubMed)。并进一步人工校验文献与性状间的关系,建立蛋白(ProteinID)-有关-性状(traits)三元组。
在Uniport 数据库中下载得到水稻、玉米、小麦和拟南芥4 个物种的蛋白质氨基酸序列,利用BLAST(序列相似度比对)工具获取跨物种间的所有同源蛋白三元组[16-17],从中选取identity>=35%且E-value<10-20的同源蛋白[18-19]。并进一步筛选出蛋白-有关-性状三元组中的已知性状蛋白,建立起蛋白-同源-蛋白三元组。在Phytozome 和RGAP下载每个物种所对应的蛋白质氨基酸序列,利用BLAST(序列相似度比对)工具建立蛋白-对应-基因三元组。
在Uniport 中获取所有蛋白实体有关的科学数据关联信息,并选取domain、subcellular location、gene symbol 和signal pathway 作为实体类型,分别建立protein-has protein domain-domain、protein-located in-subcellular location、protein-identify with-gene symbol、protein-involves in-signal pathway 三元组。在STRING 数据库中批量获取互作蛋白相关的关联数据,构建protein-interacts with-protein 三元组。在Pfam 数据库中获取蛋白家族相关的关联数据,构建protein-belongs to-protein family 三元组。并获取蛋白相应的属性信息,特别添加蛋白首次被发现的时间。
在GO 数据库中获取molecular function、cellular component 和biological process 作为实体类型,分别建 立gene-performs-molecular function、gene-located in-cellular component、gene-involves in-biological process 三元组。在KEGG 数据库中获取enzyme、metabolic pathway 作为实体类型,分别建立gene-encodes the enzyme type-enzyme 和gene-involves in-metabolic pathway 三元组。并在Ensembl plants 中获取基因相应的属性信息。最终,将构建好的多类型三元组数据存储到Neo4j 图数据库中。
2 结果
2.1 跨物种学科知识关联检索
基于以上的材料和方法,在性状调控基因本体模型为模式层的知识图谱中进行实验。实验结果表明,该图谱不仅可支持跨物种间多维度科学数据关联检索,还能细致地展示检索词及与之相关的对象属性和关联实体,从而实现跨物种间学科知识的关联发现,提高知识的获取效率。如图2-A 所示,以基因(LOC_Os01g40094)为检索词,不仅可以获取到该基因在基因水平的知识,还可通过跨物种间基因的关联,获取到与之同源的拟南芥基因AT4G26080 在基因水平、蛋白水平和富集通路水平上不同科学数据维度的知识,进而实现水稻基因LOC_Os01g40094(检索词)相关知识的发现。同时,还可以展示实体的数据属性信息。图2-B 所示为蛋白P49597 的相关数据属性信息,主要包括首次被发现时间、影响表型描述、功能描述以及蛋白标识符等。
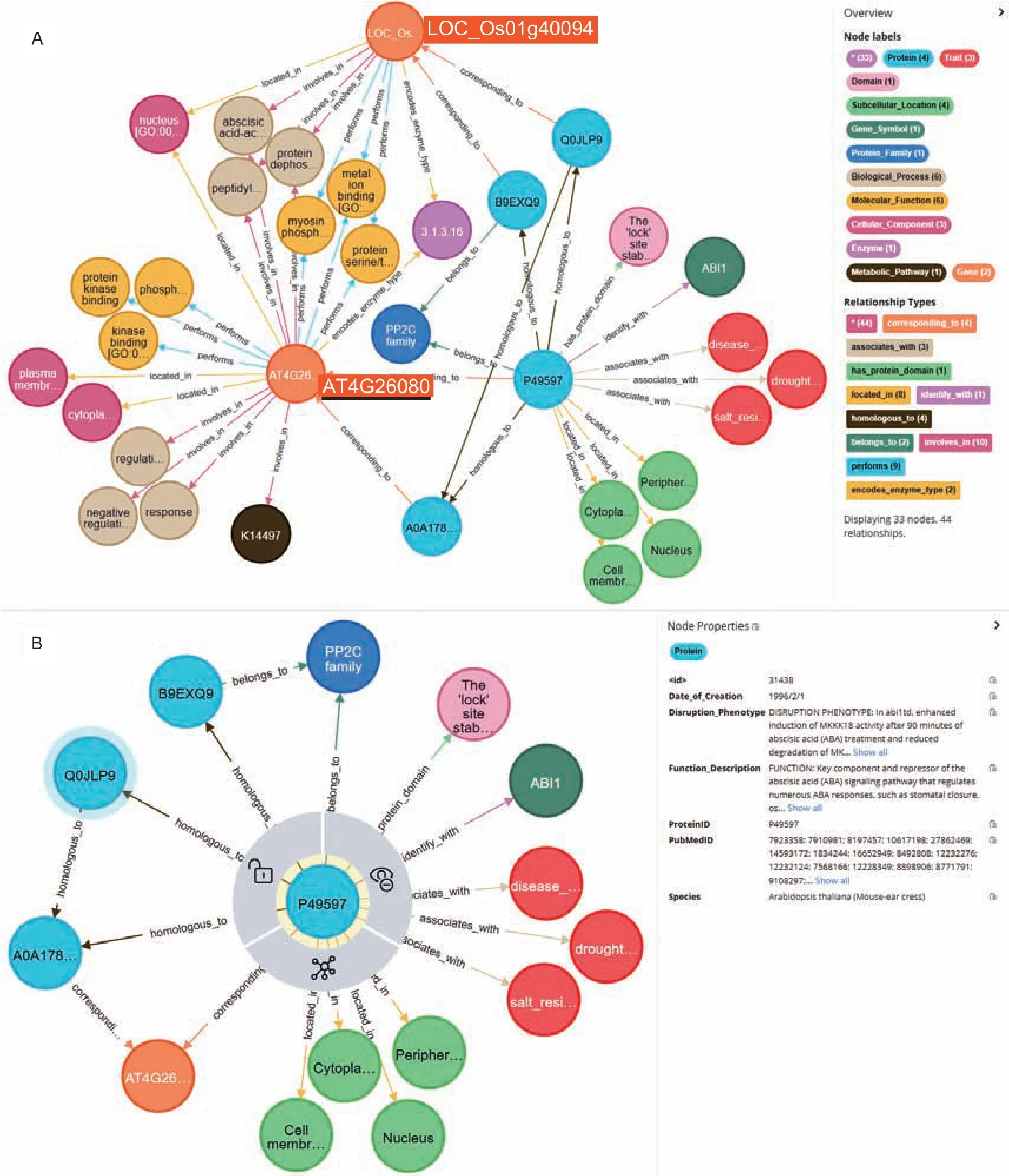
图2 性状调控基因知识图谱的层级知识结构Fig.2 Hierarchical knowledge structure of trait-regulated-genes knowledge graph
2.2 基因调控性状的预测
基于性状调控基因知识图谱,提出了综合3 种类型关联路径的基因调控性状预测方法。其中path1是通过同源蛋白路径将未知性状基因与性状关联,path2 是通过基因层面其他共联节点路径将未知性状基因与性状关联,path3 是通过蛋白层面其他共联节点路径将未知性状基因与性状关联。其中,公式中的gene1 为未知性状基因,protein1 为gene1 所对应的蛋白,protein2 为protein1 的同源蛋白,Trait 为已证实的protein2 的关联性状。
从以上3 种类型的关联路径对基因的调控性状进行推理预测,由于A1 和A2 是多种类型的实体集合,所以path2 和path3 可以有多条关联路径,而且path1、path2 和path3 的路径越多,gene1 与Trait 关联的可能性越大。因此,定义当gene1 具有path1,并且同时具有至少一条path2 或者path3 中的关联路径时,即如果sum(path1)>0 and(sum(path2)>0 or sum(path3)>0),预测gene1 与Trait 产生关联。
以小麦基因TraesCS4B02G060000 调控性状的预测为例。图3 是以基因(TraesCS4B02G060000)为检索词,下面仅以关联检索到的株高(plant height)性状为例阐述具体的路径关联原理。文献已证实图3 中玉米基因Zm00001d017742[20]与株高性状相关,在本研究所构建的性状调控基因知识图谱中,基于同源蛋白关联路径,即TraesCS4B02G060000 -[corresponding to]-A0A1D5XPT6-[homologous to]-A0A060D764 -[associates with]-plant height,建立起TraesCS4B02G060000与plant height之间的关联。在此基础上,在基因层面,通过TraesCS4B02G060000-[located in]-nucleus(GO:0005634)-[located in]-Zm00001d017742 -[corresponding to]-A0A060D764 -[associates with]-plant height 路 径可实现TraesCS4B02G060000 与plant height 之 间的关联。在蛋白层面,通过TraesCS4B02G060000-[corresponding to]-A0A1D5XPT6 -[belongs to]-GRF family-[belongs to]-A0A060D764 -[associates with]-plant height 路径也可实现TraesCS-4B02G060000 与plant height 之间的关联。由图3 可知,在性状plant height 节点和小麦基因TraesCS-4B02G060000 节点之间共有8 条路径可达,每条路径都为两个节点之间关联关系的建立提供了一条有效的数据支撑。

图3 基因调控性状的预测Fig.3 Prediction of gene-regulated-trait
2.3 优异多效基因的挖掘
基于性状调控基因知识图谱,利用上一结果中所提到的基因调控性状预测方法,实现了优异多效基因的挖掘,包括已知的多效功能基因31 个和未知的多效功能基因26 个(附表1、附表2)。如图4 所示为具有“drought resistance” “salt resistance”“grain weight” “plant height”组合性状的多效基因TraesCS3D02G078500 的挖掘。文献已证实图4 中拟南芥基因AT3G10500 与抗旱性状相关[21],水稻基因LOC_Os11g03370 与抗旱和抗盐性状相关[22],拟南芥基因AT5G39610 与抗盐性状相关[23],水稻基因LOC_Os04g38720 与粒重和株高性状相关[24-25],根据多类型关联路径的基因调控性状预测方法,图中显示小麦基因TraesCS3D02G078500 与性状“drought resistance” “salt resistance” “grain weight”“plant height”之间除了可通过同源蛋白的路径实现关联,在基因层面和蛋白层面也分别均有多条路径可建立关联。推测小麦基因TraesCS3D02G078500 很可能是一个优异的多效基因,可能与抗旱、抗盐、粒重和株高性状相关。
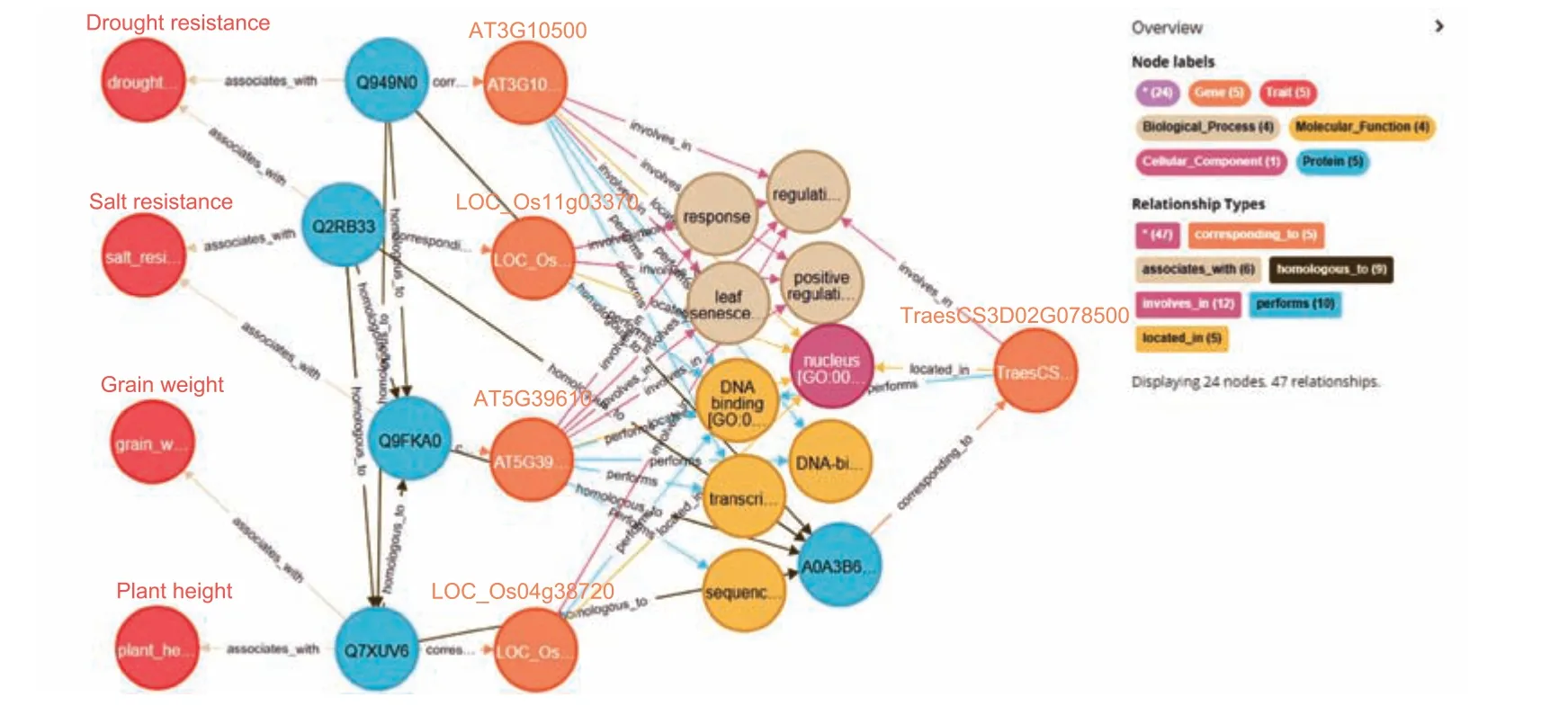
图4 多效基因TraesCS3D02G078500 的挖掘Fig.4 Mining of the elite pleiotropy gene TraesCS3D02G078500
2.4 跨物种基因功能的预测
基于性状调控基因知识图谱,利用关联规则的知识挖掘方法,可实现跨物种基因功能的高效预测。基于function identified with 关联推理规则,即
IF gene1-[corresponding to]-protein1-[identify with]-gene symbol S and
gene2-[corresponding to]-protein2-[identify with]-gene symbol S
Then gene1-[function identified with]-gene2
以小麦基因TraesCS2D02G261300 的功能预测为例,来阐述跨物种基因功能的预测。以小麦基因TraesCS2D02G261300 为检索词,如图5 所示,在本研究构建的性状调控基因知识图谱中关联检索可知,拟南芥基因AT1G48520、水稻基因LOC_Os11g34210和玉米基因Zm00001d052622 在细胞组分、分子功能、生物学过程、代谢通路和酶等数据类型层面均有相关的功能注释信息,且这些基因之间都有着共联的实体节点。此外,小麦基因TraesCS2D02G261300 与以上的功能基因有着相同的基因符号GATB。结合学科知识关联推理规则,根据所关联到的基因功能注释信息,可以对小麦基因TraesCS2D02G261300 进行基因功能的预测,为进一步的基因功能研究提供了科学的实验指导。

图5 跨物种基因功能预测Fig.5 Prediction of gene function across species
3 讨论
现有的作物领域本体模型多应用于知识关联检索,优异多效基因的发现常常受到多维度科学数据整合的挑战[8-9]。针对现有的困境,本研究构建了性状调控基因本体模型。一方面,此模型通过同源蛋白对象属性的建立,实现了跨物种间多维度科学数据的关联融合,有效建立起了跨物种实体节点间的关联关系,为跨物种间学科知识关联检索提供了关键的数据融合基础。另一方面,此模型关联整合了基因和性状间多源异构的科学数据,可直观展示多维度科学数据间的组织关联,有效地建立起了一个基因与多个性状间的隐含关联关系,为优异多效基因的发现提供了重要的知识组织框架体系。以此模型为模式层的知识图谱,可以实现已知功能多效基因和未知功能多效基因的挖掘,可实现跨物种间学科知识的关联检索。本研究较好地解决了优异多效基因发现困难与跨物种学科知识获取效率低的问题。
本研究构建的性状调控基因本体模型不仅可应用于跨物种间调控基因的关联发现,也能应用于优异多效基因的挖掘。如以某个基因ID(LOC_Os01g40094)为检索词,可通过跨物种间同源基因的关联,获取到与之关联的其他物种的已知功能基因(AT4G26080)在基因水平、蛋白水平和富集通路水平上不同科学数据维度的知识,进而实现基于多维度科学数据寻证分析的检索词(LOC_Os01g40094)相关层级知识发现结果。此外,面向目的性状(抗旱、抗盐、粒重和株高)多效基因挖掘的需求,通过多维度科学数据间的多路径关联,还可实现候选多效基因的有效挖掘。与现有的作物育种知识服务平台相比,例如常用的水稻基因组变异及功能注释平台RiceVarMap(rice variation map)[26]、水稻表 观组学 注释平 台eRice(rice epigenetic and epigenomic database)[27]、玉米多组学综合数据平台ZEAMAP[28]、小麦基因定位与基因组功能研究平台WheatGmap(wheat gene mapping)[29]等,大多是基于单一物种的数据集成来解析性状遗传调控机制,无法为科研人员提供跨物种间的性状调控基因关联检索。并且现有的作物领域本体模型多是应用于单一性状的调控基因网络知识查询,无法为科研人员提供目的性状的多效基因挖掘。例如,英国洛桑研究所提出一种开放关联的领域本体模型,最终形成了面向作物领域的知识图谱KnetMiner,以支持单一性状调控基因相关知识的查询[10]。因此,本研究所构建的性状调控基因本体模型为跨物种间多维度科学数据的融合提供了良好的语义基础,优异多效基因的发现为作物分子设计育种提供了重要的数据支撑。
本研究基于所构建的性状调控基因本体模型,实现了跨物种学科知识的高效关联检索与优异多效基因的挖掘,为新功能基因的挖掘提供了新的思路。当然,受限于试验数据的清洗效率,本研究构建的性状调控基因本体模型所涵盖的科学数据维度和对象属性还需要进一步地补充。未来研究工作将进一步扩展科学数据的类型维度,更为细粒度地描述性状和基因间的知识组织体系结构,不断完善和优化性状调控基因本体模型,以更好地应用于作物育种科学研究中。同时,面向作物育种深层次知识发现的场景需求,深入开展路径关联、规则推理和链路预测等相关知识挖掘方法的应用研究。
4 结论
本研究构建了涵盖13 种实体、16 种数据属性和14 个对象属性的性状调控基因本体模型,并在以此模型为本体层的性状调控基因知识图谱中进行实验。融合模式植物拟南芥和主粮作物水稻、玉米与小麦多维度科学数据的性状调控基因知识图谱,实现了优异多效基因的挖掘,包括已知的多效功能基因31 个和未知的多效功能基因26 个。本研究所构建的性状调控基因本体模型可应用于跨物种间学科知识高效获取、优异多效基因挖掘和跨物种基因功能高效预测,为优异多效基因的挖掘和基因功能的预测提供了一条可实现的新方法路径。
致谢:
特别感谢中国农业科学院作物科学研究所汤沙老师在数据来源方面的指导和帮助。
文章所有附表数据请到本刊官网下载(http://biotech.aiijournal.com/CN/1002-5464/home.shtml)。

