自适应模态融合双编码器MRI脑肿瘤分割网络
张奕涵,柏正尧,尤逸琳,李泽锴
云南大学信息学院,昆明 650500
0 引言
脑胶质瘤是一种常见的、死亡率较高的原发性颅脑肿瘤,严重危害患者的生命健康。脑胶质瘤按照其在病理学上的恶性程度可以分为低级别胶质瘤(low-grade gliomas,LGG)和高级别胶质瘤(highgrade gliomas,HGG),低级别胶质瘤为分化良好的胶质瘤,患者预后较好;高级别胶质瘤为恶性肿瘤,患者生存率较差。作为常见的原发性脑肿瘤,早期及时发现及治疗对患者的身体健康至关重要。由于不同级别的胶质瘤伴随不同程度的水肿和坏死,且存在肿瘤与健康组织之间边界模糊、肿瘤大小不一等问题,导致医生难以准确识别病变区域,从而严重阻碍手术进程。对异常的病变肿瘤进行准确的检测和分割,再制定对应的治疗方案,将极大地改善手术质量(Mallick 等,2019)。然而,手动分割肿瘤区域耗时耗力,且不同医生分割的结果也存在差异。因此,亟需一种自动分割技术以提高效率和分割精度。传统的MRI(magnetic resonance imaging)图像分割方法包括监督学习、无监督学习、半监督学习等机器学习方法,如马尔可夫随机场(Markov random field,MRF)、高斯混 合模型(Gaussian mixture model,GMM)(Zhang 等,2017)、支持向量机(support vector machine,SVM)(Thillaikkarasi 和Saravanan,2019)、K-均值聚类等方法常用于脑肿瘤分割,但是这些方法具有很大的局限性。随着深度学习的不断发展,卷积神经网络(convolutional neural network,CNN)已被证明在图像处理领域有着极大的潜力(Krizhevsky等,2012),基于深度学习的医学图像分割方法已成为目前的主流方法(Feng 等,2019)。Havaei 等人(2017)提出了卷积神经网络进行脑肿瘤分割,通过滑动滤波器窗口结合局部和全局特征对每个像素的标签进行预测。为降低计算成本,Long 等人(2015)基于CNN 提出了具有1 × 1 卷积核的全卷积网络(fully convolutional network,FCN)。二维和三维网络是目前医学影像分割领域中常用的深度学习网络结构,它们在多模态MRI 脑肿瘤图像分割中也得到了广泛的应用。二维卷积神经网络常用于处理多张切片 的MRI 图像序 列,如U-Net(Ronneberger 等,2015)、SegNet(Badrinarayanan 等,2017)、DeepMedic(Kamnitsas 等,2017)等模型,这些模型通过编码—解码框架并使用跳跃连接来保留图像中的细节信息和空间信息。然而,二维卷积神经网络往往会忽略图像中的体素信息,因此在处理具有空间信息的三维图像时,三维卷积神经网络受到了越来越多的关注。与二维卷积神经网络相比,三维卷积神经网络能够更好地利用图像中的三维信息,从而取得更好的性能。例如,3D U-Net(Çiçek 等,2016)是一种流行的三维卷积神经网络模型,它使用编码器—解码器框架来进行图像分割,并使用跳跃连接来保留图像中的细节和空间信息。此外,Milletari 等人(2016)针对三维图像提出了V-Net,相当于在U-Net基础上引入了残差块。但是,这些模型都存在一些缺点,如二维网络虽然参数量小且可以捕捉到局部的特征信息但无法有效地处理医学图像中的深层信息,而三维网络能够同时处理空间和时间信息,但是其计算成本较高,需要更多的存储空间和计算资源,这在实际应用中会带来不小的挑战。因此,平衡二维和三维网络的优缺点,提高医学图像的诊断精度和计算效率成为当前医学图像分割领域的首要任务。为了缓解内存占用率高和计算成本高等问题,Chandra 等人(2018)提出了一种名为CA3D(contextaware 3D CNNs)的轻量级网络,旨在提高脑肿瘤分割的计算效率,该模型使用上下文感知块在空间和通道方向上进行特征融合,此外使用局部特征下采样减少特征图的尺寸及计算量。为了有效提取不同通道之间的关联信息,Chen 等人(2019)提出一种多纤维网络结构DMF-Net(dilated multi-fiber network),该网络可以利用时间和空间信息,有效捕捉多类特征。Nuechterlein 和Mehta(2018)提出了一种轻量级网络架构3D-ESPNet,通过引入ESP(efficient spatial pyramid of dilated convolutions for semantic segmentation)模块,可以大幅减少网络参数量,同时不降低模型精度。Hu 等人(2019)提出的MCC-Net(multicascaded convolutional neural network)通过引入条件随机场(conditional random field,CRF)对CNN 的输出进行后处理来优化分割结果,相比于复杂的网络结构,MCC-Net 结构简单。Qiu 等人(2021)提出的3D EMSU-Net 采用多级并行膨胀卷积和空间注意力机制有效提取多尺度特征,并且具有较低的计算和内存开销。
多模态脑肿瘤病变图像中的异常组织往往是大小、形状不一的,在脑肿瘤分割任务中,对病灶区域进行精细、准确的分割成为目前研究的难点。多模态脑肿瘤图像中的多模态这一特点有助于进行更好的分割。不同模态图像间存在互补的信息,但直接将多种模态进行拼接作为编码器的输入可能使不同模态之间的信息不能充分交互,且网络不易关注到有用的肿瘤信息,模态融合策略对得到精确的分割结果起着重要作用。一般的网络架构分为基于单编码器和基于多模型和编码器的模型(Zhou 等,2019),当前大多数模型通常使用单编码器对图像进行处理,基于单编码器的方法直接将不同的多模态图像以通道的方式集成到输入中,无法充分利用不同模态间的互补信息,影响分割结果的准确性。因此,受到D-UNet(Zhou 等,2021)的网络结构的启发,针对目前大多数网络直接将4 种模态拼接作为网络的输入,使网络计算重复冗余的信息导致推理速度变慢和效率变低等问题,本文提出了一种具有双层编码器的模型,允许多个编码器分别提取两组特征信息,并使用特定的融合策略将它们融合,以强调对分割任务有用的信息。然后,将部分卷积块替换为扩张多纤维模块,在模型参数量较少的前提下捕获多尺度特征。此外,模型上采样过程会损失一些边缘细节特征,为了保留细节信息,在解码部分引入注意力门控来进一步优化特征的提取和融合。实验结果表明,与原始的3D U-Net、V-Net及DMF-Net相比,该模型在多个脑肿瘤数据集上取得了更加优越的分割性能。
总体来说,本文做出了以下贡献:1)提出了推理速度较快、参数量较少的自适应模态融合双编码器网络。其中上层子网络引入了扩张多纤维模块及注意力门控,通过使用扩张多纤维模块减少模型的参数量,加入注意力门控使网络更关注小肿瘤图像特征信息,对多模态图像数据进行推理时更专注有用的信息,减少对重复信息的关注。2)提出了一种新的自适应多模态特征融合模块,对两部分编码器输出的特征进行有效融合。该模块利用跨模态的交互注意力机制增强不同模态间的信息交互和融合能力。3)在BraTS2018、BraTS2019 和BraTS2020 数据集上进行实验,将分割结果与基线模型以及其他先进方法进行对比,证明本文提出的自适应模态融合双编码器网络在多模态脑肿瘤图像分割任务中的有效性。
1 网络结构及方法
1.1 D3D-Net结构
基于现有的常用深度神经网络基准模型,本文提出了一种具有双编码器结构的双层3D 融合网络D3D-Net,如图1 所示。该网络使用双编码器结构对两组模态图像进行编码,充分挖掘不同模态对的组内和组间特征信息,以保留不同模态图像的特有信息,将4 个模态分为两组,两组模态信息不同且互补,分别通过上下两层子网络提取脑肿瘤特征信息,再通过融合模块凸显有用的信息。
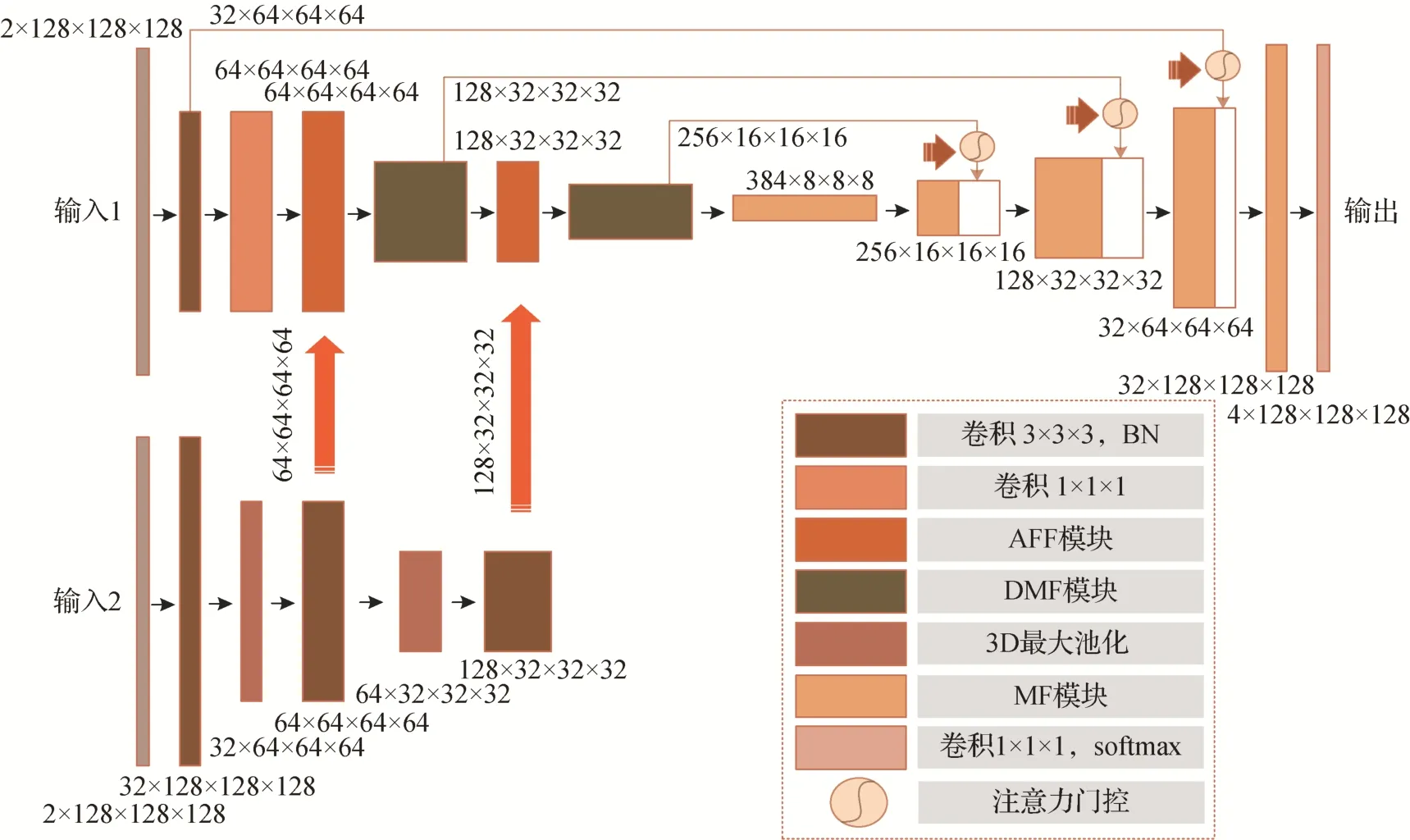
图1 本文网络结构图Fig.1 Structure of the proposed network
该网络结构主要由3 部分组成:编码器模块、特征融合模块和解码器模块。D3D-Net 分为两个子网络,上层子网络在编码和解码部分应用了扩张多纤维模块,扩张多纤维模块能够处理远距离及全局信息,且可以在不增加参数量的情况下扩大感受野大小,还可以有效减少模型的参数量,提高模型的训练速度和效率。此外,在解码器上采样部分引入了注意力门控,注意力门控可过滤跳跃连接传递的特征,帮助模型更加关注重要特征区域,更好地捕捉小肿瘤病灶区域的细节信息,从而提高分割精度。下层子网络采用卷积神经网络作为编码器对图像进行特征提取,使用3 次卷积层并使用最大池化降低特征图的尺寸,从而加快网络的训练速度。此外,由于网络早期提取特征的能力较好,且随着网络的深入,可训练的参数将会增大,所以两次特征融合均位于编码阶段,以实现组内和组间的信息融合。自适应特征融合模块的作用是将来自上、下两层子网络的特征进行融合,在编码阶段融合两次可以将不同编码器提取的特征进行更细致、更全面的融合。
1.2 扩张多纤维模块
2D 处理方法忽略了3D 图像空间上下文之间的关系,而3D 模型中3D 卷积是在整个通道上进行操作的,计算成本高,计算复杂度呈指数上升。因此,在参数量较低的情况下提高分割精度是本文的研究目标。
卷积网络达到一定规模时,标准卷积在通道和空间维度会存在冗余,解决冗余问题可极大地减少训练和推理的计算开销。分组卷积最早应用在Krizhevsky 等人(2012)的论文中,能够以训练精度小幅下降为代价大大减少模型的参数量,但简单地将普通卷积分离为多个组卷积会影响多通道之间的信息交互能力,从而损害模型的学习能力。为了改善通道间的信息交流能力,Chen 等人(2018)提出了扩张多纤维模块。如图2 所示,扩张多纤维模块将多路复用器用于多个纤维之间以促进信息交换,并且使用扩张多纤维单元捕获多尺度空间相关性。同时,扩张多纤维模块将原始的多纤维模块中的3 × 3 × 3卷积块替换为3 个3 × 3 × 3 的扩张纤维,3 个扩张纤维由3 个有着不同扩张率d的扩张卷积分支组成,3个分支被分配了3个不同的权重,最后的输出为加权相加,这种加权策略有助于从不同视野中选择最有价值的信息。为促进各纤维间的信息交互,使用包含1 × 1 × 1 卷积的多路复用器,用于不同纤维之间。此外,使用残差连接允许信息从较低层传递到较高层,增强学习能力而无需额外的参数。
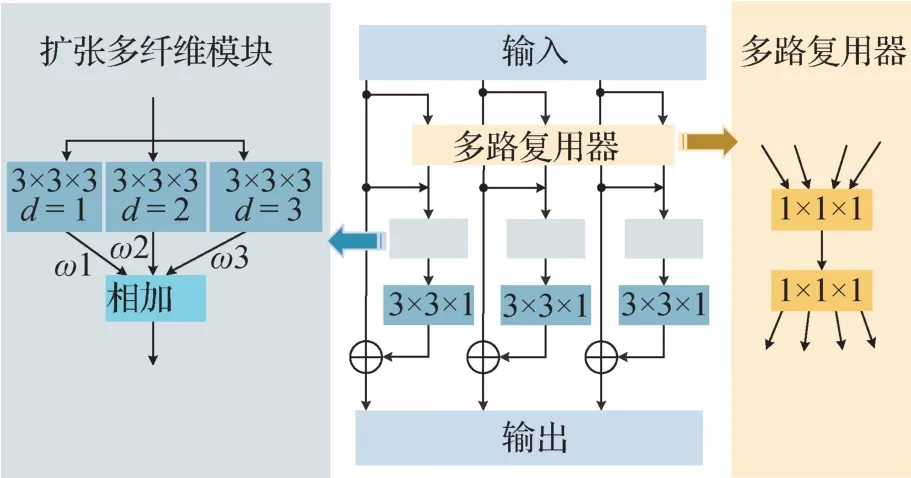
图2 扩张多纤维模块Fig.2 Dilated multi-fiber module
DMF-Net证明了扩张多纤维模块在提高网络性能和降低参数量方面的有效性,因此,本文使用扩张多纤维模块,以实现多尺度表示,在瓶颈部分和解码器中使用多纤维模块还原到原始图像大小,并输出最终的分割结果。
1.3 自适应特征融合模块
由于脑肿瘤MR 图像正、负样本像素间存在着极大的不平衡问题,训练时会关注无用的信息,因此需要引入注意力机制关注有用的特征信息。主流的注意力机制有通道注意力、空间注意力和融合了空间和通道的注意力机制。Hu 等人(2018)提出了基于压缩激励的SE(squeeze-and-excitation)模块,该模块能够自适应地学习通道间的关系,提高网络的表达能力。它由两个步骤组成:压缩(squeeze)和激励(excitation)。在压缩阶段,SE 模块通过全局平均池化来对每个通道的特征进行压缩,得到一个长度为1 的向量,表示该通道在全局特征中的重要性。这个向量可以看做是对所有特征进行的加权平均,其中权重是通过平均池化得到的。在激励阶段,SE 模块使用一个两层的全连接网络,对每个通道的重要性进行映射,得到一个激励向量,这个激励向量可以看做是一种通道权重。最后,在使用SE 模块的卷积层中,每个通道的特征都会乘以其对应的通道权重,从而强化重要的特征,抑制不重要的特征。SE 模块可以自适应地对每个通道进行加权,从而提高网络的表达能力和泛化能力。但是,SE 注意力模块的作用是对卷积神经网络的通道进行加权,在多模态三维脑肿瘤图像分割中,不同模态之间的差异很大,而SE 注意力模块只能对有限数量的通道进行加权,可能无法充分利用不同模态之间的信息。此外,脑肿瘤图像是三维图像,SE 注意力模块只考虑到了通道的信息编码,而忽略了图像的空间位置特性。为解决这一问题,Woo 等人(2018)提出了一种结合通道注意力和空间注意力机制的CBAM(convolutional block attention module)模块,旨在提高卷积神经网络对目标特征的关注能力。与SE 注意力模块类似,CBAM 也可以用于三维数据的分割任务,但也存在一些弊端,例如参数数量过多、训练过程易出现梯度爆炸或梯度消失等问题。为减少参数量,Wang等人(2020)提出了轻量级的注意力模块ECA(efficient channel attention),相比SE 模块和CBAM 模块,ECA模块在保持通道关联性的同时,不会忽略空间位置特征的信息。此外,ECA 模块相对于SE 模块和CBAM 模块计算复杂度更低,具有更高的运行效率。具体而言,如图3所示,上一层的χ特征图作为输入,对χ做全局平均池化得到1 × 1 ×C的向量g(χ),计算过程为
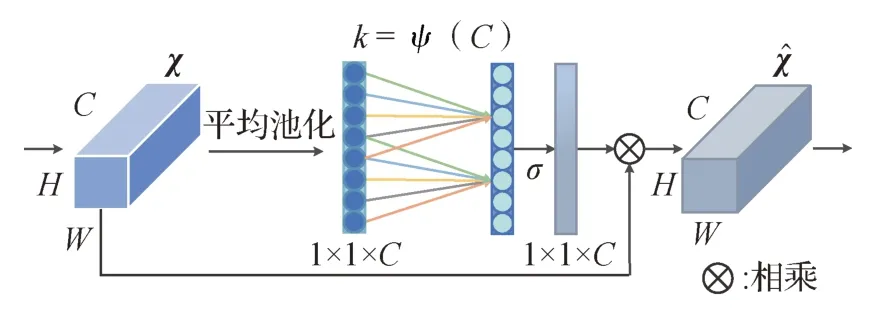
图3 ECA模块Fig.3 ECA module
式中,W和H分别代表特征图的宽度和高度,χij表示特征图的像素点。
得到g(χ)后,可以获取每个通道的权重,具体为
式中,ωi为第i个通道的权重,yi为第i个通道的特征为yi的第i通道的第j个相邻通道的特征,σ为激活函数为k个相邻通道的集合。
然后,与大小为k的卷积核进行一维卷积,具体为
式中,Conv1D为一维卷积,经过卷积使得每层的通道与相邻层的通道进行信息交互。k的大小由自适应得出,具体为
式中,取γ=2,b=1,k=5,C代表特征通道数,odd表示取奇数。
将输入的特征图χ与处理好的特征图权重相乘,权重即可加在特征图上。
本文选择ECA 模块作为自适应特征融合模块的基础模块,以实现两组模态的组内信息交互。由于网络早期提取信息的能力较强,所以在编码器部分设置两个自适应特征融合模块(adaptive feature fusion,AFF),如图4所示,该模块通过对特征通道进行注意力加权激活不同通道间的联系,然后通过残差连接对特征进行重用,最后将经过加权的两组特征相加输入到下一个模块中。
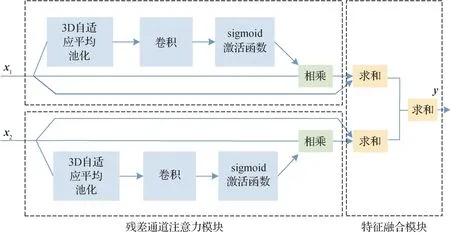
图4 自适应特征融合模块Fig.4 Adaptive feature fusion module
具体而言,AFF 模块分为残差通道注意模块和特征融合模块。其中,x1和x2分别表示来自上下两层子网络的特征图,特征图尺寸为64 × 64 × 64 ×64。两组特征图分别输入残差通道注意模块进行组内信息交互,使用残差连接将输入特征图与加权特征图相加进行特征重用,具体计算为
式中,⊗表示矩阵乘法,Δ 表示自适应平均池化,Conv表示卷积操作,σ表示sigmoid 函数,R表示残差连接。f1表示第1 个分支输出结果,f2表示第2 个分支输出结果,y表示最终的融和结果。
1.4 注意力门控
医学图像分割中,不同患者的肿瘤形状和大小差异较大时需要多级级联CNN 提取感兴趣的区域并对该区域进行密集预测,但这种方法会产生冗余的计算量和模型参数。Oktay 等人(2018)提出的新型注意力门控可用于密集标签预测,该模块可自动学习不同大小、形状的目标结构,会使注意力系数更加凸显局部区域特征,着重显示对任务有用的特征。此外,加入该模块不会产生额外的计算开销,也不需要大量的模型参数。注意力门控的具体结构如图5所示。
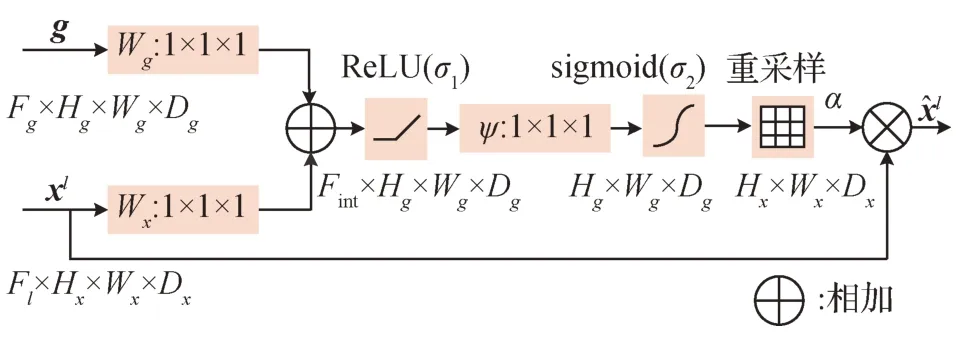
图5 注意力门控Fig.5 Attention gate
g与xl并行操作,将g和xl分别通过Wg和Wx(对g进行上采样或对xl进行下采样),将两支路输出相加,然后依次通过ReLU 操作、ψ操作、sigmoid 操作,通过重采样得到注意力权重α,最后将注意力权重α与xl相乘得到。
1.5 损失函数
医学图像分割任务中存在类别不平衡的问题,本文使用广义骰子损失函数(generalized dice loss,GDL)能够有效解决这个问题,通过对不同类别的权重进行自适应调整,使得每个类别的影响更加均匀,其计算过程为
式中,wk表示第k个类别的权重,yi,k和pi,k分别表示第i个样本属于第k个类别的真实标签和模型预测结果。在多分类问题中,yi,k通常使用one-hot编码表示,即第i个样本属于第k个类别时为1,否则为0。
2 实 验
本文模型的深度学习框架基于Pytorch,硬件环境为24 GB 显存的NVIDIA GeForce RTX 3090,实验环境为Ubuntu20.04 LTS64 位操作系统。模型训练周期为300 个epoch,批大小为6,训练时间为20 h。本文采用初始学习率为0.001 的Adam 优化器,使用L2 范数对模型正则化,权重衰减为10-5,防止过拟合,提高模型的泛化能力。
2.1 数据集
本文使用国际医学图像计算和计算机辅助干预协 会(Medical Image Computing and Computer Assisted Intervention Society,MICCAI)多模态脑肿瘤分割挑战赛的BraTS2018(brain tumor segmentation 2018)、BraTS2019和BraTS2020数据集。这3个数据集包含了来自多个医院的磁共振成像(MRI)数据,包含T1 加权、T1 加权对比增强、T2 加权和FLAIR(fluid-attenuated inversion recovery)图像(Zhou 等,2019)。每个病例都由4 种模态的MRI 图像组成,并附带有肿瘤分割标签,其中包含4 种不同的组织类别:坏死区、水肿区、非增强肿瘤和增强肿瘤。
BraTS2018 数据集包含285 个病例,每个病例图像尺寸为240 × 240 × 155 像素,其中包括210 个高级胶质瘤病例、75 个低级胶质瘤病例。验证集包含66个病例,不含人工分割标签。
BraTS2019 数据集在BraTS2018 数据集的基础上增加了49 例HGG 病例和1 例LGG 病例,本文将BraTS2018 数据集作为训练集,将增加的50 组数据集作为测试集。BraTS2020 数据集包含369 个训练集病例和125个验证集病例。分割标签包括坏疽区、浮肿区、肿瘤增强区,分割任务是由嵌套标签形成的3个子区域,分别为整个肿瘤(whole tumor,WT)、增强肿瘤(enhancing tumor,ET)和肿瘤核心(tumor core,TC)。其中,WT 包含坏死区、浮肿区和肿瘤增强区,ET包含肿瘤增强区,TC包含坏死区和肿瘤增强区。
两个数据集图像类型是nii 格式,需通过软件ITK-SNAP 读取nii 文件。4 种模态及标签的可视化结果如图6所示。
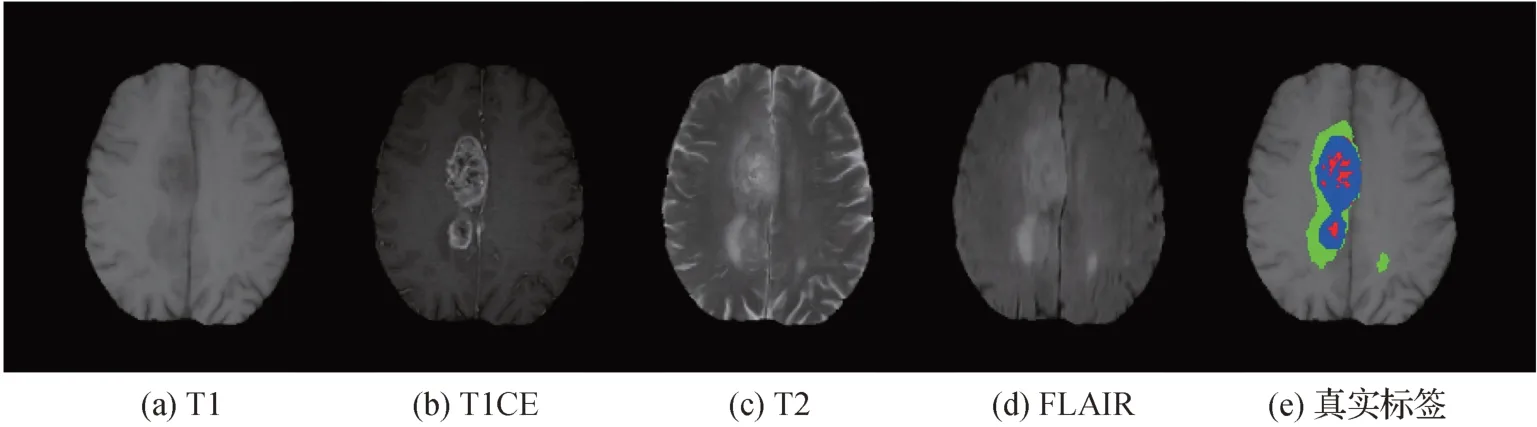
图6 脑肿瘤MRI 4种模态及标签图像Fig.6 Four modes and labels of MRI images of brain tumors((a)T1;(b)T1CE;(c)T2;(d)FLAIR;(e)ground truth)
2.2 数据预处理
由于不同模态不同病例图像大小、形态和位置有差异,且原始图像有很多无用的背景信息不利于进行推理,所以对图像进行修正减少冗余信息,从而降低图像占用显存的数据预处理操作是有必要的。通过可视化脑肿瘤图像可以发现,脑部图像约占整个MRI图像尺寸的2/3,原始图像包含大量不含肿瘤的无用背景信息,所以在分割前,将原始数据从240 × 240 × 555 体素剪裁到128 × 128 × 128 体素,从而减少无用背景信息降低计算量,剪裁后的图像作为模型的输入。此外,使用了以下数据增强技术:1)在轴面、冠状面、矢状面随机翻转概率为0.5;2)角度在[-10°,+10°]之间进行随机旋转;3)随机强度偏移在[-0.1,0.1]之间,缩放在[0.9,1.1]之间。
2.3 评价指标
为了评估网络的性能,采用Dice 系数(Dice coefficient)和Hausdorff距离(Hausdorff distance,HD)作为评价指标,对预测结果的好坏进行评估。
Dice 系数是一种集合相似度度量函数,用于计算两个样本的相似程度,分割结果最好值为1,最差值为0。
豪斯多夫距离是描述空间中任意两个集合之间相似程度的一种量度。豪斯多夫距离越小,表示分割效果越好。HD_95 是将HD 值乘以95%以消除离群值对结果的影响。
2.4 实验结果
基于BraTS2018 数据集进行实验,在BraTS2018训练集上进行模型训练,使用训练好的模型对BraTS2018 验证集进行模型验证,并将分割实验结果提交至CBICA(Center for Biomedical Image Computing and Analytics)图像处理官方在线网站得到Dice和HD95等分割评价指标。
为了验证不同模态组合对网络的影响,将FLAIR、T1、T1CE、T2 四种模态分别记为1、2、3、4。上层子网络输入模态为FLAIR 和T1,下层子网络输入模态为T1CE和T2的组合,记为D3D-Net1。同理,得到其他5 个对比网络。为了选择最优的模态组合,将6 个网络分别进行训练和验证,最终采用上层子网络输入为T1CE 和T2、下层子网络输入为FLAIR和T1的D3D-Net1作为最终的模型。
6 组网络及其他基准网络在同样实验环境下在BraTS2018验证集上的分割结果如表1所示。由表1可知,本文提出的各种组合的D3D-Net 模型在与基准模型的对比实验中表现出最佳的性能,根据分割指标,模型在增强肿瘤、整个肿瘤和肿瘤核心的Dice值分别达到了79.7%,89.5%,83.3%。与3D U-Net相比,分别提升了3.6%,1.0%,11.5%;与V-Net 相比,提升了3.0%,0.4%,2.3%;与DMF-Net 相比,提升了2.2%,0.2%,0.1%。所提出的网络在增强肿瘤部分提升的程度较大,也证明了网络在小目标分割方面的有效性。
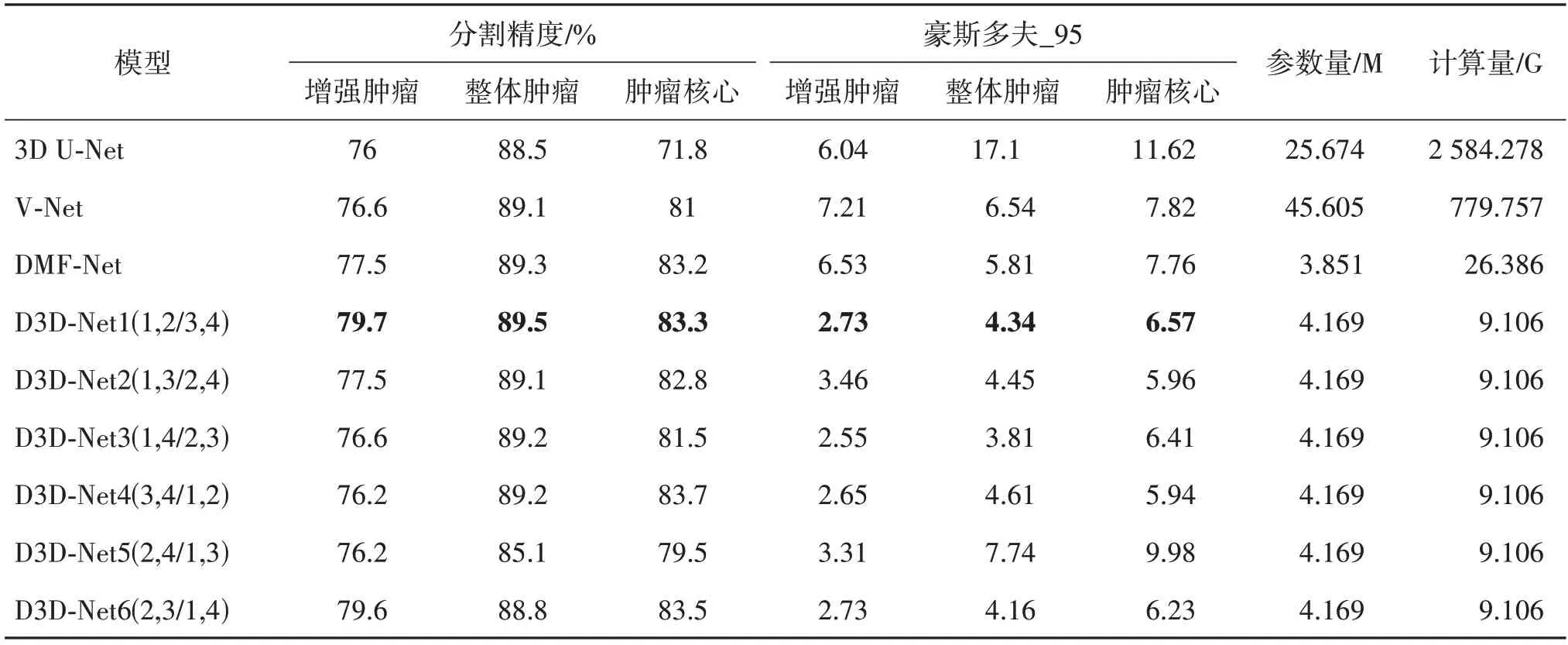
表1 在BraTS2018验证集上的分割结果对比Table 1 Segmentation results of BraTS2018 validation set
具有分割分数分布特征的箱图如图7 所示,描绘了增强肿瘤、整个肿瘤和肿瘤核心之间Dice 值和HD_95 距离的分布及样本个体值,由于部分肿瘤图像没有增强肿瘤和肿瘤核心部分,所以这部分样例的增强肿瘤和肿瘤核心的Dice 值为0,除了这部分特殊的样例和少数样例,D3D-Net对于所有这3组中的大多数对象都表现良好。少数样例的Dice 值较低可能是分割的肿瘤图像过小所导致的。
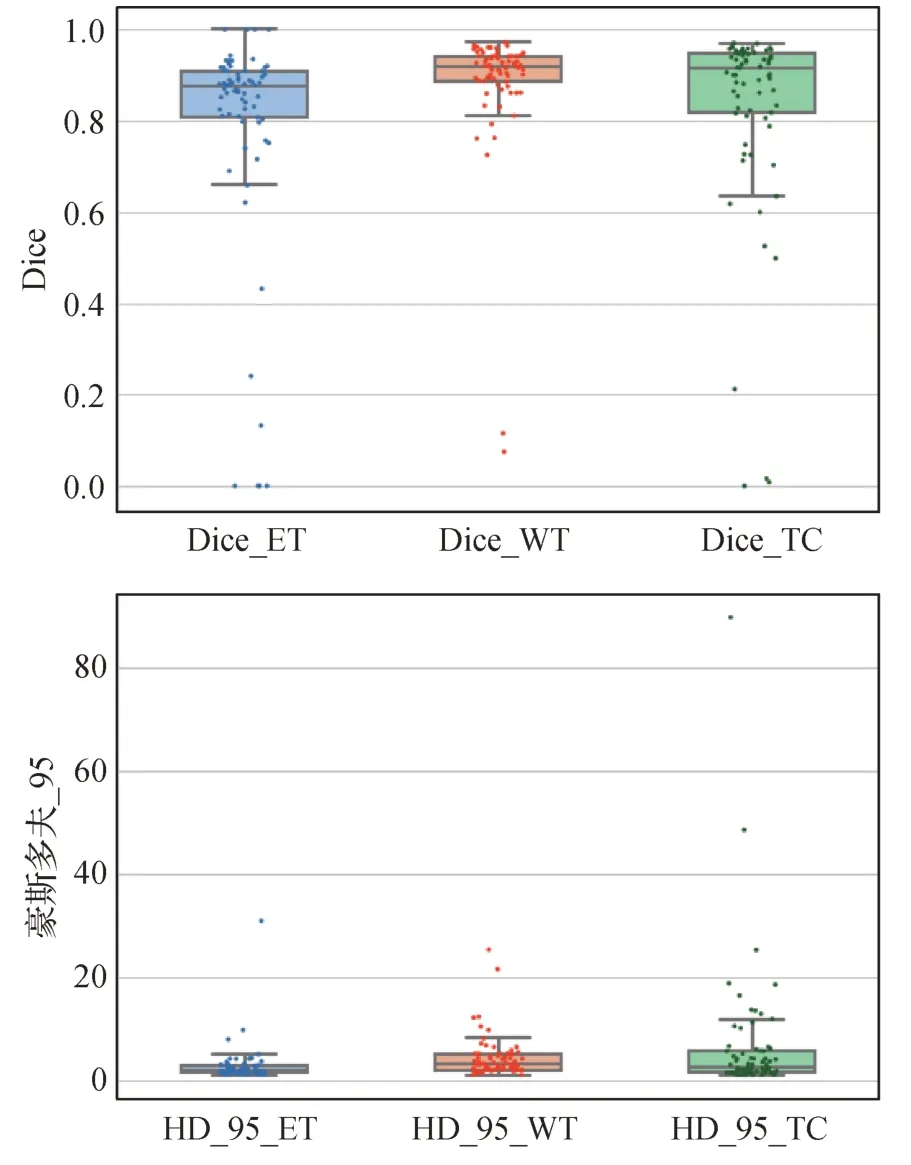
图7 ET、WT和TC数据组上的Dice和HD_95性能箱图Fig.7 The boxplot of the performance of Dice and HD_95 on the data groups of ET,WT and TC
2.5 消融实验
为了证明双层网络比单层网络更有效,本文基于BraTS2018 数据集对不同的网络结构进行实验。实验分为4 组,a 组网络结构为单层的DMF-Net 网络;b 组网络结构为没有注意力门控的双层网络;c 组网络结构为加入注意力门控的DMF-Net 网络;d组网络结构为本文提出的D3D-Net。
实验结果如表2 所示,通过对比a、b 两组,可以发现双层网络结构各个子肿瘤区域的分割精度整体要优于单层网络结构,虽然b 组肿瘤核心部分的分割精度略低于a 组,但是其增强肿瘤和肿瘤核心的豪斯多夫距离要远低于a 组,证明了双层网络比单层网络更有效。通过对比a、c 和b、c,可证明注意力门控在单层和双层网络的有效性。此外,由c、d 两组数据可证明本文提出的自适应模态融合双编码器网络的有效性。虽然d 组的豪斯多夫距离略高于c 组,但是d 组的分割精度要优于c 组,也证明了本文提出的方法对分割精度的提升有积极作用。
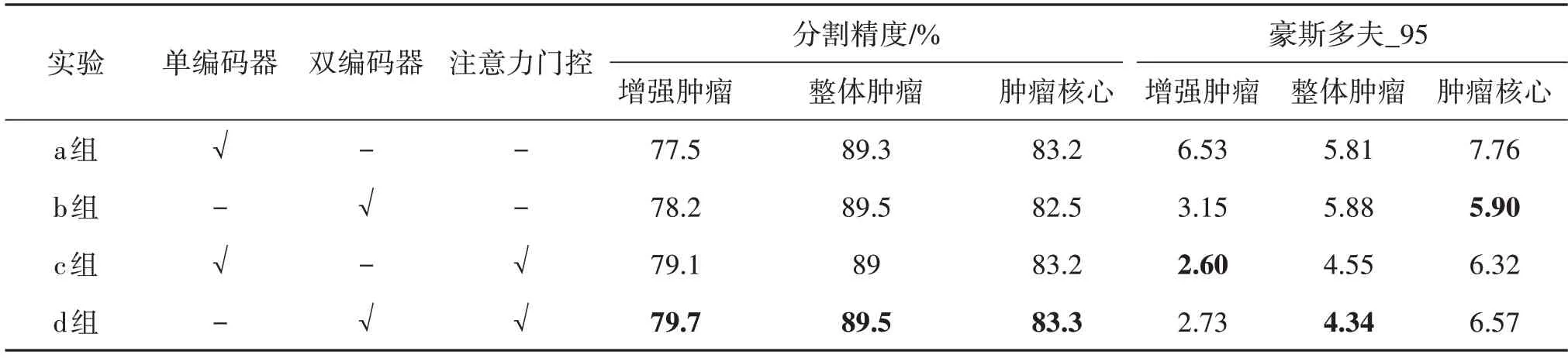
表2 网络结构消融实验结果Table 2 Results of network structure ablation experiment
此外,为了证明本文提出的自适应特征融合模块的有效性,使用几种不同的融合策略进行实验。融合策略如图8所示,分别为直接相加融合、仅有通道注意的融合和有残差结构的通道注意融合,实验结果如表3所示。与直接将输入模态进行相加和不含残差结构的通道注意融合策略相比,包含残差连接的通道注意融合AFF模块分割结果的Dice值有着明显的提高,与直接相加的融合策略相比,增强肿瘤、整个肿瘤和肿瘤核心分别提升了0.6%、0.1%、0.2%,证明了残差连接能够重用特征和引入注意力机制的有效性。
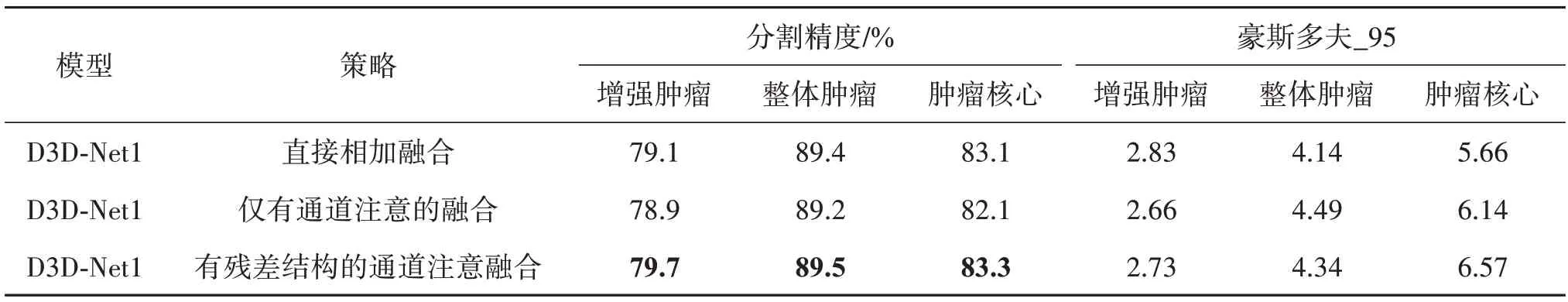
表3 不同融合策略的影响Table 3 Impact of different fusion strategies
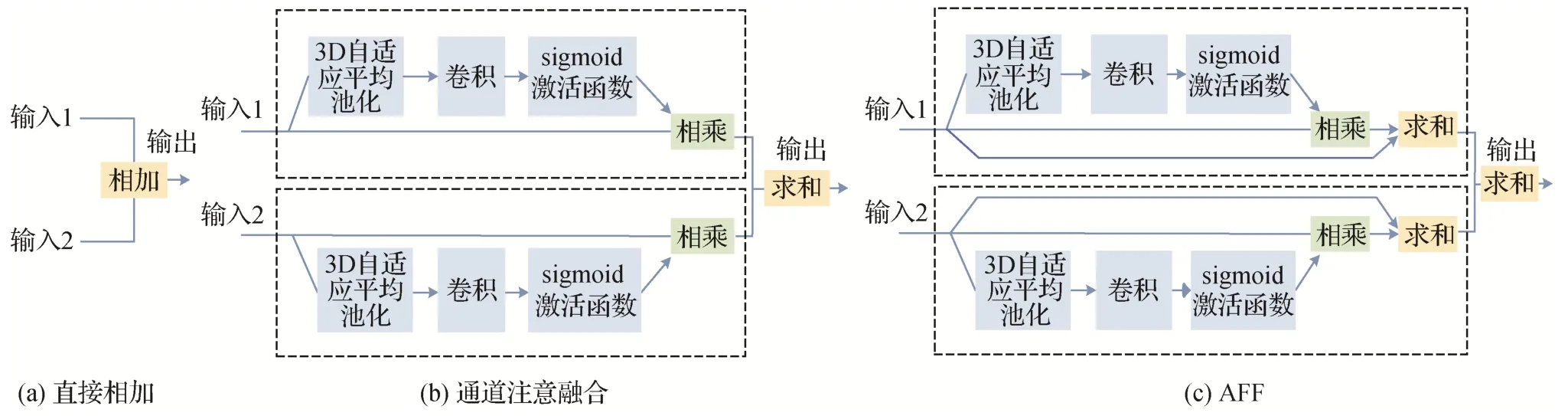
图8 3种不同的模态融合策略Fig.8 Three different modal fusion strategies((a)addition;(b)channel attention fusion;(c)AFF)
2.6 对比实验
为了证明本文所提出网络的先进性,将提出的网络与其他先进网络,如3D ESP-Net(Nuechterlein等,2018)、3D EMS-UNet(Qiu 等,2021)、CSPU-Net(夏峰 等,2022)等网络进行了比较。对比实验的结果如表4 所示,可以看出,与其他几种先进方法相比,本文网络在几个肿瘤子区域的分割精度指标都取得了最优的结果,各子区域的HD_95 值也较小。与其他模型分割结果相比,分割精度指标最少分别提升了1.8%,0.1% 和0.2%,最多分别提升了7.9%,2.3%和8.5%。这些实验结果表明本文提出的网络具有先进性。
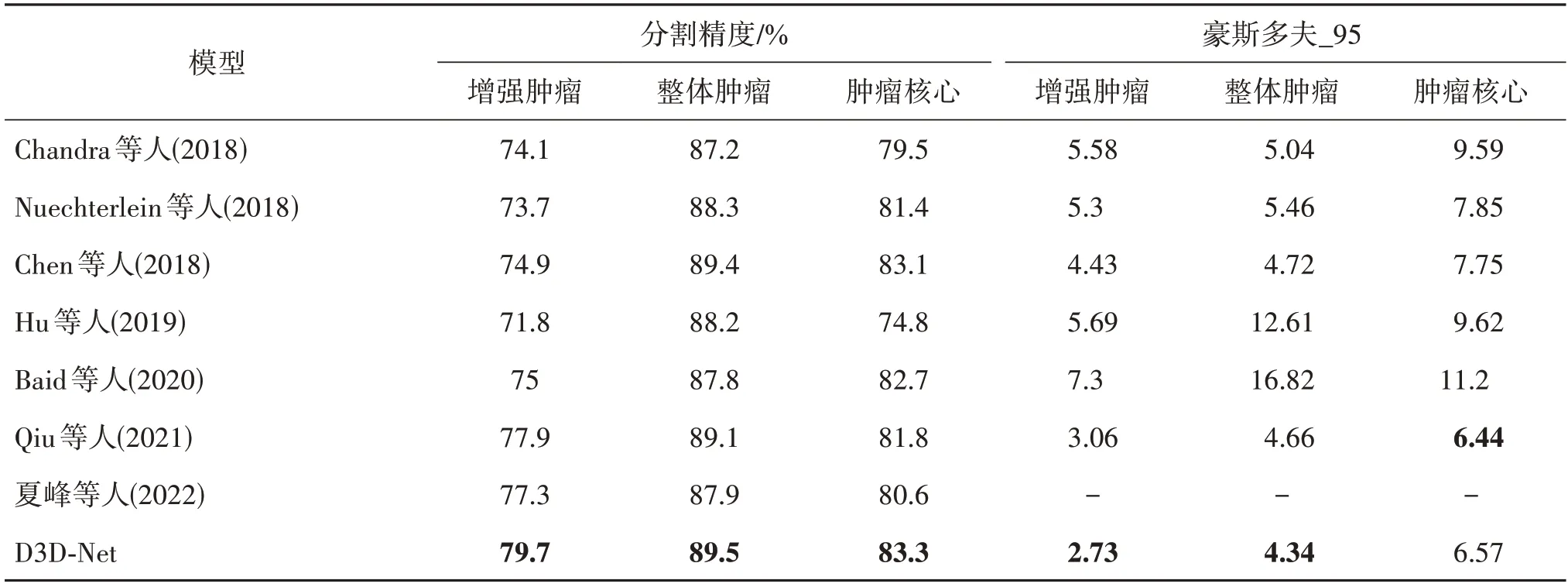
表4 在BraTS2018验证集上的分割结果与其他先进方法对比Table 4 Comparison of segmentation results on BraTS2018 validation set with other advanced methods
2.7 可视化及有效性分析
为了进一步证明本文提出的网络的有效性,在BraTS2019 数据集和BraTS2020 数据集上进行了实验。BraTS2019 数据集在BraTS2018 数据集的基础上增加了49 例HGG 病例和1 例LGG 病例,本文将BraTS2018 数据集作为训练集,将增加的50 组数据集作为测试集,不同模型的分割结果如表5所示。
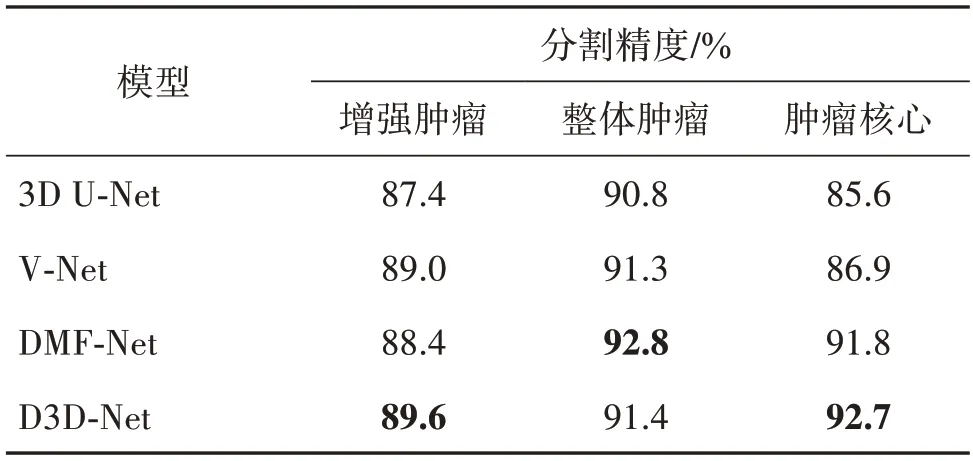
表5 在BraTS2019 50例数据集上的分割结果对比Table 5 Segmentation results of BraTS2019 50 samples
BraTS2020 训练集包含369 个病例,验证集包含125 个病例。五折交叉验证实验将数据集随机分成5 份,不重复地取其中1 份作为测试集,其他4 份作为训练集,以更全面地评估模型,五折交叉验证结果如表6所示。
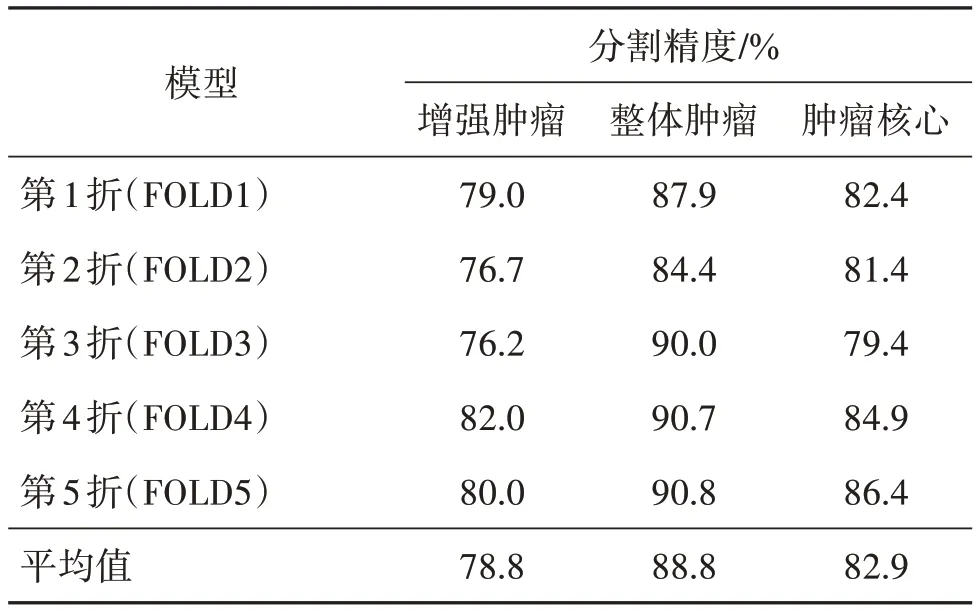
表6 在BraTS2020数据集上的五折交叉验证Table 6 Five fold cross validation results on BraTS2020
此外,为了让被评估的模型结果更加准确可信,在BraTS2020 验证集上进行了实验,验证集的分割结果如表7 所示,与其他基准网络相比,本文提出的D3D-Net 模型在3 个分割子区域的分割精度分别为78.2%、87.3%、83.8%。与DMF-Net 相比,增强肿瘤、整体肿瘤和肿瘤核心的分割精度分别提升了2.4%,0.8%,2.5%。
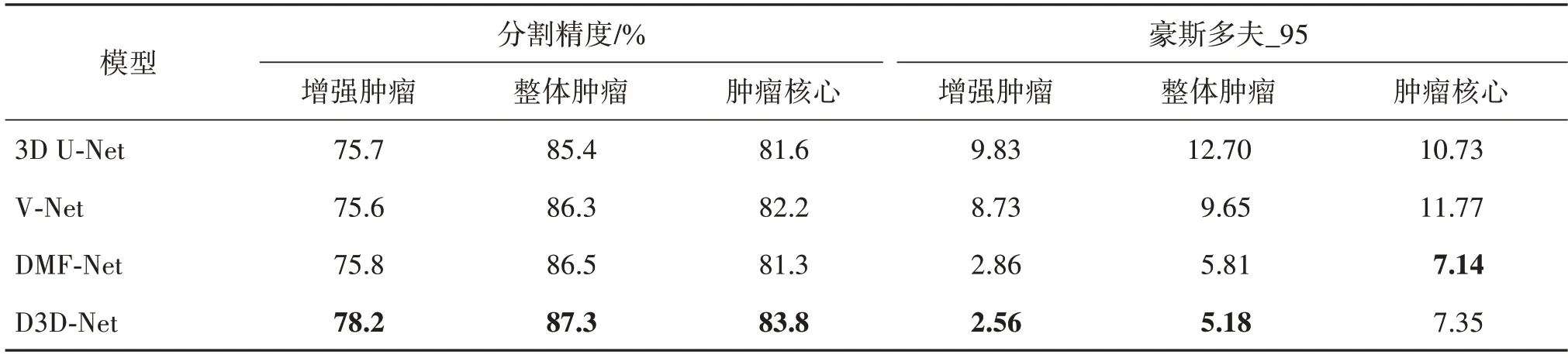
表7 在BraTS2020验证集上的分割结果对比Table 7 Segmentation results of BraTS2020 validation set
图9 是几种不同的分割方法对脑肿瘤分割的可视化结果。图9(a)—(h)分别表示4种模态图像、分割掩码(标签)、本文提出方法的分割结果、3D U-Net分割结果和DMF-Net 分割结果。其中,红色代表非增强肿瘤及坏死肿瘤,绿色代表肿瘤附近的水肿区域,蓝色代表增强型肿瘤。从5 组不同病例图像的分割结果可以明显看出,相比于3D U-Net 和DMFNet 的分割结果,本文提出的D3D-Net 模型分割的3 个子区域与手动标注的标签更加接近,尤其是小病灶及肿瘤核心的分割效果较好。
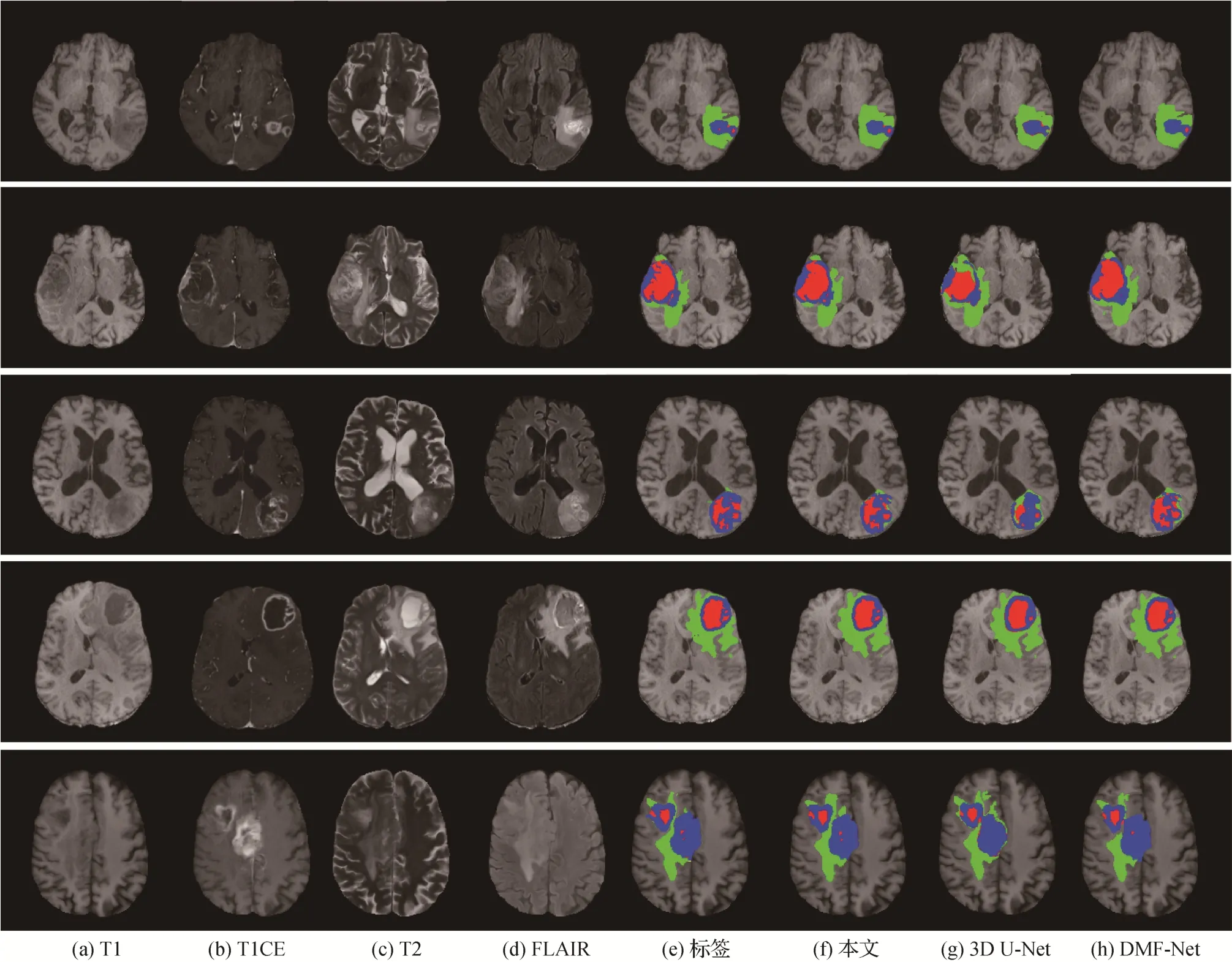
图9 各模态、标签及不同模型分割结果对比Fig.9 Comparison of modalities,labels and segmentation results of different models((a)T1;(b)T1CE;(c)T2;(d)FLAIR;(e)label;(f)ours;(g)3D U-Net;(h)DMF-Net)
3 结论
本文针对大多数单编码器网络存在的特征融合不充分等问题,提出了一种自适应模态融合双编码器结构——D3D-Net,通过使用特定的融合策略将两组特征信息融合,有效地强调了对分割任务有用的信息。同时,通过使用扩张多纤维模块替换部分卷积块,该模型在参数量较少的情况下成功地捕获了多尺度特征。另外,在解码部分引入注意力门控,进一步优化了特征的提取和融合,从而成功地保留了一定的边缘细节特征。为了证明本文提出方法的有效性,使用BraTS2018、BraTS2019 和BraTS2020 数据集进行实验。实验结果表明,该模型在多个脑肿瘤数据集上的分割性能显著优于3D U-Net、V-Net、DMF-Net等网络,在增强肿瘤、整体肿瘤和肿瘤核心的分割精度上有较显著的提升。因此,该模型在医学影像分割任务中具有良好的应用前景。
虽然本文提出的网络具有较好的分割效果,但仍存在一些问题:首先,本文只使用了双层编码器提取两组模态信息,但是在实际的医学影像分割任务中,4 种模态的图像通常具有不同的特征信息。其次,虽然本文提出的是参数量较少的轻量化模型,但是由于模型是双编码器结构,因此在训练时占用显存较大。后续工作可以考虑如何更好地充分利用多模态图像的特征信息以及如何减少模型的计算复杂度。此外,对小肿瘤边缘信息提取的准确性的提高有待进一步研究。